基于线粒体细胞色素b基因序列探讨长江流域斑点蛇种群遗传结构和地理分化
李小兵, 唐琼英, 俞丹, 刘焕章*
(1. 中国科学院水生生物研究所,水生生物多样性与保护重点实验室,武汉430072; 2. 中国科学院大学,北京100049)
长江是我国第一大河流,跨越我国大陆地势的三大阶梯,地貌类型多样,气候差异显著,形成了多样的生境,呈现出丰富的鱼类多样性及较高程度的种群分化(Chengetal. ,2015a,2015b)。如,基于线粒体细胞色素b(Cytb)基因的研究表明,长江流域的宽鳍Zaccoplatypus包含4个独立进化的线粒体谱系,且谱系间存在较大的遗传分化,可能对应4个不同物种(Perdicesetal. ,2004)。类似的研究结果在铜鱼Coreiusheterodon、圆口铜鱼C.guichenoti、长薄鳅Leptobotiaelongata和中华倒刺鲃Spinibarbussinensis中也有报道(袁希平等,2008;田辉伍等,2013;任泷等,2014)。这些研究充分体现了长江流域鱼类丰富的种群遗传多样性(Zhangetal. ,2015)。
线粒体DNA(mtDNA)具有分子结构简单、严格的母系遗传、不同区域进化速度存在差异等特点,已被广泛应用于系统发育及种群遗传研究(Patwardhanetal. ,2014;Lakshmananetal. ,2015)。其中,Cytb基因由于进化速度适中,且易扩增,已被广泛用于种群遗传多样性的研究(Chiuetal. ,2015)。本文拟通过对采自长江上游赤水河江段、长江上游干流江段(包括合江江段和宜宾江段)及长江中游江段(洞庭湖、赣江及鄱阳湖)的斑点蛇种群,进行Cytb基因的序列测定,分析不同地理种群的遗传结构,探讨其分化过程,以期为长江鱼类资源的保护提供科学的理论依据。
1 材料与方法
1.1 材料
本研究所用样本于2010—2015年采集于长江流域20个样点,共计303尾。根据地理分布,将其分为3个地理种群。来自长江上游赤水河江段10个样点的为赤水河种群,四川省宜宾市和合江县2个样点属于长江上游干流种群,其余8个样点分别来自湖南洞庭湖水系、江西赣江及鄱阳湖水系,属于长江中游种群。标本均用95%乙醇固定保存于中国科学院水生生物研究所淡水鱼类博物馆(图1,表1)。
1.2 方法
1.2.1基因组DNA的提取、PCR扩增及测序基因组DNA的提取采用高盐抽提法。基因序列片段通过PCR扩增所得。扩增所用引物为L14724和H15915。PCR反应的总体积为60 μL,约50 ng基因组DNA为模板,反应体系包括:10×Buffer 6 μL,dNTPs 0.75 μL(10 mmol·L-1),引物各1.5 μL(10 μmol·L-1),Taq DNA聚合酶1.5 U,加灭菌双蒸水至60 μL。PCR反应条件为:94 ℃预变性3 min;94 ℃变性45 s,52~58 ℃退火45 s,72 ℃延伸1 min,共35个循环;最后72 ℃延伸8 min。将获得的PCR产物送生工生物工程(上海)股份有限公司完成测序。
1.2.2数据分析为确保序列准确性,对获得的序列,通过DNASTAR中的Seqman手动拼接。用Clustal X 2.1进行序列比对(Larkinetal. ,2007)。为避免序列中存在测序误读等错误,采用MEGA6.0将其翻译成相应的氨基酸序列进行校正(Tamuraetal. ,2013)。使用PAUP* 4.0b10(Swofford,2002)和MEGA6.0对序列进行初步分析,包括碱基组成及核苷酸同质性检验等。
采用DnaSP 5.10.1统计种群遗传多样性参数,并获得单倍型序列数据组。基于单倍型序列数据,采用邻接法(NJ)、最大似然法(ML)和贝叶斯法(BI)构建分子系统发育树。使用MrMTgui、Modeltest 3.7和PAUP* 4.0b10选择最适合的碱基替代模型,对数据的拟合度通过AIC来检测。NJ分析在PAUP* 4.0b10中进行,系统树的分支置信度采用自引导法进行1 000次重复检测。BI分析用MrBayes 3.2.5,以后验概率来表示各分支的可信性(Ronquist & Huelsenbeck,2003)。ML分析用PhyML 3.0,通过1 000次自引导法检验各分支的置信度(Guindon &

表1 长江流域斑点蛇采样点地理信息、样本数量及单倍型信息Table 1 Sample location, size and haplotype information of Saurogobio punctatus in the Yangtze River basin
1.贵州省叙永县赤水镇, 2. 贵州省大方县田坎乡, 3.贵州省仁怀市茅台镇, 4. 贵州省赤水市葫市镇, 5. 贵州省赤水市复兴镇, 6. 贵州省赤水市大同镇, 7. 贵州省赤水市, 8. 四川省合江县先市镇, 9. 四川省合江县密溪乡, 10. 贵州省赤水市长沙镇, 11. 四川省合江县, 12. 四川省宜宾市, 13. 湖南省岳阳市, 14. 江西省武宁县, 15. 江西省上饶市, 16. 江西省新余市, 17. 江西省都昌县, 18. 江西省鄱阳县, 19.江西省鹰潭市, 20. 江西省婺源县
1. Chishui town, Xuyong county, Guizhou province, 2. Tiankan town, Dafang county, Guizhou province, 3. Maotai town, Renhuai city, Guizhou province, 4. Hushi town, Chishui city, Guizhou province, 5. Fuxing town, Chishui city, Guizhou province, 6. Datong town, Chishui city, Guizhou province, 7. Chishui city, Guizhou province, 8. Xianshi town, Hejiang county, Sichuan province, 9. Mixi town, Hejiang county, Sichuan province, 10. Changsha town, Chishui city, Sichuan province, 11. Hejiang county, Sichuan province, 12. Yibin city, Sichuan province, 13. Yueyang city, Hunan province, 14. Wuning county, Jiangxi province, 15. Shangrao city, Jiangxi province, 16. Xinyu city, Jiangxi province, 17. Duchang county, Jiangxi province, 18. Poyang county, Jiangxi province, 19. Yingtan city, Jiangxi province, 20. Wuyuan county, Jiangxi province
Gascuel,2003)。利用Network 4.613中的中接法构建单倍型的网络关系图,分析单倍型之间的进化关系。
使用Arlequin 3.5进行分子方差分析(AMOVA),计算遗传分化指数(FST)、错配分布值及Tajima’sD和 Fu’sFs值(Excoffier & Lischer,2010)。使用BEAST 1.8.2进行种群动态的Bayesian skyline plot(BSP)分析(Drummondetal. ,2012)。
2 结果
2.1 Cyt b基因的序列变异分析
2.2 单倍型系统发育关系及进化网络分析
单倍型进化网络关系图(图3)显示:整体上,单倍型间的网络进化关系呈星状分布,单倍型间彼此相连、相差一步或多步的突变距离。单倍型Hap3(为长江上游干流种群和赤水河种群共享)为较原始的单倍型,位于星状网络图最中心的位置。其他单倍型均由Hap3直接演化或逐步演化而来。其中,经过Hap3多次演化后的Hap11(为长江上游干流种群和赤水河种群共享)又演化出更多的单倍型。
2.3 不同地理种群的遗传多样性、遗传分化及分子方差分析

图2 基于线粒体Cyt b基因单倍型构建的长江流域 斑点蛇不同地理种群的邻接树、最大似然树、贝叶斯树Fig. 2 Neighbor-Joining/Maximum-Likelihood/Bayesian inference tree of Saurogobio punctatus populations based on mtDNA Cyt b gene in the Yangtze River basin
节点处的数值代表NJ/ML/BI分析获得的支持率, 支长代表分歧度
Values at the node represent support values in NJ/ML/BI analysis, respectively; the length of branch indicates the percentage of divergence
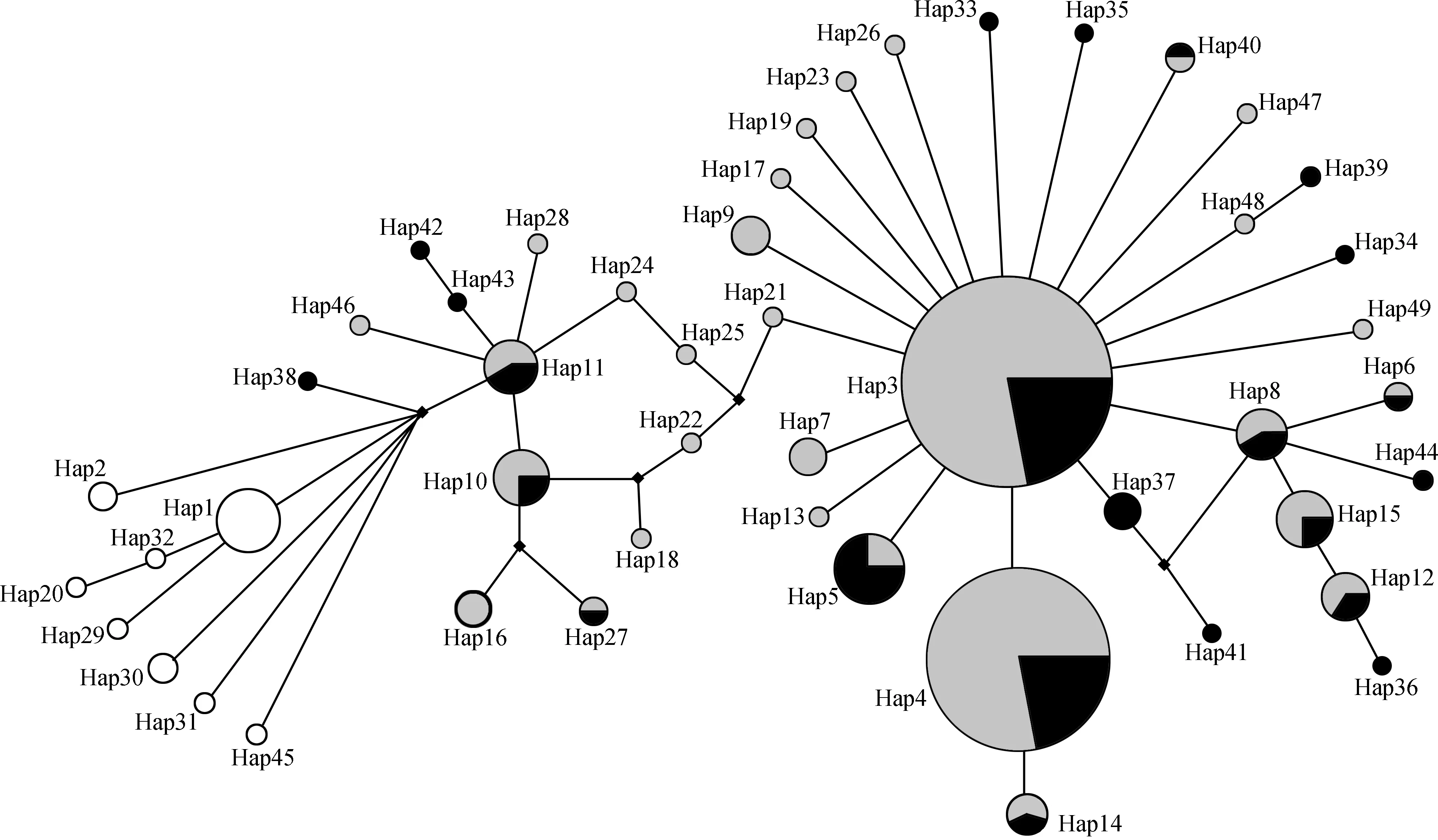
图3 基于线粒体Cytb基因的长江流域斑点蛇不同地理种群的单倍型网络图
Fig. 3 The haplotype network ofSaurogobiopunctatusbased onCytbgene in the Yangtze River basin
灰色部分、黑色部分和白色部分分别表示赤水河种群、长江上游干流种群及长江中游种群所拥有的单倍型; 圆圈的大小表示单倍型包含的个体数; 中间小黑点代表缺失的单倍型
Gray, black and white portions represent the Chishui River population, the upper Yangtze River main stream population, and the middle Yangtze River population, respectively; black dot represents the lacked haplotype

表2 长江流域斑点蛇不同地理种群的遗传多样性统计参数Table 2 Genetic diversity parameters of Saurogobio punctatus in different populations of the Yangtze River basin
注Notes:*P<0.05,**P<0.01; 下同the same below

表3 长江流域斑点蛇不同地理种群的遗传分化指数Table 3 Genetic differentiation index among differentgeographic populations of Saurogobio punctatusin the Yangtze River basin

表4 基于线粒体Cyt b基因对长江流域斑点蛇的分子方差分析Table 4 AMOVA analysis based on Cyt b of Saurogobio punctatus among 3 populations in the Yangtze River basin
2.4 长江流域斑点蛇种群的动态分析
3 讨论
3.1 长江流域斑点蛇不同地理种群的遗传分化与变异
由于地理隔离,同一物种的不同地理种群可能会产生遗传分化(Brownetal. ,1979)。本研究结果显示,长江上游干流和赤水河斑点蛇种群间的FST值较低,为0.029 4。而长江中游与长江上游干流和赤水河种群的FST值均较高,且差异有高度统计学意义。Wright(1978)认为,若种群的FST值为0~0.05,说明不存在分化;0.05~0.15为中度分化;0.15~0.25为高度分化。因此,斑点蛇长江上游干流种群与赤水河种群间未发生遗传分化或分化程度较低,而长江中游种群与上游种群则已发生高度分化。长江上游不同江段同种种群间不存在遗传分化的现象在其他鱼类中也有报道,如,田辉伍等(2013)发现长江干流与赤水河、岷江及嘉陵江的长薄鳅种群间基因交流频繁。但长江中游与上游的斑点蛇间遗传分化显著,种群间缺乏广泛的交流,这与长江流域Hemiculterleucisculus的研究结果一致(范启,何舜平,2014)。

图4 长江流域斑点蛇种群动态随时间变化 的Bayesian skyline plots
Fig. 4 Bayesian skyline plots for population dynamics with time ofSaurogobiopunctatusin the Yangtze River basin
(a) 长江流域种群, (b) 长江上游干流种群, (c) 长江中游种群; 黑色线表示中值, 阴影部分为95%的置信区间
(a) the population from the Yangtze River basin, (b) the population from the upper Yangtze River main stream, (c) the population from the middle Yangtze River; the black line indicates the median, the shadow part shows the 95% highest probability density
3.2 长江流域斑点蛇不同地理种群的遗传多样性
作为生物多样性的重要组成部分,遗传多样性是经历突变及长时间自然选择后产生的结果,也是一个物种生存适应和发展进化的前提(Taoetal. ,2013)。其中,Hd和Pi是衡量种群遗传变异程度的2个重要指标(Brooksetal. ,2015)。Hd和Pi较高说明种群遗传多样性高,遗传资源丰富(Chen & He,2002)。在长江流域斑点蛇3个种群中共检测到49个单倍型,整体的Hd和Pi平均值分别为0.803和0.003 71,呈现较高的单倍型多样性和较低的核苷酸多样性,暗示该种群可能经历过瓶颈效应或奠基者效应后迅速扩张(Goodall-Copestakeetal. ,2012;Yangetal. ,2014)。在扩张后的群体中,遗传变异虽导致了单倍型多样性的增加,但核苷酸多样性依然维持在相对较低的水平(Arundelletal. ,2015)。
3.3 长江流域斑点蛇的种群历史
在种群动态分析中,Tajima’sD值和 Fu’sFs值是2种通过中性检验研究种群在历史上是否发生过扩张的较常用参数。Tajima’sD值和Fu’sFs值接近0时,表明种群处于稳定状态,两者为负且P值小于0.05被认为种群在历史上有扩张迹象。此外,对于发生过扩张的种群,错配分布图一般呈现单峰(Ramos-Onsins & Rozas,2002)。本研究中,中性检验及错配分布图均表明,整体上,长江流域斑点蛇经历过种群的快速扩张;长江上游干流种群和赤水河种群曾发生过扩张现象,而长江中游种群则未能检测到种群扩张,处于稳定状态。此外,斑点蛇种群动态随时间变化的BSP图显示,整体上,长江流域斑点蛇从0.20 Ma BP开始发生种群扩张,一直持续到末次间冰期(MIS5)晚期(75~128 ka BP)(Thieletal. ,2015);而后种群得到迅速扩张,一直持续到0.05 Ma BP左右;此后,种群保持相对稳定。另外,据单倍型网络图,Hap3(长江上游干流和赤水河种群共享的单倍型)是最原始的单倍型,而长江中游种群的单倍型均由其间接演化而来。因此推测,斑点蛇的起源中心可能在长江上游,然后通过种群扩张逐渐扩散到长江中游,进化成遗传分化较大的种群。
致谢:感谢课题组刘飞助理研究员、但胜国高级实验师及张富斌等同学在采样方面给予的帮助。贵州省赤水市畜牧水产局的娄必云、黎良等同志在采样方面提供诸多便利,一并致以谢忱!
:
陈宜瑜. 1998. 中国动物志 硬骨鱼纲 鲤形目(中卷)[M]. 北京: 科学出版社: 379-389.
丁淑荃, 万全, 李飞, 等. 2013. 斑点蛇精子结构研究[J]. 安徽农业大学学报, 40(6): 959-963.
范启, 何舜平. 2014. 长江流域种群遗传多样性和遗传结构分析[J]. 水生生物学报, 38(4): 627-635.
任泷, 吴金明, 李雷, 等. 2014. 赤水河中华倒刺鲃的形态分化及Cytb基因序列比较研究[J]. 淡水渔业, 44(6): 58-64.
田辉伍, 段辛斌, 汪登强, 等. 2013. 长江上游长薄鳅Cytb基因的序列变异与遗传结构分析[J]. 淡水渔业, 43(6): 12-18.
袁希平, 严莉, 徐树英, 等. 2008. 长江流域铜鱼和圆口铜鱼的遗传多样性[J]. 中国水产科学, 15(3): 377-385.
Arundell K, Dunn A, Alexander J,etal. 2015. Enemy release and genetic founder effects in invasive killer shrimp populations of Great Britain[J]. Biological Invasions, 17(5): 1439-1451.
Brooks TM, Cuttelod A, Faith DP,etal. 2015. Why and how might genetic and phylogenetic diversity be reflected in the identification of key biodiversity areas?[J]. Philosophical Transactions of the Royal Society B, 370: 20140019. DOI: 10.1098/rstb.2014.0019.
Brown WM, George M, Wilson AC. 1979. Rapid evolution of animal mitochondrial DNA[J]. Proceedings of the National Academy of Sciences of USA, 176(4): 1967-1971.
Chen YF, He DK. 2002. Biodiversity in the Yangtze River basin fauna and distribution of fishes[J].Journal of Ichthyology, 42: 161-171.
Cheng F, Li W, Castello L,etal. 2015a. Potential effects of dam cascade on fish: lessons from the Yangtze River[J]. Reviews in Fish Biology and Fisheries, 3: 569-585.
Cheng F, Li W, Klopfer M,etal. 2015b. Population genetic structure and its implication for conservation ofCoreiusguichenotiin the upper Yangtze River[J]. Environmental Biology of Fishes, 98(9): 1999-2007.
Chiu TH, Kuo CW, Lin HC,etal. 2015. Genetic diversity of ivory shell (Babyloniaareolata) in Taiwan and identification of species using DNA-based assays[J]. Food Control, 48: 108-116.
Drummond AJ, Suchard MA, Xie D,etal. 2012. Bayesian phylogenetics with BEAUti and the BEAST 1.7[J]. Molecular Biology and Evolution, 29: 1969-1973.
Excoffier L, Lischer HEL. 2010. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 10: 564-567.
Goodall-Copestake WP, Tarling GA, Murphy EJ. 2012. On the comparison of population-level estimates of haplotype and nucleotide diversity: a case study using the gene cox1 in animals[J]. Heredity, 109: 50-56.
Guindon S, Gascuel O. 2003. A simple, fast and accurate method to estimate large phylogenies by maximum-likelihood[J]. Systematic Biology, 52: 696-704.
Lakshmanan LK, Gruber J, Halliwell B,etal. 2015. Are mutagenic non D-loop direct repeat motifs in mitochondrial DNA under a negative selection pressure?[J]. Nucleic Acids Research, 43(8): 4098-4108.
Larkin MA, Blackshields G, Brown NP,etal. 2007. Clustal W and clustal X version 2.0[J]. Bioinformatics, 23: 2947-2948.
Patwardhan A, Ray S, Roy A. 2014. Molecular markers in phylogenetic studies-a review[J]. Phylogenetic and Evolutionary Biology, 2: 131.
Perdices A, Cunhaa C, Coelhoa MM. 2004. Phylogenetic structure ofZaccoplatypus(Teleostei, Cyprinidae) populations on the upper and middle Chang Jiang (=Yangtze) drainage inferred from cytochrome b sequences[J]. Molecular Phylogenetics and Evolution, 31(1): 192-203.
Ramos-Onsins SE, Rozas J. 2002. Statistical properties of new neutrality tests against population growth[J]. Molecular Biology and Evolution, 19(12): 2092-2100.
Ronquist F, Huelsenbeck JP. 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 19: 1572-1574.
Swofford DL. 2002. PAUP*: phylogenetic analysis using parsimony (* and other methods), version 4[M]. Sunderland, Massachusetts: Sinauer Associates.
Tamura KG, Stecher D, Peterson AF,etal. 2013. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 30: 2725-2729.
Tang QY, Li XB, Yu D,etal. 2018.Saurogobiopunctatussp. nov. , a new cyprinid gudgen (Teleostei: Cypriniformes) from the Yangtze River, based on both morphological and molecular data[J]. Journal of Fish Biology, 92(2): 347-364.
Tao W, Jin XX, Xu TJ. 2013. The first complete mitochondrial genome fromBostrychusgenus (Bostrychussinensis) and partitioned Bayesian analysis of Eleotridae fish phylogeny[J]. Journal of Genetics, 92(2): 247-257.
Thiel C, Tsukamoto S, Tokuyasud K,etal. 2015. Testing the application of quartz and feldspar luminescence dating to MIS 5 Japanese marine deposits[J]. Quaternary Geochronology, 29: 16-29.
Wright S. 1978. Evolution and the genetics of populations volume 4: variability within and among natural populations[M]. Chicago: University of Chicago Press.
Yang M, Tian C, Liang XF,etal. 2014.Genetic structure and diversity in natural and stocked populations of the mandarin fish (Sinipercachuatsi) in China[J]. Genetics and Molecular Research, 14(2): 5153-5160.
Zhang X, Gao X, Wang JW,etal. 2015. Extinction risk and conservation priority analyses for 64 endemic fishes in the upper Yangtze River, China[J]. Environmental Biology of Fishes, 98(1): 261-272.
——美丽赤水河

