川滇蔷薇的种下分类系统和表型变异
吴 ,李慧敏,杨 维,杨锦红,陈 敏,李进昆,蹇洪英
(云南省农业科学院花卉研究所,云南昆明650205)
蔷薇属(Rosa L.)的分类系统非常复杂,这种复杂性是由演化过程如杂交、基因渐渗等导致的表型、基因型和生态型的变异和可塑性所共同产生的[1],这些因素之间又相互关联[2]。蔷薇属植物的表型可塑性给其分类带来了很大的麻烦,不同的分类系统对物种数的认定也各不相同,其种下分类更是复杂。
东喜马拉雅-横断山地区是蔷薇属植物的分布中心之一,横断山地区分布有蔷薇属植物45个种、18个变种和10个变型[3-4]。川滇蔷薇(R.soulieana Crép)是横断山地区的蔷薇属代表植物之一,广泛分布在海拔2 500~3 700 m的灌丛、山坡、溪边及农地旁,蕴藏着对月季品种改良和种质创新所需的耐干旱[5]、耐贫瘠和花香等重要的优异性状及相关基因,是一种非常重要的月季种质资源[6]。根据花序中小花数量、叶片大小及有无毛和有无腺等特征分为川滇蔷薇原变种(R.soulieana var.soulieana)、毛叶川滇蔷薇(R.soulieana var.yunnanensis)、大叶川滇蔷薇(R.soulieana var.sungpanensis)和小叶川滇蔷薇(R.soulieana var.microphylla),小叶川滇蔷薇和大叶川滇蔷薇分别只在西藏八宿和四川松藩有分布,毛叶川滇蔷薇主要分布在云南,原变种则在横断山地区广泛分布[7-8]。《中国植物志》和《Flora of China》均认为安徽省金寨县也有川滇蔷薇原变种分布。实际上,在野外调查和标本查阅中可以发现川滇蔷薇各变种之间的分类特征界限并不明显,过渡类型较多。那么,现有的种下分类系统是否真实地反映了川滇蔷薇种内的遗传变异?来自安徽的川滇蔷薇居群与来自横断山地区的川滇蔷薇居群之间的相互关系如何?川滇蔷薇种内的表型变异究竟如何?为了弄清这3个问题,有必要对来自横断山地区的川滇蔷薇居群以及来自安徽金寨的川滇蔷薇居群进行种下分类系统和表型变异研究。
形态标记是检测遗传多样性大小和分布的传统标记,形态特征多样性常被用来估计居群间或居群内的遗传变异[9]。单拷贝核基因遵循双亲遗传方式,能提供大量信息位点,且能减少旁系同源(paralog)的影响[10]。本研究利用2个单拷贝核基因片段:3-磷酸甘油醛脱氢酶基因(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、叶绿体表达的谷氨酰胺合成酶基因(chloroplast-expressed glutamine synthetase,ncpGS)以及43个形态特征对川滇蔷薇来自横断山地区的38个居群和来自安徽金寨的1个居群进行聚类分析,探讨川滇蔷薇的种下分类系统,同时研究其表型多样性及表型变异特征。
1 材料与方法
1.1 研究材料
根据标本记录,在有标本和分布记录的横断山地区以及安徽省金寨县进行川滇蔷薇的标本和分子材料采集。采样策略遵循居群生态学采样的原理和方法[11],同时用GPS记录居群的详细信息,如经度、纬度和海拔(表1)。
1.2 研究方法
1.2.1 基于单拷贝核基因片段的居群间系统关系 (1)总DNA提取和PCR扩增。每个居群随机选择1个个体,用植物总DNA提取试剂盒(Plant DNA-Easy kit,北京百泰克公司)进行所有样品[包括丽江蔷薇(R.lichiangensis Yüet Ku)和野蔷薇(R.multiflora Thunb)2个外类群]总DNA的提取。选择GAPDH和ncpGS这2个单拷贝核基因作为目的片段。GAPDH的引物序列为:GPDX7-F,5′-GATAGATTTGGAATT GTTGAGG-3′;GPDX11-R,5′-GACATTGAATGAGATAAA CC-3,PCR反应程序参照 Joly等的方法[12]。ncpGS的引物序列为:GScp687-F,5′-GATGCTCACTACAAGGCTTG-3′;GScp994-R,5′-AATGTGCTCTTTGTGGCGAAG-3′[13],PCR反应程序参照Meng等的方法[14]。用2%琼脂糖凝胶检测PCR扩增产物的质量和浓度,最终产物送上海美吉生物技术公司进行序列测定。
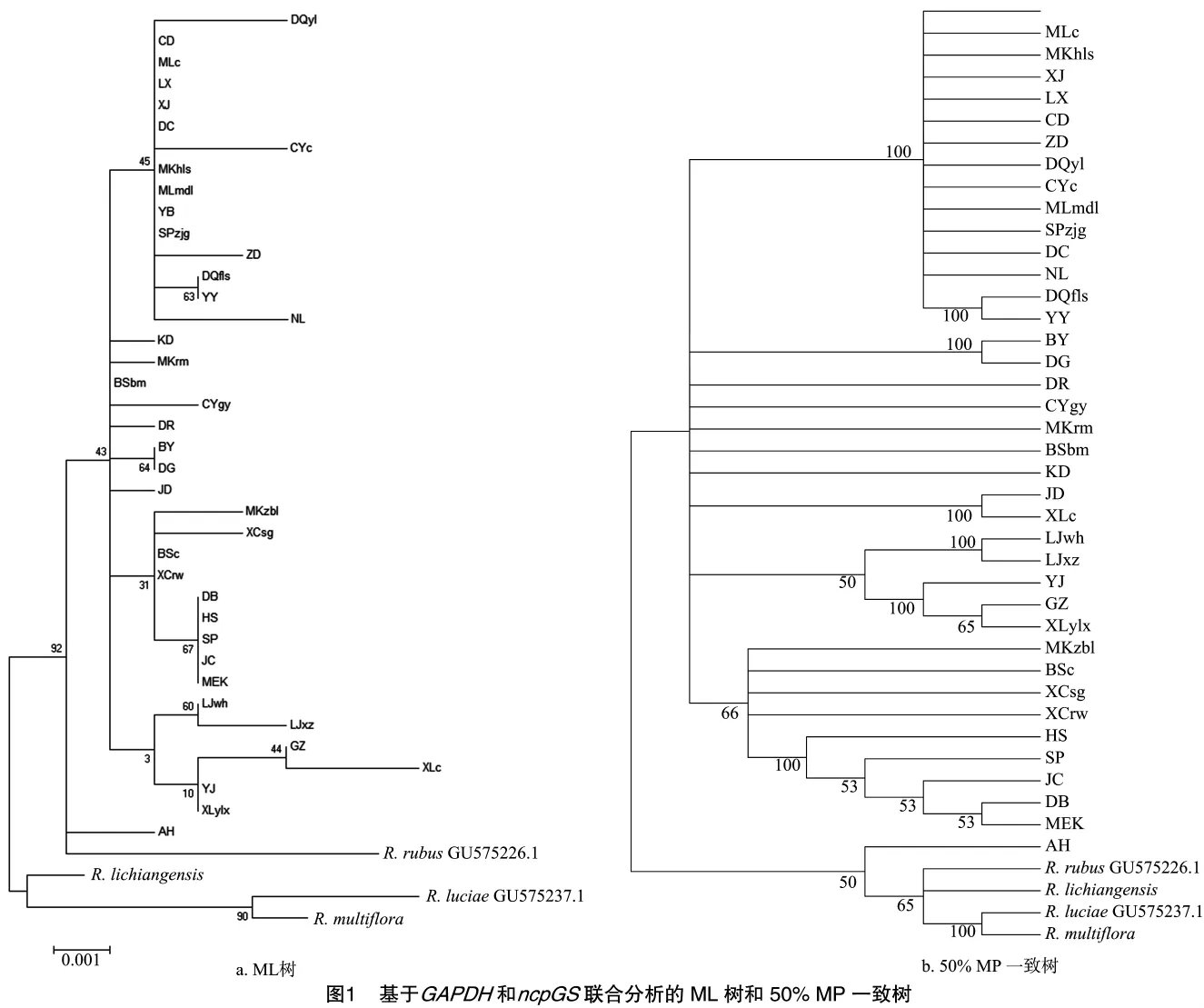
(2)数据分析。采用Chromas软件对测序结果进行核对,核对后的单条序列用 DNAStar(gene codes corporation,USA)进行序列拼接,对于测序结果中存在的杂合位点用简并碱基进行代替。从 GeneBank(http://www.ncbi.nlm.nih.gov/genebank)下载光叶蔷薇(R.luciae Franch.et.Roch.)和悬钩子蔷薇(R.rubus Lével.)的相应序列。采用 Clustal ver.181[15]对所有序列进行比对,用 BioEdit ver.7.0.4.1软件[16]进行修正。采用 Mega 6.0软件[17]分别构建基于最大似然法的ML树和最大简约法的50%一致树。
1.2.2 基于形态数据的居群间聚类关系 形态数据的编码及测量:根据《中国植物志》对蔷薇主要是川滇蔷薇及其变种的分类特征的描述,选取与枝、刺、叶和花等形态相关的43个表型性状进行编码,共包含14个二元性状、2个多元性状和27个直接数量性状。对含安徽金寨居群在内的花和叶形态特征较完善的31个居群的所有个体进行形态特征测量,测定值或计数值即为编码(表2)。每个个体每个数量性状重复3次,与叶相关的性状以花枝上中间部位的复叶为测量对象;与花相关的性状则以完全展开的花朵为测量对象。用游标卡尺测定花梗长、萼筒的长与宽,并计算萼筒的长宽比。
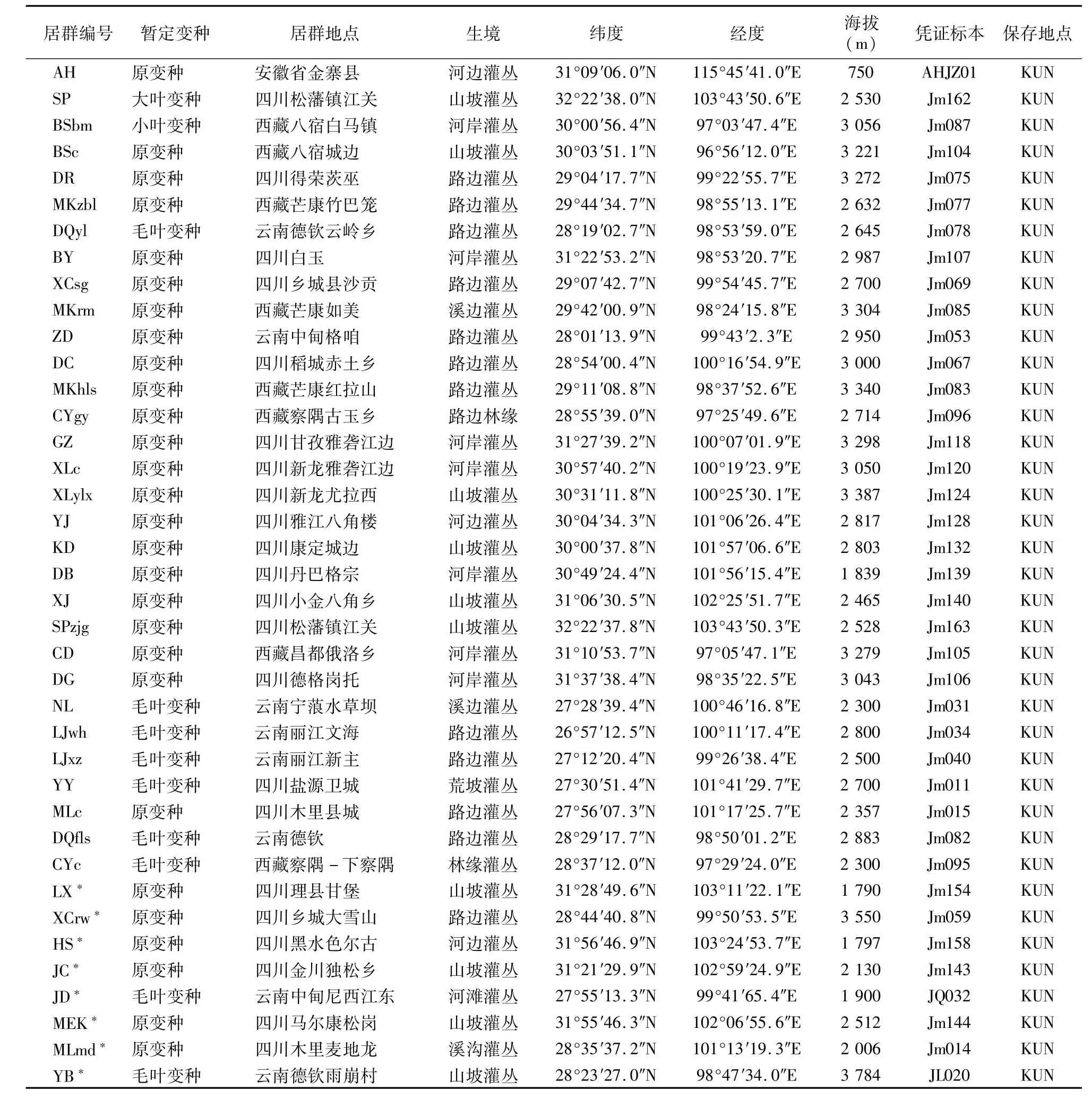
表1 川滇蔷薇居群信息
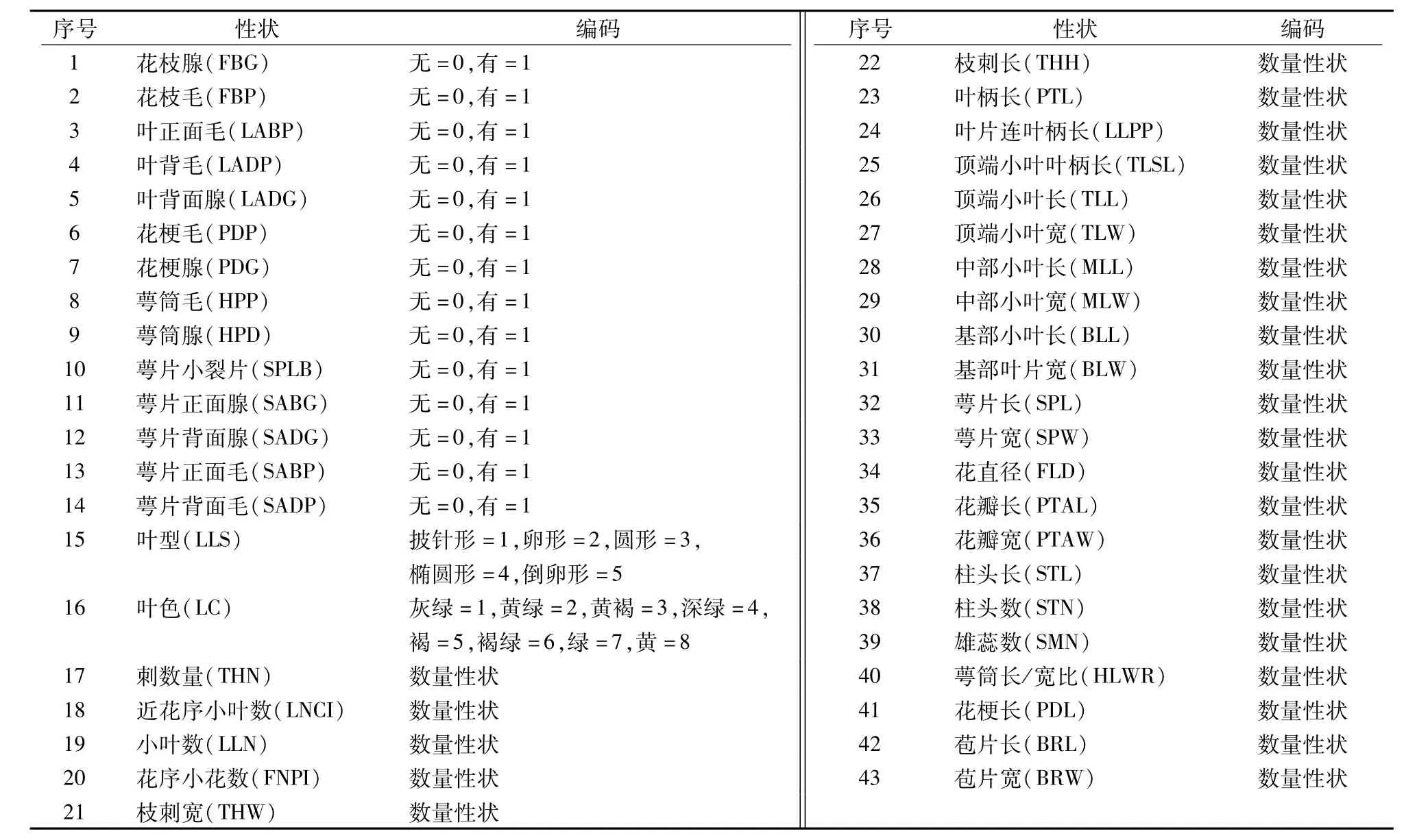
表2 表型性状及编码
形态数据分析方法:采用NTSYS pc ver.2.1对形态数据进行标准化,然后进行居群的PCA分析和居群间基于表型的UPGMA聚类。
1.2.3 表型多样性和居群间表型变异的研究方法 形态数据的测量方法见“1.2.2”节,主要用27个数量性状对根据单拷贝核基因和表型聚类结果已确定为川滇蔷薇的来自横断山的30个居群的所有个体进行测量。采用SPSS13.0软件进行居群各数量性状的均值(Mean)、标准差(SD)和变异系数(CV)的统计,用SAS 9.0软件进行各直接数量性状在居群间和居群内的方差分析。其中,表型分化系数的计算同杨维泽等的方法[18]。
2 结果与分析
2.1 基于GAPDH和ncpGS居群间亲缘关系
2.1.1 序列特征 比对后ncpGS基因数据矩阵长700 bp,有
21个变异位点,其中13个是信息位点。比对后的GAPDH基因数据矩阵长719 bp,有57个变异位点,其中28个是信息位点。2个片断联合后形成的数据矩阵长1 419 bp,有78个变异位点,其中41个是信息位点。
2.1.2 居群间系统关系 从图1可以看出,来自横断山的所有川滇蔷薇居群总是单独地成为一支,在ML树中与来自安徽(AH)的材料和悬钩子蔷薇一起成为并系,说明来自安徽的材料可能不属于川滇蔷薇。50%MP一致树也表明,来自横断山的川滇蔷薇居群单独成为一支,而来自安徽的材料则以50%的靴带值(bootstrap值)与来自合柱组的其他种单独构成一支。因此,来自安徽的材料不是川滇蔷薇。在川滇蔷薇内,来自横断山的居群并没有严格地按照地理距离或种下分类系统的4个变种进行聚类。如根据形态特征被定名为大叶川滇蔷薇的松藩居群(SP)在ML树和50%MP一致树中都与被定名为川滇蔷薇原变种的丹巴(DB)、黑水(HS)、金川(JC)和马尔康(MEK)等居群聚在一起;被定名为原变种的小金(XJ)和理县(LX)等居群又与被定名为毛叶变种的宁蒗(NL)、德钦云岭(DQyl)等居群聚在一起。
2.2 基于表型性状的居群间相互关系
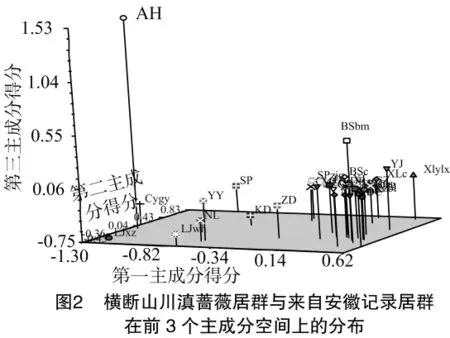
2.2.1 不同居群基于表型性状的主成分分析 对含安徽居群在内的39个居群的主成分分析,结果(表3)表明,对第一主成分贡献最大的是顶端小叶长(TLL)、中部小叶长(MLL)、叶片连柄长(LLPP)、顶端小叶柄长(TLSL)、基部小叶长(BSL)、叶柄长(PTL)、中部小叶宽(MLW)、顶端小叶宽(TLW)、基部小叶宽(BSW)以及花序的小花数(FNPI)。对第二主成分贡献最大的是花直径(FLD)、花瓣宽(PTAW)、花瓣长(PTAL)和萼片宽(SPLW)。对第三主成分贡献最大的是苞片长(BRL)、苞片宽(BRW)、柱头长(STL)以及柱头数量(STN)。第一主成分的贡献率为33.26%,第二和第三主成分的贡献率分别为18.15%和10.82%,前3个主成分的累计贡献率为62%,说明可以应用主成分分析法进行39个居群的表型聚类分析。从图2可以看出,AH居群与来自横断山地区的川滇蔷薇居群形态差异较大。AH居群的顶端小叶叶柄长可达1.06 cm,花梗长1.64 cm,均远大于来自横断山的川滇蔷薇居群。虽然其叶片、顶端小叶叶柄长及花梗长等均较大,但其花部特征偏小,主要是花的直径小(2.26 cm)、花瓣窄(1.17 cm)、花柱短(0.381 cm)、萼筒近圆形。来自横断山地区的丽江文海(LJwh)、丽江新主(LJxz)、察隅古玉(CYgy)、宁蒗(NL)、盐源(YY)、康定(KD)、中甸(ZD)和松藩(SP)等居群间关系较松散,而其他居群之间的表型差异较小、亲缘关系较近。
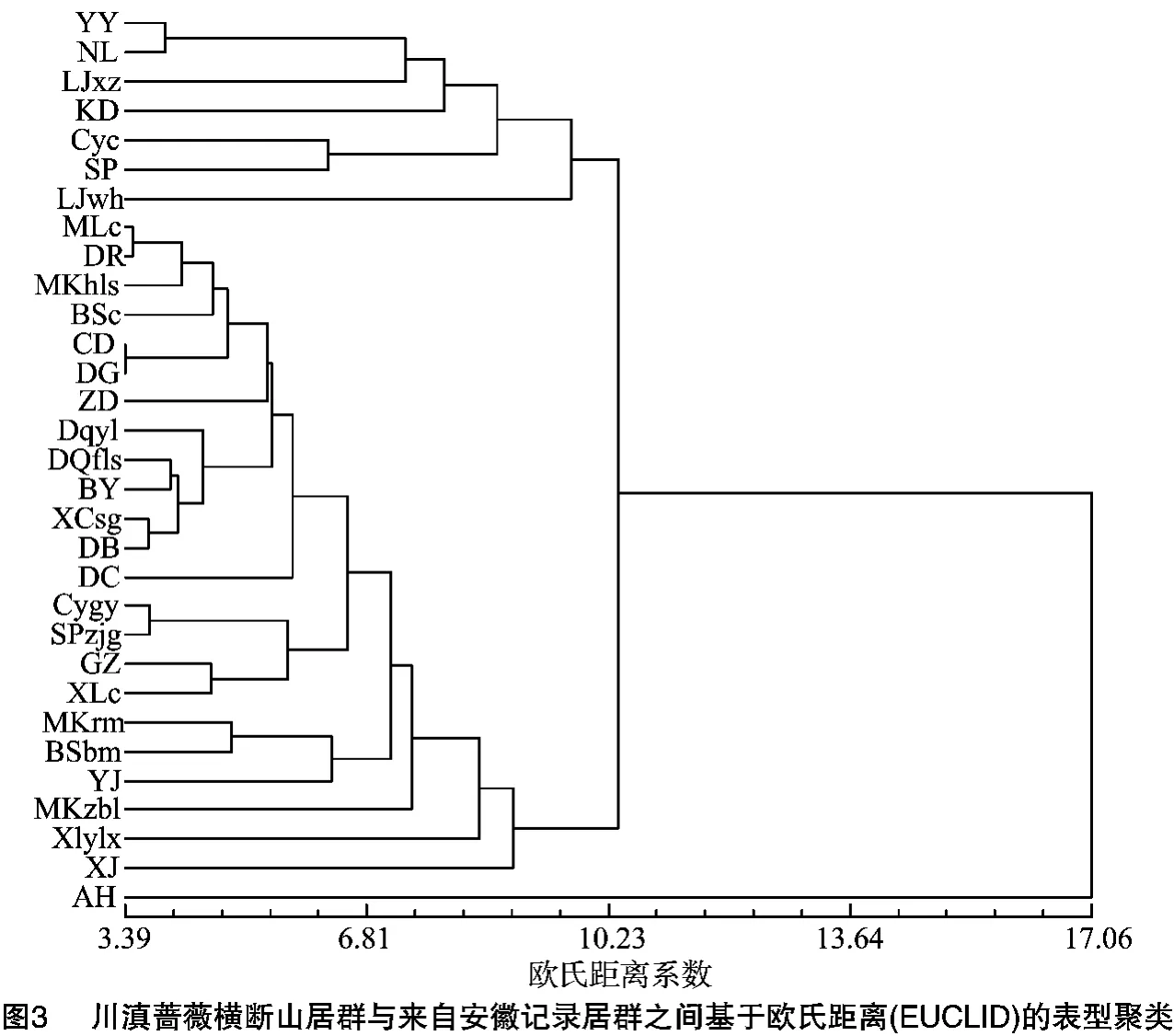
2.2.2 不同居群基于表型性状的UPGMA聚类 各居群基于欧氏距离(EUCLID)的聚类结果(图3)表明,安徽(AH)群体离其他居群的遗传距离最远,而分布于横断山地区的其他居群并未严格按地理距离进行聚类。虽然部分居群间由于地理距离近且表型相近而聚为一支,但不是所有地理距离相近的居群聚在一起,另有其他与之距离很远的居群与之聚在一起,如盐源、宁蒗和丽江新主居群形成了一个小分支,与来自康定的居群聚为一支;来自松藩和察隅的居群聚为一支;这2支最后又与来自丽江文海的居群聚为一大支,与来自藏东南和川西的其他居群所构成的另一大支并列。来自藏东南和川西的其他居群所构成的另一大支也同样存在类似的情况。由此可见,基于表型性状的居群间也没有根据种下变种进行聚类。
2.3 川滇蔷薇居群的表型多样性及表型分化
2.3.1 川滇蔷薇表型性状的变异特征 川滇蔷薇27个直接数量性状在居群间的差异均达到极显著水平(P<0.01,表4)。与叶相关的性状在居群内的差异均达到了极显著水平(P<0.01),而除花序小花数(FNPI)外,与花相关的性状在居群内的差异均不显著(P>0.05)。花序小花数和皮刺数量(THN)在居群内的变异最大,30个居群的平均变异系数分别为59.8%和45.29%,萼片宽(SPW)和叶片小叶数(LLN)在居群内最稳定,30个居群的平均变异系数分别为11.79%和12.58%。
2.3.2 川滇蔷薇群体间的表型分化 川滇蔷薇27个数量性状的平均表型分化系数为66.36%,说明居群间的表型变异是川滇蔷薇表型变异的主要来源。刺数量、近花序叶片小叶数、萼片长度的表型分化系数小于50%,表明这些性状的遗传变异主要来自于居群内,而其他性状的遗传变异则主要来源于居群间(表5)。
3 讨论与结论
3.1 川滇蔷薇是横断山地区的特有种,安徽没有川滇蔷薇分布
《中国植物志》和《Flora of China》都认可安徽有川滇蔷薇的分布[7-8],但王春景等通过对有关资料记录和标本的查看,实地调查走访却并未采到实物[19]。本研究所采用的安徽居群采自北京植物所标本管(PE)的川滇蔷薇标本记录地点。基于2个单拷贝核基因的居群间系统关系表明,来自安徽的材料与其他38个来自横断山地区的川滇蔷薇居群的遗传距离较远。虽然支持率不高,但ML树、50%的MP严格一致树均显示其不属于川滇蔷薇,与川滇蔷薇、悬钩子蔷薇、野蔷薇和丽江蔷薇成为并系。在基于表型性状的聚类分析和主成分分析中,来自安徽的材料与其他来自横断山地区的川滇蔷薇相距很远。此外,由于有报道的安徽省内分布的蔷薇属合柱组全缘托叶系只有川滇蔷薇、软条七蔷薇、琅琊蔷薇(R.langyashanica)和悬钩子蔷薇[19],其中琅琊蔷薇的叶片边缘有较深的尖锐锯齿,软条七蔷薇的小叶多为3~5个,且较大、容易识别,悬钩子蔷薇与川滇蔷薇从现有的检索表中进行检索时常因叶背有无毛等容易误判。因此,川滇蔷薇是横断山地区的特有种,安徽没有川滇蔷薇分布,以前被认为是川滇蔷薇的来自安徽的标本可能是悬钩子蔷薇。
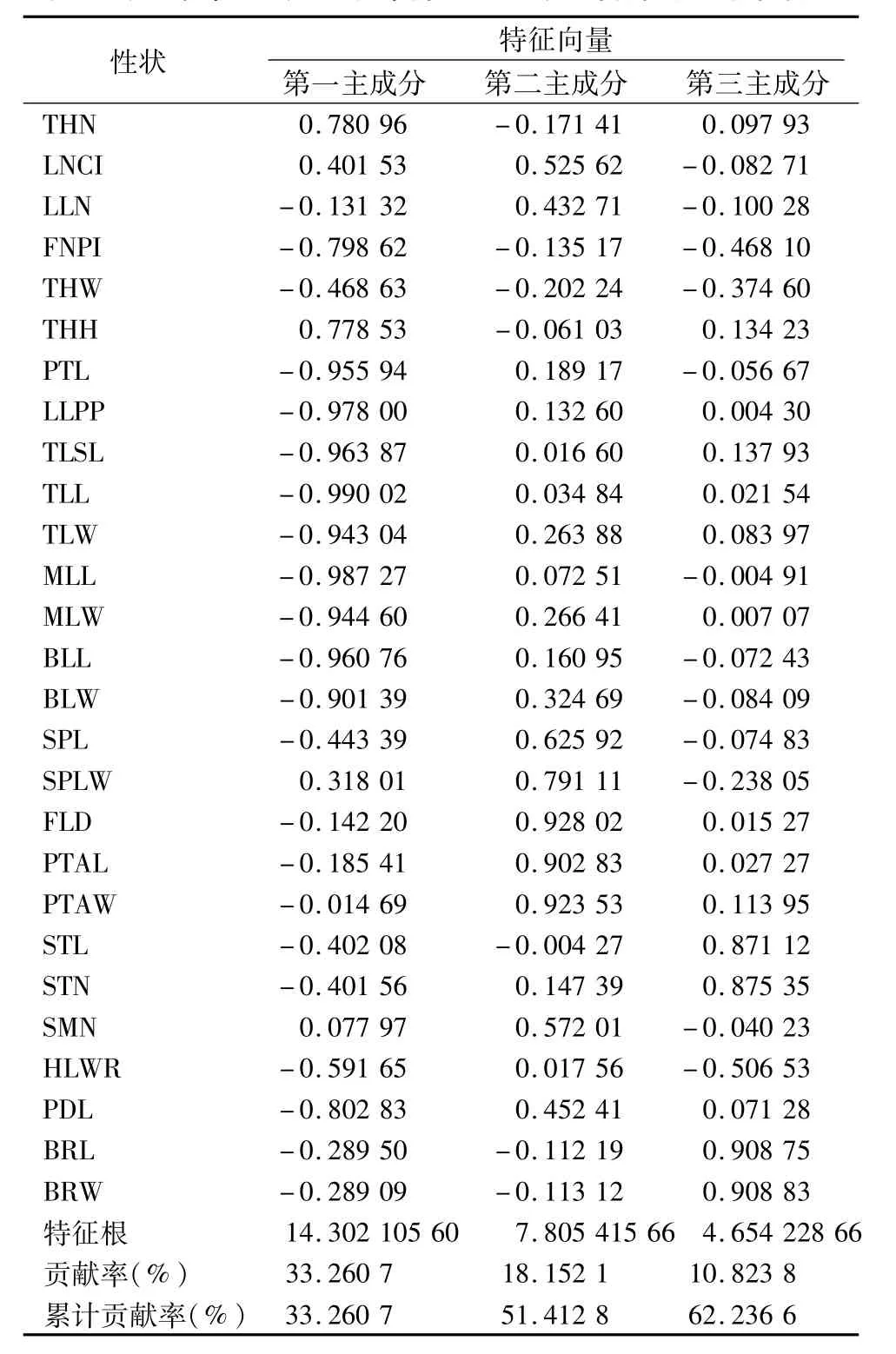
表3 表型性状主成分分析中前3位主成分的特征根和特征向量
3.2 川滇蔷薇基于单拷贝核基因和形态特征的种下分类系统
Stebbins指出,“植物分类引用了亚种、变种、变型等种下单位,这种分类方法所造成的混乱多于它所要澄清的混乱”[20]。其中,变种是指具有形态生理、遗传特征差异但在地理分布上可能重叠的群体,有时也指未弄清地理分布的亚种。
随着进化植物学的发展,种下分类越来越比较客观地反映出植物各类群间的亲缘关系,尤其是种下分类单位,从居群遗传和变异的角度阐释得更加清楚[21]。根据居群间基于核基因和形态特征的聚类分析以及形态特征的PCA分析可知,根据叶片等表型性状来进行的川滇蔷薇种下分类系统并不能反映其种内的遗传变异。表型聚类除来自安徽的材料外,其他30个居群直接聚类成两大支。第一大支与第二大支的主要区别在于花序上的花朵数和小叶片大小。第一大支的花朵数都在10朵以上,叶片中部和顶端小叶片长于2 cm,然而居群间基于核基因的聚类既没有反映形态变异,也没有按照地理距离进行。这可能是由于川滇蔷薇主要分布在横断山地区的干暖河谷及其支流河岸,有时也延伸到海拔3 700 m以下的山坡路边和灌丛中。虽然河流和峡谷有利于川滇蔷薇同一流域的居群间基因交流和迁移,但分布于同一流域不同纬度、不同海拔即使具有相同或相近的遗传背景(基因型)的居群及个体,也会由于对环境的适应而形成许多不同的形态特征。
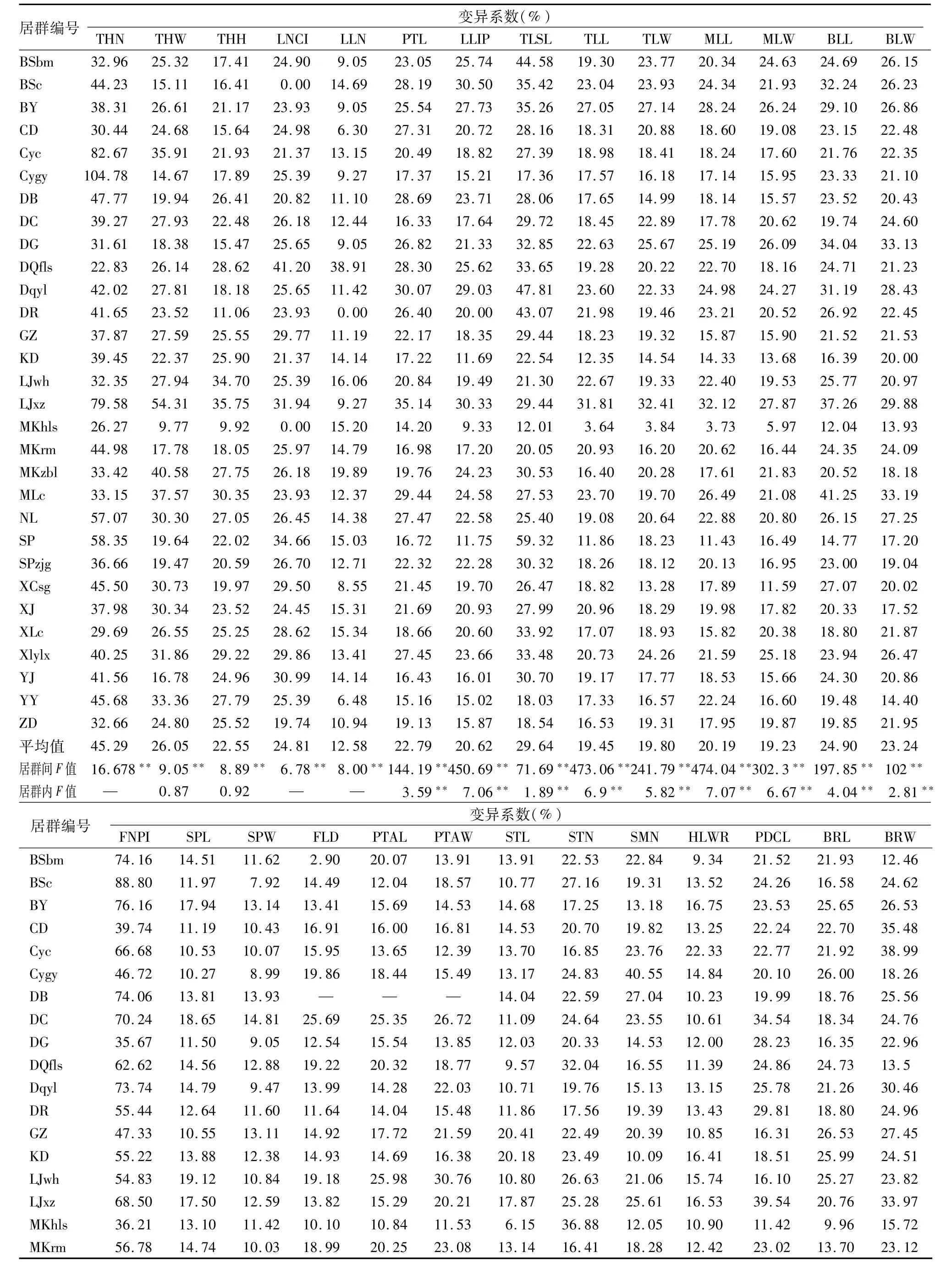
表4 川滇蔷薇各居群表型性状的变异系数
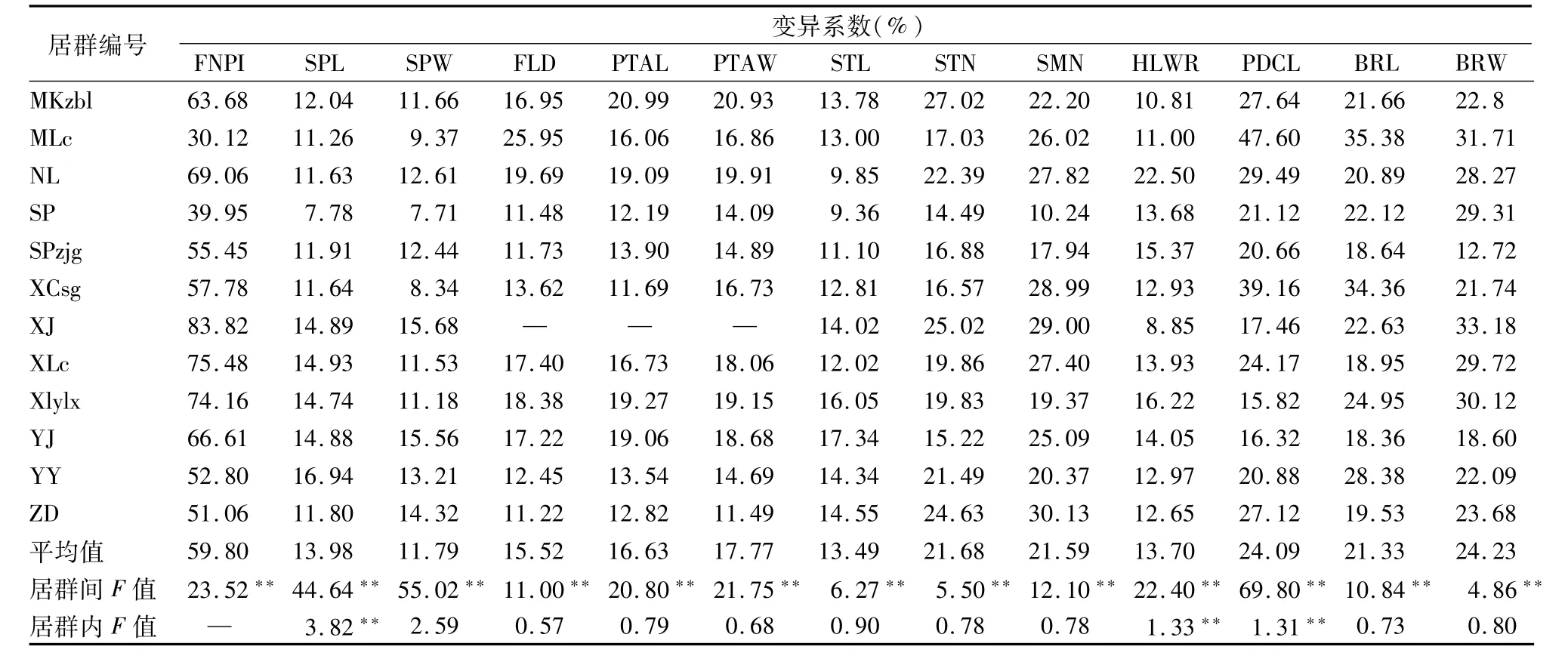
续表4
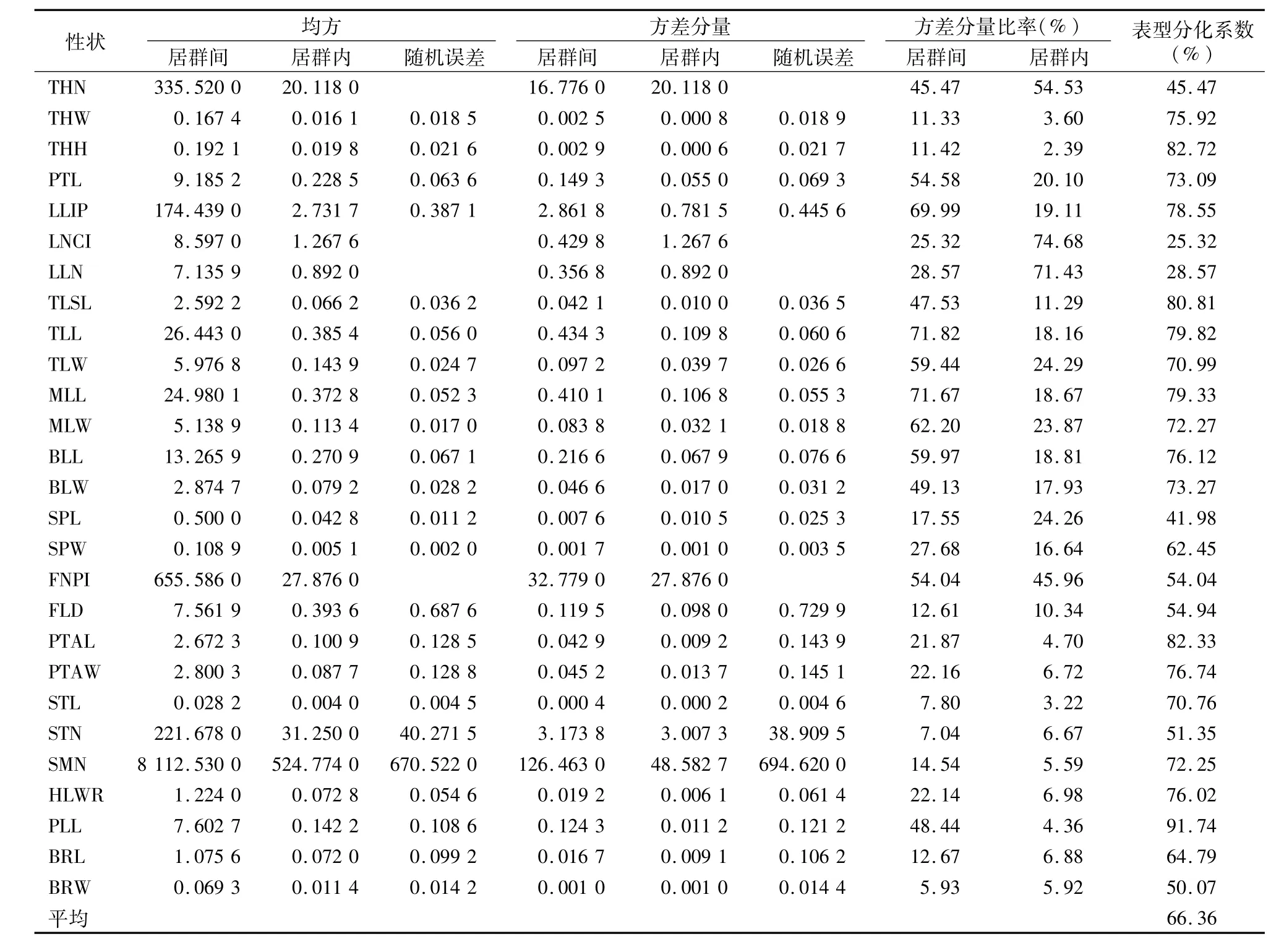
表5 川滇蔷薇直接表型数量性状的方差分量及群体间分化系数
3.3 川滇蔷薇的表型多样性和表型分化
表型差异可能并不与潜在的基因突变数目成比例。性状的表达主要取决于性状本身,取决于是单基因或是多基因控制。此外,对蔷薇属植物来说,其发育阶段、对环境的可塑性程度等对表型差异的影响并非一成不变[22]。唐开学等分别对川滇蔷薇、峨眉蔷薇(R.omeiensis Rolfe)、大花香水月季[R.odorata(Andr.)Sweet var.gigantea(Crép.)Rehd.etWils.]和中甸刺玫(R.praelucens Byhouwer)等分布于云南的居群进行基于表型性状的多样性研究,结果显示,它们的表型在居群间和居群内均存在丰富的变异,但不同物种的表型变异与地理距离之间的关系不同。其中,川滇蔷薇在云南省内几个居群的表型多态性丰富,变异主要来源于居群间[23-26]。本研究中,川滇蔷薇整个分布区30个居群的27个表型性状观测值差异均达到极显著水平,变异系数变幅为 11.79% ~5980%。川滇蔷薇存在丰富的表型多样性,表型分化系数为66.36%,说明居群间的表型变异是川滇蔷薇表型变异的主要来源,再次表明在横断山地区因其特殊的环境特点,广泛分布的川滇蔷薇表型在种内分化剧烈,居群间存在显著的表型变异。这种变异是否是其遗传变异的完全体现,尚待进一步通过分子水平的遗传多样性进行研究验证。
参考文献:
[1]Cock K D.Genetic diversity ofwild roses(Rosa spp.)in Europe,with an in depth morphological study of Flemish populations[D].Ghent:Ghent University,2008.
[2]Wissemann V.Evolution by hybridisation.The influence of reticulate evolution of biosymmetrical patterns and processes in plants[J].Theory in Biosciences,2005,123(3):223-233.
[3]中国科学院青藏高原综合科学考察队.横断山区维管植物(下册)[M].北京:科学出版社,1994.
[4]Jian H Y,Li SF,Li SB,et al.Diversified and valuable wild roses in the Hengduan Mountains, Southwestern China[J]. Acta Horticulturae,2015(1064):39-45.
[5]周志琼,包维楷,吴福忠,等.岷江干旱河谷黄蔷薇和川滇蔷薇更新能力及其限制因素[J].生态学报,2009,29(4):1931-1939.
[6]蹇洪英.川滇蔷薇的遗传分化及地理分布格局形成研究[D].昆明:中国科学院大学,2015.
[7]谷粹芝.中国蔷薇科植物分类之研究(六)[J].植物研究,1990,10(1):1-15.
[8]Ku T C,Robertson K R.Rosa(rosaceae)[M].Louis:Missouri Botanical Garden Press,2003:339-381.
[9]Ayele T B,Gailing O,Finkeldey R.Assessment and integration of genetic,morphological and demographic variation in Hagenia abyssinica(Bruce)J.F.Gmel to guide its conservation[J].Journal for Nature Conservation,2011,19(1):8-17.
[10]Duarte JM,Wall P K,Edger P P,et al.Identification of shared single copy nucear genes in Arabidopsis,Populus,Vitis and Oryza and their phylogenetic utility across various taxonomic levels[J].BMC Evolutionary Biology,2010,10(1):61-78.
[11]钱迎倩,马克平.生物多样性研究的原理和方法[M].北京:中国科学技术出版社,1994.
[12]Joly S,Starr JR,LewisW H,etal.Polyploid and hybrid evolution in roses eastof the Rocky Mountains[J].American Journal of Botany,2006,93(3):412-425.
[13]Emshwiller E,Doyle J J. Chloroplast- expressed glutamine synthetase(ncpGS):potential utility for phylogenetic studieswith an example from Oxalis(Oxalidaceae)[J].Molecular Phylogenetics and Evolution,1999,12(3):310-319.
[14]Meng J,Fougere-Danezan M,Zhang L B,et al.Untangling the hybrid origin of the Chinese tea roses:evidence from DNA sequences of single-copy nuclear and chloroplastgenes[J].Plant Systematics and Evolution,2011,297(3/4):157-170.
[15]Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface:flexible strategies formultiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876-4882.
[16]Hall T A.BioEdit:a user friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J].Nucleic Acids Symposium Series,1999,41:95-98.
[17]Tamura K,Stecher G,Peterson D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[18]杨维泽,金 航,杨美权,等.云南滇龙胆居群表型多样性及其与环境关系研究[J].西北植物学报,2011,31(7):1326-1334.
[19]王春景,周守标,黄文江,等.安徽蔷薇属植物资源及其开发利用[J].中国野生植物资源,2005,24(1):32-35.
[20]Stebbins G L.Variation and evolution in plants[M].New York:Columbia University Press,1970.
[21]沈显生,张锡亭.试论系统植物学和进化植物学中的种下分类单位[J].湘潭师范学院学报(社会科学版),1996,17(3):43-46.
[22]Werlemark G.Genetic variability and reproductive strategies on Nordic dogroses,Rosa section Caninae[D].Uppsala:Swedish University of Agricultural Sciences,2000.
[23]唐开学.云南蔷薇属种质资源研究[D].昆明:云南大学,2009.
[24]周宁宁,唐开学,邱显钦,等.云南峨眉蔷薇天然居群的表型多样性[J].西南农业学报,2009,22(6):1732-1736.
[25]邵珠华,李名扬,邱显钦,等.大花香水月季天然群体表型多样性研究[J].江苏农业科学,2010(2):184-187.
[26]李树发,李纯佳,蹇洪英,等.云南香格里拉特有易危植物中甸刺玫的表型多样性[J].园艺学报,2013,40(5):924-932.

