一种简单的gRNA体外筛选方法
成大欣, 徐丽然, 于清清, 高守翠, 王晓靖, 刘 懿, 刘恩岐, 赵四海
(1. 陕西省人民医院新生儿科, 西安 710068; 2. 西安交通大学医学部动物中心, 西安 710061)
规律成簇的间隔短回文重复及其相关蛋白9(Clustered regularly interspaced short palindromic repeats/ CRISPR associated protein 9, CRISPR/Cas9)基因编辑技术是目前最热门的基因编辑技术,科学家们可利用引导核糖核酸(guide ribonucleic acid,gRNA)引导Cas9核酸酶在多种细胞的特定基因组位点上进行切割、修饰[1-3]。很长时间以来, 胚胎干细胞(embryonic stem cell, ES细胞)只有在小鼠中比较成功地进行了体外培养建系, 在其他动物, 比如家兔等, 虽然取得一定进展, 但离实际应用仍有距离[4]。在基于ES细胞的基因敲除技术应用上, 由于除小鼠外的动物缺乏ES细胞, 故难在实现在体的基因敲除[4-6]。由于CRISPR/Cas9技术的高效性(成功率可达90%以上), 可以直接在胚胎上进行操作, 不用借助ES细胞, 这就突破了基于ES细胞同源重组技术的种属限制[1,7-9]。CRISPR/Cas9技术使得在不同物种进行基因编辑成为可能,基因修饰技术实施起来变得更便利,基因修饰动物的应用得到进一步扩大[1-3]。除小鼠外的基因修饰动物模型, 如家兔[7-11]、大鼠[12,13]、猪[14]和灵长类[15]基因修饰动物模型, 也快速发展起来。CRISPR/Cas9技术的实施需要研究者自行设计gRNA, 引导Cas9核酸酶进行切割。如何筛选gRNA,在体外验证其结合靶序列引导Cas9核酸酶进行切割的能力, 往往在实验前(如受精卵注射)需要进行筛选。现有利用体外培养细胞验证gRNA效率的方法相对较复杂, 花费较大。本实验以家兔扭转蛋白2A(torsin family 2 member A,TOR2A)基因的gRNA设计和筛选为例, 介绍一种简单的gRNA体外筛选方法(图1)。相比传统的gRNA制备方法, 该方法简单易行, 不用将打靶序列克隆进质粒[16]。
1 材料与方法
1.1 试剂与仪器
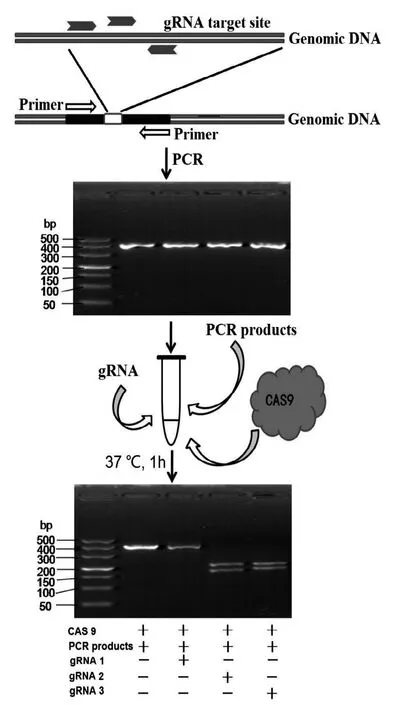
图 1 gRNA打靶效率的筛选Figure 1 In vitro target efficiency screening of gRNAs
pX330 (编号71707) 质粒由密歇根大学张继峰博士惠赠; 琼脂糖(西班牙Biowest公司); Tris和EDTA(美国Genview公司); 基因组DNA提取试剂盒和胶回收试剂盒(美国Omega Bio-Tek公司); T7-ScribeTMStandard RNA IVT Kit(美国 CELLSCRIPT公司); miRNeasy Mini Kit(美国QIAGEN公司); Cas9内切酶(美国NEB公司); PCR mix(中国擎科新业生物公司); PCR仪(德国Eppendorf公司); 电泳仪(中国北京六一生物科技有限公司); NanoDrop超微量紫外-可见光分光光度计(美国Thermo公司); 凝胶成像系统(中国上海培清科技有限公司); 其它试剂均为国产分析纯。
1.2 gRNA的在线设计
以家兔TOR2A(TOR2A ENSOCUG00000006907)为例,根据分析,将2号外显子序列用在线软件设计打靶用gRNA,可选用如http://crispr.mit.edu/或http://zifit.partners.org/ZiFiT/ChoiceMenu.aspx等在线工具。
1.3 gRNA的扩增与纯化
以pX330质粒为模板,正向引物为(用以上选定gRNA序列替换引物中N): TGTAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNN-gttttagagctagaaatagc; 反向引物: AGCACCGACTCGGTGCCACT。PCR程序为: 98 ℃预变性 2 min, 94℃变性10 s, 68 ℃退火 30 s,72 ℃延伸1min,30个循环,72 ℃延伸10 min。质量分数2%琼脂糖凝胶电泳,切割目的片段的凝胶,按胶回收试剂盒说明回收PCR产物。分别以回收产物为模板,按照T7-ScribeTMStandard RNA IVT Kit说明书, 选用20 μL体系, 将3个含gRNA序列的PCR产物转录为RNA。转录产物用miRNeasy Mini Kit纯化gRNA,纯化后的gRNA用NanoDrop定量,-80℃备用。
1.4 含打靶序列的PCR扩增与胶回收
抽取普通级家兔(西安交通大学医学部实验动物中心,SCXK(陕)2012-003)血液200 μL,按基因组提取试剂盒说明,提取基因组DNA用做模板。一般PCR目的片段可设计在300~1 000 bp。在TOR2A目的基因扩增中, 正向引物: GCCGTCTGGAAGGCTCT; 反向引物: GGAGTGACTGTCACCAGCC,产物为386 bp。98 ℃预变性 2 min,94 ℃变性 10 s,67 ℃退火 30 s,72 ℃延伸 1 min,30个循环,72 ℃延伸10 min。质量分数2%琼脂糖凝胶电泳,切割目的片段凝胶,按胶回收试剂盒说明书回收PCR产物。
1.5 体外gRNA引导的Cas9酶切效率鉴定
2 结果
2.1 gRNA的设计、扩增与与纯化
根据评分和脱靶评估情况,最终选了3个gRNA打靶序列。分别为gRNA1: GGACCTTAAGAGCTGGGTCC; gRNA2: GCTTGTCCATCTCGTCGAAG; gRNA3: TCTCTTCCTCTTCGACGAGA。用以上序列替换正向引物中的N(TGTAATACGACTCACTATAGGNNNNNNNNNNNNNNNNNNNN-gttttagagctagaaatagc)gRNA的扩增和转录均按试剂盒数说明进行,纯化回收良好。
2.2 gRNA的引导酶切效率的鉴定
结果显示, gRNA2和gRNA3引导的Cas9酶切效率比较理想, gRNA1效果不佳(图1)。这提示在体内实验时, gRNA2和gRNA3可能是更为理想的选择。
3 讨论
CRISPRs广泛分布于细菌和古菌基因组中的一种序列[17]。研究[18,19]证实CRISPR通过产生RNA(crRNA)与CRISPR-associated protein相结合形成识别催化结构,识别外源DNA序列并通过切除突变的方式达到防御自身的作用,又称CRISPR防御体系。科学家将crRNA和转录激活transactivating RNA(tracrRNA),它们一起形成双链RNA并与Cas9结合,由RNA识别DNA序列驱动Cas9定位后,由 Cas9内切酶对基因组DNA进行切割[1]。虽然CRISPR/Cas9技术的切割效率很高,而且对于gRNA设计的要求也不高,但研究人员往往面临如何筛选多条符合基因打靶要求的gRNA。筛选一条或几条有效的gRNA, 也是实验进行顺利的保证[8,9]。
本文根据以往经验建立的这种体外筛选方法,不需特殊仪器和试剂,实用性强。通过将设计好的gRNA转录后,与含目的打靶片段的PCR产物,Cas9酶共同体外反应,筛选其指导酶切的效率。相比转染细胞后再进行筛选,受精卵或胚胎显微操作后培养至囊胚再基因组扩增并测序等检验策略,本方法更简单易行,适合初步筛选[20]。而且判定方法也简单,不需要特殊仪器和试剂,可以在不同研究层次的实验室进行。本文中,我们以针对家兔TOR2A基因的gRNA设计与检测的过程为例,阐述了该种方法。关于gRNA的体外筛选方法,也可在培养细胞系来进行,但往往需要荧光检测仪器,对于一些实验室来说不是很方便,而且实验成本高。本文阐述的方法,实验条件限制小,可操作强,可作为基因打靶gRNA的体外筛选,是细胞系筛选的有益补充, 也是后期在体实验的保证。
当然, 本法是在体外/细胞外进行的筛选, 其反应条件与体内存在一定差异。但基于CRISPR/Cas9技术的高效率和高特异性,经过本法筛选后,基本达到了实验要求。尤其是在排除相对低效的gRNA方面,有很重要意义。根据我们的后期体内实验也基本验证了初筛的效果。例如,在制作腺苷酸环化酶9(ADCY9)基因敲除兔时,通过体外筛选后,我们对新生仔兔体内验证表明其敲除率为76.5% (数据尚未发表)。
综上所述,我们描述了一种快速体外筛选gRNA引导Cas9酶切效率的方法,该方法简单可行,为研究者筛选gRNA提供了便利。
参考文献:
对甲状腺功能求得的TGI指数为:≥51岁TGI=242,女性TGI=102,已婚TGI=104。可知年龄最大的一组对甲状腺功能的偏好性最强,且远超其它年龄段。在不同性别和婚姻状况中,差异较小。
[1] Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121):819-823.
[2] Hwang WY, Fu Y, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nat Biotechnol,2013, 31(3):227-229.
[3] Moreno-Mateos MA, Vejnar CE, Beaudoin JD, et al.CRISPRscan: designing highly efficient sgRNAs for CRISPRCas9 targetingin vivo[J]. Nat Methods, 2015, 12(10):982-988.
[4] Wang Y, Zhao S, Bai L, et al. Expression systems and species used for transgenic animal bioreactors[J]. Biomed Res Int,2013, 2013:580463.
[5] Zhao S, Li Y, Gao S, et al. Autocrine human urotensin II enhances macrophage-derived foam cell formation in transgenic rabbits[J]. Biomed Res Int, 2015, 2015:843959.
[6] 高守翠, 成大欣, 赵四海, 等. 胆固醇酯转移蛋白转基因家兔制作及生物学特性分析[J]. 中国实验动物学报, 2015,22(4):331-335.
[7] Yang D, Song J, Zhang J, et al. Identification and characterization of rabbit ROSA26 for gene knock-in and stable reporter gene expression[J]. Sci Rep, 2016, 6:25161.
[8] Song J, Yang D, Xu J, et al. RS-1 enhances CRISPR/Cas9- and TALEN-mediated knock-in efficiency[J]. Nat Commun,2016, 7:10548.
[9] Yang D, Xu J, Zhu T, et al. Effective gene targeting in rabbits using RNA-guided Cas9 nucleases[J]. J Mol Cell Biol, 2014,6(1):97-99.
[10] Niimi M, Yang D, Kitajima S, et al. ApoE knockout rabbits:A novel model for the study of human hyperlipidemia[J].Atherosclerosis, 2016, 245:187-193.
[11] Yang D, Zhang J, Xu J, et al. Production of apolipoprotein CIII knockout rabbits using Zinc finger nucleases[J]. J Vis Exp, 2013, 81:e50957.
[12] Ma Y, Zhang X, Shen B, et al. Generating rats with conditional alleles using CRISPR/Cas9[J]. Cell Res, 2014, 24(1):122-125.
[13] Suzuki K, Tsunekawa Y, Hernandez-Benitez R, et al.In vivogenome editing via CRISPR/Cas9 mediated homology-independent targeted integration[J]. Nature, 2016, 540(7631):144-149.
[14] Huang L, Hua Z, Xiao H, et al. CRISPR/Cas9-mediated ApoE-/-and LDLR-/-double gene knockout in pigs elevates serum LDL-C and TC levels[J]. Oncotarget, 2017, 8(23):37751-37760.
[15] Niu Y, Shen B, Cui Y, et al. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos[J]. Cell, 2014, 156(4):836-843.
[16] 魏泓. 医学动物实验技术[M]. 北京: 人民卫生出版社, 2016:462-469.
[17] Grissa I, Bouchon P, Pourcel C, et al. On-line resources for bacterial micro-evolution studies using MLVA or CRISPR typing[J]. Biochimie, 2008, 90(4):660-668.
[18] Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science,2007, 315(5819):1709-1712.
[19] Marraffini LA. CRISPR-Cas immunity in prokaryotes[J].Nature, 2015, 526(7571):55-61.
[20] 何玉婷, 李娟, 孙冉冉, 等. 靶向 HIF-1α 基因的 CRISPR/Cas9基因敲除质粒的构建与鉴定[J]. 郑州大学学报: 医学版, 2016, 51(3):293-297.

