基于基因组学的天然产物研究
王 涛
(济宁医学院生物科学学院,日照 276826)
先导化合物的发掘与鉴定是新药研发过程中一项耗时费力、极具挑战性的科研活动。从历史上看,来源于植物和微生物的天然产物及其结构衍生物曾一度是新药研发中先导化合物的主要来源,也是当前临床用药的重要组成部分,特别是抗肿瘤和抗菌药物。据统计(1981-2014年),临床上使用的药物有超过50%来源于天然产物。近几年,仍有诸多有代表性的天然产物(结构衍生物)进入临床试验阶段甚至被批准进入市场,例如:Trabectedin,Halaven及Bryostatin等。究其原因,一方面是天然产物本身通常具有相对复杂的化学结构,具有大量的药效团结构和高度的立体选择性,导致了很多天然产物具有十分重要的生物活性;另一方面,研究表明:成功的药物分子通常会具有类代谢产物性质(metabolite-likeness)[1]。因此,作为次级代谢产物的天然化合物通常不仅具有目标生物活性,而且极有可能是一种或者几种转运蛋白的底物,因而,更容易进入胞内环境发挥作用。以往,大多数的天然产物研究基本上都遵从一条自上而下的研究策略:基于目标生物活性筛选化合物、产物提取及结构鉴定。由于大部分的微生物(例如,海产动物海兔,黑色软海绵)不能在常规的实验条件下实现分离和培养,因而传统的自上而下的探索方法仅能接触到微生物一小部分生物合成潜力,而一些原本可能十分有价值的药用天然产物往往被忽略。这导致自20世纪以来,传统的技术手段造成天然产物的再发现率急剧升高,难以发现新结构天然产物。
由于DNA测序成本的不断降低及高通量测序技术的发展,使基因组学取得重大进步,这也导致天然产物研究再次引起研究者的兴趣。急剧膨胀的微生物基因组和宏基因组数据显示自然界中存在着大量的生物合成基因簇(biosynthetic gene clusters,BGCs),据估计这些基因簇能够编码合成的化合物将远超过现阶段鉴定的化合物数量[2]。理论上通过DNA测序和基因组分析,几乎可以鉴定出微生物中所有负责天然产物合成的BGCs,因此,也能获得所有潜在的这些次级代谢产物信息。如何基于这些生物信息顺利获得这些潜在的化合物及如何高效生产目标化合物使其满足产业化的要求,将是未来天然产物研究的主要方向。本文重点阐述基于基因组的天然产物发现的主要技术与概念,包括生物信息学指导的GGCs鉴定,沉默基因簇的激活以及组合生物合成生产新结构天然产物。
1 后基因组时代的药物发现
随着基因测序技术的飞速发展、分子生物学及遗传学等相关操作技术的成熟为新型天然产物发现提供了新的研究思路与开发途径。不断降低的测序成本,使得通过高通量基因组测序技术获得不同物种的基因组序列信息变得越来越方便可行。通过对这些基因组数据进行分析,可以发现、筛选并鉴定出潜在的、具有新颖结构的活性化合物相关的“沉默”基因簇基因。因此,如何基于海量的基因组数据,挖掘发现新的活性天然产物已成为近年来国内外研究的重点和热点。
通过基因组学分析微生物代谢谱,定性和定量的分析机体在特定的时空条件下所有的代谢产物,不仅能够直接的监测基因的功能和机体的生理生化状态,而且能够有选择性地优化特定的生物合成途径,进而生产获得有活性的次级代谢产物。基于基因组学的药物发现通常包含3个主要的步骤:1)BGCs的发现与鉴定;2)沉默基因簇的激活;3)组合生物合成生产目标化合物。见图1。
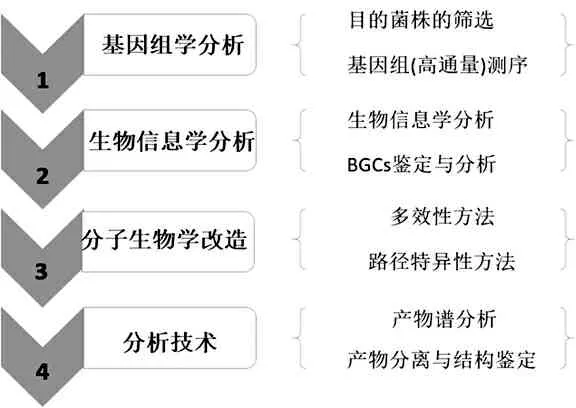
图1 基于基因组学的药物发现过程
2 BGCs的发现与鉴定
来源于微生物及植物的天然产物通常直接与其生物合成基因簇相关,通过编码相应的酶,调控次级代谢产物的生物合成、自身免疫及转运过程。如何从海量的基因序列中优先处理并鉴定出有价值的基因簇,是基于基因组学的药物发现过程中第一个关键步骤[3-4]。基因组挖掘(genome mining)技术可以通过分析孤儿基因簇(orphan BGCs)的基因序列确定其是否与新型的天然产物生产有关。目前,相关的一些生物信息学软件已能够在一定程度上满足基因组挖掘的目的,例如:HMMER,GOLD,NORINE,SBSPKS,SEARCHPKS,NRPSpedictor,plantiSMASH。其中,AntiSMASH(antibiotics and secondary metabolite analysis shell)是在分析抗生素合成基因簇当中使用最广泛的一种生物信息学软件之一[5]。它能够进行自动的基因组鉴定,并能对生物合成基因簇进行分析。其中3.0版本中完整的整合了最新版本的ClusterFinder算法,利用这种基于概率的算法,用户能够发现并识别一些未知类型的基因簇。这在通过基因组挖掘发现新型天然产物的研究中具有十分重要的应用。随着这些预测软件/程序的不断改善,基因挖掘也变得相对简单,研究者或许不再需要具备大量的经验,但必须正视与注意这些分析软件的应用限制与不足。由于大多数的预测软件都是基于现有的、有代表性的代谢途径而开发的,因此,它们可能更容易鉴定出一些比较成熟的BGCs(例如聚酮合酶复合物等等),但是对一些不常见的代谢途径,其鉴定潜在BGCs的能力可能就要大打折扣。虽然大多数预测软件都不能很好地预测次级代谢产物的化学结构,也存在一定的应用限制,但是基于计算机软件预测潜在的BGCs仍是指导后续实验研究的一个切实可行的出发点。
3 沉默基因簇的激活
天然产物通常是通过机体的次级代谢产生,这是一个途径特异性、多效性,涉及全局性调控基因在内的多层次严格调控的过程。研究发现,大量的BGCs在常规的实验室培养条件下处于沉默或者弱表达状态。因此,探索如何激活这些沉默的基因簇将对获得潜在的活性天然产物具有十分重要的价值。基于不同的作用机制,基因簇激活的方法也有不同的分类,实际操作中主要有两种不同的激活策略:物理化学方法和代谢工程改造方法。见图2。
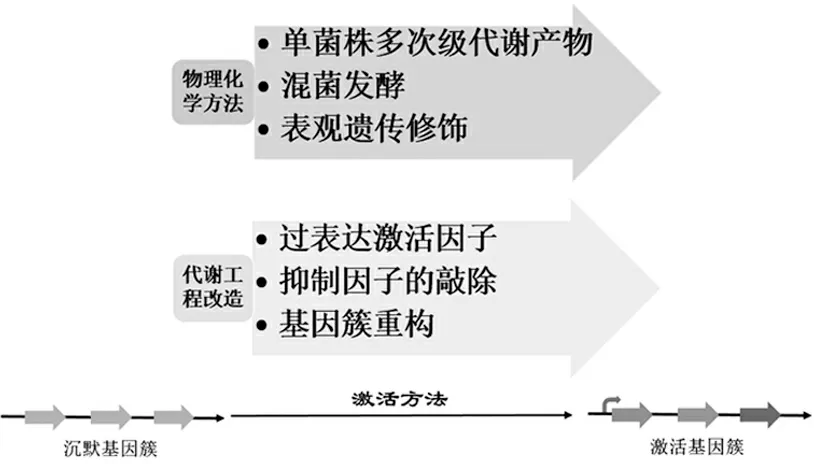
图2 沉默基因簇激活的主要策略
3.1 物理化学法激活沉默基因簇
物理化学方法主要是改变微生物的培养条件,通过研究培养条件的变化刺激微生物做出某些应激反应,同时产生更多类型或更大数量的次级代谢产物来应对这一环境变化。One strain many compounds(OSMAC)法是实施这一策略的一种便捷有效的方法。该方法主要通过改变发酵过程中相关参数(改变培养基成分、培养条件、添加酶抑制剂及混菌发酵等),激活不同功能基因簇,进而改变其代谢途径,产生新的次级代谢产物。利用此方法,研究人员成功地发现了多种新结构化合物,例如:桉烷型倍半萜类[6],fusarielin[7]。但该方法在许多情况下并不能使微生物生产新的次级代谢产物,并且该方法的使用具有一定的盲目性,各种成本较高,因此应用范围受到极大限制。针对这一限制,研究者通过将该方法与基因组扫描技术相结合,大大提高了发现新结构化合物和活性天然产物的概率。iChip(isolation chip)是OSMAC方法的一个重要扩展,该技术的使用对于研究常规条件下无法培养的微生物具有十分重要的应用价值。
表观遗传修饰是通过 DNA 甲基化、组蛋白共价修饰和非编码 RNA 调控等手段干扰特定基因的正常表达,从而定向调控特定的代谢途径。利用表观遗传修饰激活某些沉默的BGCs及其相关的代谢途径、发现结构新颖和具有生物活性的化合物成为目前研究的热点。在Aspergillusnidulans中,通过敲除表观遗传修饰酶(组蛋白脱乙酰基酶及组蛋白甲基转移酶)能够激活数个沉默基因簇,并且显著的改变其代谢产物谱[8]。另外,化学干扰真菌的表观遗传同样能够改变机体的次级代谢,产生新的代谢产物。该方法虽然在激活沉默基因簇方面具有重要的应用价值,但是目前的技术手段对于表观遗传的修饰会导致不可预测的代谢调控的全局改变[9],因此会存在一定的盲目性,增加工作的难度。
3.2 代谢工程方法激活沉默基因簇
基于代谢工程策略激活沉默基因簇的方法是通过调节对沉默BGCs转录和翻译过程起关键作用的调控基因,实现沉默基因簇的表达。随着DNA测序技术的不断进步和蛋白组学的发展,应用该方法激活沉默基因簇具有目的性强的特点,是以后新药发现与研发的主要方向。
3.2.1野生宿主沉默基因簇的激活 理论上,野生宿主细胞内含有必要的次级代谢产物生物合成的调控因子(如前体化合物,路径调控因子及转运蛋白等)。因此,通过基因组编辑和基因工程技术,对野生宿主进行一定的代谢工程改造,能在一定程度上激活沉默基因簇或者提高弱表达基因簇的表达量。一方面可以通过增加正调控因子(转录激活因子)的表达量激活沉默基因簇,生产新型的次级代谢产物(如新结构51环糖基化大环内酯[10])。另一方面,可以通过抑制负调控因子(如途径特异性阻遏物)的表达量激活沉默基因簇,生产新型次级代谢产物[11]。需要注意的是,在一些生物合成途径中没有明显的调控因子,此时通过全局调控基因的表达会是一个比较有帮助的方案。例如:通过定点突变RNA聚合酶或者核糖体蛋白能够影响基因的表达进而诱导机体产生新结构化合物[12]。
3.2.2异源宿主沉默基因簇的激活 对于一些无法培养的或者遗传信息尚不明确的微生物,在异源宿主体内操作特定的基因簇将是一条具有操作性的方法[13]。在选择异源表达宿主时,针对特定的BGCs,必须充分考虑宿主是否含有必要的代谢前导物、催化酶以及恰当的调控系统。为了提高次级代谢产物的产量,通常需要对异源宿主进行优化。
在实际的操作中,在异源宿主中的表达BGCs之前,通常需要对其进行重构处理,例如:启动子替换,插入恰当的核糖体结合位点(RBS位点)以及终止子的插入等。直接获得目的基因簇,并在适宜的宿主中异源表达是最常见也是相对最容易的策略。例如在大肠杆菌体内,通过RecET介导的线性重组可以将巨大合成酶(megasynthetase)插入表达载体进而成功表达[14]。结果表明,即使是超过50kb的基因簇也能够成功的构建表达载体并实现高效异源表达,获得目标表达产物。CRISPR/Cas9系统是目前在基因重组领域研究的热点工具,利用RNA介导的Cas9核酸酶,通过Gibson 组装[15]或者TAR(transformation-associated recombination)[16]可以更加方便地直接克隆大片段基因簇(>100kb)。见图3。

图3 一种基因簇重构激活沉默基因簇激活的方法示意图
4 组合生物合成生产目标化合物
组合生物合成基于酶底物多功能性(substrate promiscuity),通过利用工程改造的催化酶或者代谢途径生产新型的‘非天然’天然化合物。因此,如何在异源宿主细胞内高效地表达组合生物合成途径将对提高化合物的产量具有重要的意义,能够最终实现降低药物研发成本的目的。由于聚酮合酶(polyketide synthase,PKS)和非核糖体肽合成酶(non-ribosomal peptide synthetases,NRPSs)内在的模块化属性及其催化产物的可预测性,使其成为这一领域的研究热点。目前已经成功的应用组合生物学生产聚酮类化合物、非核糖体肽以及皂苷的生物合成[17-20]。
组合生物合成的实施主要包括3种策略,即:1)基于前体化合物的组合生物合成;2)基于酶的修饰;3)基于代谢通路的重组。
4.1 基于前体化合物的生物合成
基于前体化合物的组合生物合成,不仅能够成功应用于PKSs及NRPSs复合物的研究,而且对于PKS/NRPS 杂合的酶复合物也具有十分重要的应用价值(见图4)。目前,大量的具有重要研究价值的酶未被结构鉴定,这往往给后续研究造成比较大的障碍。而基于前体化合物的组合生物合成,能够有效解决这一难题,只要知道其前体化合物即可。
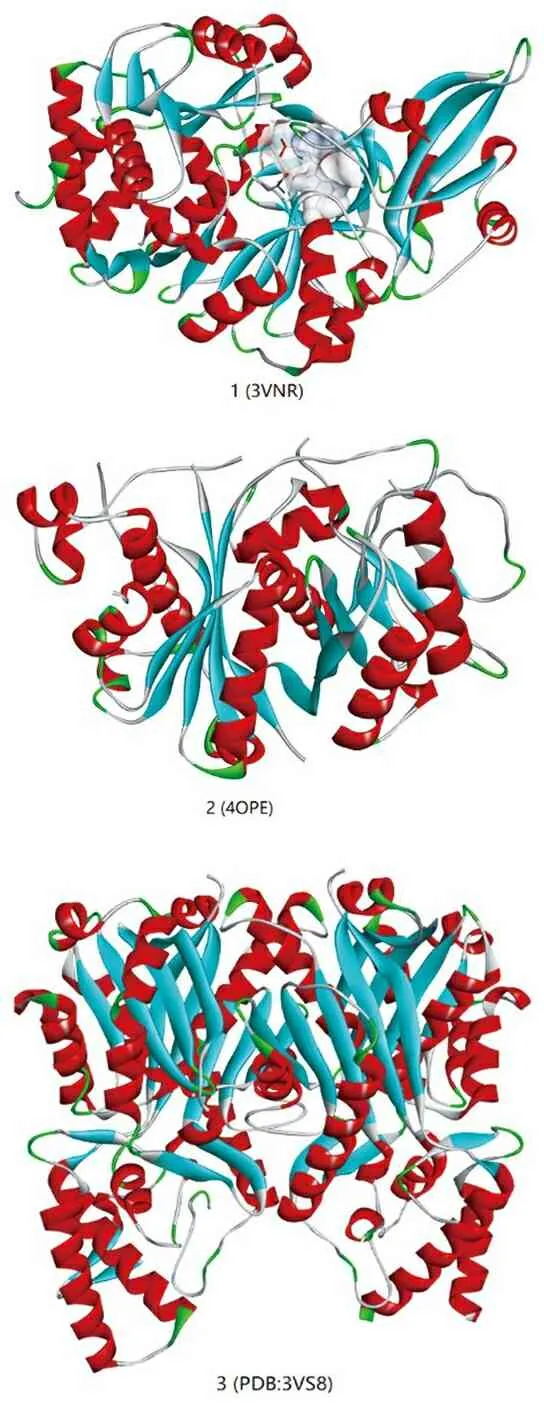
注:1.来源于链霉菌的NRPS腺苷酰化蛋白 CytC1 2.恶唑霉素酮基合成酶区域 3.III 型PKS ArsC
图4 几种典型的PKS蛋白酶
在天然产物组装过程中,单体的多样性很大程度上决定了其结构的多样性。基于前体化合物的组合生物合成利用天然产物生物合成过程中酶底物的多样性,通过吸收不同的单体从而催化合成不同的天然产物类似物。模块化I类聚酮合酶(mPKSs)包含连续的催化模块,而每一模块具有不同的催化区域能够完成C链延伸的一个循环。例如,在Streptomycescinnamonensis体内,聚醚抗生素-monensin就是通过mPKS的作用生物合成。脂酰基转移酶区域(AT 区域)是monensin合成PKS复合物中的第5个模块,能够吸收非天然丙二酸衍生物作为单体,合成新的monensin前体衍生物。基于AT区域的计算机模型,Bravo-Rodriguez等预测该酶的AT活性区域能够吸收体积较大的丙炔基基团作为底物单体,通过添加人工合成化合物-propargyl-malonyl-N-acetyl-cysteamine,经过StreptomycescinnamonensisA495发酵生成了丙炔基-monensin前体化合物[18]。与多模块PKSs不同,III型PKSs结构上比较简单,仅含有一个活性区域重复催化聚酮化合物合成的启动、延伸及环化反应。由于其显著的底物耐受能力,III型PKSs复合物为组合生物合成新型的“非天然”天然产物提供了一个极好的平台。来自于原始石松Huperziaserrata中的HsPKS1能够接受不同的起始单元生产大量的芳香族丁烯酮化合物。出乎意料的是,HsPKS1也能接受大型的起始单元(4-methoxycinnamoyl-coenzyme A(CoA),N-methylanthraniloyl-CoA),通过添加这些单体成功获得了 4-methoxy-2′,4′,6′-trihydroxychalcone 及1,3-dihydroxy-N-methylacridone。
除了mPKSs,NRPSs能够组装大量的多肽化合物天然产物,包括:抗生素(actinomycin,daptomycin),免疫抑制剂(cyclosporine A),及抗肿瘤药物(bleomycin)等。尿苷肽类抗生素是通过NRPSs合成的一类具有较好抗菌效果的肽类抗生素,但其合成酶的底物特异性比较差,能够催化合成新的sansanmycin衍生物[21]。Zhang等[22]鉴定出了一条9酶 ‘组装线’,能够生物合成pacidamycin。由于该酶复合物中的PacI和A区域的底物特异性差,从而生产出其它的9种pacidamycin结构类似物。见图5。

注:AT,酰基转移酶;ACP,酰基载体蛋白 KS,酮基合成酶;KR,酮基还原酶 DH,脱水酶
图5 典型的PKS复合物参与催化反应示意图
4.2 基于酶的修饰
在聚酮化合物生物合成过程中,I类PKS(mPKSs)中每一模块催化一步特定的反应,然后将成熟的产物传递给下一模块,这一特性使得通过“排列与组合”这些酶复合物来设计并合成新产物成为可能。
通过对mPKSs中特定的酶进行修饰(包括整个功能域、模块或者亚基的替换),进而使其所催化的底物发生变化产生新化合物结构,已经成为组合生物合成方法的常规实验手段。例如,利用雷帕霉素合成途径中的乙酰转移酶区域替换红霉素合成系统中的乙酰转移酶区域,能够获得61种6-deoxyerythronolide B(6-DEB)类似物,其中包含多种新结构化合物[23]。研究发现:对于NRPSs,功能域及模块替换同样能够达成相似的目的[24],研究者通过敲除A54145 NRPSs中的lptI甲基转移酶基因,并对其进行异源表达获得多种新结构化合物。与mPKSs不同,I型PKSs(iPKSs)反复利用一套催化区域合成特定产物。通过理性功能域重构,不仅能够产生新型的“非天然”天然产物,而且有助于深入研究该类酶的催化机理。例如,用来自于StcA的SAT功能域替换AfoE中的 ACP酰基转移酶(SAT)能够生产一种新型的非天然聚酮类产物。
作为酶工程的经典方法,定点突变与定向进化技术同样可以用在组合生物学中。这些方法的使用不仅能够实现对特定酶结构与动能的改造,而且能够实现对其本身的催化活性影响最小的目标。例如,通过定点突变使特定的还原酶区域失活,能够产生22种premonensin结构衍生物[25]。为了改变NRPS AT区域的底物特异性,通过在AT区域种引入K278Q单突变,能够将其底物特异性从谷氨酸变成谷氨酰胺,产生含谷氨酰胺的CDA结构类似物[26]。与定点突变相比,定向进化能在引起功能域底物特异性强烈变化的同时,最大限度地减少酶活的损失。Zhang等[27]通过酵母细胞表面展示技术显著地改变了bacillibactin NRPS 复合物中DhbE AT区域底物的特异性,使其识别非天然底物的特异性提高了200多倍,并且合成了非天然芳香族单体。
4.3 基于代谢通路的重组
分子生物学和合成生物学的不断进步使得在遗传代谢清晰的宿主细胞中异源表达不同物种的生物合成基因簇成为一种切实可行的实验操作。自1985年以来,‘杂交’代谢途径便被广泛地应用在新型天然产物的生物合成中。例如三萜皂苷,就已经被成功的利用组合生物合成的方法异源合成。首先在酵母细胞中分别异源表达β-香树精合成酶、细胞色素 P450 还原酶、 β-香树精氧化酶CYP93E2 和 CYP72A61v2以及β-香树精合成酶、细胞色素 P450 还原酶、 β-香树精氧化酶CYP716A12 和CYP72A68v2,通过这些酶的共同作用分别成功合成大豆皂醇 B 和丝石竹酸。并且后一种方法中得到的工程菌能够生产非天然三萜类化合物。
糖类化合物通常对药物-靶点的相互作用起着重要的作用,糖基化过程会对药物溶解性和生物活性产生显著影响。因此,化合物的糖基化给新药发现提供了一个新的方向。基于糖生物合成质粒(“sugar biosynthesis plasmids”)[28]的方法,能够方便地获取新型的糖-修饰药物,特别是针对那些具有良好抗肿瘤活性却存在严重的细胞毒性化合物,例如普卡霉素。Núez等[29]在工程菌S.argillaceusM3W1中表达糖基化NDP-d-digitoxose合成质粒,通过改变糖分子轮廓或者同时改变其分子轮廓和3-侧链,获得七种新结构普卡霉素结构类似物。其中的一种(demycarosyl-3D-β-ddigitoxosyl-mithramycin SK)表现出高效的抗肿瘤活性,而其细胞毒性却比mithramycin低很多。Han 等[30]开发了一种高效的、基于Streptomycesvenezuelae的系统。研究者首先在宿主S.venezuelae中敲除苦霉素的生物合成基因簇,然后在工程菌细胞中通过异源表达不同的基因组合,依次实现脱氧糖的激活、ε-罗多新醌配基的转化以及糖基化后修饰过程,从而将阿霉素的ε-罗多新醌配基转化成糖基化阿霉素类似物。
5 小结与展望
天然产物目录(the Dictionary of Natural Products)已经收录了大约20万种植物次级代谢产物,其中包括大约17万种独特结构。ClinicalTrials.gov数据库中,大约15%的药物来源于植物。虽然植物化学在药物发现中取得了巨大的成功,但是面对着如此巨大的植物种类,研究者们确信在现代药物研发中仍有许多资源值得深入开发。因此,如何系统、全面、有针对性地对来源于植物的天然产物进行深入的研究,是在后基因组时代急需解决的一个科学问题。微生物次级代谢产物是新药发现的另一个重要的来源,其生物多样性要远超过真核生物的代谢产物。然而,由于大量的微生物不能在实验室成功培养,导致仅有不到1%的微生物得到充分的研究。一方面,虽然海洋微生物中物种丰富,但其中相当多的物种在实验室中无法正常培养,因而,是新药研发过程中亟待开发的领域,具有重要的研究价值。另一方面,海洋特殊生态系统决定了海洋生物次级代谢产物的多样性,必定存在着大量的化学结构新颖、生物活性多样、作用机制独特的天然化合物。因而在后续的新药研发过程中,必须对海洋生物资源更加重视,基于高通量测序获得的DNA序列大数据,更加有针对性地进行开发,发现重要先导药物。
以基因组挖掘为导向,深入研究复杂天然产物的生物合成机制、调控机制、关键酶反应机制等是加快天然产物研究的一个必需步骤。通过在微生物体内针对代谢途径进行遗传操作,不仅能够获得更多新结构、高活性的化合物,还能发现不同种类化合物的生物合成途径,这将为新药研发提供崭新的途径。在新药发现和研发领域,组合生物合成是以天然产物的生物合成及代谢为基础,从分子水平对天然产物的生物合成途径进行合理化的遗传修饰和重组,建立结构复杂的天然产物类似物库可以从中开发出更具有临床应用价值的药物。
参考文献:
[1] O Hagan S,Swainston N,Handl J,et al.A 'rule of 0.5' for the metabolite-likeness of approved pharmaceutical drugs[J].Metabolomics,2015,11(2): 323-339.DOI:10.1007/s11306-014-0733-z.
[2] Rutledge PJ,Challis GL.Discovery of microbial natural products by activation of silent biosynthetic gene clusters[J].Nat Rev Microbiol,2015,13(8): 509-523.DOI:10.1038/nrmicro3496.
[3] Cimermancic P,Medema MH,Claesen J,et al.Insights into secondary metabolism from a global analysis of prokaryotic biosynthetic gene clusters[J].Cell,2014,158(2): 412-421.DOI:10.1016/j.cell.2014.06.034.
[4] Land M,Hauser L,Jun SR,et al.Insights from 20 years of bacterial genome sequencing[J].Funct Integr Genomics,2015,15(2): 141-161.DOI:10.1007/s10142-015-0433-4.
[5] Weber T,Blin K,Duddela S,et al.antiSMASH 3.0-a comprehensive resource for the genome mining of biosynthetic gene clusters[J].Nucleic Acids Res,2015,43(W1): W237-W243.DOI:10.1093/nar/gkv437.
[6] Hussain A,Rather MA,Dar MS,et al.Novel bioactive molecules from Lentzea violacea strain AS 08 using one strain-many compounds (OSMAC) approach[J].Bioorg Med Chem Lett,2017,27(11): 2579-2582.DOI:10.1016/j.bmcl.2017.03.075.
[7] Hemphill CFP,Sureechatchaiyan P,Kassack MU,et al.OSMAC approach leads to new fusarielin metabolites from Fusarium tricinctum[J].J Antibiot,2017,70(6): 726-732.DOI:10.1038/ja.2017.21.
[8] Bok JW,Chiang YM,Szewczyk E,et al.Chromatin-level regulation of biosynthetic gene clusters[J].Nat Chem Biol,2009,5(7): 462-464.DOI:10.1038/nchembio.177.
[9] Zhang MM,Qiao Y,Ang EL,et al.Using natural products for drug discovery: the impact of the genomics era[J].Expert Opin Drug Discov,2017,12(5): 475-487.DOI:10.1080/17460441.2017.1303478.
[10] Laureti L,Song L,Huang S,et al.Identification of a bioactive 51-membered macrolide complex by activation of a silent polyketide synthase in Streptomyces ambofaciens[J].Proc Natl Acad Sci USA,2011,108(15): 6258-6263.DOI:10.1073/pnas.1019077108.
[11] Sidda J D,Song L,Poon V,et al.Discovery of a family of γ-aminobutyrate ureas via rational derepression of a silent bacterial gene cluster[J].Chem Sci,2014,5(1): 86-89.DOI:10.1039/c3sc52536h.
[12] Hosaka T,Ohnishi-Kameyama M,Muramatsu H,et al.Antibacterial discovery in actinomycetes strains with mutations in RNA polymerase or ribosomal protein S12[J].Nat Biotechnol,2009,27(5): 462-464.DOI:10.1038/nbt.1538.
[13] Nah H J,Pyeon H R,Kang S H,et al.Cloning and heterologous expression of a large-sized natural product biosynthetic gene cluster in streptomyces species[J].Frontiers in Microbiology,2017,8.DOI:10.3389/fmicb.2017.00394.
[14] Fu J,Bian X,Hu S,et al.Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J].Nat Biotechnol,2012,30(5): 440-446.DOI:10.1038/nbt.2183.
[15] Wang JW,Wang A,Li K,et al.CRISPR/Cas9 nuclease cleavage combined with Gibson assembly for seamless cloning[J].BioTechniques,2015,58(4): 161-170.DOI:10.2144/000114261.
[16] Lee NC,Larionov V,Kouprina N.Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J].Nucleic Acids Res,2015,43(8): e55.DOI:10.1093/nar/gkv112.
[17] DeSieno M A,Denard C A,Zhao H.Drug discovery and development by combinatorial biosynthesis[J].Enzyme Technologies,2011: 251-283.DOI:10.1002/9780470627303.ch8.
[18] Bravo-Rodriguez K,Ismail-Ali AF,Klopries S,et al.Predicted incorporation of non-native substrates by a polyketide synthase yields bioactive natural product derivatives[J].Chembiochem,2014,15(13): 1991-1997.DOI:10.1002/cbic.201402206.
[19] Moses T,Pollier J,Almagro L,et al.Combinatorial biosynthesis of sapogenins and saponins in Saccharomyces cerevisiae using a C-16 hydroxylase from Bupleurum falcatum[J].Proceedings of the National Academy of Sciences,2014,111(4): 1634-1639.DOI:10.1073/pnas.1323369111.
[20] Xu Y,Zhou T,Zhang S,et al.Thioesterase Domains of Fungal Nonreducing Polyketide Synthases Act as Decision Gates during Combinatorial Biosynthesis[J].Journal of the American Chemical Society,2013,135(29): 10783-10791.DOI:10.1021/ja4041362.
[21] Zhang N,Liu L,Shan G,et al.Precursor-directed biosynthesis of new sansanmycin analogs bearing para-substituted-phenylalanines with high yields[J].J Antibiot,2016,69(10): 765-768.DOI:10.1038/ja.2016.2.
[22] Grüschow S,Rackham EJ,Elkins B,et al.New pacidamycin antibiotics through precursor-directed biosynthesis[J].Chembiochem,2009,10(2): 355-360.DOI:10.1002/cbic.200800575.
[23] McDaniel R,Thamchaipenet A,Gustafsson C,et al.Multiple genetic modifications of the erythromycin polyketide synthase to produce a library of novel “unnatural” natural products[J].Proc Natl Acad Sci USA,1999,96(5): 1846-1851.DOI:10.1073/pnas.96.5.1846.
[24] Baltz RH.Biosynthesis and genetic engineering of lipopeptide antibiotics related to daptomycin[J].Curr Top Med Chem,2008,8(8): 618-638.DOI:10.1128/aac.01307-09.
[25] Kushnir S,Sundermann U,Yahiaoui S,et al.Minimally invasive mutagenesis gives rise to a biosynthetic polyketide library[J].Angew Chem Int Ed Engl,2012,51(42): 10664-10669.DOI:10.1002/anie.201202438.
[26] Thirlway J,Lewis R,Nunns L,et al.Introduction of a non-natural amino acid into a nonribosomal peptide antibiotic by modification of adenylation domain specificity[J].Angew Chem Int Ed Engl,2012,51(29): 7181-7184.DOI:10.1002/anie.201202043.
[27] Zhang K,Nelson KM,Bhuripanyo K,et al.Engineering the substrate specificity of the DhbE adenylation domain by yeast cell surface display[J].Chem Biol,2013,20(1): 92-101.DOI:10.1016/j.chembiol.2012.10.020.
[28] Olano C,Méndez C,Salas JA.Harnessing sugar biosynthesis and glycosylation to redesign natural products and to increase structural diversity[M].John Wiley & Sons,Inc.2014:317-339.
[29] Núez L E,Nybo S E,González-Sabín J,et al.A novel mithramycin analogue with high antitumor activity and less toxicity generated by combinatorial biosynthesis[J].Journal of Medicinal Chemistry,2012,55(12): 5813-5825.DOI:10.1021/jm300234t.
[30] Han AR,Park JW,Lee MK,et al.Development of a Streptomyces venezuelae-based combinatorial biosynthetic system for the production of glycosylated derivatives of doxorubicin and its biosynthetic intermediates[J].Appl Environ Microbiol,2011,77(14): 4912-4923.DOI:10.1128/AEM.02527-10.

