茶树CsMYB123转录因子的克隆及表达特性研究
郭亚飞,马煜明,水刘媛,郭 飞,倪德江
(华中农业大学 园艺林学学院,园艺植物生物学教育部重点实验室,武汉 430070)
花青素是一种天然水溶性色素,具有抗氧化、抗衰老、抗消炎、抗突变等多种生理功能。‘紫娟’茶树是一种新稍富含花青素的特异种质资源,表现为芽、嫩叶和嫩茎均呈紫色。花青素在植物体内的积累受到外界环境因子(如光、氧、温度等因素)影响,但主要受自身基因的调控[1]。调控植物花青素生物合成的基因主要分为两类:结构基因和调控基因,结构基因编码花青素生物合成的催化酶;调控基因编码的转录因子应答外界环境刺激,调控结构基因的时空表达。高等植物体内的基因表达是一个复杂精密的系统,在这个过程中,调控基因作为开关和枢纽控制着很多基因的表达[2]。目前在很多植物中都已研究表明MYB类转录因子参与调控花青素的生物合成。
MYB类转录因子是植物中最重要的一类含有MYB结构域的转录因子。MYB结构域是一段约51~52个氨基酸的肽段,含有高度保守的氨基酸和间隔序列,这些高度保守的氨基酸可以使MYB蛋白的结构域折叠成螺旋-螺旋-转角-螺旋的结构。根据MYB结构域的数量,将其分为4类:R1/R2-MYB、R2R3-MYB、3R-MYB和4R-MYB。植物中MYB基因编码的蛋白质大多数是R2R3-MYB蛋白,该类蛋白一般在N端都有2个MYB结构域(R2、R3),在C端有转录激活区域,它们广泛参与调节植物初生和次生代谢、应答激素刺激、生物和非生物胁迫应答等[3]。已有研究表明,拟南芥的第4亚族、第5亚族、第6亚族和第7亚族中的R2R3-MYB型转录因子成员参与调控花青素的生物合成途径[4-5]。龙胆草中R2R3-MYB型的GtMYBP3和GtMYBP4调节龙丹花花冠中黄酮的合成[6]。葡萄中的VvMYBF1促进葡萄中类黄酮的合成[7]。Vimolmangkang等[8]发现苹果中的MdMYB3参与花青素的生物合成。Mano等[9]发现番薯中IbMYB1特异调节果肉和块根中CHS、CHI、F3H、DFR、ANS和3GT基因的表达及花青素的积累。
本研究基于转录组数据,从‘紫娟’茶树一芽一叶中成功克隆出了CsMYB123基因的完整开放阅读框,进行了生物信息学分析,并探讨了该基因在茶树不同组织和不同激素处理下的表达特性,以期为下一步深入研究CsMYB123的生物学功能奠定一定的理论基础。
1 材料和方法
1.1 材 料
供试材料为‘紫娟’5年生无性系茶树,种植于华中农业大学茶树种质资源圃。于2016年7月,选择长势一致、无病虫害、无环境污染的划定区域,逐次随机采摘一芽一叶、第二叶、第三叶、第四叶、嫩茎、老茎,分别混匀,装入已做标记的样品袋,液氮固定,放入-80 ℃冰箱保存备用。
1.2 方 法
1.2.1总RNA的提取和cDNA的合成按照EASYspin Plus植物RNA快速提取试剂盒说明书提取供试样品总RNA。用DS-11超微量紫外/可见分光光度计(美国丹诺尔公司)检测所提RNA纯度和浓度,用1%琼脂糖凝胶电泳检测RNA质量。使用TRUEscript RT MasterMix试剂盒合成cDNA,用于RT-PCR和qRT-PCR。
1.2.2茶树CsMYB123基因的克隆基于本实验室前期测转录组筛选得到的CsMYB123基因序列,用Primer 5.0软件设计克隆引物CsMYB123-QF(5′-ATGGGGAGGAGTCCATGCTGCT-3′)和CsMYB123-QR(5′-TCATGGCCAGTCCTCAGAATCAAGA-3′)。扩增体系:2 × Ultra-Pfu Master Mix 酶(Dye Plus) 5 μL、ddH2O 2.4 μL、模板1 μL和正反引物各0.8 μL。反应条件:94 ℃预变性3 min;94 ℃ 20 s,59 ℃ 20 s,72 ℃ 1 min进行32个循环;72 ℃ 7 min。以第一次的PCR产物为模板1 μL、2 × Ultra-Pfu Master Mix酶(Dye Plus) 12.5 μL、正反引物各0.8 μL、ddH2O 9.9 μL,相同反应程序,进行第二轮PCR。用1%琼脂糖凝胶电泳分离PCR产物,经回收、连接、转化、测序,最后得到CsMYB123的cDNA序列。
1.2.3生物信息学分析利用 NCBI 网站对CsMYB123 的 cDNA 序列在线查找 ORF 和同源性分析;利用 EMBL 的SMART 分析CsMYB123 基因编码氨基酸序列的保守结构域;多物种氨基酸序列的比对,亲水性和疏水性分析用 DNAMAN 6.0软件;利用在线工具包(SMS) 和 Prot Param 分析氨基酸序列的组成及理化性质;运用在线工具 TMHMM server 2.0对 CsMYB123蛋白的跨膜结构进行预测分析;用MEGA7.0构建系统进化树;运用在线工具 SignalP4.1 server 进行信号肽的分析和预测。
1.2.4花青素含量的检测准确称取1.0 g磨碎茶样,加10 mL沸水,沸水浴中浸提30 min,过滤至容量瓶,加水定容至50 mL作为供试液。吸取2~4 mL供试液放在刻度试管中,加酸性乙醇溶液至10 mL,摇匀立即显出刚果红色,显色30 min后,将澄明的红色溶液进行比色测定。选用535 nm波长,1 cm比色杯,以酸性乙醇为空白对照,测定光密度。花青素含量的计算公式为:花青素(mg/g)=(光密度/吸取溶液量×101.83)/样品干重。
1.2.5茶树CsMYB123基因的表达特性分析将资源圃中的‘紫娟’茶树,按照5点取样法设取样点,分别用0.1 mmol/L脱落酸(ABA)、1 mmol/L赤霉素(GA3)、1 mmol/L生长素(IAA)、1 mmol/L乙烯利(ETH)、1 mmol/L茉莉酸甲酯(MeJA)、1 mmol/L水杨酸(SA)处理生长健康、正常、无病虫害植株的新稍第二叶,处理0(CK)、2、4、8和24 h,处理结束时立即取样,混匀,分别装入已标记的样品袋,-80 ℃冰箱保存备用。
采用实时荧光定量PCR,研究CsMYB123基因在‘紫娟’茶树不同组织和不同激素处理下的表达模式。CsMYB123基因的检测引物为CsMYB123-JF(5′-GCTCCAAGGAAGGACTGAAC-3′)和CsMYB123-JR(5′-GCCTGCAACTCTTACCACAT-3′);内参基因GAPDH的引物为GAPDH-JF(5′-TTGGCATCGTTGAGGGTCT-3′)和GAPDH-JR(5′-CAGTGGGAACACGGAAAGC-3′)。实时荧光定量PCR的操作步骤参照2×Sbyr Green qPCR Mix的说明书进行,采用2-ΔΔCT方法[10]进行相对定量分析。
2 结果与分析
2.1 茶树 CsMYB123 基因的克隆及序列分析
以‘紫娟’茶树一芽一叶cDNA为模板,经PCR扩增后获得CsMYB123基因的片段,琼脂糖凝胶电泳检测与目的条带大小相符(图1)。对扩增结果进行测序和分析后表明,该基因的开放阅读框(ORF)长度为915 bp,共编码304个氨基酸。
2.2 茶树 CsMYB123 基因序列的生物信息学分析
2.2.1茶树CsMYB123基因编码氨基酸序列分析对茶树CsMYB123基因编码的氨基酸序列进行SMART分析,结果表明,在该序列的N端包含2个典型的MYB结构域(R2、R3)(图2),R2结构自第17个氨基酸开始(图3),每隔19个aa有1个保守的疏水性氨基酸残基色氨酸W(17、37和57 aa);R3结构第一个W被异亮氨酸(I,70 aa)所取代,此后每隔18个aa有1个W残基(89 和108 aa)。该结构域存在于核受体共阻遏物和许多染色质重塑复合物的亚基中,由3个α-螺旋的串联重复序列组成,主要参与DNA结合。在115~225 aa,存在一个CTD结合域。该区域对于转录延伸因子蛋白Spt5与Spt4结合形成功能性复合物是必需的,该功能性复合物可通过RNA聚合酶Ⅱ调节早期转录延伸,并且还可能与mRNA加帽酶相结合共同参与前体mRNA加工。故CsMYB123基因属于MYB家族R2R3-MYB型转录因子,在转录调控中发挥重要作用。
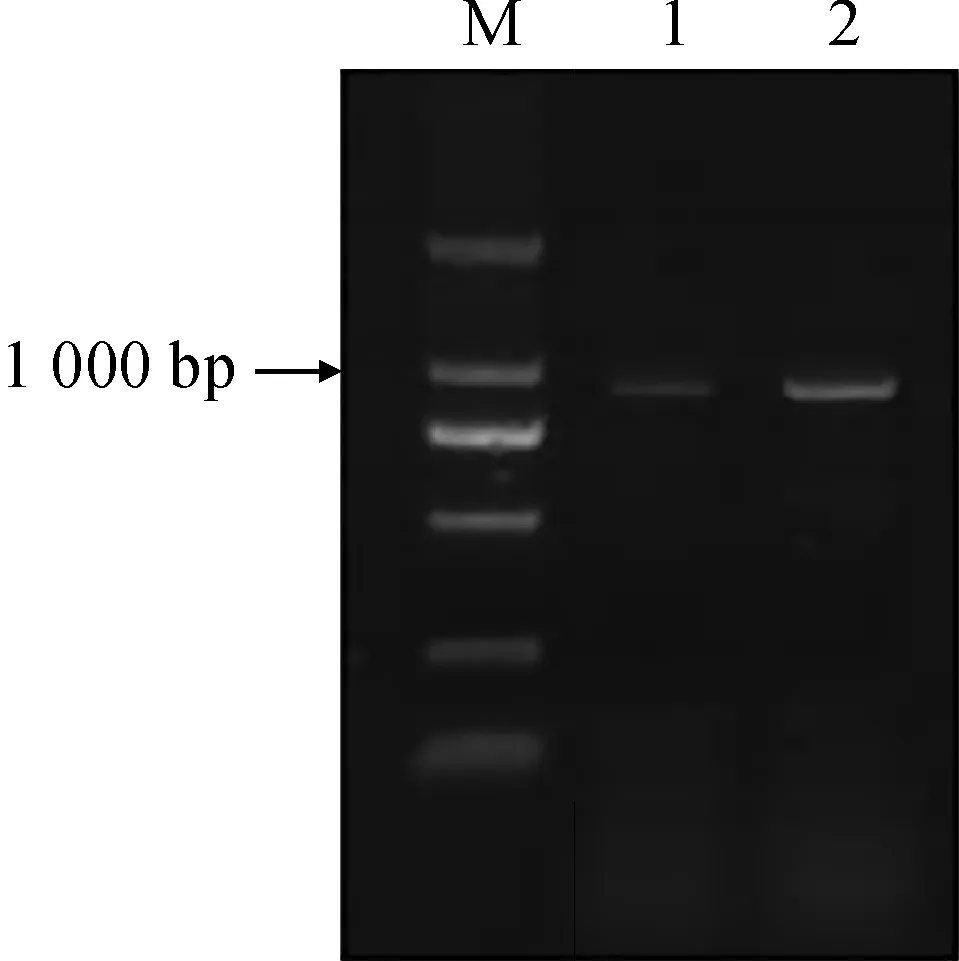
M.DNA marker;1.退火温度57 ℃;2.退火温度59 ℃图1 茶树CsMYB123基因PCR扩增结果M. DNA marker; 1. Annealing temperature 57 ℃; 2. Annealing temperature 59 ℃Fig.1 PCR application of CsMYB123
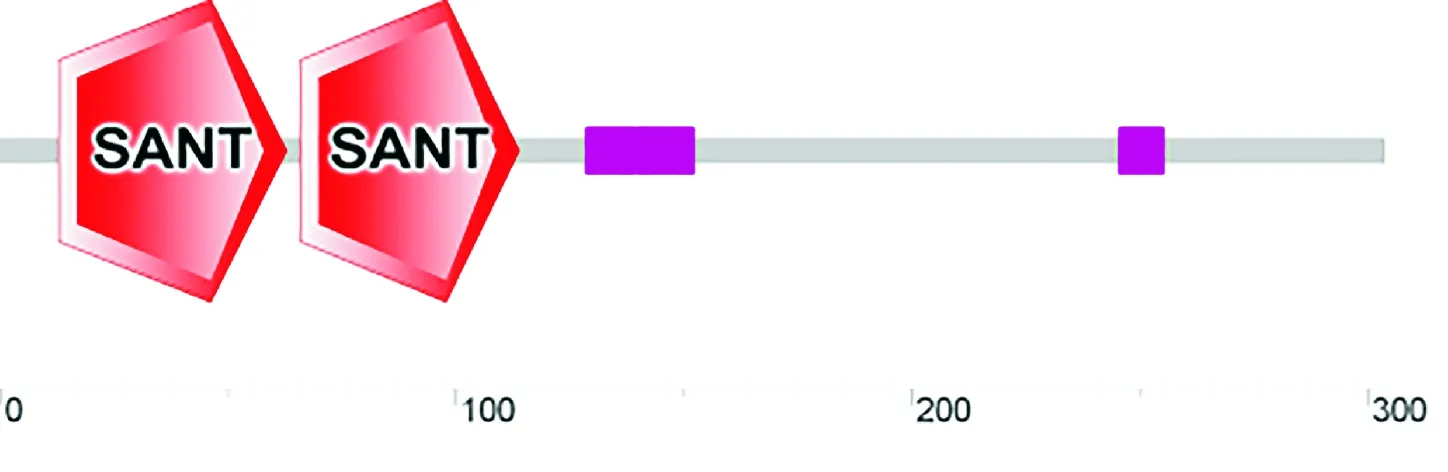
图2 CsMYB123 氨基酸序列的保守结构域预测Fig.2 A putative conserved domain of CsMYB123 amino acid sequence

MYB结构域用阴影标出,保守氨基酸用加粗体标出图3 茶树CsMYB123基因核酸序列与推测的氨基酸序列MYB domains are marked in shadow, and the conserved amino acids are denoted in bold font typeFig.3 The nucleotide and putative amino acid sequences of CsMYB123 gene from tea plant
将CsMYB123基因编码的氨基酸序列与其他物种R2R3-MYB型转录因子进行序列比对,结果如图4所示,它们在MYB结构域所在位置处于高度保守。
2.2.2茶树CsMYB123基因编码氨基酸序列的进化树构建为了预测CsMYB123 基因的功能,利用 MAGE7.0软件对CsMYB123基因编码的蛋白和拟南芥 MYB 家族基因编码的蛋白进行同源性比对,构建系统进化树(图5)。结果显示,CsMYB123蛋白与拟南芥中 MYB 家族成员 AtMYB123 聚集在一个单独的进化枝,属同一亚组,亲缘关系最接近。
2.2.3茶树CsMYB123基因推导氨基酸组成及理化性质用ExPASy的ProtParam在线分析软件对同源性较高的MYB蛋白的氨基酸序列进行分析。结果如表1所示,这些植物中的MYB蛋白的氨基酸残基数在262~304之间;相对分子量为(2.977~3.407)×104Da;理论等电点在8.05~9.05之间;脂肪族氨基酸的平均含量为31%,芳香族氨基酸的平均含量是6%,酸性氨基酸和碱性氨基酸的平均含量分别是12%和17%。各物种MYB基因编码氨基酸序列的GRAVY均为负值,说明CsMYB123为亲水性蛋白。亲/疏水性分析结果表明:CsMYB123的第248位(谷氨酸Glu)、第249位(蛋氨酸Met)、第250位(谷氨酸Glu)和第352位(谷氨酸Glu)亲水性最强,且亲水氨基酸残基的比例高于疏水氨基酸残基。推测CsMYB123属于亲水性蛋白,这与该基因编码氨基酸序列的分析结果预测一致。
2.2.4茶树CsMYB123蛋白的信号肽、跨膜结构及亚细胞定位预测分析运用在线工具SignalP4.1 server进行信号肽的分析和预测,结果表明,C值、S值、Y值均小于0.2,推测CsMYB123蛋白无信号肽结构,属于非分泌蛋白。蛋白跨膜结构预测分析的结果表明:该蛋白不跨膜。此外,亚细胞定位预测分析结果显示,CsMYB123主要定位于细胞核。

图4 茶树CsMYB123基因编码的氨基酸序列与其他植物MYB蛋白序列的保守域比较Fig.4 Comparison of the putative amino acid sequences for CsMYB123 transcription factor in tea plant with other plant MYB proteins
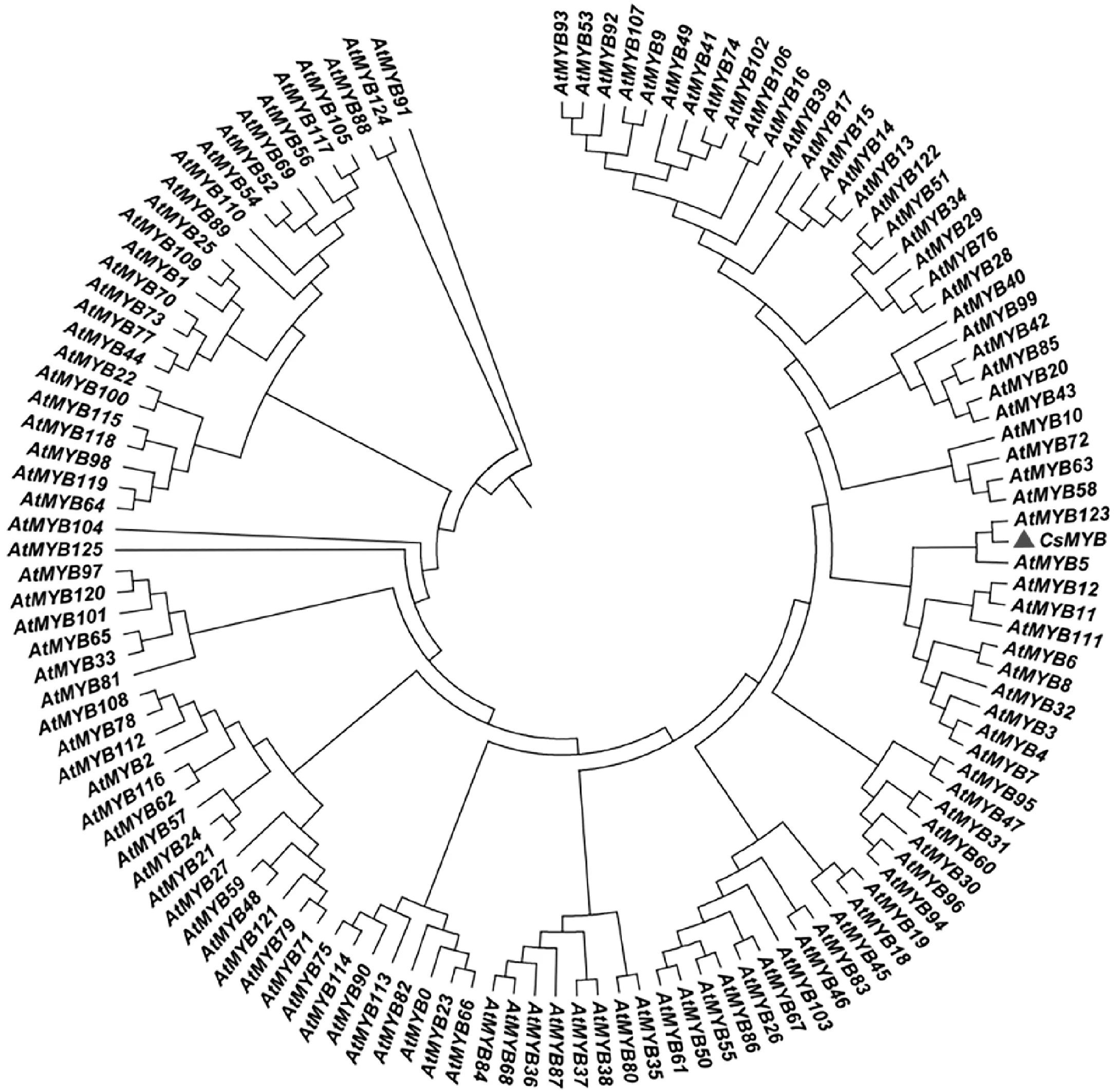
图5 茶树 CsMYB123蛋白与拟南芥 MYB 基因家族的系统进化分析Fig.5 Phylogenetic tree analysis of CsMYB123 protein in tea plant and MYB gene family from A. thaliana
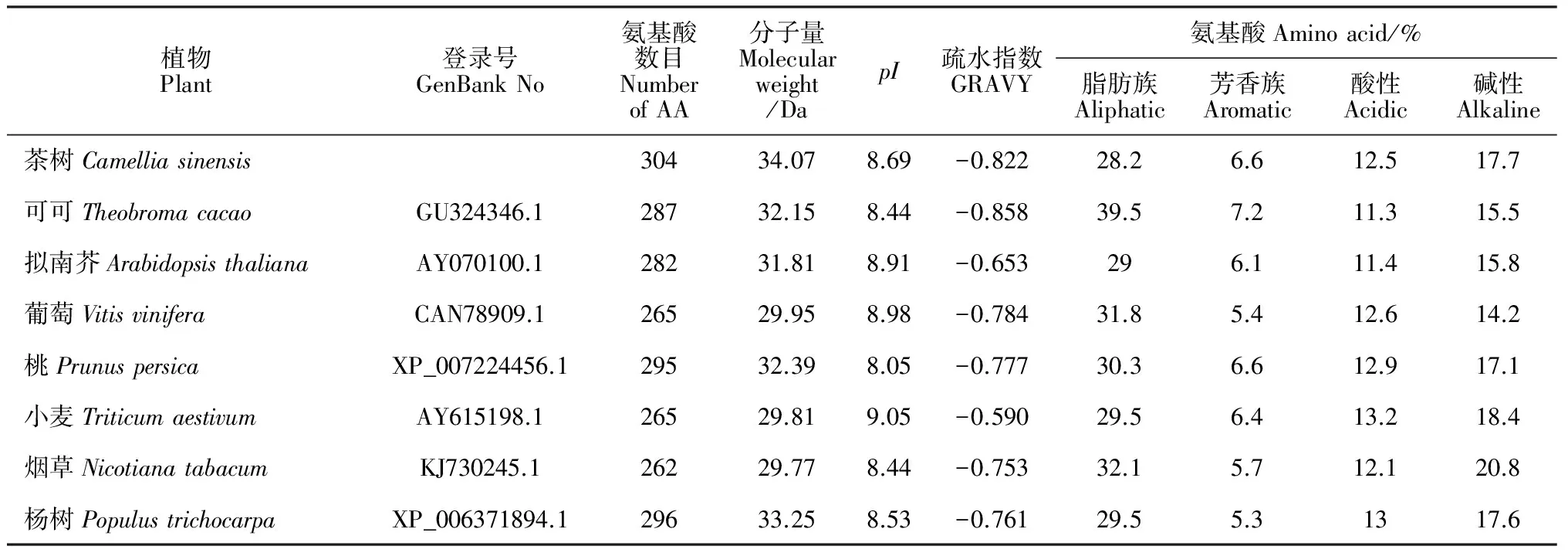
表1 不同物种 R2R3-MYB转录因子氨基酸组成成分及理化性质分析

Ⅰ. 一芽一叶;Ⅱ. 二叶;Ⅲ.三叶;Ⅳ.四叶;Ⅴ. 嫩茎;Ⅵ. 老茎;不同小写字母表示在0.05水平上差异显著,下同图6 不同组织中 CsMYB123 基因表达水平及花青素含量Ⅰ. First leaf and a bud;Ⅱ. Second leaf;Ⅲ.Third leaf;Ⅳ.Fourth leaf;Ⅴ. Tender stem;Ⅵ. Old stem;Different normal letters indicate significant difference at 0.05 level,the same as belowFig.6 Analysis on expression of CsMYB123 gene and anthocyanin content in different tissues
2.3 不同组织中 CsMYB123 基因表达水平及花青素含量检测
检测不同组织的花青素含量,发现第二叶中的花青素含量最高(7.36 mg/g),显著高于其他组织;其次是一芽一叶,为5.36 mg/g;老茎中的花青素含量最低,为0.49 mg/g(图6,A)。随嫩度降低,花青素含量呈先上升后下降的趋势。
检测CsMYB123基因在不同组织的表达水平,结果如图6,B所示,表现为一芽一叶>第二叶>第三叶>第四叶>老茎>嫩茎。CsMYB123基因在一芽一叶中的表达量是嫩茎的15.68倍。整体上,表达量随着嫩度的降低呈下降趋势。
综合各组织中的花青素含量与CsMYB123基因表达水平分析,发现新稍中的花青素呈高含量累积,且CsMYB123基因在新稍中呈高水平表达,二者之间存在一定程度的正相关关系。
2.4 茶树CsMYB123基因受不同激素处理后的表达水平分析
图7显示,ABA处理‘紫娟’茶树新稍第二叶2、4和8 h时,CsMYB123基因表达量逐渐下降,在24 h时,表达量恢复至未处理水平,说明ABA在一定时间内下调茶树CsMYB123基因的表达;IAA处理后,CsMYB123基因的表达量在2 h内迅速下降,2 ~ 8 h上升后,在24 h又下降,不同处理时间点的表达量均低于对照,表明IAA下调该基因的表达;GA3处理后,CsMYB123基因的表达量在2 h内迅速下降,4 h上升后在8 h出现下降,在24 h时又升高,但不同处理时间点的表达水平均低于对照,说明GA3下调该基因的表达;ETH处理后,CsMYB123基因的表达量在2 h内迅速下降,4 h上升后在8 h出现下降,在24 h时迅速升高,且只在该时间点的表达量高于对照,说明ETH在一定时间内下调该基因的表达;MeJA处理后,CsMYB123基因的表达量在0~4 h内逐渐升高,在4 h时达到最大,之后又下降,说明MeJA在一定时间内上调该基因的表达;SA处理后,CsMYB123基因的表达量在2 h内迅速上降,之后逐渐降低,表明SA在短时间内下调该基因的表达。综合来看,茶树CsMYB123基因响应多种激素刺激,受激素种类和处理时间不同,其表达水平也存在差异。
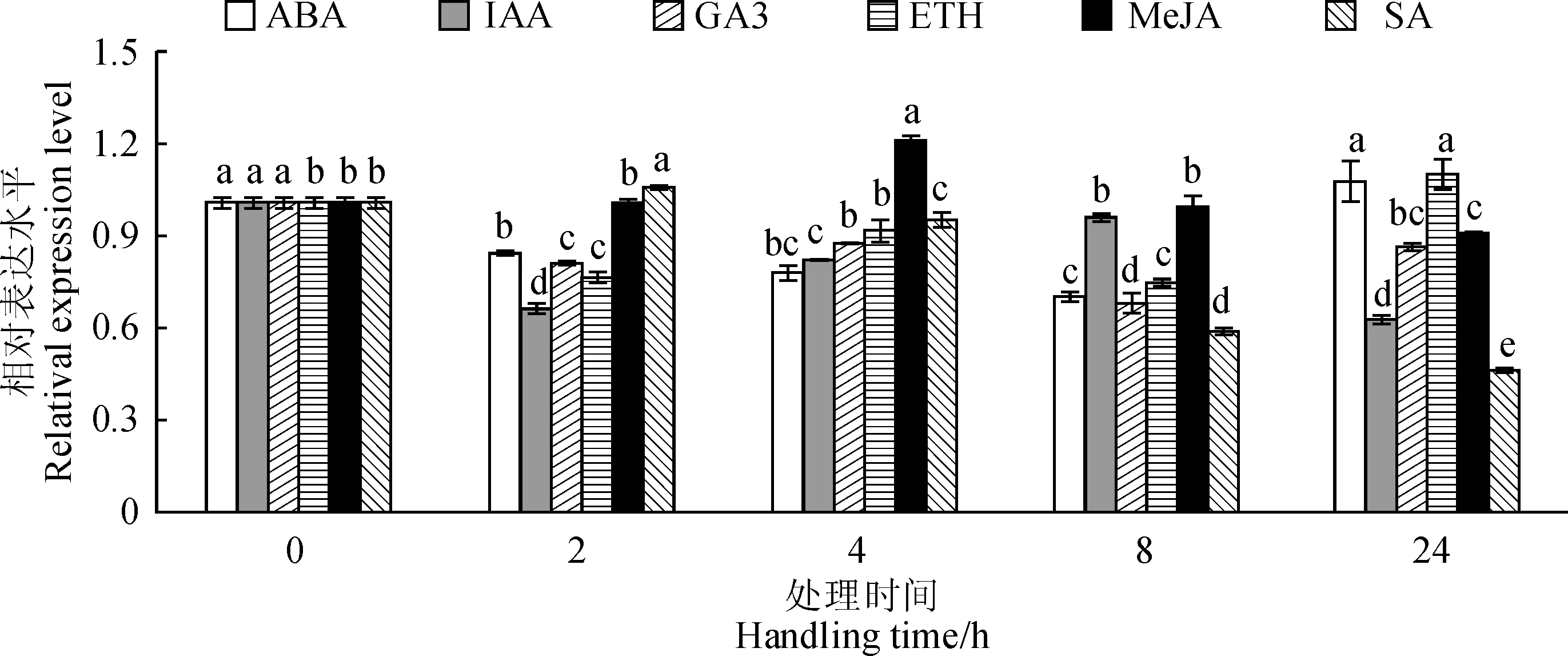
图7 茶树CsMYB123 基因在不同激素处理下的表达分析 Fig.7 Expression analysis of CsMYB123 gene under different hormonal treatments
3 讨 论
本实验从‘紫娟’茶树中克隆得到CsMYB123基因。序列分析表明,CsMYB123基因编码的蛋白属R2R3-MYB型转录因子。而R2R3-MYB型转录因子已被证明广泛参与调控植物中花青素生物合成途径,VvMYB5a转化烟草可诱导其体内酚类化合物花青素和黄酮醇主要组分(花青素鼠李葡萄糖苷和槲皮素-3-鼠李葡萄糖甙)的大量积累[11];柿树cvFuyu中DkMYB4被抑制后,原花青素通路相关基因和原花青素生物合成出现实质性的下调[12]。Stracke等[13]和 Matus等[14]基于同一个亚组中MYB基序相同的现象,将拟南芥的R2R3-MYB分为25个亚组,AtMYB123 (TT2,Q9FJA2)属于第5亚组(其成员还包括:VvMYB5a、GhMYB38和DkMYB4)。同源性分析结果表明:CsMYB123蛋白与拟南芥AtMYB123蛋白在同一分支,进化关系最近,推测它们具有类似的生物功能。Ban等[15]研究发现AtMYB123基因参与调节拟南芥种皮中原花青素的生物合成。Nesi等[16]在拟南芥种子发育阶段研究发现,AtMYB123基因与原花青素生物合成途径中参与酶基因的表达模式完全一致。因此,我们推测CsMYB123基因参与茶树花青素生物合成的调控。‘紫娟’茶树不同组织的花青素含量表现为第二叶>一芽一叶>第三叶>第四叶>嫩茎>老茎,这与蔡丽等研究‘紫娟’茶树新稍第一叶到第四叶的花青苷含量随叶片嫩度降低而呈显著下降[17]的结论相一致。此外,CsMYB123基因在‘紫娟’茶树新稍中高水平表达,且其表达模式与不同组织中的花青素含量呈较好的正相关,这也从一定程度上说明CsMYB123基因极有可能参与茶树花青素生物合成的调控。
激素能通过信号转导启动或阻遏下游基因的表达,影响植物体内次生代谢途径中相关基因的表达,进而影响植物的生长发育[18]。在本研究中,IAA、ETH、GA3和ABA 4种激素处理‘紫娟’茶树新稍第二叶2 h后,CsMYB123基因的表达量均出现显著下降。结合前人研究,单红等[19]研究发现部分R2R3-MYB转录因子可以响应激素诱导;Ji等[20]通过高通量测序技术揭示了与IAA功能相同的人工合成的生长调节剂萘乙酸(NAA)抑制花青素合成途径中调节基因的表达;Jeong等[21]发现ETH可以诱导拟南芥花青素生物合成途径中相关的负调控基因MYBL2表达,且抑制相关正调控基因(如GL3、TT8和PAP1)的表达,从而抑制花青素的生物合成;Ilan等[22]发现胡萝卜悬浮细胞经GA3处理后,花青素的累积量显著下降;Yanhui等[23]通过反向Northern印记研究发现,AtMYB123基因经IAA、ABA、ETH和GA3处理,其表达水平仅为b-tubulin的5%或更少。综合来看,推测茶树CsMYB123基因很有可能参与了激素响应刺激,进而影响茶树花青素的生物合成。
总体上,本研究克隆了茶树CsMYB123基因,并对该基因在不同组织中以及受不同激素处理的表达模式进行了分析,推测出CsMYB123基因在茶树花青素代谢的调控中起重要作用。后续将通过瞬时表达、转基因等实验技术鉴定CsMYB123基因的功能,并深入研究该基因与花青素生物合成途径相关关键酶基因之间的互作关系,进而全面解析其参与茶树花青素代谢调控的分子机制。
参考文献:
[1]周琼琼, 孙威江. 茶树芽叶紫化的生理生化分析及其关键酶基因的表达[J]. 生物技术通报, 2015,31(1): 86-91.
ZHOU Q Q, SUN W J. Physiological and biochemical analysis of young shoot purple-related and gene expression of key enzymes in tea plant (Camelliasinensis) [J].BiotechnologyBulletin, 2015,31(1): 86-91.
[2]房栋, 吕俊宏, 郭旺珍, 等. 一个新的棉花MYB类基因(GhTF1)的克隆及染色体定位分析[J]. 作物学报, 2008,34(2): 207-211.
FANG D, LÜ J H, GUO W Z,etal. Cloning and mapping of a new MYB transcription factor (GhTF1) in cotton [J].ActaAgronomicaSinica, 2008,34(2): 207-211.
[3]万小荣, 李玲. 植物的MYB蛋白[J]. 植物生理学通讯, 2002,38(2): 165-170.
WAN X R, LI L, MYB proteins in plants [J].PlantPhysiologyCommunications, 2002,38(2): 165-170.
[4]STRACKE R, ISHIHARA H, HUEP G,etal. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of theArabidopsisthalianaseedling[J].ThePlantJournal, 2007,50(4): 660-677.
[5]HICHRI I, BARRIEU F, BOGS J,etal. Recent advances in the transcriptional regulation of the flavonoid biosynthetic pathway[J].JournalofExperimentalBotany. 2011,62(8):2 465-2 483.
[6]NAKATSUKA T, SAITO M, YAMADA E,etal. Isolation and characterization of GtMYBP3 and GtMYBP4 orthologues of R2R3-MYB transcription factors that regulate early flavonoid biosynthesis in gentian flowers [J].JournalofExperimentalBotany, 2012,63(18): 6 505-6 517.
[7]CZEMMEL S, STRACKE R, WEISSHAAR B,etal. The grapevine R2R3-MYB transcription factor VvMYBF1 regulates flavonol synthesis in developing grape berries [J].PlantPhysiology, 2009,151(3): 1 513-1 530.
[8]VIMOLMANGKANG S, HAN Y, WEI G,etal. An apple MYB transcription factor MdMYB3 is involved in regulation of anthocyanin biosynthesis and flower development [J].BMCPlantBiology, 2013,13(1): 176-189.
[9]MANO H, OGASAWARA F, SATO K,etal. Isolation of a regulatory gene of anthocyanin biosynthesis in tuberous roots of purple-fleshed sweet potato[J].PlantPhysiology, 2007,143(3): 1 252-1 268.
[10]SCHAFFER A, ARAVIND L, MADDEN T,etal. Improving the accuracy of PSI-BLAST protein database searches with composition-based statistics and other refinements [J].NucleicAcidsResearch, 2001,14(29): 2 994-3 005.
[11]BARRIEU F, LAUVERGEAT V, CARDE J P. Characterization of a grapevine R2R3-MYB transcription factor that regulates the phenylpropanoid pathway [J].PlantPhysiology, 2006,140(2): 499-511.
[12]AKAGI T, IKEGAMI A, TSUJIMOTO T,etal. DkMYB4 is a MYB transcription factor involved in proanthocyanidin biosynthesis in persimmon fruit[J].PlantPhysiology, 2009,151(4): 2 028-2 045.
[13]STRACKE R, WERBER M, WEISSHAAR B. The R2R3-MYB gene family inArabidopsisthaliana[J].CurrentOpinioninPlantBiology. 2001,4(5): 447-456.
[14]MATUS J T, AQUEA F, ARCE-JOHNSON P. Analysis of the grape MYB R2R3, subfamily reveals expanded wine quality-related clades and conserved gene structure organization acrossVitis, andArabidopsis, genomes[J].BMCPlantBiology, 2008,8(1): 83-98.
[15]BAN T, ISHIMARU M., KOBAYASHI S,etal. Abscisic acid and 2,4-dichlorohenoxyacetic acid affect the expression of anthocyanin biosynthetic pathway genes in‘Kyoho’grape berries[J].JournalofHorticulturalScience&Biotechnology, 2003,78(4): 586-589.
[16]NESI N, JOND C, DEBEAUJON I,etal. TheArabidopsisTT2 gene encodes an R2R3 MYB domain protein that acts as a key determinant for proanthocyanidin accumulation in developing seed [J].ThePlantCell, 2001,13(9): 2 099-2 114.
[17]蔡丽, 梁名志, 夏丽飞,等. “紫娟”茶外观表象差异研究[J]. 西南农业学报, 2010,23(3): 700-703.
CAI L, LIANG M Z, XIA L F,etal. Study on exterior appearance difference of ‘Zijuan’[J].SouthwestChinaJournalofAgriculturalSciences. 2010,23(3): 700-703.
[18]岳川, 曾建明, 章志芳,等. 茶树中植物激素研究进展[J]. 茶叶科学, 2012,32(5):382-392.
YUE C, ZENG J M, ZHANG Z F,etal. Research process in the phytohormone of tea plant (Camelliasinensis)[J].JournalofTeaScience, 2012,32(5): 382-392.
[19]SHAN H, CHEN S, JIANG J,etal. Heterologous expression of the chrysanthemum R2R3-MYB transcription factor CmMYB2 enhances drought and salinity tolerance, increases hypersensitivity to ABA and delays flowering inArabidopsisthaliana[J].MolecularBiotechnology, 2012,51(2): 160-173.
[20]JI X H, ZHANG R, WANG N,etal. Transcriptome profiling reveals auxin suppressed anthocyanin biosynthesis in red-fleshed apple callus (Malussieversiif.niedzwetzkyana)[J].PlantCellTissue&OrganCulture, 2015,123(2): 389-404.
[21]JEONG S W, DAS P K, JEOUNG S C,etal. Ethylene suppression of sugar-induced anthocyanin pigmentation inArabidopsis[J].PlantPhysiology, 2010,154(3): 1 514-1 531.
[22]ILAN A, DOUGALL D K. The effect of growth retardants on anthocyanin production in carrot cell suspension cultures [J].PlantCellReports, 1992,11(5-6): 304-309.
[23]CHEN H Y, YANG X Y, HE K,etal. The MYB transcription factor superfamily ofArabidopsis: expression analysis and phylogenetic comparison with the rice MYB family [J].PlantMolecularBiology, 2006,60(1): 107-124.

