植物腺苷5′-磷酰硫酸激酶(APSK)研究进展
谷 宁,杨宇平,蒲首丞,孙梅好
(浙江师范大学化学与生命科学学院,浙江金华 321004)
APS激酶磷酸化APS以形成PAPS, 这种酶较近才引起植物研究界的关注,因为不像酵母、真菌和许多细菌,植物直接使用APS而不是PAPS进行硫还原代谢[1]。因此,在这些生物体中,APS激酶是硫酸盐同化的重要组成部分(图1)。在植物中,它实际上主要从同化物中吸收硫以合成PAPS,因此是初级和次级硫代谢的分支点,其活性的强弱决定植物细胞内硫酸盐的不同代谢方向[2]。
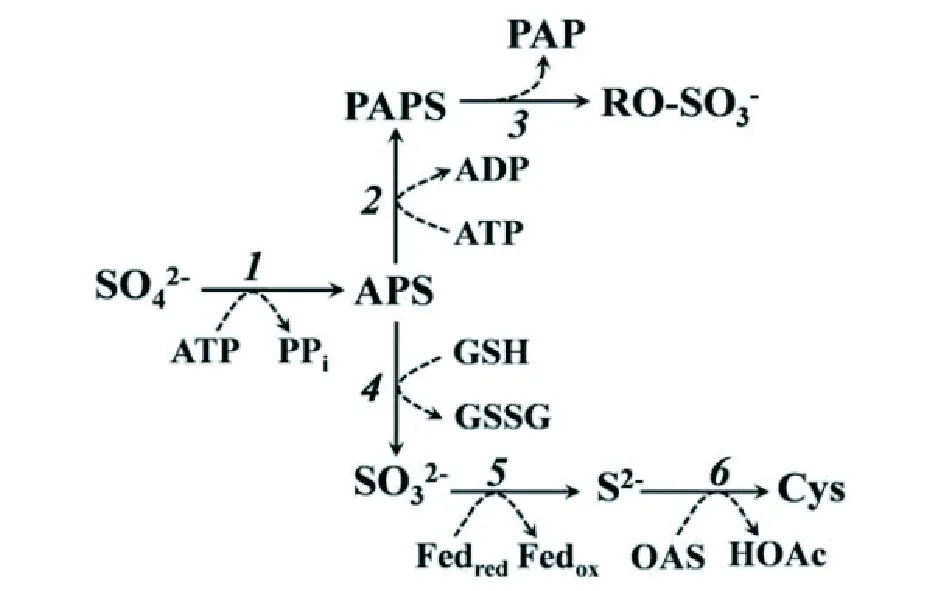
图1 硫酸盐在植物细胞内的同化过程Fig.1 Scheme of sulfate assimilation pathway in plant cells
在拟南芥中发现4种APSK亚型,水稻和杨树有3种APK同种型,苔藓卷毛藓有4种,青藻衣藻和牛蒡属各自含有1种APSK基因[3]。因此,APSK在目前分析的大多数植物中都由小型多基因家族编码。然而,其衍生蛋白质的特定靶向存在差异。虽然水稻似乎只编码具有叶绿体靶向肽的APSK同种型,但在小立陶宛藓中,所有的APSK似乎都是胞质的。虽然水稻和杨树基因组编码多个SOT基因,但在这种类型的磺基转移酶中没有同系物存在于小立陶宛藓或莱茵衣藻中[4]。
在拟南芥中,沉默APK1和APKII亚型,得到的双突变体表现出半矮型表型,说明APS激酶不仅是植物硫酸盐代谢的关键酶之一,而且影响植物生长素的积累[4]。但APS激酶的重要性目前尚不清楚,因为没有硫代葡萄糖苷的植物也没有显示出任何显著的生长表型,并且即使缺失植物激素和PSY1受体以及酪氨酸蛋白质磺基转移酶也不致死。
2 植物腺苷5-磷酸-硫酸激酶(APSK)的结构与反应机理
2.1APSK的总体结构APSK催化γ-磷酸酯从ATP向APS腺嘌呤环的3′-羟基转移,得到PAPS和ADP。来自产黄青霉、大肠杆菌和拟南芥的APSK的动力学研究提出的反应顺序遵循APS底物抑制的有序机制[5-7]。 虽然没有植物APSK的结构信息可用,但已经确定来自人、嗜热菌和脱硫硫杆菌的产黄青霉APSK和双功能ATP硫化酶-APSK的X射线晶体结构,并揭示典型的α/β嘌呤核苷酸结合折叠。对于双功能人类酶,APS对APSK活性的底物抑制与低序列同源性的20-aa 左右N末端环连接。为了更好地了解植物APSK的分子功能,确定了与β,γ-亚氨基腺苷-5′-三磷酸(AMP-PNP)、Mg2+和APS复合的ATAPSK异构体,一个缺乏叶绿体定位的晶体结构(图2)。
缺少77-aa N末端叶绿体定位序列的ATAPSK同种型1(残基78-276)[8]用于蛋白质晶体学。 与AMP-PNP、Mg2+和APS复合的ATAPSK结构通过使用产黄青霉APSK[9]的分子替代作为不对称单位中的3种单体的搜索模型来解决。链A形成结晶二聚体,链B和C产生第二非结晶二聚体。二聚体中每个单体的总体折叠由可变的N-末端区域(残基80-98)组成,并且覆盖核苷酸结合位点。其包括α1和相邻单体,典型的α/β-嘌呤核苷酸结合结构域(β1-α2-β2-α3 -β3-α4-β4-α7)以及α5和α6的小结构域。虽然ATAPSK与来自人类的双功能PAPS合成酶(1.2 Å rmsd for 187 Cα atoms; 51% sequence identity)和产黄青霉APSK(1.3 Å rmsd.for 176 Cα atoms; 55% sequence identity)共享共同的折叠,但这些结构上,N端定位不同[12]。该区域的特征是各种APSK结构中具有约7-aaα(α1)的非结构化环[9-13]。在ATAPSK结构的初始2F o-Fc图中,每个半胱氨酸的硫原子之间的距离落在二硫键的预期键长(~2.05Å)的范围内。Cys86(A)-Cys119(A)和Cys86(B)-Cys119(C)二硫化物被完全氧化,Cys86(C)-Cys119(B)二硫化物被部分氧化。形成二硫化物的半胱氨酸在来自植物和苔藓的APSK上是不变的,但是在其他生物的同源物中通常缺失。
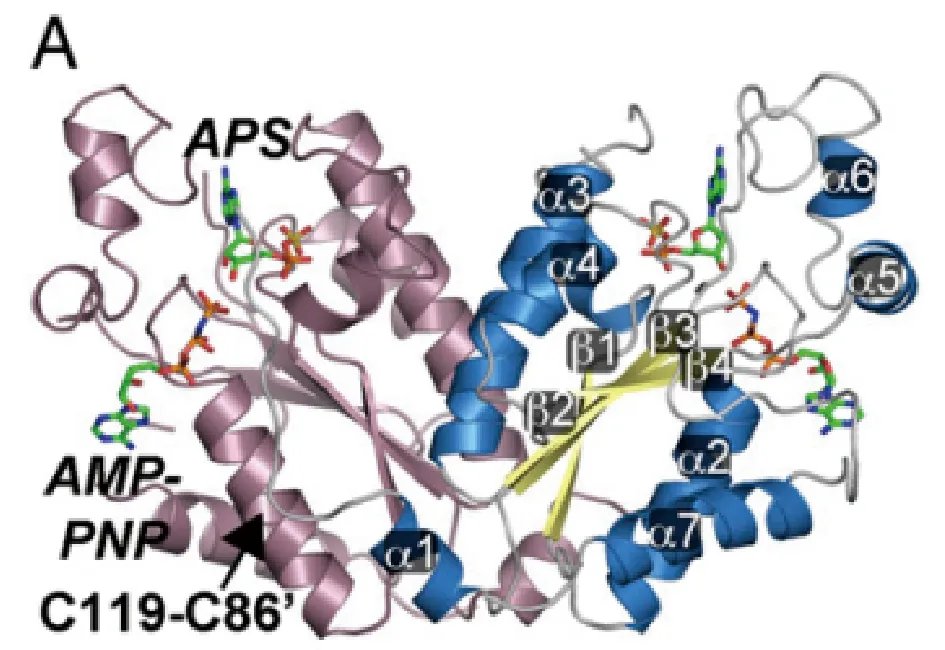
注:每个单体分别以粉色和蓝色着色。 AMP-PNP和APS的位置显示为棒状分子。 C86·C119二硫键的位置在左单体中表示。 二级结构特征标记在右单体Note:Each monomer is colored in pink and blue, respectively.The positions of AMP-PNP and APS are shown as stick molecules.The position of the C86·C119 disulfide bond is indicated in the left monomer.Secondary structure features are labeled in the right monomer图2 ATAPSK的二聚体带状图Fig.2 Ribbon diagram of ATAPSK’s dimer
2.2APSK的反应机制植物可以通过2种途径代谢硫酸盐,这2种途径在腺苷5′-磷酰硫酸(APS)的水平上分支。APS可以还原成硫化物,并且被并入主要的硫酸盐同化途径的Cys中,或者被APS激酶磷酸化成3-磷酸腺苷5-磷酸硫酸盐,作为硫酸化反应的硫酸盐供给。
ATP-硫酸化酶(MgATP:硫酸腺苷酰转移酶,EC 2.7.7.4)和APS激酶(MgATP:腺苷酸3′-磷酸转移酶,EC 2.7.1.25)催化硫酸盐活化的初始步骤:
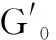
+46 kJ/mol
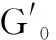
-21 kJ/mol
2种酶是能量耦合的,包括无机焦磷酸酶(△G′0:-6 kcal/mol)的焦磷酸盐水解将无机硫酸盐转化成高反应性磺酰化合物PAPS.[14]。APS激酶是将磷酸基团从ATP转移到腺苷酸(APS)中核糖部分的3-OH基团的小的磷酸转移酶(22-27kDa)。体外研究的所有APS激酶对10-6M范围内Km的APS具有非常高的亲和力。APS激酶的另一个特征是在没有ATP的情况下可以看到有效的底物抑制。对APS激酶的反应机理已从产黄青霉菌和大肠杆菌中广泛地进行研究,但是植物APS激酶研究较少[15-17],尽管在几个较高的植物的叶组织或质体中发现了APS激酶活性,但纯化酶的动力学研究仅报道于绿藻[17]。目前的观点是植物在质体中使用APS进行半胱氨酸生物合成,使用PAPS形成硫酸酯[18]。在细胞质中合成硫酸酯类如硫酸化黄酮醇、硫代葡萄糖苷、植物类固醇和植物素[19],并定位于液泡和高尔基体系。磺脂酰(磺基喹诺基糖基二酰基甘油酯)的生物合成也取决于作为磺酰基供体的体外APS[20],至于质体APS-激酶的功能尚不清楚。质体中APS的稳定浓度是未知的,但是当通过合成,结合和代谢APS的纯化酶的体外测定估算时,质体中游离APS的浓度可能在纳摩尔范围内[21-22]。如果在相同隔室中的同种条件下活跃,则APSkinase将积累PAPS并剥夺其他APS消耗底物的反应。 研究表明,在PAPS离开酶后,在ExMgADP复合物解离前,APS可占据磺酰基核苷酸结合位点。这种机制与从大肠杆菌报道的APS激酶的机制不同,其中APS在ATP之前结合形成酶-APS死端复合物。
3 APSK的功能
硫是所有活生物体的必需元素,是生物合成多种代谢物和大分子所必需的。植物和原核生物是将周围环境中的无机硫酸盐(SO42-)转化为生理有用形式的硫的主要同化生物体。硫同化的代谢组织因植物和微生物而异,在酵母、真菌和肠杆菌(包括大肠杆菌)中,将硫酸盐掺入腺苷-5′-磷酸(APS)中,然后转化为3′-磷酸APS(PAPS)作为生物学“活化”化合物,其被还原成硫化物。在其他硫酸盐同化细菌如铜绿假单胞菌中,APS可用于还原硫化物。 植物具有分叉的硫醇代谢途径,其反映能适应环境胁迫和营养波动的固着生物的代谢需要。这些途径在APS形成后分化。植物中主要硫代谢途径使用APS作为活化的高能化合物用于硫还原和半胱氨酸的生成,这对于蛋白质、蛋氨酸、铁-硫簇、维生素辅因子和保护抗氧化应激的化合物(包括谷胱甘肽和植物螯合素肽)的合成至关重要,或者APS可以转化成PAPS,以提供硫酸盐供体,用于修饰多种天然产物,油菜素类固醇和茉莉酮酸激素,植物鸟嘌呤和其他磺化分子。APS还原酶(APSR)和APSK分别控制植物中初级(即还原性)和次级代谢途径之间分支点的硫酸盐分离。APSR催化APS对亚硫酸盐(SO32-)和AMP的谷胱甘肽依赖性还原。 植物中APSR的大量研究表明,该酶在通过初级硫同化途径调节通量方面起关键作用[23]。拟南芥(ATAPSR)的APSR氧化还原反应改变的活性调节通过调节二硫键发生,其在还原时减弱活性。与APSR相反,APSK在植物硫醇代谢中的作用才开始被检验到。
对拟南芥最近的研究表明,腺苷-5磷酸硫酸激酶(APK)提供活性硫酸盐用于次级代谢物的硫酸化,腺苷5′-磷酰硫酸激酶的破坏降低硫酸次代谢物水平,包括硫代葡萄糖苷。已经成功地隔离这个家族的4种可能的三重纯合突变体组合中的3种。单独的APK1同种型足以维持WT的生长和发育水平。 分析APK1 APK2 APK3和APK2 APK3 APK4的突变显示,APK3和APK4功能上是冗余的,尽管分别位于细胞溶质和质体。然而,无法分离出APK1 APK3 APK4的突变体,最有可能是因为APK1 APK3 APK4的三倍变体组合是花粉致死的。 因此,APS激酶对植物繁殖和生存力至关重要[4,24-26]。
4 APSK氧化还原调控
在植物中,需要腺苷5′-磷酰硫酸激酶(APSK)的生殖活力,并且在专门的代谢中作为PAPS的硫供体。以前对拟南芥APSK的研究(ATAPSK)鉴定N末端结构域(NTD)和核心支架上半胱氨酸之间形成的调节二硫键。在植物中,APSK酶活性通过其N末端环中的Cys86和Cys119间的亚基间二硫键进行生物化学调节。这种硫醇开关是苔藓、裸子植物和被子植物独有的[27]。在植物中,分支点从APS进入亚硫酸盐或PAPS可以通过氧化还原条件来调节。硫醇基氧化还原开关差异性调节APS还原酶和APSK的活性[23,28-29,40]。还原减弱APS还原酶活性并增强APSK活性,而氧化加速APS还原酶并减弱APSK产生的PAPS[23,28-29,40]。他们的相互氧化还原调节提供了一种根据细胞条件将硫通量引导到初级硫代谢(APS还原酶)或专门代谢(APSK)的方法。
植物中的氧化还原敏感性的APSK在绿色植物谱系硫同化途径分叉后进化,提供一种通过APS将硫流分配成质体中的初级和特异性硫醇代谢途径的控制机制[23,28-30]。来自拟南芥APSK的早期生物化学研究表明,大肠杆菌硫氧还蛋白可以介导类似的氧化还原调节[31],这与通过硫氧还蛋白还原二硫化物的共同靶标识别机制一致[32]。在植物中,氧化应激增加对半胱氨酸和谷胱甘肽的需求,并在导致这些分子的主要途径中激活2种关键酶(APS还原酶和谷氨酸-半胱氨酸连接酶)[33-36]。氧化条件减弱APSK活性,以限制APS在次级途径中的使用。 因此,APS分支点的相互调节可以通过细胞氧化还原状态来控制。最近研究表明,2,8-二羟甲基二苯并噻吩S-氧化物在光氧化过程中产生的O(3P)能够调节硫醇残基氧化APSK[28]。已经证明该方法提供一种调节APSK氧化的有效手段及合适的光化学手段来调控APSK酶的活性。
5 APSK重组酶动力学分析
合成重组APK作为融合蛋白与谷胱甘肽-S-转移酶(GST-APK)或作为与麦芽糖结合蛋白的融合蛋白。 我们怀疑植物APS激酶可能通过污染重组蛋白质制备物或商业凝血酶和因子Xa制剂中的蛋白酶而对降解非常敏感。而纯化的APK融合蛋白非常稳定,在-70 ℃储存1年或在室温下孵育24 h后没有催化活性损失。
由于发现融合蛋白是具有功能性的,所以可以直接用融合蛋白进行动力学试验。 我们使用酶偶联测定法测定APS激酶的活性。Lee等[37]研究表明,将APS滴定至含有5 mmol/L ATP的反应混合物中显示速度线性增加至10 mmol/L APS,但在较高的APS浓度下被抑制。在此试验中,最大速度为0.3 U/mg。与4种不同的酶制剂(2种MAL-APK和2种GST-APK)得到的结果相同,尽管比活度范围为0.3~0.5 U/mg。 该分析表明,理论Vmax为1.2 U/mg,动力学常数(Km)对于APS为3.6 mmol/L,ATP为1.9 mmol/L。所有制备物显示相同的动力学常数 。 最大观察速度和理论Vmax之间的差异可能是由于在高APS浓度下观察到的酶抑制。 这些结果类似于产气荚膜梭菌和大肠杆菌报道的结果。其中它被解释为APS是与ATP相关的非竞争性抑制剂。应该注意的是,由于活性测量使用其中65%是麦芽糖结合蛋白(MAL-APK)或谷胱甘肽转移酶(GST-APK)的APK融合蛋白进行,更实际的比活性可以是3倍以上 ,即0.9~1.5 U/mg[37]。这个值远低于产黄青霉APS激酶(24 U/mg)和大肠杆菌(153 U/mg)的比活性,但接近来自莱茵衣藻的纯APS激酶(2.2 U/mg )和酿酒酵母(0.8 U/mg)的比活性。
6 讨论
植物中的硫同化途径支持硫原子初级代谢中硫的还原和半胱氨酸的合成以及次级代谢的硫化分子的产生。通过APSR和APSK的活性平衡植物中初级和次级代谢途径间硫酸盐的分离。最近的工作揭示APSK对拟南芥生殖生存力的重要作用,但是这种酶在植物中的生物化学调节尚不清楚[23-26]。AtAPSK的结构和功能研究提供PAPS形成的分子基础和这一重要代谢分支酶的氧化还原控制的见解。提出APSK的反应顺序首先遵循ATP·Mg2+结合的顺序机制,然后进行APS催化,释放PAPS,释放ADP·Mg2+[38]。
APS激酶通过硫代葡萄糖苷网络的MYB因子在转录水平上调节[39]。转录物水平也对硫酸盐饥饿有反应,并与硫代葡萄糖苷的其他基因网络相结合,抑制mRNA。此外,在APSK酶活的氧化还原调节中,植物APS激酶具有氧化还原活性半胱氨酸对,当其减少时,增加催化效率并降低APS的抑制作用。由于APS还原酶被氧化激活,氧化还原调节可能有助于控制初级和次级代谢之间的硫分配[23,40-41]。然而,这需要在体内证明。 来自其他生物体的APS激酶与植物蛋白质类似,作为PAPS合成酶蛋白质的一部分或作为独立的酶[42]。除植物外,APS激酶尚未被证明是高度受调节的,也不被认为是硫代谢的关键调节步骤。这与植物特异性的氧化还原敏感性半胱氨酸对一致。
[1] KOPRIVA S,KOPRIVOVA A.Plant adenosine 5′-phosphosulphate reductase:The past, the present, and the future[J].Journal of experimental botany,2004,55(404):1775-1783.
[2] LI X,ZHONG W D,ALPHONES A,et al.Pilot-aided channel equalization in RGI-PDM-CO-OFDM systems[J].IEEE Photonics Technology Letters,2013,25(23):2304-2307.
[3] KOPRIVA S,FRITZEMEIER K,WIEDEMANN G,et al.The putative moss 3′-phosphoadenosine-5′-phosphosulfate reductase is a novel form of adenosine-5′-phosphosulfate reductase without an iron-sulfur cluster[J].Journal of biological chemistry,2007,282(31):22930-22938.
[4] MUGFORD S G,YOSHIMOTO N,REICHELT M,et al.Disruption of adenosine-5′-phosphosulfate kinase inArabidopsisreduces levels of sulfated secondary metabolites[J].Plant cell,2009,21(3):910-927.
[5] SATISHCHANDRAN C,MARKHAM G D.Adenosine-5′-phosphosulfate kinase fromEscherichiacoliK12.Purification,characterization, and identification of a phosphorylated enzyme intermediate[J].Journal of biological chemistry,1989, 264(25):15012-15021.
[6] LILLIG C H,SCHIFFMANN S,BERNDT C,et al.Molecular and catalytic properties ofArabidopsisthaliana,adenylyl sulfate (APS)-kinase[J].Archives of biochemistry & biophysics,2001,392(2):303-310.
[7] MACRAE I J,SEGEL I H.Adenosine 5′-phosphosulfate (APS) kinase:Diagnosing the mechanism of substrate inhibition[J].Archives of biochemistry & biophysics, 1999,361(2):277-282.
[8] PHARTIYAL P,KIM W S,CAHOON R E,et al.Soybean ATP sulfurylase, a homodimeric enzyme involved in sulfur assimilation, is abundantly expressed in roots and induced by cold treatment[J].Archives of biochemistry & biophysics,2006, 450(1):20-29.
[9] MACRAE I J,SEGEL I H,FISHER A J.Crystal structure of adenosine 5′-phosphosulfate kinase fromPenicilliumchrysogenum[J].Biochemistry,2000,39(7):1613-1621.
[10] YU Z H,LANSDON E B,SEGEL I H,et al.Crystal structure of the bifunctional ATP sulfurylase-APS kinase from the chemolithotrophic thermophileAquifexaeolicus[J].Journal of molecular biology,2007,365(3):732-743.
[11] GAY S C,SEGEL I H,FISHER A J.Structure of the two-domain hexameric APS kinase fromThiobacillusdenitrificans:Structural basis for the absence of ATP sulfurylase activity[J].Acta crystallographica,2009,65(10):1021-1031.
[12] SEKULIC N,DIETRICH K,PAARMANN I,et al.Elucidation of the active conformation of the APS-kinase domain of human PAPS synthetase 1[J].Journal of molecular biology,2007,367(2):488-500.
[13] SEKULIC N,KONRAD M,LAVIE A.Structural mechanism for substrate inhibition of the adenosine 5′-phosphosulfate kinase domain of human 3′-phosphoadenosine 5′-phosphosulfate synthetase 1 and its ramifications for enzyme regulation[J].Journal of biological chemistry,2007,282(30):22112-22121.
[14] SCHWENN J D,SCHRIEK U.A new role for thioredoxin in assimilatory sulphate reduction[J].FEBS Letters,1984,170(1):76-80.
[15] BURNELL J N,ANDERSON J W.Adenosine 5′-sulphatophosphate kinase activity in spinach leaf tissue[J].Biochemical journal,1973,134(2):565-579.
[16] JENDER H G,SCHWENN J D.Purification and properties of the ATP:Adenylylsulphate 3′-phosphotransferase fromChlamydomonasreinhardii[J].Archives of microbiology,1984,138(1):9-14.
[17] LEE S,LEUSTEK T.APS kinase fromArabidopsisthaliana:Genomic organization, expression, and kinetic analysis of the recombinant enzyme[J].Biochemical & biophysical research communications,1998,247(1):171-175.
[18] BICK J A,LEUSTEK T.Plant sulfur metabolism-the reduction of sulfate to sulfite[J].Current opinion in plant biology,1998, 1(3):240-244.
[19] VARIN L,MARSOLAIS F,RICHARD M,et al.Sulfation and sulfotransferases 6: Biochemistry and molecular biology of plant sulfotransferases[J].FASEB Journal:Official Publication of the Federation of American Societies for Experimental Biology,1997,11(7):517-525.
[20] SANDA S,LEUSTEK T,THEISEN M J,et al.RecombinantArabidopsisSQD1 converts udp-glucose and sulfite to the sulfolipid head group precursor UDP-sulfoquinovoseinvitro[J].Journal of biological chemistry,2001, 276(6):3941-3946.
[21] RENOSTO F,PATEL H C,MARTIN R L,et al.ATP sulfurylase from higher plants:Kinetic and structural characterization of the chloroplast and cytosol enzymes from spinach leaf[J].Archives of biochemistry & biophysics,1993,307(2):272-285.
[22] MACRAE I J,ROSE A B,SEGEL I H.Adenosine 5′-phosphosulfate kinase from Penicillium chrysogenum.site-directed mutagenesis at putative phosphoryl-accepting and ATP P-loop residues[J].Journal of biological chemistry, 1998,273(44):28583-28589.
[23] RAVILIOUS G E,NGUYEN A,FRANCOIS J A,et al.Structural basis and evolution of redox regulation in plant adenosine-5′-phosphosulfate kinase[J].Proceedings of the national academy of sciences of the United States of America,2012, 109(1):309-314.
[24] KOPRIVA S,MUGFORD S G,MATTHEWMAN C,et al.Plant sulfate assimilation genes:Redundancy versus specialization[J].Plant cell reports,2009,28(12):1769-1780.
[25] MUGFORD S G,MATTHEWMAN C A,HILL L,et al.Adenosine-5′-phosphosulfate kinase is essential forArabidopsisviability[J].FEBS Letters,2010,584(1):119-123.
[26] YATUSEVICH R,MUGFORD S G,MATTHEWMAN C,et al.Genes of primary sulfate assimilation are part of the glucosinolate biosynthetic network inArabidopsisthaliana[J].Plant journal, 2010, 62(1):1-11.
[27] HERRMANN J,NATHIN D,LEE S G,et al.Recapitulating the structural evolution of redox-regulation in adenosine-5′-phosphosulfate kinase from cyanobacteria to plants[J].Journal of biological chemistry,2015,290(41):24705-24714.
[28] ZHANG M,RAVILIOUS G E,HICKS L M,et al.Redox switching of adenosine-5′-phosphosulfate kinase with photoactivatable atomic oxygen precursors[J].Journal of the American chemical society,2012,134(41):16979-16982.
[29] RAVILIOUS G E,WESTFALL C S,JEZ J M.Redox-linked gating of nucleotide binding by the N-terminal domain of adenosine 5′-phosphosulfate kinase[J].Journal of biological chemistry,2013,288(9):6107-6115.
[30] BICK J A,SETTERDAHL A T,KNAFF D B,et al.Regulation of the plant-type 5′-adenylyl sulfate reductase by oxidative stress[J].Biochemistry, 2001,40(30):9040-9048.
[31] LILLIG C H, SCHIFFMANN S, BERNDT C, et al.Molecular and catalytic properties ofArabidopsisthaliana,adenylyl sulfate (APS)-kinase[J].Archives of biochemistry & biophysics, 2001, 392(2):303-310.
[32] PALDE P B,CARROLL K S.A universal entropy-driven mechanism for thioredoxin-target recognition[J].Proceedings of the national academy of sciences of the United States of America,2015,112(26):7960-7965.
[33] JEZ J M,CAHOON R E,CHEN S.Arabidopsisthalianaglutamate-cysteine ligase: functional properties, kinetic mechanism, and regulation of activity[J].Journal of biological chemistry,2004,279(32):33463.
[34] HOTHORN M,WACHTER A,GROMES R,et al.Structural basis for the redox control of plant glutamate cysteine ligase[J].Journal of biological chemistry,2006,281(37):27557-27565.
[35] HICKS L M,CAHOON R E,BONNER E R,et al.Thiol-based regulation of redox-active glutamate-cysteine ligase fromArabidopsisthaliana[J].Plant cell,2007,19(8):2653-2661.
[36] GROMES R,HOTHORN M,LENHERR E D,et al.The redox switch of -glutamylcysteine ligase via a reversible monomer-dimer transition is a mechanism unique to plants[J].Plant J,2008,54:1063-1075.
[37] LEE S,LEUSTEK T.APS kinase fromArabidopsisthaliana:Genomic organization, expression, and kinetic analysis of the recombinant enzyme[J].Biochemical & biophysical research communications,1998,247(1):171-175.
[38] RENOSTO F,SEUBERT P A,SEGEL I H.Adenosine 5′-phosphosulfate kinase fromPenicilliumchrysogenum.Purification and kinetic characterization[J].Journal of biological chemistry,1984,259(4):2113-2123.
[39] MUGFORD S G,LEE B R,KOPRIVOVA A,et al.Control of sulfur partitioning between primary and secondary metabolism[J].Plant J,2011,65(1):96-105.
[40] BICK J A,SETTERDAHL A T,KNAFF D B,et al.Regulation of the plant-type 5′-adenylyl sulfate reductase by oxidative stress[J].Biochemistry,2001,40(30):9040-9048.
[41] PATRON N J,DURNFORD D G,KOPRIVA S.Sulfate assimilation in eukaryotes:Fusions, relocations and lateral transfers[J].BMC Evolutionary Biology,2008,8(1):1-14.

