重要养殖鱼类经济性状QTL定位研究进展
(中国水产科学研究院黑龙江水产研究所,淡水鱼类育种国家地方联合工程实验室,黑龙江 哈尔滨 150070)
地球上大约有3 000种硬骨鱼类,据联合国粮食及农业组织(FAO)统计,养殖水产物种近400种。水产养殖业是农业领域发展最迅速的分支之一,随着水产养殖业的快速发展,产业中出现一系列问题,如产量、肉质和抗逆性下降、病害严重、性早成熟等,需要对水产养殖动物进行遗传改良和选育,而了解表型变异的遗传基础是最大的生物学挑战。大多数的表型性状是由多个数量性状位点(QTL)等位基因分离导致的数量遗传变异引起[1]。这种基因型-表现型复杂的关系包括环境的效应,应将标记辅助选择育种和自然群体的适应性变异给出重要的解释[2,3]。过去几十年对动植物的大量研究就是为了剖析基因型-表现型之间的关系。水产养殖动物的经济性状多为数量性状,传统的选育方法难以做到准确选择,需要借助各种分子生物学手段,如构建遗传连锁图谱进行QTL定位和性状-标记间关联分析等,发掘与重要经济性状相关的基因和标记,开展标记辅助育种,以提高育种效率[4]。
有关酵母、果蝇、小鼠和斑马鱼Danio rerio等模式生物的基因型-表现型关系的研究很多[5]。近年来,高通量测序技术的优势和应用使非模式物种的相关研究发展很快[6,7],连锁图谱和QTL定位在水产养殖物种中的研究也快速提升,使养殖鱼类重要性状的QTL研究取得了喜人的成果[8]。本文综述了近年来几种主要养殖鱼类QTL研究的现状及存在问题,并展望了未来发展趋势。
1 主要养殖鱼类QTL定位分析的主要经济性状
水产养殖动物的大多数经济性状诸如生长、肌肉质量和抗病性等都由多个基因、环境及其互作所控制。QTL是染色体上决定一个数量性状的区域,可能包含一个或多个基因。QTL作图的目的是弄清决定一个性状的基因的数量和效应,以有助于选择育种,加速重要经济性状的遗传改良速度。连锁分析、关联分析和连锁不平衡分析方法已经广泛用于发掘数量性状的基因位点和图谱定位[9]。尽管水产养殖动物的QTL定位研究明显晚于其他农业动植物,但在过去20年也有了长足的进步[10]。目前养殖鱼类的QTL定位研究主要集中于下列性状:
生长性状:包括体质量和体长的生长速率、饲料系数和利用率等,是水产物种最重要的经济性状。这些性状容易测量,在大多数水产物种中具有高遗传力,可以用传统的选择方法进行改良。几乎所有水产物种的生长性状都进行QTL定位分析。
肌肉品质性状:包括脂肪含量和分布、脂肪酸和氨基酸类型和比例、肌肉颜色、纹理和屠宰率等。多数情况下,这些性状只有在宰杀后才能精确测量,更需要分子标记辅助手段进行性状改良。
性成熟时间:可以导致一些水产物种生长减慢,鱼片质量降低,已在一些物种中开展了晚熟品种的选育。该性状表型选择困难,耗费时间,因此在早期阶段进行标记辅助选择(MAS,Marker-assisted selection)更适合。
性别决定:由一个或几个遗传因素、环境及其互作控制。性别决定因子在性染色体或常染色体上。有一些物种的性别与生长显著相关。例如雄性罗非鱼、黄颡鱼较雌性生长快很多。
抗病性:是水产物种QTL定位研究较多的性状,每个个体对病原的抗性分级,需要做攻毒试验,记录死亡和存活时间等。在大西洋鲑Salmo salar、虹鳟Oncorhynchus mykiss和牙鲆Paralichthys olivaceus等水产养殖动物中已经开展抗病性QTL研究。
温度耐受性、耐盐碱以及环境胁迫:这些性状主要在罗非鱼、虹鳟、大西洋鲑及亚洲海鲈Lates calcarifer中开展,目的是筛选在高盐碱度环境、超高/超低温度以及其他胁迫条件下生存的鱼类品种。
一些模式生物已经开展了数量性状的基因表达 QTL(eQTL)分析[11-13],而水产物种却尚未见报道。
2 几种重要养殖鱼类经济性状QTL定位的研究进展
目前已对20多种鱼类、贝类和甲壳类的重要经济性状开展了QTL定位分析[14]。养殖量大的鱼类主要有六种:罗非鱼是世界上最重要的淡水养殖鱼类,鲤是世界上养殖最广泛的淡水品种,虹鳟和大西洋鲑是广泛养殖的冷水性鱼类,而亚洲海鲈和牙鲆是新的重要海水养殖种类。这些重要养殖鱼类很多经济性状的QTL已经定位在图谱上(表1)。
2.1 虹鳟QTL定位
虹鳟QTL定位研究主要针对环境胁迫、抗病性、生长以及生殖等相关性状。
虹鳟是最早开展QTL定位研究的养殖鱼类。1998年,Jackson等[15]首次鉴定了虹鳟耐热性(Upper thermal tolerance,UTT)相关 QTL。三个 UTT相关QTL被定位在微卫星标记Omy325UoG、Ssa14DU和Ssa20.19NUIG附近,分别位于不同的连锁群上,验证了不同群体中Ssa20.19NUIG标记,证实与热耐受显著相关[16]。而Danzmann等[17]研究发现,一个热耐受相关标记的等位基因在一种遗传背景下对温度耐受起加强作用,而在另一种遗传背景下则相反。
了解胁迫反应对于改善动物福利和提高农业生产效率至关重要。Drew等[18]采用区间作图和复合区间作图对虹鳟家系OSU×Arlee杂交双单倍体群体对应急后皮质醇水平进行QTL定位,发现两个相关的QTL位点对皮质醇水平有正向作用,解释了43%的表型变异。Rexroad等[19]测定了64个虹鳟家系的拥挤应激性,发现与该性状显著相关的QTL位点位于第16染色体上,解释了40%~43%的表型变异。Rexroad等[20]利用F2家系定位了10个环境胁迫相关的QTLs,其中位于第12染色体上的一个QTL位点,解释表型变异为25%。Quillet等[21]利用SSR和SNP标记在两个虹鳟家系中分别鉴定了8个和5个应急反应QTLs,平均解释表型变异为8.3%,两个群体在第30染色体上有一个共享QTL。Liu等[22]采用RAD测序(Restriction-site associated DNA tag se-quencing)方法对虹鳟拥挤应激性进行全基因组关联分析,获得26个标记与该性状显著相关,其中16个位于第12染色体上。QTL定位分析表明,在染色体8和12上定位到两个QTL位点,分别解释表型变异为9%和16%,并鉴定了相关候选基因。
抗病性是另一个主要的QTL定位较多的性状。传染性造血细胞坏死(Infectious hematopoietic necrosis,IHN)是由病毒感染造成虹鳟大量死亡的疾病。Ozaki等[23]首次采用51个微卫星标记,初步定位了两个IPN抗病相关QTL位点。Rodriguez等[24]利用抗病的雄硬头鳟和雌虹鳟杂交产生的雄性F1,再回交得到70个家系(BC1)。从每个F1和BC1家系中取近100个个体感染IHN病毒。利用AFLP和微卫星标记构建了遗传连锁图谱,并进行了QTL定位,获得了16个AFLP标记和6个微卫星标记和IHN抗性有关。Khoo等[25]利用5个回交群体在LG2上鉴定到了一个新的QTL区域与IHN抗病性相关。Ozakil等[26]利用回交群体定位了7个传染性胰坏死 QTLs,分别位于第 11、12、17、23、26、29 和 31连锁群上。Verrier等[27]用两个DH群体对抗病毒性败血症(Rhabdovirus,VHSV)QTL做定位分析,分别获得了4个和3个QTL,共获得7个存活相关的QTL位点,分布于6个不同的连锁群上,在两个家系中均鉴定出其中一个QTL位点。Wiens等[28]在第19染色体上,鉴定出一个与抗细菌性冷水病(Bacterial cold water disease resistance,BCWD)的 QTL 位点。Vallejo等[29]用三个F2和回交群体研究BCWD抗性的QTL定位,在7个染色体上鉴定到9个QTLs,最大解释表型变异为40%,最低为13%。
生长是个涉及基因和环境互作的复杂过程。O’Malley等[30]利用两个远缘的虹鳟家系的回交群体首次鉴定了三个与体质量相关的QTLs。Wringe等[31]采用微卫星标记对10个亲本产生的两个回交群体和6个半同胞家系的体质量进行QTL定位,共获得10个体质量相关的QTL位点,仅有6个QTL位点在不同家系间共享。更为有趣的是,在雄性个体中鉴定出的QTL多于雌体,暗示家系背景对生长相关基因的影响。Gonzalez-Pena等[32]利用57K高密度SNP芯片对体质量和出肉率(Fillet Yield,FY)两个性状进行了全基因组关联分析,分别鉴定到12个和13个SNP与这两个性状相关。这些标记均位于第9染色体上,但解释遗传变异只有1.0%~1.5%,暗示这两个性状由微效多基因位点控制。
产卵季节与虹鳟的产量紧密相关。Sakamoto等[33]对虹鳟产卵期(Spawning time,SPT)性状进行QTL分析,鉴定到13个QTLs,分别位于7个连锁群上。O’Malley等[29]鉴定了4个回交群体SPT相关的QTLs。Haidle等[34]采用6个回交群体鉴定4个全基因组水平的性早熟相关的QTLs,其中两个与雌、雄个体显著相关。Colihueque等[35]用微卫星标记对产卵期与每年两次产卵行为之间的关联性进行了相关QTL和标记验证,发现三个控制产卵时间的QTLs影响每年两次产卵行为。Easton等[36]对胚胎的孵化率进行QTL定位,在第8连锁群上定位了两个QTL位点,其中一个位点(RT-8)解释表型变异达23.7%。
Le Bras等[37]还定位了渗透调节能力相关QTL位点,Norman等[38]研究耐盐性的QTL定位,定位了胚胎发育速率相关的QTLs[39,40]。
2.2 罗非鱼QTL定位
罗非鱼主要进行了性别控制、生长、耐寒及耐盐碱等性状相关的QTL定位研究。
关于罗非鱼性别决定(Sex determination,SD)基因QTL定位的研究很多。尼罗罗非鱼Oreochromis niloticus和莫桑比克罗非鱼O.mossambicus的性别由XX/XY(雄配子)系统控制,而蓝色罗非鱼O.aureus的性别控制由WZ/ZZ(雌配子)决定。几个性别连锁标记已经定位在罗非鱼不同连锁群上[41-47]。纯种尼罗罗非鱼的性别决定QTL位点定位在第1和第23连锁群[42,46,47],而尼罗罗非鱼与奥利亚罗非鱼的杂交种性别控制QTL被定位在第3连锁群上。Lee等[43,44]报道了位于尼罗罗非鱼第1连锁群上的两个微卫星标记UNH995和UNH104距离主性别决定QTL位点仅几个厘摩。位于第3连锁群上的三个标记(UNH168、GM271和 UNH31)距离 SD位点也很近。位于LG1和LG3上的SD相关QTL互作分析表明,位于第3染色体上的(W单倍型)QTL位点充当显性雌性的抑制者角色,而位于第1染色体上的(Y单倍型)QTL的功能是雌性的主要决定者。Eshel等[47]报道了一个主效SD QTL位于第23联锁区SSR标记的ARO172和ARO177之间。雌性个体中位于QTL附近的12个标记为纯合的,而在雄性个体中是杂合,即纯合位点发生了等位基因替换。这个染色体区段被定义为性别决定区域,这似乎表明尼罗罗非鱼的不同染色体上至少有两个主要性别控制位点。这两个位点之间是否存在互作或如何互作尚不清楚,需进一步研究。蓝罗非鱼的SD由位于第3染色体上的一个主要QTL控制。Liu等[48]利用微卫星标记构建了莫桑比克罗非鱼和红罗非鱼F1家系的连锁图谱,将莫桑比克罗非鱼XY性别决定QTL定位在连锁群LG1上,而在红色罗非鱼中XY则定位在LG22上。Palaiokostas等[49]采用RAD测序方法,构建了SNP遗传连锁图谱,定位两个SD相关QTL位点在LG1性别决定区域内,在LG20上发现一个新位点与性别决定相关。
尽管普遍认为,罗非鱼的性别由几个主要位点决定,但其他位点的微小效应及环境因子也影响性别[44]。罗非鱼的性别也可能是由基因和环境因素的网络调控机制决定[50,51]。基因之间及其与环境因子的互作也许非常复杂,需进一步研究。
Liu等[52]定位了罗非鱼生长相关性状体质量、全长和体长性状的QTL,分别获得10个、7个和8个QTLs,其中一个位于LG1上的生长相关QTL定位在性别决定位点上,解释表型(体质量、全长和体长)变异达到了71.7%、67.2%和64.9%,暗示了罗非鱼性别和生长之间的关系。Lin等[53]对耐盐碱罗非鱼家系的体质量性状进行QTL定位,获得5个雄性特异体质量QTL位点和三个雌性特异体质量QTL位点,分别位于不同染色体上。Cnaani等[54]定位了体长相关的QTLs。
Cnaani等[54,55]利用微卫星标记对耐寒、体长和免疫性状进行QTL定位研究,定位一个耐寒相关QTL(UNH879)、一个体长相关QTL和 6个免疫相关QTLs。Moen等[56]鉴定了一个位于第23连锁群上的耐寒相关标记UNH130。Rengmark等[57]将耐盐碱的候选基因,例如β血红蛋白、Ca2+转运质膜ATP酶及β-肌动蛋白等,定位在尼罗罗非鱼已有的图谱中。
2.3 鲤QTL定位
自从2004年Sun等[58]构建了鲤Cyprinus carpio的第一张遗传连锁图谱,并首次定位了抗寒性状基因位点以来,相继开展了其他性状的QTL定位研究,如生长、肌肉、生理、体型状和食物转化率等性状。鲤是进行QTL定位性状最多的养殖鱼类。
生长性状一直是养殖鱼类重要的经济性状之一,而鱼体质量、体长、体高、体厚、食物转化率等是鲤育种的主要目标性状。在不同类型的作图群体、家系和品种的鲤中获得了大量生长相关的QTLs[59-72]。Zhang等[59]利用DH家系获得6个体长相关的QTLs。Li等[60]利用全同胞家系鉴定了14个体质量相关QTLs,解释表型变异介于14.1%~45.5%之间,其中贡献率超过20%的主效QTLs有9个。张天奇等[61]对镜鲤体长性状进行了QTL定位,获得12个QTLs,解释表型变异为13.8%~64.9%。郑先虎等[62]基于父系QTL分析,鉴定5个与体质量相关QTLs,3个与体长相关;基于母系,鉴定6个体质量QTLs,5个体长QTLs。在第LG26连锁群上存在一个QTL区间与两个性状均相关。Lashari等[63]利用回交群体定位了体质量、体长等生长相关性状,共检测到14个QTLs与这些性状显著相关。Lashari等[64]定位了16个生长相关性状的QTL,解释表型变异17.0%~32.1%,其中有三个体质量相关QTLs、两个体长相关和两个体厚相关的QTLs在不同遗传背景的群体中一致。Wang等[65]对瓯江彩鲤生长性状进行QTL定位,获得25个QTLs,解释表型变异0.05%~61.40%之间。Gu等[66]采用微卫星标记构建了建鲤遗传连锁图谱,并对体质量性状进行QTL定位,获得8个QTLs,解释表型变异介于12.9%~17.3%之间。普通鲤家系体质量QTL定位比较结果表明,有3个QTLs一致。Lv等[67]对8个全同胞家系522个子代进行QTL定位分析,获得10个全基因组水平显著和28个染色体水平显著的QTLs位点,鉴定了多个家系共享的QTL位点。Peng等[68]利用鲤250KSNP芯片,构建了黄河鲤108个个体的家系高密度遗传连锁图谱,并对生长性状进行QTL定位分析,获得22个QTLs,鉴定一些生长相关的候选基因(KISS2,IGF1,SMTLB,NPFFR1和 CPE)。其他生长相关性状如眼径、眼间距、尾鳍长度及生长速率等的QTLs也被定位在不同染色体上[69-72]。
Kuang等[73]采用双重定位策略定位了镜鲤8个全同胞家系肌肉脂肪含量相关QTLs,获得一个位于第31连锁群上的基因组水平显著QTL,解释表型变异为36.2%;还鉴定了3个染色体水平的QTLs,分别位于LG3、LG42和LG45上,解释表型变异介于15.3%~19.5%之间。Zheng等[74]利用鲤250KSNP芯片对背、腹部肌肉脂肪含量、腹部脂肪重量及腹部脂肪重量占去内脏后体质量的百分比等四个肌肉脂肪含量性状进行全基因组关联分析,共检测到这四个性状18个位点达到全基因组显著水平,其中有一个位点对腹部脂肪重量及腹部脂肪重量占去内脏后体质量的百分比两个性状均达到了全基因组显著水平。10个QTL位点在一些已知基因附近,其中5个背部肌肉脂肪含量基因在高、低基因型中进行了验证。定量分析表明,其中4个基因(ankrd10a、tanc2、fjx1 以及 chka)呈现明显的差异表达,可作为肌肉脂肪含量的候选基因。
饲料转化率是水产养殖的重要性状。但它难于测量,很多水产养殖种类都没有开展相关工作,仅在鲤中有报道,获得了与饲料转化率显著相关的标记、QTL区间及候选基因[75-78]。王宣鹏等[76]获得了15个饲料转化率相关QTLs,解释表型变异17.7%~52.2%。Lu等[77]在两个群体中分别定位了9个QTLs,解释表型变异为17.0%~35.6%之间,并获得候选基因。
Zhang等[79]还首次鉴定了肌纤维性状相关QTLs。其他性状如游泳能力相关性状[80]、乳酸脱氢酶活性[81]、超氧化物酶歧化酶活性[82]及酸、碱性磷酸酶活性[83]等生理生化指标相关性状的QTLs也被定位。
2.4 大西洋鲑QTL定位
大西洋鲑QTL的研究主要集中在抗病性、性成熟和生长性状,体脂含量和肌肉颜色等其他性状也有少量研究。
抗病性一直是大西洋鲑QTL定位研究的热点。Moen等[84]利用340个AFLP标记对含有79个个体的全同胞家系进行攻毒试验,定位了两个传染性鲑贫血病抗性QTL位点。Houston等[85]首次对10个全同胞家系大西洋鲑传染性胰脏坏死病进行了QTL分析,在雄性中发现了两个全基因组水平的QTL位点,位于第21和26连锁群上。其中位于第21连锁群上的QTL位点解释表型变异达到20.9%。Moen等[86]利用10个家系定位了IPN抗性相关QTL,鉴定了一个主效QTL位点和14个微效QTL位点。主效QTL位于第21连锁群上,解释表型变异达24.6%。之后,Houston等[87]利用不同家系也获得了相同的QTL定位结果,鉴定出了位于第LG21连锁群的一个IPN抗性主效QTL位点,解释表型变异达50.9%。LG21上的三个DNA标记(SSa0285BSFU、Alu333和SSa0374BSFU/II)与抗IPN的QTL位点紧密连锁,可能是主效IPN抗性位点。Gonen等[88]利用两个群体病毒(Alphavirus,SAV)引起的胰腺病进行QTL定位分析,鉴定出6个抗病相关QTLs,其中位于第3染色体上的QTL位点达到了全基因组显著水平,在两个群体中结果一致。Gilbey等[89]利用39个微卫星标记在回交群体中鉴定出10个三代虫病抗性(Gyrodactylus salaris resistance)相关的QTL位点,解释表型变异占总变异的27.3%。Cauwelier等[90]鉴定出5个增生性肾病(Proliferative kidneydisease,PKD)相关 QTLs。
生长是大西洋鲑另一个进行QTL定位研究较多的性状。Reid等[91]鉴定出两个体质量相关QTLs,与虹鳟连锁群上的同源区域具有相似的效应[29]。Baranski等[92]构建了6个F2家系基于父系的遗传连锁图谱,对体质量、体长和体色三个性状进行QTL分析,共鉴定出13个和32个全基因组水平和染色体水平显著相关的QTLs。Gutierrez等[93]采用6.5KSNP芯片分型技术,对大西洋鲑体质量相关QTL进行定位分析,检测到染色体水平显著相关的QTL位点4个,分别位于第9、15、19和20染色体上,其中位于第9染色体上的一个QTL位点达到了全基因组水平,但仅在一个家系中检测到,解释表型变异达25.2%。Tsai等[94]利用20个父本的1695个子代的混合家系进行了QTL分析。在父系图谱的第13、18、19和20染色体上鉴定到全基因组显著水平的生长相关QTL位点;在母系图谱的第13、18和20染色体上检测到生长相关QTLs。Besnier等[95]利用6.5KSNP芯片对50个全同胞家系的生长性状进行GWAS分析,获得6个QTL与体长、体质量和环境因子相关。Gutierrez等[97]利用大西洋鲑6.5K SNP芯片,对471个个体的生长速率(体质量达到5kg所用的时间)和性成熟性状进行GWAS分析,仅在第13染色体上获得一个生长相关的位点(Ssa13),达到染色体显著水平。
Pedersen等[97]采用EST-SNP标记对大西洋鲑性成熟性状进行QTL定位分析,发现7个全基因组水平显著的位点,其中位于第21和23连锁群上的两个位点与虹鳟第22和4连锁群上的性成熟相关位点同源、类似。Gutierrez等[98]采用6.5KSNP芯片分型技术,鉴定了位于第10和21染色体上两个性早熟QTL位点,解释表型变异分别为16.7%和25%。仅发现一个QTL位点与性晚熟显著相关,位于第18染色体上,解释表型变异为14.7%。这些位点与之前鉴定到的生长相关的QTL位点相互独立。Ayllon等[99]对6条河流120个样本进行高通量全基因组测序,通过GWAS分析,鉴定了138个SNPs与性成熟显著相关,其中74个位于第25染色体上,覆盖约370Kb染色体区域。为了验证这一结果,分别在野生和驯化群体中加以验证。结果在驯化群体中缩小了该单倍型区域,发现一段长2 386bp含有4个SNP标记的基因组序列与该性状显著相关。此区域有一个vgll3基因(该基因与人类青春期年龄有关,暗示脊椎动物之间在成熟控制方面存在保守机制),解释表型变异33%~36%。
Derayatz等[100]对体脂含量性状进行全基因组扫描鉴定QTL,发现一个脂肪含量相关QTL位点,该位点1.3cM有一微卫星标记Ssa0016NVH与其紧密连锁。Sodeland等[101]利用SNP芯片对肌肉脂肪含量和肌肉紧实度进行GWAS分析,发现位于第9和10染色体上的两个位点对脂肪含量显著相关,而位于第3和11染色体上的两个位点与肌肉紧实度显著相关。Boulding等[102]对色素沉着和体型性状进行QTL定位分析。
2.5 亚洲海鲈QTL定位
亚洲海鲈QTL定位的主要是生长性状及少量肌肉品质和抗病性状等。
Yue等[103]对380个体的F1家系进行生长性状QTL定位分析,经过区间作图和多重性状模型分析,鉴定了32个QTL分布在10个连锁群上,其中三个与体质量、全长和体长相关的主效QTL被定位在LG2上的微卫星标记Lca287附近,解释表型变异分别为28.8%、58.9%和59.7%。另两个体质量相关QTL被定位在LG2(微卫星标记Lca287附近)和LG3(微卫星标记Lca154附近)上,分别解释表型变异6.4%和8.8%。Wang等[104]在不同的群体中验证了之前Yue等[103]鉴定到的体质量和体长紧密连锁的标记,其中位于LG2上的Lca371在多个家系中得到验证。而其他的相关标记仅在一个或两个家系中检测到。这暗示亚洲鲈生长相关QTL大多数为家系特异性。Feng等[105]利用加密图谱精细定位了生长性状QTL在染色体的相关区域,将位于连锁群2和3上的QTL区间缩小至0.2~1.4cM,并利用比较基因组分析鉴定了生长相关的候选基因。Wang等[106]定位了尾鳍长以及尾鳍长和体长的比例性状,发现了6个QTL位点,解释表型变异5.5%~16.6%。Xia等[107]对359个F2个体6月龄和9月龄的体质量进行全基因组扫描分析,发现9个QTL与6月龄体质量相关,另有9个QTL与9月龄体质量相关。其中两个月龄体质量性状有5个QTLs一致,分别位于 LG5、LG7、LG15、LG21 和 LG24 上,解释表型变异介于2.8%~10%之间。而位于LG2、LG3、LG6、LG9、LG11、LG13、LG18 和 LG23 上的 QTL 位点仅在其中一个年龄段被检测到。这些结果暗示不同发育阶段的生长相关QTL可能不同。位于LG5上的一个显著相关的QTL区间有一IFABP-a基因,为生长性状候选基因。Shen等[108]利用BAC克隆测序技术在染色体2区域鉴定了11个生长相关基因,其中三个基因(ctsb、skp1和 ppp2ca)上的 SNPs标记可作为快速生长的选择标记。
Xia等[109]定位了4个肌肉欧米伽-3多不饱和脂肪酸含量相关的QTL位点,分别定位在第LG6和LG23上,解释表型变异3.8%和6.3%。Liu等[110,111]利用RAD法进行SNP分型,对43个家系1 127个个体的病毒性神经坏死病(Viral nervous necrosis,VNN)和存活率两个性状进行QTL分析,分别获得4个QTLs,解释表型变异介于7.8%~11.0%之间。
2.6 牙鲆QTL定位
对牙鲆的QTL定位研究主要集中在对各种抗病相关标记和基因的鉴定工作,对其他性状只有少量研究。
Fuji等[112]采用50个微卫星标记对回交群体淋巴囊肿(Lymphocystis disease,LD)抗病位点进行QTL定位分析,在第15连锁群上,检测到一个主效QTL位点。位于该位点附近的微卫星标记Poli.9-8TUF解释表型变异达到总变异的50%。微卫星标记Poli.9-8TUF与抗病性QTL紧密连锁,该标记一个等位基因与淋巴囊肿病抗性显著相关,且具有正向作用,含有该等位基因的雌性个体被选作抗病性亲本。Fuji[113]等利用Poli.9-8TUF标记辅助选择选育了一个新的群体。利用选择的一个携带优势基因型的纯合雌性个体为母本,以快速生长、理想体型但不具抗病性等位基因的雄性个体为父本进行交配。抗病等位基因由母本传递给子代,所有的子代为LD-R杂合基因型。池塘养殖试验表明,子代对LD具有抗病性,而不具有该等位基因的对照组淋巴囊肿病存活率仅有4.5%~6.3%。这些结果表明,MAS是非常有效的育种策略。Xu等[114]利用76个MHC抗病等位基因,在牙鲆高抗(HR)和低抗
(LR)家系进行基因分型筛选,鉴定了一个标记Paol-DAB*4301在HR家系中出现的频率远远大于LR家系。
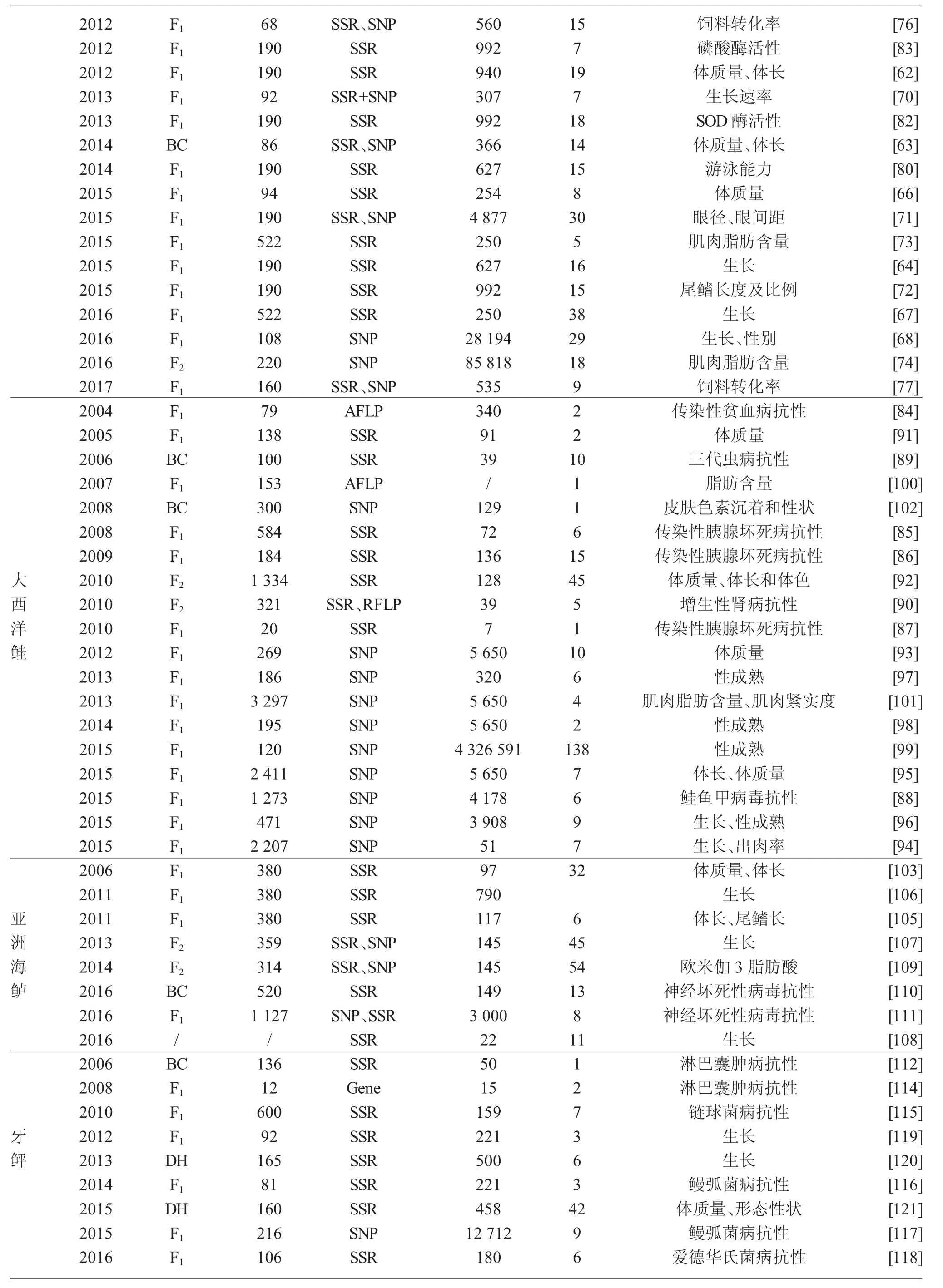
表1 (续)
Ozaki等[115]利用微卫星标记对牙鲆链球菌Streptococcus病抗性进行QTL分析,在第7、10、11和17连锁群上鉴定出7个相关位点。Wang等[116]定位了鳗弧菌(Vibrio anguillarum,VA)抗病性QTL位点,通过全基因组扫描鉴定出4个多态性标记,其中位于LG7上的两个标记在抗病和感病群体间差异显著。该连锁群上的一个主效QTL位点解释表型变异高达65%。Shao等[117]采用RAD测序法定位分析了鳗弧菌病抗性的QTL,鉴定出9个QTL。这些QTLs在第6和21连锁群形成两个簇,解释表型变异在5.1%~8.38%之间,对应的基因组区域含有12个免疫相关基因,其中4个基因与鳗弧菌病显著相关。Wang等[118]对牙鲆迟钝爱德华氏菌Edwardsiella tarda抗病性进行QTL分析,获得6个QTLs,解释表型变为16.0%~89.5%,其中两个QTLs是主效QTLs。
近年来,我国学者对牙鲆生长性状进行了QTL定位研究。牛余泽等[119]采用牙鲆日本和韩国群体杂交的F1群体,构建了牙鲆的遗传连锁图谱,对全长等三个生长性状进行QTL定位,获得三个QTLs(qgt-f4、qgt-m20和qgt-f20),可解释表型变异率分别为27.60%、3.74%和10.27%。刘奕等[120]对体长等6个生长相关性状进行QTL定位,检测到22个QTLs,分布在10个连锁群上。Yan等[121]对牙鲆体质量性状和形态特征进行QTL分析,采用贝叶斯模型剖析了性状的遗传结构,共鉴定42个主效QTL位点和59个互作QTL位点,其中第6染色体和第9染色体之间存在一个互作QTL,解释表型变异25.19%。
3 小结
近十几年鱼类QTL定位研究取得了显著进展。从研究方法看:连锁分析占绝对优势,仅有少量关联分析。标记使用:2010年以前,微卫星标记是最常用的标记类型;从2011年开始,SNP标记取代微卫星标记成为主流,到2015年,绝大多数研究都采用SNP标记。作图软件:比较常用的为MapQTL、GridQTL和各种R软件包。群体类型:鱼类QTL研究所使用的作图群体最多的是双亲杂交(F1)群体,也是最简单的实验设计,且与大多数软件兼容。总之,过去15年,QTL定位的图谱标记密度逐步增长,而到2011年,标记呈现急剧增加,相比较而言,群体样本量增长有限。
4 存在的问题及展望
尽管养殖鱼类重要经济性状的QTL定位研究进展迅速,但仍存在一些问题:一些多基因控制性状较小效应的QTLs检测能力缺乏、上位作用以及环境因素对QTL定位的影响、复杂表型性状之间的互作采取什么样的QTL定位策略等[122-124]。QTL定位的目的是鉴定给定性状的遗传变异,解释遗传成分。对微效QTL的检测能力和QTL定位的准确性、更高的样本容量和标记密度是设计实验时讨论最多的两个问题[125,126]。养殖鱼类用于QTL分析的样本量较农作物和畜禽小,多数样本小于200个,2010年之前构建图谱所用的标记也少,一般<150个标记,仅覆盖一部分基因组,导致一些重要的QTL未检测到。2011年之后,标记有了很大程度的提高,但很少研究能做到精细定位并鉴定候选基因或QTNs(Quantitative Trait Nucleotides)[14]。受一些因素限制,诸如缺乏研究资金和时间等,对多数QTL结果未进行确认和验证,仅有一小部分研究结果进行了确认,如亚洲鲈和虹鳟的生长性状、大西洋鲑的抗病性状以及日本牙鲆的抗病性状[30,86,104,113]。最近几年一些新研究手段的出现,使得基因分型技术发生了革命性的变化,但表型分析技术的进展却很缓慢。
针对上述问题,建议未来养殖鱼类QTL定位研究首先应加强鱼类重要经济性状表现型性状的鉴定。先进的表型组学有助于解开表型、基因型和环境之间的互作关系,减少背景变异,提高QTL的研究力量。加强更高基因组覆盖度的标记和加大样本量,以增强微小效应QTLs的检测能力。用多基因遗传分析鉴定不同位点之间的互作,更好地反应表型变异的多基因遗传性。用更高效数据分析方法增强在QTL分析中对复杂表型、表观遗传和环境数据的处理能力,不断构建高质量基因组测序组装,加速不同物种间比较基因组研究和QTLs共享。
[1] MackayTF C.The genetic architecture ofquantitative traits[J].Annual Review of Genetics,2001,35(3):303-339.
[2] Dekkers J C M.Application of genomics tools to animal breeding[J].Current Genomics,2012,13:207-212.
[3] Savolainen O,LascouxMand Merilä J.Ecological genomics of local adaptation[J].Nature Reviews Genetics,2013,14(11):807-820.
[4] Gjedrem T and Baranski M.Selective Breeding in Aquaculture:An Introduction[M].Netherlands:Springer,2009.
[5] Kahraman A,AvramovA,NashevLG,et al.PhenomicDB:a multi-species genotype/phenotype database for comparative phenomics[J].Bioinformatics,2005,21(3):418-420.
[6] Elshire R J,Glaubitz J C,Sun Q,et al.A robust,simple genotyping-by-sequencing(GBS)approach for high diversityspecies[J].Plos One,2011,6(5):e19379.
[7] Ellegren H.Genome sequencing and population genomics in non-modelorganisms[J].Trendsin Ecology&Evolution,2014,29(1):51-63.
[8] Tong J and Sun X.Genetic and genomic analyses for economicallyimportant traits and their applications in molecularbreedingofculturedfish[J].ScienceChina-LifeSciences,2015,58(2):178-186.
[9] Rockman MV.The QTN program and the alleles that matter for evolution:all that's gold does not glitter[J].Evolution,2012,66(1):1-17.
[10] Kocher T D and Kole C.Genome Mapping and Genomics in Fishes and Aquatic Animals[M].Berlin Heidelberg:Springer,2008.
[11] West MA L,Kim K,Kliebenstein D J,et al.Global eQTL mapping reveals the complex genetic architecture of transcript-level variation in Arabidopsis[J].Genetics,2007,175(3):1441-1450.
[12] Franke L and Jansen R C.eQTL analysis in humans[J].Methods in Molecular Biology,2009,573(May):311.
[13] Zhu Z,Zhang F,Hu H,et al.Integration of summary data from GWAS and eQTL studies predicts complex trait gene targets[J].Nature Genetics,2016,48(5):481-487.
[14] Yue GH.Recent advances ofgenome mappingand marker-assisted selection in aquaculture[J].Fish&Fisheries,2014,15(3):376-396.
[15] Jackson T R,Ferguson MM,Danzmann R G,et al.Identification of two QTL influencing upper temperature tolerance in three rainbow trout(Oncorhynchus mykiss)half-sib families[J].Heredity,1998,80(2):143-151.
[16] PerryG ML,Danzmann R G,Ferguson MM,et al.Quantitative trait loci for upper thermal tolerance in outbred strains of rainbow trout (Oncorhynchus mykiss)[J].Heredity,2001,86(3):333-341.
[17] Danzmann R G,Jackson TR and Ferguson MM.Epistasis in allelic expression at upper temperature tolerance QTLin rainbowtrout[J].Aquaculture,1999,173(1-4):45-58.
[18] Drew R E,Schwabl H,Wheeler P A,et al.Detection of QTL influencing cortisol levels in rainbow trout(Oncorhynchus mykiss)[J].Aquaculture,2007,272(2):S183-S194.
[19] Rexroad C E,Vallejo R L,Liu S,et al.QTL affecting stress response to crowding in a rainbow trout broodstock population[J].BMCGenetics,2012,13(1):1-12.
[20] Rexroad C E,Vallejo R L,Liu S,et al.Quantitative trait loci affecting response to crowding stress in an F2generation of rainbow trout produced through phenotypic selection[J].Marine Biotechnology,2013,15(5):613-627.
[21] Quillet E,Krieg F,Dechamp N,et al.Quantitative trait loci for magnitude of the plasma cortisol response to confinement in rainbowtrout[J].Animal Genetics,2014,45(2):223-234.
[22] Liu S,Vallejo R L,Gao G,et al.Identification of single-nucleotide polymorphism markers associated with cortisol response to crowding in rainbow trout[J].Marine Biotechnology,2015,17(3):328-337.
[23] Ozaki A,Sakamoto T,Khoo S,et al.Quantitative trait loci(QTLs)associated with resistance/susceptibility to infectious pancreatic necrosis virus(IPNV)in rainbow trout(Oncorhynchus mykiss)[J].Molecular Genetics&Genomics,2001,265(1):23-31.
[24] RodriguezMF,Lapatra S,Williams S,et al.Genetic markers associated with resistance to infectious hematopoietic necrosis in rainbow and steelhead trout(Oncorhynchus mykiss) backcrosses[J].Aquaculture,2004,241(1):93-115.
[25] Khoo S K,Ozaki A,Nakamura F,et al.Identification of a novel chromosomal region associated with infectious haematopoietic necrosis(IHN)resistance in rainbowtrout Oncorhynchus mykiss[J].Fish Pathology,2004,39(2):95-101.
[26] Ozaki A,KhooSK,Yoshiura Y,et al.Identification ofadditional quantitative trait loci(QTL)responsible for susceptibility to infectious pancreatic necrosis virus in rainbowtrout[J].Fish Pathology,2007,42(3):131-140.
[27] Verrier E R,Dorson M,Mauger S,et al.Resistance to a rhabdovirus(VHSV)in rainbowtrout:identification of a major QTL related to innate mechanisms[J].Plos One,2013,8(2):e55302.
[28] Wiens G D,Vallejo R L,Leeds T D,et al.Assessment of genetic correlation between bacterial cold water disease resistance and spleen index in a domesticated population of rainbow trout:identification of QTL on chromosome Omy19[J].Plos One,2013,8(10):e75749.
[29] Vallejo R L,Palti Y,Liu S,et al.Detection of QTL in rainbow trout affecting survival when challenged with Flavobacterium psychrophilum[J].Marine Biotechnology,2014,16(3):349-360.
[30] O'MalleyK G,SakamotoT,Danzmann R G,et al.Quantitative trait loci for spawning date and body weight in rainbow trout:testing for conserved effects across ancestrally duplicated chromosomes[J].Journal ofHeredity,2003,94(4):273-284.
[31] Wringe B F,Devlin R H,Ferguson M M,et al.Growth-related quantitative trait loci in domestic and wild rainbow trout (Oncorhynchus mykiss)[J].BMC Genetics,2010,11(1):63.
[32] Gonzalez-pena D,GaoG,Baranski M,et al.Genome-wide association study for identifying loci that affect fillet yield,carcass,and body weight traits in rainbow trout(Oncorhynchus mykiss)[J].Frontiers in Genetics,2016,7(Pt2):1-14.
[33] Sakamoto T,Danzmann R G and Okamoto N.Linkage analysis of quantitative trait loci associated with spawning time in rainbowtrout(Oncorhynchus mykiss)[J].Aquaculture,1999,173(173):33-43.
[34] Haidle L,Janssen J E,Gharbi K,et al.Determination of quantitative trait loci(QTL)for earlymaturation in rainbowtrout (Oncorhynchus mykiss)[J].Marine Biotechnology,2008,10(5):579-592.
[35] Colihueque N,Cárdenas R,Ramírez L,et al.Analysis of the association between spawning time QTL markers and the biannual spawning behavior in rainbow trout(Oncorhynchus mykiss)[J].Genetics&Molecular Biology,2010,33(3):578-582.
[36] Easton A A,Moghadam H K,Danzmann R G,et al.The genetic architecture of embryonic developmental rate and genetic covariation with age at maturation in rainbowtrout Oncorhynchus mykiss[J].Journal of Fish Biology,2011,78(2):602-623.
[37] Le Bras Y,Nicolas D,Francine K,et al.Detection of QTL with effects on osmoregulation capacities in the rainbow trout(Oncorhynchus mykiss)[J].BMC Genetics,2011,12(1):1-14.
[38] Norman J D,Robinson M,Glebe B,et al.Genomic arrangement of salinity tolerance QTLs in salmonids:a comparative analysis of Atlantic salmon(Salmo salar)with Arctic charr (Salvelinus alpinus)and rainbowtrout(Oncorhynchus mykiss)[J].BMC Genomics,2012,13(1):420.
[39] Robison B D,Wheeler P A,Sundin K,et al.Composite interval mapping reveals a major locus influencing embryonic development rate in rainbowtrout(Oncorhynchus mykiss)[J].Journal ofHeredity,2001,92(1):16-22.
[40] Sundin K,Brown K H,Drew R E,et al.Genetic analysis of a development rate QTL in backcrosses of clonal rainbow trout,Oncorhynchus mykiss[J].Aquaculture,2005,247(1):75-83.
[41] Shirak A,Palti Y,Cnaani A,et al.Association between loci with deleterious alleles and distorted sex ratios in an inbred line of tilapia (Oreochromis aureus)[J].Journal ofHeredity,2002,93(4):270-276.
[42] Lee B Y,Penman D J and Kocher T D.Identification of a sex-determining region in Nile tilapia(Oreochromis niloticus) using bulked segregant analysis[J].Animal Genetics,2003,34(5):379-383.
[43] Lee B Y,Hulata G and Kocher T D.Two unlinked loci controlling the sex of blue tilapia(Oreochromis aureus)[J].Heredity,2004,92(6):543-549.
[44] Lee B Y,Lee WJ,Streelman J T,et al.A second-generation genetic linkage map of tilapia(Oreochromis spp.)[J].Genetics,2005,170(1):237-244.
[45] Shirak A,Seroussi E,Cnaani A,et al.Amh and Dmrta2 genes map totilapia(Oreochromis spp.)linkage group 23 within quantitative trait locus regions for sexdetermination[J].Genetics,2006,174(3):1573-1581.
[46] Eshel O,Shirak A,Weller J I,et al.Fine mapping ofa locus on linkage group 23 for sex determination in Nile tilapia (Oreochromis niloticus)[J].Animal Genetics,2011,42(2):222-224.
[47] Eshel O,Shirak A,Weller J I,et al.Linkage and physical mapping of sex region on LG23 of Nile tilapia(Oreochromis niloticus)[J].G3:Genes Genomes Genetics,2012,2(1):35-42.
[48] Liu F,Sun F,Li J,et al.A microsatellite-based linkage map of salt tolerant tilapia(Oreochromis mossambicus×Oreochromis spp.)and mapping of sex-determining loci[J].BMCGenomics,2013,14(1):58.
[49] Palaiokostas C,Bekaert M,Khan M G,et al.A novel sex-determiningQTL in Nile tilapia(Oreochromis niloticus)[J].BMCGenomics,2015,16(1):171.
[50] Cnaani A,Lee B Y,Zilberman N,et al.Genetics of sex determination in tilapiine species[J].Sexual Development GeneticsMolecularBiology,Evolution,Endocrinology,Embryology&Pathology of Sex Determination&Differentiation,2008,2(1):43-54.
[51] Lühmann L M,Knorr C,Hörstgen-Schwark G,et al.First evidence for family-specific QTL for temperature-dependent sex reversal in Nile tilapia(Oreochromis niloticus)[J].Sexual Development,2012,6(5):247-256.
[52] Liu F,Sun F,Xia J H,et al.A genome scan revealed significant associations of growth traits with a major QTL and GHR2 in tilapia[J].Scientific Reports,2014,4(4):7256.
[53] Lin G,Chua E,Orban L,et al.Mapping QTL for sex and growth traits in salt-tolerant tilapia(Oreochromis spp.×O.mossambicus)[J].Plos One,2016,11(11):e0166723.
[54] Cnaani A,Hallerman E M,Ron M,et al.Detection of a chromosomal region with twoquantitative trait loci,affecting cold tolerance and fish size,in an F2 tilapia hybrid[J].Aquaculture,2003,223(1):117-128.
[55] Cnaani A,Zilberman N,Tinman S,et al.Genome-scan analysis for quantitative trait loci in an F2tilapia hybrid[J].Molecular Genetics&Genomics,2004,272(2):162-172.
[56] Moen T,Agresti J J,Cnaani A,et al.A genome scan of a four-way tilapia cross supports the existence of a quantitative trait locus for cold tolerance on linkage group 23[J].Aquaculture Research,2004,35(9):893-904.
[57] Rengmark A H,Slettan A,Lee W J,et al.Identification and mapping of genes associated with salt tolerance in tilapia[J].Journal of Fish Biology,2007,71(Supplement sc):409-422.
[58] Sun X and Liang L.A genetic linkage map of common carp(Cyprinus carpio L.)and mapping ofa locus associated with cold tolerance[J].Aquaculture,2004,238(1):165-172.
[59] Zhang Y,Liang L Q,Chang Y M,et al.Mapping and genetic effect analysis ofquantitative trait loci related tobody size in common carp (Cyprinus carpio L.)[J].Heredity,2007,29(10):1243-1248.
[60] Li W and Cui L.Mapping QTLs related to body weight of mirror carp (Cyprinus carpio L.)[J].Genomics&Applied Biology,2011,30(4):316-324.
[61] 张天奇,张晓峰,谭照君,等.镜鲤体长性状的QTL定位分析[J].遗传,2011,33(11):1245-1250.
[62] 郑先虎,曹顶臣,匡友谊,等.镜鲤体质量和体长的QTL定位研究[J].中国水产科学,2012,19(2):189-195.
[63] Laghari MY,Lashari P,Zhang X,et al.Mapping quantitative trait loci(QTL)for bodyweight,length and condition factor traits in backcross (BC1)family of Common carp(Cyprinus carpio L.)[J].Molecular BiologyReports,2014,41(2):721-31.
[64] Laghari MY,Lashari P,Zhang X,et al.QTL mapping for economically important traits of common carp(Cyprinus carpio L.)[J].Journal of Applied Genetics,2014,56(1):65-75.
[65] Wang J.Genetic map construction and quantitative trait locus(QTL)analysis on growth-related traits in common carp (Cyprinus carpio L.)[J].African Journal of Biotechnology,2012,11(31):7875-7884.
[66] Gu Y,Lu C,Zhang X,et al.Genetic mapping and QTL analysis for body weight in Jian carp(Cyprinus carpio var.Jian)compared with mirror carp (Cyprinus carpio L.)[J].Chinese Journal of Oceanology and Limnology 33(3):636-649.
[67] LvW,ZhengX,KuangY,etal.QTLvariationsforgrowth-relatedtraitsineightdistinctfamiliesofcommoncarp(Cyprinuscarpio)[J].BMCGenetics,2016,17(1):65.
[68] Peng W,Xu J,Zhang Y,et al.Erratum:an ultra-high density linkage map and QTL mapping for sex and growth-related traits of common carp(Cyprinus carpio)[J].Scientific Reports,2016,6:26693.
[69] Jin S,Zhang X,Jia Z,et al.Genetic linkage mapping and genetic analysis ofQTLrelated toeye cross and eye diameter in common carp (Cyprinus carpio,L.)using microsatellites and SNPs[J].Aquaculture,2012,358-359(6):176-182.
[70] Laghari MY,Yan Z,Lashari P,et al.Quantitative trait loci(QTL)associated with growth rate trait in common carp(Cyprinus carpio)[J].Aquaculture International,2013,21(6):1373-1379.
[71] Jin S B,Zhang X F,Lu J G,et al.Genetic analysis of QTL for eye cross and eye diameter in common carp(Cyprinus carpio L.)usingmicrosatellites and SNPs[J].Genetics&Molecular Research GMR,2015,14(2):3557.
[72] Lu C,Zhang Y,Zheng X,et al.Mapping QTLs of caudal fin length in common carp(Cyprinus carpio L.)[J].New Zealand Journal of Marine&Freshwater Research,2014,49(1):96-105.
[73] Kuang Y,Zheng X,Lv W,et al.Mapping quantitative trait locifor flesh fatcontentin common carp(Cyprinus carpio)[J].Aquaculture,2015,435(435):100-105.
[74] Zheng X,Kuang Y,Lv W,et al.Genome-wide association study for muscle fat content and abdominal fat traits in common carp(Cyprinus carpio)[J].Plos One,2016,11(12):e0169127.
[75] 张丽博,张晓峰,曹顶臣,等.利用SSR及EST标记对鲤鱼饲料转化率的QTL分析[J].农业生物技术学报,2010,18(5):963-967.
[76] 王宣朋,张晓峰,李文升,等.鲤饲料转化率性状的QTL定位及遗传效应分析[J].水生生物学报,2012,36(2):177-196.
[77] Lu C,Laghari MY,Zheng X,et al.Mapping quantitative trait loci and identifying candidate genes affecting feed conversion ratio based onto two linkage maps in common carp (Cyprinus carpio,L)[J].Aquaculture,2017,468(468):585-596.
[78] 张晓峰,李超,鲁翠云,等.鲤饲料转化率性状的关联分析及优异等位变异挖掘[J].水产学杂志,2017,30(3):11-18.
[79] Zhang Y,Xu P,Lu C,et al.Genetic linkage mapping and analysis of muscle fiber-related QTLs in common carp(Cyprinus carpio L.)[J].Marine Biotechnology,2011,13(3):376-92.
[80] Laghari MY,Lashari P,ZhangX,et al.Mapping QTLs for swimming ability related traits in Cyprinus carpio L.[J].Marine Biotechnology,2014,16(6):629-637.
[81] 毛瑞鑫,刘福军,张晓峰,等.鲤鱼乳酸脱氢酶活性的QTL 检测[J].遗传,2009,31(4):407-411.
[82] Xu Y,Zhang X,Zheng X,et al.Studies on quantitative trait loci related to superoxide dismutase in mirror carp(Cyprinus carpio L.)[J].Aquaculture Research,2013,44(12):1860-1871.
[83] 尹森,于倩,郑先虎,等.镜鲤碱性磷酸酶和酸性磷酸酶QTL 定位分析[J].上海海洋大学学报,2012,21(3):351-357.
[84] Moen T,Fjalestad K T,Munck H,et al.A multistage testing strategy for detection of quantitative trait loci affecting disease resistance in Atlantic salmon[J].Genetics,2004,167(2):851-858.
[85] Houston R D,Haley C S,Hamilton A,et al Major quantitative trait loci affect resistance to infectious pancreatic necrosis in Atlantic salmon (Salmo salar)[J].Genetics,2008,178(2):1109-1115.
[86] Moen T,Baranski M,Sonesson A K,et al.Confirmation and fine-mapping of a major QTL for resistance to infectious pancreatic necrosis in Atlantic salmon(Salmo salar):population-level associations between markers and trait[J].BMCGenomics,2009,10(1):368.
[87] Houston R D,Haley C S,Hamilton A,et al.The susceptibility of Atlantic salmon fry to freshwater infectious pancreatic necrosis is largely explained by a major QTL[J].Heredity,2010,105(3):318-327.
[88] Gonen S,Baranski M,Thorland I,et al.Mappingand validation ofa major QTLaffectingresistance topancreas disease (salmonid alphavirus)in Atlantic salmon(Salmo salar)[J].Heredity,2015,115(5):405-414.
[89] Gilbey J,Verspoor E,Mo T A,et al.Identification of genetic markers associated with Gyrodactylus salaris resistanceinAtlanticsalmonSalmo salar[J].DiseasesofAquatic Organisms,2006,71(2):119-129.
[90] Cauwelier E,Gilbey J,Jones C S,et al.Genotypic and phenotypic correlates with proliferative kidney disease-induced mortalityin Atlantic salmon[J].Diseases of Aquatic Organisms,2010,89(2):125-135.
[91] Reid D P,Szanto A,Glebe B,et al.QTL for body weight and condition factor in Atlantic salmon (Salmo salar):comparative analysis with rainbow trout(Oncorhynchus mykiss) and Arctic charr (Salvelinus alpinus)[J].Heredity,2005,94(2):166-172
[92] Baranski M,Moen T and Vage D I.Mapping of quantitative trait loci for flesh colour and growth traits in Atlantic salmon (Salmo salar)[J].Genetics Selection Evolution,2010,42(1):1-14.
[93] Gutierrez A P,Lubieniecki K P,Davidson E A,et al.Genetic mapping of quantitative trait loci (QTL) for body-weightinAtlanticsalmon(Salmo salar)usinga6.5K SNParray[J].Aquaculture,2012,358-359(2):61-70.
[94] Tsai H Y,Hamilton A,Guy D R,et al.The genetic architecture ofgrowth and fillet traits in farmed Atlantic salmon(Salmo salar)[J].BMCGenetics,2015,16(1):51.
[95] Besnier F,Glover K A,Lien S,et al.Identification of quantitative genetic components of fitness variation in farmed,hybrid and native salmon in the wild[J].Heredity,2015,115(1):47-55.
[96] Gutierrez A P,Lubieniecki K P,Fukui S,et al.Detection of quantitative trait loci(QTL)related to grilsing and late sexual maturation in Atlantic salmon (Salmo salar)[J].Marine Biotechnology,2014,16(1):103-110.
[97] Pedersen S,Berg P R,Culling M,et al.Quantitative trait loci for precocious parr maturation,early smoltification,and adult maturation in double-backcrossed trans-Atlantic salmon (Salmo salar)[J].Aquaculture,2013,410-411(2):164-171.
[98] Gutierrez A P,Yáñez J M,Fukui S,et al.Genome-wide association study(GWAS)for growth rate and age at sexual maturation in Atlantic salmon(Salmo salar)[J].Plos One,2015,10(3):e0119730.
[99] Ayllon F,E Kjærnersemb E,Furmanek T,et al.The vgll3 locus controls age at maturity in wild and domesticated Atlantic salmon(Salmo salar L.)males[J].Plos Genetics,2015,11(11):e1005628.
[100] Derayat A,Houston R D,Guy D R,et al.Mapping QTL affecting body lipid percentage in Atlantic salmon(Salmo salar)[J].Aquaculture,2007,272 (1):S250-S251.
[101] Sodeland M,Gaarder M,Moen T,et al.Genome-wide association testing reveals quantitative trait loci for fillet texture and fat content in Atlantic salmon[J].Aquaculture,2013,408-409(2):169-174.
[102] Boulding E G,Culling M,Glebe B,et al.Conservation genomics ofAtlantic salmon:SNPs associated with QTLs for adaptive traits in parr from four trans-Atlantic backcrosses[J].Heredity,2008,101(4):381.
[103] Yue G,Zhu Z,LoongL,et al.A genome scan for quantitative trait loci affecting growth-related traits in an F1 familyofAsian seabass(Lates calcarifer)[J].BMC Genomics,2006,7(1):274.
[104] WangCM,LoLC,FengF,et al.Identification and verification of QTL associated with growth traits in two genetic backgrounds of Barramundi(Lates calcarifer)[J].Animal Genetics,2008,39:34-39.
[105] Feng F,Loong L,Fei S,et al.A high-resolution linkage map for comparative genome analysis and QTL fine mapping in Asian seabass,Lates calcarifer[J].BMC Genomics,2011,12(1):174.
[106] Wang C M,Lo L C,Zhu Z Y,et al.Mapping QTL for an adaptive trait:the length of caudal fin in Lates calcarifer[J].Marine Biotechnology,2011,13(1):74-82.
[107] Xia J H,Lin G,He X,et al.Whole genome scanning and association mapping identified a significant association between growth and a SNP in the IFABP-a gene of the Asian seabass[J].BMCGenomics,2013,14(1):295.
[108] Shen X,Si Y N,Thevasagayam N M,et al.BAC-pool sequencing and analysis confirms growth-associated QTLs in the Asian seabass genome[J].Scientific Reports,2016,6:36647.
[109] Xia J H,Lin G,He X,et al.Mapping quantitative trait loci for Omega-3 fattyacids in Asian seabass[J].Marine Biotechnology,2014,16(1):1-9.
[110]Liu P,Wang L,Wan Z Y,et al.Mapping QTL for resistance against viral nervous necrosis disease in Asian seabass[J].Marine Biotechnology,2016,18(1):107-116.
[111] Liu P,WangL,Sek-Man W,et al.Fine mappingQTLfor resistance to VNN disease using a high-density linkage map in Asian seabass[J].Scientific Reports,2016,6:32122.
[112] Fuji K,Kobayashi K,Hasegawa O,et al.Identification of a single major genetic locus controlling the resistance to lymphocystis disease in Japanese flounder(Paralichthys olivaceus)[J].Aquaculture,2006,254(1):203-210.
[113] Fuji K,Hasegawa O,Honda K,et al.Marker-assisted breeding of a lymphocystis disease-resistant Japanese flounder (Paralichthys olivaceus)[J].Aquaculture,2007,272(1-4):291-295.
[114] Xu T J,Chen S L,Ji X S,et al.MHC polymorphism and disease resistance to Vibrio anguillarum in 12 selective Japanese flounder(Paralichthys olivaceus)families[J].Fish&Shellfish Immunology,2008,25(3):213-221.
[115] Ozaki A,Okamoto H,Yamada T,et al.Linkage analysis ofresistance to Streptococcus iniae infection in Japanese flounder(Paralichthys olivaceus)[J].Aquaculture,2010,308(1):S62-S67.
[116] Wang L,Fan C,Liu Y,et al.A genome scan for quantitative trait loci associated with Vibrio anguillarum infection resistance in Japanese flounder(Paralichthys olivaceus)bybulkedsegregantanalysis[J].MarineBiotechnology,2014,16(5):513-521.
[117] ShaoC,Niu Y,Rastas P,et al.Genome-wide SNP identification for the construction of a high-resolution genetic map of Japanese flounder(Paralichthys olivaceus):applications to QTL mapping of Vibrio anguillarum disease resistance and comparative genomic analysis[J].DNA Research,2015,22(2):161-170.
[118] Wang X X,Xu W T,Liu Y,et al.Quantitative trait loci detection of Edwardsiella tarda resistance in Japanese flounder Paralichthys olivaceus using bulked segregant analysis[J].Chinese Journal of Oceanology&Limnology,2016,34(6):1297-1308.
[119] 牛余泽,廖小林,宋文涛,等.牙鲆遗传作图及生长性状QTL 定位[J].水产学报,2012,36(11):1640-1649.
[120] 刘奕,王桂兴,周丹,等.牙鲆身体纵轴生长相关性状QTL 定位[J].中国水产科学,2013,20(3):514-520.
[121] Yan C,Hongwei W,Xuemei Q,et al.Bayesian analysis for genetic architectures ofbody weights and morphological traits using distorted markers in Japanese flounder Paralichthys olivaceus[J].Marine Biotechnology,2015,17(6):693-702.
[122] Mackay T F,Stone E A and Ayroles J F.The genetics of quantitative traits:challenges and prospects[J].Nature Reviews Genetics,2009,10(8):565-577.
[123] Slate J.From beavis to beak color:a simulation study to examine how much QTL mapping can reveal about the genetic architecture of quantitative traits[J].Evolution,2013,67(5):1251-1262.
[124] Wellenreuther M and Hansson B.Detecting polygenic evolution:problems,pitfalls,and promises[J].Trends in Genetics,2016,32(3):155-164.
[125] Hu Z and Xu S.A simple method for calculating the statistical power for detectinga QTLlocated in a marker interval[J].Heredity,2008,101(1):48-52.
[126] CeCile M,Henk B,Chris H,et al.QTL mapping designs for aquaculture[J].Aquaculture,2008,285(1):23-29.

