面向肿瘤精准医学的综合数据资源TCGA及其相关在线分析工具推荐
, ,睿,, ,,
癌症是一种极为复杂的人类疾病,涉及基因组的多种动态变化[1]。每种类型的癌症,发生的遗传畸变都是独特的,包括体细胞突变、拷贝数变异、基因表达谱差异和表观遗传改变。因此需要更好地理解肿瘤的各种遗传变化,才能更好地对其进行诊断、治疗和预防。全基因组测序和生物信息技术的发展为癌症基因组研究提供了新的线索[2]。典型的综合数据资源是癌症基因组图谱(The Cancer Genome Atlas,TCGA)项目,它收集整理了大量癌症基因组数据,并利用新的基因组分析技术以加速对癌症的全面了解。
TCGA数据库的目标是完成一套完整的与所有癌症基因组改变相关的“图谱”,旨在获得癌症生物学的新见解,从而有助于癌症的治疗。该项目是2006年由美国国立卫生研究院牵头的一项大型癌症基因组计划,自2008年开始有阶段性成果发表[3],2009年继续投资2.75亿美元,增加了多种类型的癌症数据,到2014年已收集了36类癌症数据,包括临床数据、DNA、RNA、蛋白质等多层次的数据。在数据生成方面,该项目取得了无可争议的成功。随着样品采集、测序和分析技术的快速发展,TCGA收录的肿瘤相关数据呈指数增长。目前,新成立的NCI Genomics Data Commons将TCGA的数据整合在该门户网站中,并且为基因组数据用户提供了交互式支持和更清晰友好的界面。
我们可以用前所未有的微观视角来看待癌症,但是还没有达到能够解释这种疾病的全貌的程度,对其发病机制亦不完全清楚。而TCGA数据已被用于发现新的突变,确定内在的肿瘤类型,确定泛癌相似性和差异性,同时收集肿瘤演变的证据。目前已经开发了大量针对TCGA数据的生物信息学工具,反映出TCGA数据资源的重要性。
1 TCGA数据介绍
为了全面分析癌症基因组图谱,TCGA应用基于微阵列和下一代测序方法的高通量技术,产生了癌症的多种数据类型信息。
TCGA中的癌症数据通过各种标识符(ID)进行识别和编目(表1),每种癌症类型都包括体细胞突变、拷贝数、基因表达、miRNA表达、DNA甲基化、逆转蛋白相位阵列(RPPA)和临床信息。除原始排序文件外(表2),每种数据类型都包括可供公开下载的原始数据和已处理的数据。

表1 TCGA数据库中的ID号

表2 数据类型和可获取水平
2 TCGA数据在线分析工具
目前TCGA数据分析很复杂,涉及多个步骤,为获得有意义的生物学结果,需要仔细考虑分析每个步骤,并将特定工具应用于某些实验模型。为现有数据开发相关的探索工具,需要实验科学家和计算科学家之间的协调。然而,实验科学家很难使用计算科学家开发的计算工具,因为这些计算工具需要数据准备以及安装和使用打包软件,而且某些软件往往只适用于某些特定平台或操作系统。一些更高级的计算工具往往难以理解或使用,从而限制了其应用。不过有基于网络的工具可以提供方便的计算解决方案,帮助实验科学家使用和分析复杂的癌症基因组数据。这些工具帮助无生物信息学背景的生物学家和医学家获得更多的生物学和医学见解,但是选择适当的工具并不是一项简单的任务,对于没有经验的用户来说尤其如此。
本文整理了一个基于网络的可用于分析TCGA数据的公开工具列表,并将这些工具进行分类以便更好地进行查询和使用。
表3显示了基于网络工具的32个在线分析资源,它们代表了当前可用于分析TCGA数据的主要资源。为了进一步区分和指导这些工具的选择,本文将所有资源工具分为全局分析工具、目标分析工具和辅助分析工具三大类。
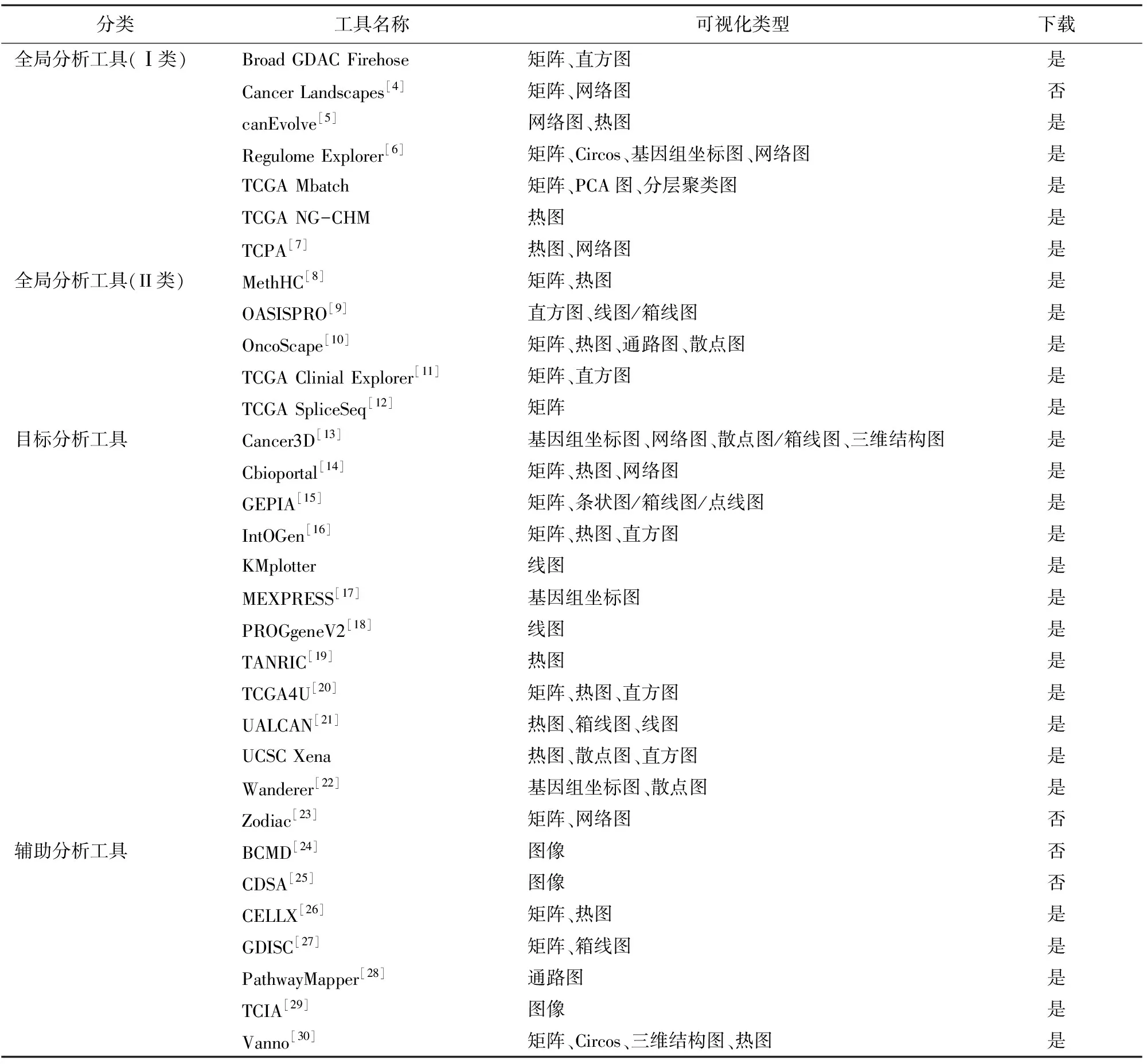
表3 针对TCGA数据的在线分析资源
全局分析工具能够检查癌症基因组的整体特征,可以成为刚刚开始研究癌症基因组数据研究人员的宝贵资源。全局分析工具有两种类型即Ⅰ型和Ⅱ型,前者仅提供全局分析,后者则提供除全局分析之外的选定目标分析。
目标分析工具是研究人员最常使用的基于网络的公共工具。这些工具可以令研究人员深入分析具体的基因或者基因集,甚至miRNA等研究对象,方便使用者调查癌症数据中自己感兴趣的目标。
基于公共网络的辅助分析工具可以将TCGA数据转换为易于访问、浏览和下载的在线资源。这些数据可以帮助用户补充实验结果或者提供额外的证据和解释,帮助研究人员更全面地分析自己的研究和促进生物学发现。
3 TCGA数据在线分析工具推荐
首先可以由本文的分类区分不同工具的使用类型,缩小选择范围;然后根据实际需要结合具体研究(如数据来源、数据类型、分析方法、研究目的),选择具体的工具进行进一步的分析。以下是对TCGA数据进行不同分析时建议选择的一些工具,但这些工具都不能完全取代先进的计算和统计方法,只是为研究人员提供一些使用帮助,扩展他们癌症组学、癌症复杂性和癌症网络等方面的相关知识。
3.1 突变分析
有10种在线工具(Broad GDAC Firehose,Cancer3D,cbioportal,CELLX,IntOGen,TANRIC,TCGA Clinical Explorer,TCGA4U,UCSC Xena和Vanno)可以进行突变分析。一般来说,推荐使用cbioportal,因其包含多种癌症类型和多种可视化分析功能,功能强且易于使用。
3.2 相关性分析
有17种在线工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,cbioportal,CELLX,GDISC,GEPIA,MethHC,MEXPRESS,OASISPRO,Regulome Explorer,TANRIC,TCGA Clinical Explorer,TCGA NG-CHM,TCPA,Wanderer和Zodiac)可以进行相关性分析。总的来说,推荐使用麻省理工学院和哈佛大学Broad研究所研发的Broad GDAC Firehose,因其有多种分析算法供用户使用,功能全面,且包含多种分析工具。
3.3 差异分析
有12种在线工具(Broad GDAC Firehose,canEvolve,cbioportal,CELLX,GEPIA,MEXPRESS,OncoScape,TANRIC,TCGA4U,TCPA,UALCAN和Wanderer)可以进行差异分析,一般推荐使用分析基因表达谱的工具GEPIA。差异分析是该工具的主要分析功能,其在线分析界面简单易懂,非常易于理解和使用。
3.4 通路分析
有8种在线工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,MethHC,OncoScape,PathwayMapper,Regulome Explorer和TCGA NG-CHM)可以进行通路分析。推荐使用Broad GDAC Firehose和OncoScape,前者分析方法丰富,后者简单直观。
3.5 生存分析
有16种在线工具(Broad GDAC Firehose,Cancer Landscapes,canEvolve,cbioportal,CELLX,GDISC,GEPIA,KMplotter,OASISPRO,PROGgeneV2,TANRIC,TCGA Clinical Explorer,TCGA4U,TCPA,UALCAN和UCSC Xena)可以进行生存分析。如果仅想进行单一的生存分析,推荐使用PROGgeneV2,因其具有广泛的数据来源和多种可选参数设置。
3.6 泛癌分析
有8种在线工具(Broad GDAC Firehose,CancerLandscapes,cbioportal,IntOGen,Regulome Explorer,TCGA NG-CHM,UCSC Xena和Zodiac)可以进行泛癌症分析(pan-cancer analysis)。一般来说,推荐使用cbioportal和Cancer Landscapes,前者收集了来自泛癌研究的大量样本且拥有强大的分析能力;后者的癌症图谱模型中包含了泛癌模型,可以直接用于分析。
4 总结
科学家们开发出多种生物信息学工具进行数据挖掘和分析,以便寻找新发现。不久的将来,新发现将有助于诊断、治疗和预防癌症。TCGA提供的癌症基因组学数据可以系统地揭示癌症分子生物学的新图景。这些大量公开可用的数据,为世界各地的研究人员提供了癌症遗传学的知识来源,结合多种分析有助于开发个性化癌症药物。本文全面整理了基于网络的公共可用的在线分析资源和工具,可以帮助研究人员方便地查找和使用合适的工具,增进他们对癌症基因组学的理解。

