烟草野火病菌特异性检测引物筛选及应用
陈瑞朋,卢 凯,张 敏,刘元德,宗 浩,牛纪军,刘文涛,徐后娟*
烟草野火病(Tobacco Wildfire)是由丁香假单胞菌烟草致病变种(Pseudomonas syringaepv.Tabaci)危害引起的一种烟草中后期叶部病害,在世界范围内广泛分布。该病害在我国各主产烟区均有报道,近几年在云南、山东等主要产烟区的发生危害普遍且呈上升趋势,气候适宜时极易造成大面积流行,从而影响到烟草的优质高效生产[1-2]。目前对于烟草野火病的检测方法一般是基本对发病症状的观察,此类方法必须是在发病之后,无法对病害进行提前的预报[3-6],且田间病害症状发病初期和其他几种叶部病害相似,生产上容易导致误诊。利用分子生物学病害诊断法具有准确、灵敏、快速等优点,已经广泛应用于植物病害的诊断[7-9]。本研究利用Mauve软件,对不同的假单胞菌属以及丁香假单胞菌不同致病变种的全基因组进行比对,找到了丁香假单胞菌烟草致病变种的特异性区段,并据此进行了丁香假单胞菌烟草致病变种的检测引物的筛选和应用研究[10]。
1 材料与方法
1.1 试验材料
供试菌株:烟草野火病标准菌株由牡丹江烟草研究所提供。其他烟草野火病菌分别采集于山东省诸城市、高密市、临朐县、沂水县、蒙阴县、沂南县、日照岚山以及贵州省、湖北省、福建省、云南省。供试对照菌株均为实验室保存,分别为虫媒假单胞杆菌(P. entomophila)、类黄色假单胞杆菌(P.parafulva)、恶臭假单胞杆菌(P. putida)、嗜盐假单胞杆菌(P. psychrotolerans)、荧光假单胞杆菌(P.fluorescens)、青枯假单胞杆菌(P. solanacearum)、巨大芽胞杆菌(Bacillus megaterium)、枯草芽胞杆菌(B. subtilis)、尖孢镰刀菌(Fusarium oxysporum)、链格孢菌(Alteraria alternata)、立枯丝核菌(Rhizoctonia solani)、沙福芽孢杆菌(B. safensis)、嗜麦芽寡养单胞菌(Stenotrophomonas maltophilia)、稻黄单胞菌(Xanthomonas oryzae)、欧文氏杆菌(Erwinia carotovora);DNA聚合酶等购置于诺唯赞生物科技有限公司,引物由上海生工生物科技有限公司合成,PCR测序由青岛擎科梓熙生物技术有限公司完成,DL1000 maker购置于大连宝生物工程有限公司。
1.2 试验方法
1.2.1 烟草野火病菌引物设计 从NCBI基因组数据库中下载全基因组数据,包括假单胞菌属 25个不同菌种以及127个丁香假单胞菌属不同致病变种,利用软件Mauve 2.3.1进行全基因组比对,获得特异性区段之后,利用Primer 6.0软件进行引物设计。
1.2.2 细菌DNA的提取 DNA模板的简易制备参照赫然等的煮沸法[11]。基因组 DNA的提取使用CTAB 法[12]。
1.2.3 PCR鉴定引物特异性 首先将设计的13对引物与烟草野火病标准菌株进行PCR扩增,筛选出能够产生单一的目的大小条带的引物,之后利用筛选出来的引物与对照菌株进行PCR扩增,观察其能否与其他病原菌产生阳性反应。PCR扩增条件为:94 ℃预变性10 min; 94 ℃变性30 s,5 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃再延伸10 min;PCR加样体系如表1。
1.2.4 烟草感病叶片中野火病模板的制备 取大田中疑似感病的烟草叶片 10片,进行表面消毒,将病斑以及病斑周围1 cm以内的叶片剪下,以病斑圆心为中心点,每3 mm将叶片切条,放于100 μL超纯水中浸泡两天两夜。取50 μL浸泡液于金属浴100 ℃加热 10 min作为模板备用。感病叶片检测PCR加样体系如表2。
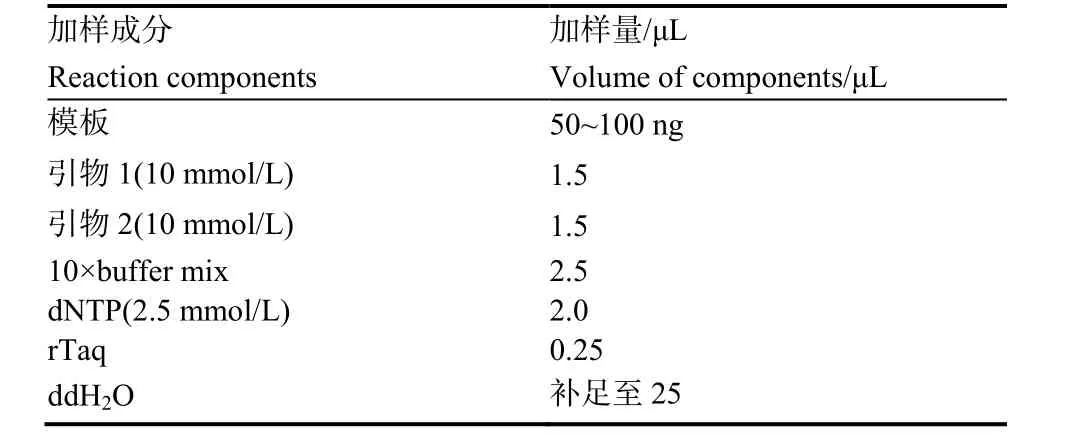
表1 PCR加样体系Table 1 PCR system
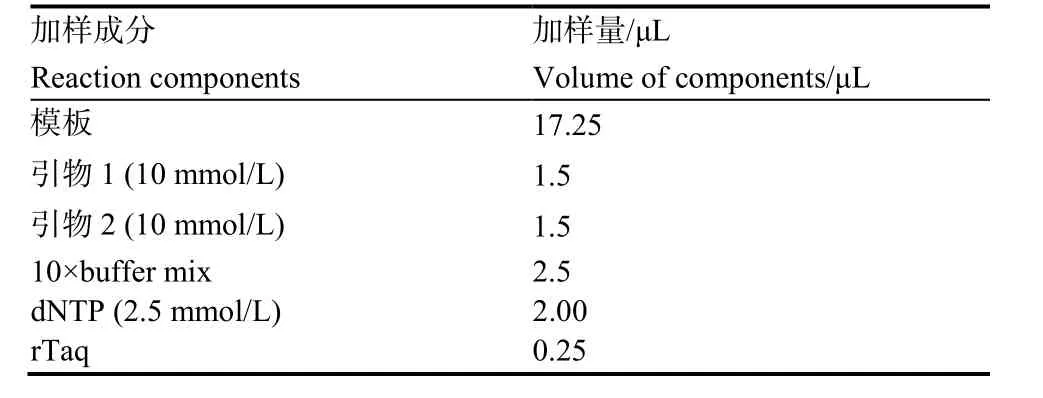
表2 PCR加样体系Table 2 PCR system
2 结 果
2.1 引物设计
利用软件Mauve 2.3.1进行全基因组比对,共找到P. syringae pv.tabaciATCC 11528的特异性序列段51个,进一步利用Primer Premier 6.0对这些特异性序列段进行引物的设计,共设计引物13对,如表3所示。
2.2 引物筛选
将设计好的13对引物分别进行PCR反应,模板为烟草野火病标准菌株,结果如图1所示。其中引物5455、48016、40429、33239以及27344均能扩增出目的大小条带,测序验证为烟草野火病菌,其余8对引物扩增条带不理想,杂带过多。

表3 烟草野火病菌引物Table 3 Primers of Pseudomonas syringae pv. Tabaci

图1 烟草野火病菌引物筛选结果Fig. 1 Amplification results of P. syringae pv. Tabaci with different primers
2.3 引物的特异性筛选
将筛选出的5对引物分别与供试15种对照菌株进行普通PCR扩增,检测初筛的5对引物的特异性。结果如图2所示,引物5455、48016、33239、27344对于部分其他菌株能够扩增出条带,引物40429对于15种相近其他病菌株均未扩增出条带。因此,确定引物40429为最佳烟草野火病菌的最佳引物。

图2 不同引物对照菌种的PCR扩增结果Fig. 2 Amplification results of control bacteria with different primers
2.4 引物的特异性检验
用实验室保存的来自 11个不同地区和标准的烟草野火病病菌,对引物 40429进行特异性检测。如图3所示:引物40429均能够对于来自不同地区的烟草野火病扩增出单一的目的条带,条带大小为203 bp,测序之后在NCBI进行Blast比对显示为烟草野火病菌,说明引物40429能够对烟草野火病菌进行良好的特异性扩增。
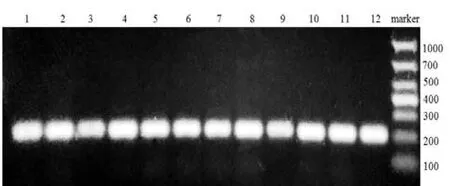
图3 引物40 429对不同地区烟草野火病鉴定结果电泳图Fig. 3 Amplification results of P. syringae pv. tabaci from different regions with primer pair 40 429
2.5 烟草野火病病菌最低检测浓度的测定
用设计的最优引物40 429对烟草野火病菌进行PCR扩增,模板取1 μL。将烟草野火病菌分别稀释为 1010、109、108、107、106、105、104、103、102、10 cfu/mL 10个数量级梯度浓度。电泳结果如图4所示:当模板浓度降低到106cfu/mL时,PCR扩增产物开始明显降低,当模板菌液浓度为104cfu/mL时,PCR扩增产物较少,但是仍然能够检测到,因此,最低检测浓度为104cfu/mL。
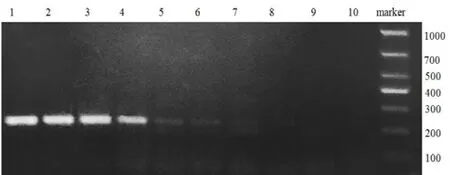
图4 不同模板浓度PCR电泳图Fig. 4 Amplification results of different template concentrations
2.6 烟草感病叶片中野火病分子检测
取上述制备的模板进行PCR扩增,PCR结果如图5所示,当距离分别为3 mm和6 mm时候,进行PCR反应能够产生特异性扩增,当距离大于6 mm时,由于模板浓度过低未产生反应。说明该引物能够对大田中感病的烟草叶片能够进行较好的检测。
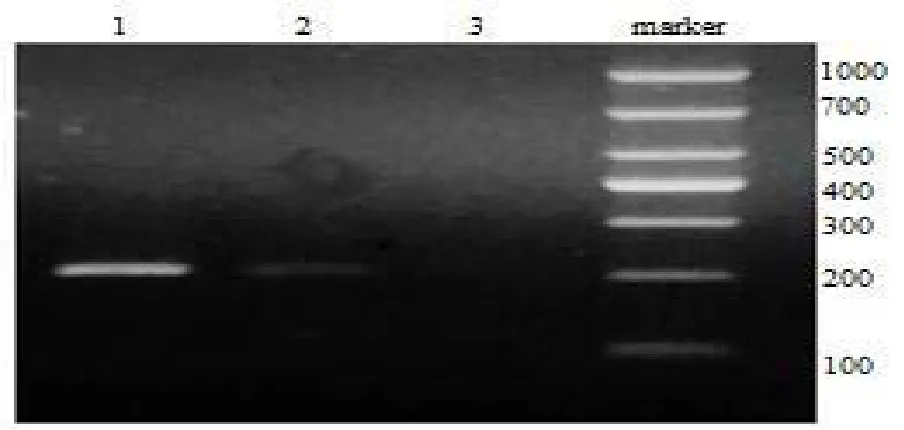
图5 烟草感病叶片PCR鉴定电泳图Fig. 5 Amplification results with infected tobacco leaves
3 讨 论
烟草野火病严重危害烟草叶片的完整度以及烤后烟叶的外观质量,给烟草农业生产造成巨大的经济损失。在实验室使用烟草野火病菌作为PCR反应模板时,本试验最终获得的分子检测引物能够对烟草野火病进行精确的检测,PCR反应特异性强,并且对于相近的其他杂菌不产生反应。与前人设计的引物相比较,本试验设计的引物条带大小较小,扩增产物仅为203 bp,并且对于反应体系的要求比较低,在进行PCR扩增时候能够大大的缩短分子检测所用时长,该引物设计时获得的特异性区段,能为其他试验的引物设计提供思路。同时在对烟草表面的优势菌群进行同步检测时,未见扩增出任何的条带,所以本试验获得的引物对于烟草野火病能够较好地检测。
在大田实际应用中,试验获得的引物能够对烟草叶片中的烟草野火病菌进行直接检测,但是检测的条件比较严格,如果直接使用烟草病叶浸出液作为PCR扩增模板,只有距离病斑部位很近时候才会产生目的条带,这是由于烟草叶片病斑部位的病菌含量较低,本试验检测到的病菌最低检测浓度为104cfu/mL,只有当病菌的浓度达到这个临界值时,PCR才能够进行较好的扩增并产生明显的条带。叶片中的病菌的数量由于达不到检测的最低限度,需要较多的叶片累加进行模板病菌的获取才能够进行扩增。
4 结 论
本试验利用Mauve 2.3.1和Primer Premier 6.0获得了多对烟草野火病菌引物,通过PCR筛选验证得到1对烟草野火病菌特异性引物。利用烟草野火病菌和感病烟叶浸出液为模板进行PCR扩增,得到单一目的条带,证明该引物对于烟草野火病菌能够进行特异性扩增,对烟草野火病菌进行良好的检测。
[1] 王振国,丁伟. 烟草野火病发生与防治的研究进展[J].中国烟草学报,2012,18(2):101-106.WANG Z G, DING W. Research progress in tobacco wildfire disease’s occurrence and control[J]. Acta Tabacaria Sinica, 2012, 18(2)∶ 101-106.
[2] 王铁霖,李晶,杨玉文,等. 烟草野火病菌Pseudomonas syringaepv.tabaciyuexi-1信号肽预测及分析[J]. 中国烟草学报,2016,22(1):92-100.WANG T L, LI J, YANG Y W, et al. Analysis of coding region for proteins containing signal peptides ofPseudomonas syringaepv.tabaciyuexi-1 strain[J]. Acta Tabacaria Sinica, 2016, 22(1)∶ 92-100.
[3] 张成省,孔凡玉,李多川,等. 烟草叶围细菌的分离及其对Alternaria alternata的拮抗作用[J]. 中国烟草学报,2005,11(4):17-20.ZHANG C S, KONG F Y, LI D C, et al. Screening of antagonistic bacteria from phylloshere and its resistance toAlternaria alternata[J]. Acta Tabacaria Sinica, 2005,11(4)∶ 17-20.
[4] 陈赟娟,刘雅婷,李永忠,等. 云南烟草野火病菌菌系分化研究[J]. 云南农业大学学报,2007,22(4):491-494.CHEN Y J, LIU Y T, LI Y Z, et al. Study on strains differentiation ofPseudomonas syringaepv.tabaciin Yunnan[J]. Journal of Yunnan Agricultural University,2007, 22(4)∶ 491-494.
[5] ZITOUDI K, MARGARITOPOULOS J T, MAMURIS Z, et al. Genetic variation inMyzus persicaepopulations associated with host-plant and life cycle category.[J].Entomologia Experimentalis EtApplicata, 2001, 99(3)∶303-311.
[6] RONEAL J, GUTARRA L, PRIOU S. Rapid differentiation of strains ofRalstonia solanacearumby restriction analysis of PCR-amplified fragments[J].Bacterial wilt newsletter, 1999, 16(5)∶ 7-10.
[7] 徐秀红. 烟草野火病的抗性鉴定及其抗性基因的分子标记与辅助选择[D]. 泰安:山东农业大学, 2005.XU X H. Research on resistance identification of tobacco to wildfire and RAPD marker of the resistant gene and its application in marker-assisted selection[D]. Taian∶Shandong Agricultural University, 2005.
[8] 杨姣弟,陈武,王运生,等. 烟草青枯菌特异性引物筛选及其应用[J]. 中国农学通报,2012,28(10):254-258.YANG J D, CHEN W, WANG Y S, et al. Screening and application ofRalstoniasolanacearumspecific-primers[J]. Chinese Agricultural Science Bulletin, 2012, 28(10)∶ 254-258.
[9] 魏兰芳,姬广海,张世光. 云南马铃薯青枯病菌的PCR检测[J]. 西南农业大学学报,2002,24(1):72-74.WEI L F, JI G H, ZHANG S G. Specific detection ofRalstonia solanacearumin potato by PCR[J]. Journal of Southwest Agricultural University, 2002, 24(1)∶ 72-74.
[10] 刘冰,褚小静,焦娇,等. 皖南烟田三种土传病原物分子检测[J]. 中国烟草科学,2013,34(5):100-103.LIU B, CHU X J, JIAO J, et al. Molecular detection of three soil borne pathogens from tobacco field in Anhui province[J]. Chinese Tobacco Science, 2013, 34(5)∶100-103.
[11] 赫然,张颖,王强,等. 利用微波炉和煮沸法快速制备大肠杆菌基因组DNA PCR模板[J]. 中山大学学报(自然科学版),2004,43(增刊):80-81.HE R, ZHANG Y, WANG Q, et al. Rapid preparation of PCR templates of genomic DNA fromEscherichia coliby using microwave and boiling[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2004, 43(s1)∶80-81.
[12] Bidlack W R. Proteins of iron metabolism[J]. Journal of the American College of Nutrition, 2002, 21(3)∶ 290-291.

