滇池沉积物中反硝化细菌及产甲烷菌的分子鉴别研究
尹利方,陈泽斌,夏体渊,王定康,姚丽媛,华金珠,徐胜光,王家银
(1.昆明学院 农学院,云南 昆明 650214;2.云南省高校生物炭工程研究中心,云南 昆明 650214; 3.云南省农业科学院农业经济与信息研究所,云南 昆明 650205)

表1 所用PCR引物
【研究意义】随着中国工业和农业的快速发展,城市污水的入湖量不断增加,河口和近岸海域氮磷负荷逐年增加,近岸水质持续下降,富营养化问题日趋严重,导致了一系列的环境问题,如频繁的赤潮和水华。滇池是中国著名的高原淡水湖泊,水污染严重,氮磷有机污染严重超标,水质为V类水体,严重富营养化。【前人研究进展】在过去几十年里,围绕滇池开展的研究多为非点源污染控制、地球化学和生态学方面,而水体和沉积物中微生物的研究十分匮乏。甲烷(CH4)是产甲烷菌在厌氧条件下分解有机物产生的温室气体,它对全球变暖的贡献仅次于二氧化碳[1]。20世纪80年代以来,甲烷的温室效应已达20 %,并以每年0.8 %~1 %的速度增长,在温室效应中起着越来越重要的作用[2]。目前对湖泊甲烷释放总量的研究主要集中在源和汇上,在低温下,大型淡水湖泊是大气CH4的来源,来源于沉积物中的产甲烷古菌,它在厌氧条件下产生和释放甲烷[3]。因此,为全面了解甲烷排放的来源。有必要研究湖泊沉积物中甲烷菌群落结构,为湖泊沉积物CH4排放的准确估算提供数据支持。反硝化作用在水体氮循环系统中起着重要作用。在此过程中,反硝化细菌受到缺氧外部条件的影响,将硝酸盐或亚硝酸盐转化为氮气或一氧化二氮,并将其释放到大气中,减少了水体中氮元素。【本研究切入点】因此,对滇池沉积物中反硝化细菌的多样性进行研究,可为微生物对反硝化作用的调控、反硝化作用的时空耦合效应和区域氮素生物地球化学循环模式的建立提供理论依据。【拟解决的关键问题】文章以滇池沉积物为研究对象,采用克隆文库的分子克隆方法,研究产甲烷菌和反硝化菌的群落结构,从而使人们对滇池富营养化和甲烷排放的机制有更全面的认识,为滇池水体修复提供生物学支撑。
1 材料与方法
1.1 样品采集
沉积物样品DC6于2017年1月采集自滇池草海,采用抓斗式底泥采样器在纬度N24°55′59″,经度E102°41′8″位点分别进行3次重复采样,重复之间间隔10 m,重复样品混匀后装入灭菌的250 mL锥形瓶,用于总DNA的提取。
1.2 DNA提取与PCR扩增
按陈泽斌等[4]的方法提取沉积物总DNA,采用引物NirS1F / NirS6R和Me1 / Me2分别对沉积物总DNA进行PCR扩增(表1),得到反硝化细菌和产甲烷菌特异性片段。PCR反应体系及条件参照周丽[5]和杨秀清[6]的文献,取5 μl PCR产物,用1 %琼脂糖凝胶电泳进行检测。
1.3 克隆与测序
回收PCR产物连接到pMD19-T载体(TaKaRa),按载体说明书进行连接转化。用菌落PCR验证阳性克隆(PCR反应体系和条件同1.2),阳性克隆测序委托上海桑尼生物科技有限公司完成。
1.4 16S rDNA系统发育分析
使用Mothur软件按相似性(>97 %)将测得序列划分为不同操作分类单元(OTU)并计算文库的覆盖率(C)[7];用BLASTN程序在GenBank数据库中完成序列比对[8];用MEGA软件生成序列系统发育树[9]。
2 结果与分析
2.1 反硝化细菌和产甲烷菌特异性片段的电泳检测
用表1列出的引物对反硝化细菌和产甲烷菌特异性片段进行PCR扩增,产物经电泳检测(图1),分别得到长度分别为1000和760 bp的单一片段,这与周丽[5]和杨秀清[6]的报道一致。反硝化细菌特异性片段浓度较低,低于50 ng/μl,产甲烷菌特异性片段浓度较高,高于100 ng/μl。试验证实了滇池沉积物中存在反硝化细菌和产甲烷菌,且产甲烷菌的丰度高于反硝化细菌。
2.2 克隆文库评价
分别在2个克隆文库中随机挑取100个菌落,进行菌落PCR验证,获得的阳性克隆子数分别为55和99个(表2),说明反硝化细菌克隆文库中克隆子阳性率偏低,约为55.00 %,这可能与2.1中PCR扩增得到的反硝化细菌特异性片段的浓度较低有关,导致其与pMD19-T载体连接率低,大量空载体被转化入大肠杆菌;2.1中PCR扩增得到的产甲烷菌特异性片段的浓度高(>100 ng/μl),其克隆文库的阳性率也高,约为99.00 %。Mothur软件按大于97 %的相似性将2个克隆文库的测得序列分别划分为10个和5个OUT(表2),说明尽管反硝化细菌的丰度低于产甲烷菌,但物种多样性高于产甲烷菌。2个文库的覆盖率分别为81.82 %和94.95 %(表2),均大于80 %,说明构建的2个文库有效,测序深度适宜,可以代表沉积物中反硝化细菌和产甲烷菌的物种多样性。
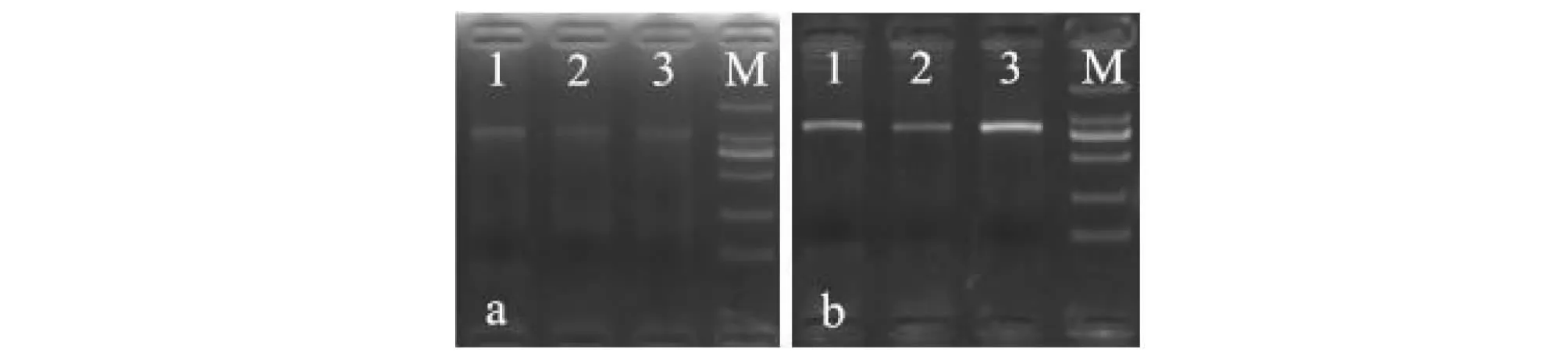
a:反硝化细菌特异性片段电泳图;b:产甲烷菌特异性片段电泳图;M:5 μl DL2000 DNA Marker;1、2、3-泳道a:Specific fragment electrophoresis of denitrifying bacteria; b:Specific fragment electrophoresis map of methanogenic bacteria; M:5 μl DL2000 DNA Marker; 1,2,3:Lane图1 反硝化细菌和产甲烷菌特异性片段电泳Fig.1 Specific fragment electrophoresis of denitrifying bacteria and methanogenic bacteria
2.3 反硝化细菌克隆文库中代表序列的BLASTN比对结果及系统发育分析
结合MEGA软件生成的系统发育树(图2),以及Mothur软件划分OTU的结果(表3),从55个阳性克隆子中选出10个代表序列(图2),并运用BLASTN程序在GenBank数据库中进行序列比对,发现55个阳性克隆子可分为10个种类:OTU1(代表序列为1-6-1,占比1.82 %)类序列与从污泥中一株未培养细菌CYCU的以亚硝酸盐还原酶基因序列相似性达91 %[10],说明该类菌株具有反硝化活性,但其分类地位未能准确鉴定;OTU2(1-6-22,1.82 %)类序列与Pandoraeafaecigallinarum的相似性达94 %;OTU3(1-6-20,1.82 %)类序列与水稻土中对异化铁还原反应的贡献最大的优势菌群Geoalkalibactersubterraneus的相似性达81 %[11],是潜在的未知种类;OTU4(1-6-33,10.91 %)类序列与废水活性污泥细菌Steroidobacterdenitrificans的相似性达82 %[12];OTU5(1-6-15,1.82)类序列与有光异养制氢特性的细菌Rubrivivaxgelatinosus相似性达80 %[13];OTU6(1-6-27,7.28 %)类序列与Azoarcussp.的相似性达80 %;OTU7(1-6-38,7.28 %)类序列与Polymorphumgilvum的相似性达81 %;OTU8(1-6-41,10.91 %)类序列与Sulfuritaleahydrogenivorans的相似性达79 %;OTU9(1-6-84,3.64 %)类序列与具有甲苯降解特性的Thauerasp.相似性达82 %[14];OTU10(1-6-4,52.73)类序列与污水处理厂尾水中细菌Dechloromonasaromatica的相似性达86 %[15],该类群是反硝化细菌中的优势类群;OTU1、OTU2、OTU3、OTU5、OTU9类序列的发现频率较低,为反硝化细菌中的低丰度类群。

表2 反硝化细菌和产甲烷菌特异性片段克隆文库分析

表3 反硝化细菌特异性片段克隆文库中各OUT代表序列分析
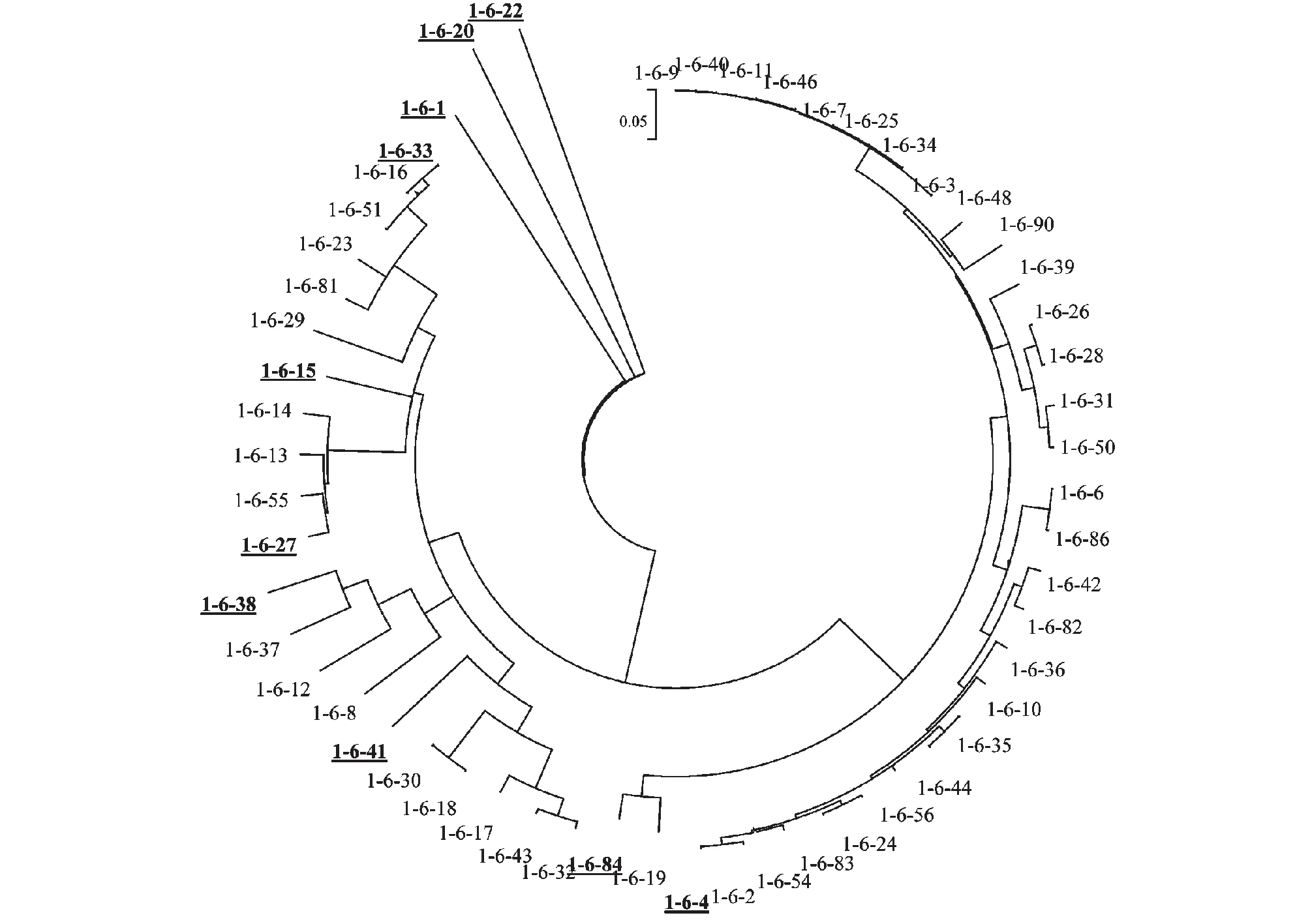
粗体和下划线标记的序列为代表序列,下同Bold and underlined sequences were representative sequences. The same as below图2 反硝化细菌的系统发育树Fig.2 Phylogenetic tree of denitrifying bacteria
2.4 产甲烷菌克隆文库中代表序列的BLASTN比对结果及系统发育分析
结合MEGA软件生成的系统发育树(图3),以及Mothur软件划分OTU的结果(表4),从99个阳性克隆子中选出5个代表序列(图3),并运用BLASTN程序在GenBank数据库中进行序列比对,发现99个阳性克隆子可分为5个种类:OTU1(I073,1.01 %)类序列与水稻土及污泥中大量存在的Methanobacteriumoryzae相似性达85 %[16];OTU2(H10,1.01 %)类序列与利用氢和甲酸的产甲烷菌甲酸甲烷杆菌(Methanobacteriumformicicum)相似性达85 %[17];OTU3(E12,3.03 %)类序列与酸性泥炭沼泽中大量存在的CandidatusMethanoregulaboonei相似性达85 %[18];OTU4(E07,3.03 %)类序列与采油废水中检测到的Methanolineatarda相似性达86 %。OTU5(H012, 91.92 %)类序列与甲酸甲烷规则菌(Methanoregulaformicicum)相似性达86 %,该类群是反硝化细菌中的优势类群,该类群也是最早被报道的产甲烷微生物;OTU1、OTU2为反硝化细菌中的低丰度类群。
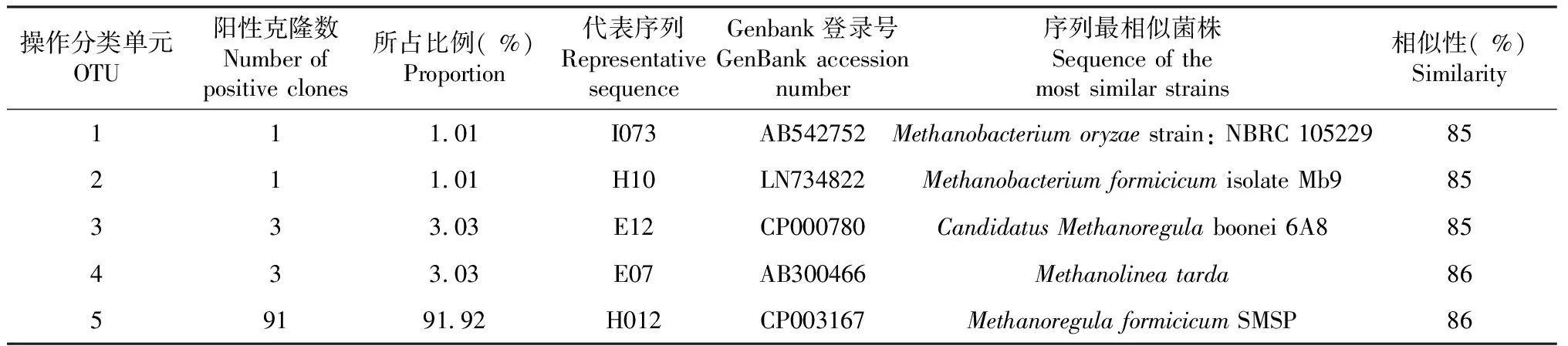
表4 产甲烷菌特异性片段克隆文库中各OUT代表序列分析
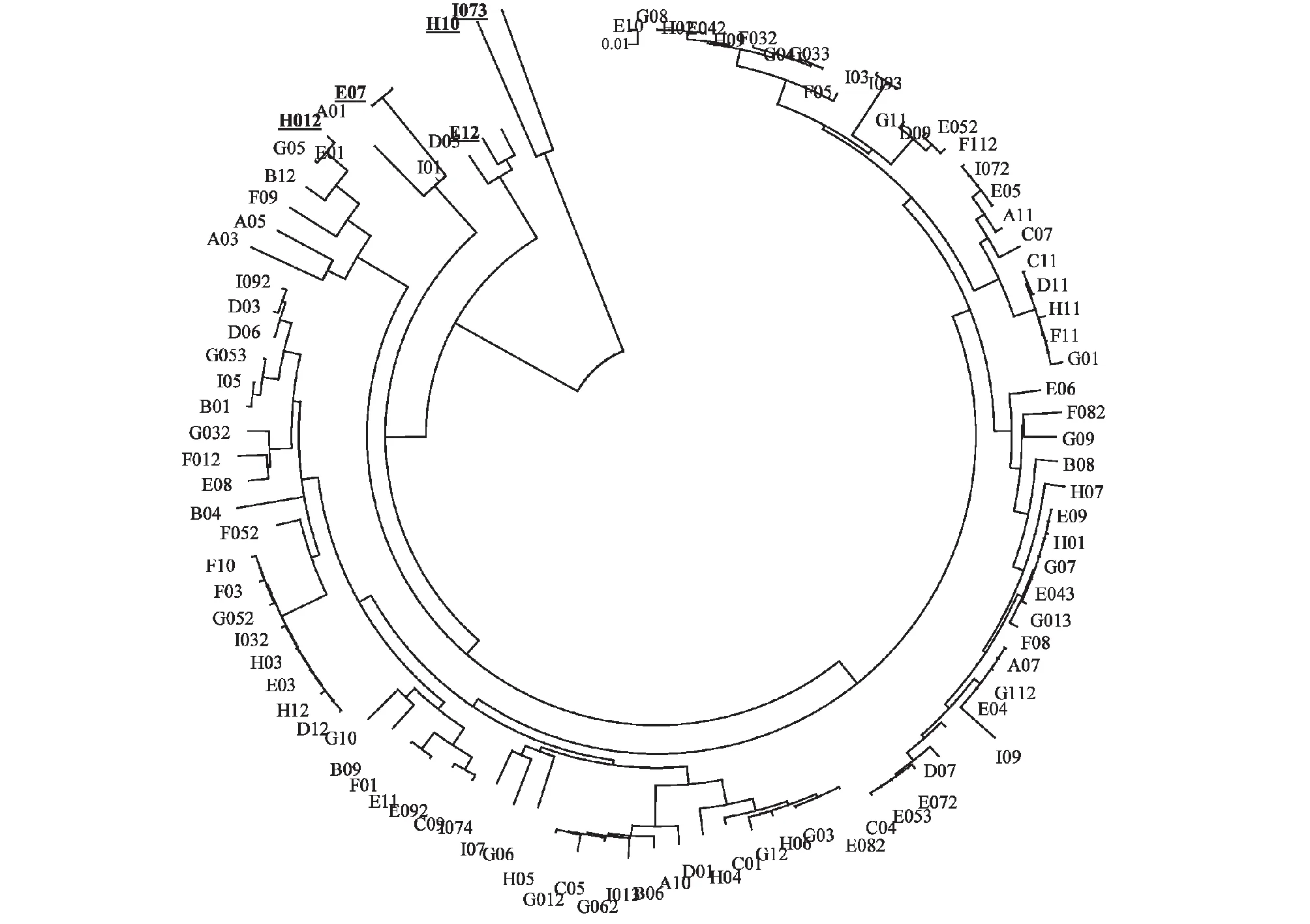
图3 反硝化细菌的系统发育树Fig.3 Phylogenetic tree of denitrifying bacteria
3 讨 论
本研究测得的154条克隆子序列与GenBank数据库中已知可培养菌株的相似性在79 %~94 %,98.7 %的序列与可培养菌株的相似性低于90 %,说明滇池沉积物中的反硝化细菌与产甲烷菌多为潜在的未被培养的新种,相似的可培养群落多来自于污泥、水稻土、废水和沼泽,这与前人的研究报道一致。
研究得到的反硝化细菌序列全部归属于Proteobacteria门,这与大量反硝化菌属于Betaproteobacteria纲的报道相符合[19]。与文库中序列相似的可培养微生物属于Pandoraea属(1.82 %)、Thauera属(3.64 %)、Geoalkalibacter属(1.82 %)、Steroidobacter属(1.82 %)、Rubrivivax属(1.82 %)、Azoarcus属(7.28 %)和Polymorphumgilvum属等,在以往的研究中,这些属也作为反硝化细菌被报道[20]。
在《伯杰系统细菌学手册》第九版中,产甲烷菌的分类包括甲烷杆菌目(Methanobacteriales)、甲烷球菌目(Methanococcales)、甲烷八叠球菌目(Methanosarcinales)、甲烷微菌目(Methanomicrobiales)和甲烷火菌目(Methanopyrales)共5个目,本研究得到的所有序列都归属于甲烷杆菌目,未检测到其他目的产甲烷菌种类。据报道,甲烷杆菌目微生物能以H2、CO2、甲酸盐和甲醇为代谢底物,产生甲烷[21]。本研究发现的Methanoregula属微生物被报道是泥炭藓湿地土壤中优势的产甲烷菌类群[22]。
本试验中,通过对目的片段的克隆得到了启示:通过PCR获得的目的片段如果要克隆到载体上,连接体系中目的片段的浓度必须要达到要求。例如反硝化细菌特异性片段浓度过低导致其与pMD19-T载体连接率低,大量空载体被转化入大肠杆菌,文库中克隆子阳性率偏低。相反,产甲烷菌特异性片段的浓度高(>100 ng/μl),其克隆文库的阳性率也高,约为99.00 %。
本研究只采用引物Me1/Me2对产甲烷菌的多样性进行研究,尚且存在不足。Banning[23]和Juottonen[24]等人研究指出,Mcr引物和Me引物在检测产甲烷菌检测中具有不同的灵敏度。Me引物无法检测到产甲烷菌中的Methanosaetaceae和Methanobacteriaceae类群,导致研究结果不能覆盖全部物种,这与本研究的结果一致。因此,为了全面准确的评估环境中的产甲烷菌的多样性,需要选用多对引物进行研究,其次,还应结合传统的分离培养方法,生理生化鉴定方法,才能得到准确的鉴定结果。
4 结 论
随着厌氧培养和分子生物学技术的发展,人们对反硝化细菌和产甲烷菌的群体研究会更加详细和全面。今后的相关研究可以集中在以下2个方面:首先,可以通过改进微生物分类技术发现更多的产甲烷菌群;其次,对产甲烷菌的代谢特征进行更详细的研究,为环境治理和生物能源开发提供理论依据。
[1]张坚超, 徐镱钦, 陆雅海. 陆地生态系统甲烷产生和氧化过程的微生物机理[J]. 生态学报, 2015, 35(20): 6592-6603.
[2]周 炜, 张岳芳, 朱普平, 等. 种植制度对长江下游稻田温室气体排放的影响[J]. 江苏农业学报, 2017, 33(2): 35-41.
[3]王琛瑞, 黄国宏, 梁战备, 等. 大气甲烷的源和汇与土壤氧化(吸收)甲烷研究进展[J]. 应用生态学报, 2002, 13(12): 1707-1712.
[4]陈泽斌, 夏体渊, 王定康, 等. 滇池沉积物中厌氧氨氧化细菌分布的时空差异[J]. 水生生物学报, 2016, 40(2): 425-430.
[5]周 丽, 付智丹, 杜 青, 等. 减量施氮对玉米/大豆套作系统中作物氮素吸收及土壤氨氧化与反硝化细菌多样性的影响[J]. 中国农业科学, 2017, 50(6): 1076-1087.
[6]杨秀清, 吴瑞薇, 韩作颖, 等. 基于mcrA基因的沁水盆地煤层气田产甲烷菌群与途径分析[J]. 微生物学通报, 2017, 44(4): 795-806.
[7]严立恩, 陈志荣, 王晶晶, 等. 南极乔治王岛陆地土壤和潮间带放线菌多样性及其抑菌活性研究[J]. 中国海洋药物, 2016, 35(1): 19-28.
[8]尹利方, 任 禛, 夏体渊, 等. 应用克隆文库构建法研究枇杷根系AMF多样性[J]. 西南农业学报, 2017, 30(5): 1009-1015.
[9]尹利方, 陈泽斌, 夏体渊, 等. 一株铁皮石斛组织培养污染内生细菌的分离与鉴定[J]. 西南农业学报, 2016, 29(8): 1982-1986.
[10]You S J. Identification of Denitrifying Bacteria Diversity in an Activated Sludge System by using Nitrite Reductase Genes[J]. Biotechnology Letters, 2005, 27(19): 1477-1482.
[11]王媛媛. 磷酸盐对水稻土中异化铁还原菌丰度和群落结构的影响[D]. 西北农林科技大学, 2014.
[12]蒙小俊, 李海波, 曹宏斌, 等. 焦化废水活性污泥细菌菌群结构分析[J]. 环境科学, 2016, 37(10): 3923-3930.
[13]Ru Y L, Fang H H P. Hydrogen production characteristics of photoheterotrophicRubrivivaxgelatinosus, L31[J]. International Journal of Hydrogen Energy, 2008, 33(3): 974-980.
[14]徐爱玲, 任 杰, 宋志文, 等. 污水处理厂尾水细菌群落结构分析[J]. 环境科学, 2014(9): 3473-3479.
[15]Joulian C, Patel B K C, Ollivier B, et al.Methanobacteriumoryzaesp. nov. a novel methanogenic rod isolated from a Philippines ricefield.[J]. International Journal of Systematic & Evolutionary Microbiology, 2000, 50 Pt 2(2): 525-528.
[16]穆维娜, 李玉成, 武 超, 等. 秸秆蓝藻混合发酵产沼气过程中微生物群落分析[J]. 微生物学杂志, 2017, 37(1): 70-77.
[17]Bräuer S L, Cadillo-Quiroz H, Ward R J, et al.Methanoregulabooneigen. nov. sp. nov. an acidiphilic methanogen isolated from an acidic peat bog[J]. International Journal of Systematic & Evolutionary Microbiology, 2011, 61(Pt1): 45.
[18]穆 剑, 匡 丽, 高迎新, 等. 采油废水厌氧处理系统的微生物群落特征[J]. 环境工程学报, 2014, 8(3): 807-814.
[19]洪 璇, 洪有为, 陈仲巍, 等. 九龙江河口区nirS型反硝化细菌多样性及系统发育学分析[J]. 微生物学通报, 2015, 42(9): 1639-1650.
[20]方晶晶, 马传明, 刘存富. 反硝化细菌研究进展[J]. 环境科学与技术, 2010(s1): 206-210.
[21]马 凯. 产甲烷菌的分子系统学和生态功能研究[D]. 中国科学院微生物研究所, 2006.
[22]曾志华. 闽江河口区盐度梯度下潮汐沼泽湿地产甲烷菌群落结构及对环境因子的响应[D]. 福建师范大学, 2014.
[23]Banning N, Brock F, Fry J C, et al. Investigation of the methanogen population structure and activity in a brackish lake sediment[J]. Environmental Microbiology, 2005, 7(7): 947.
[24]Juottonen H, Galand P E, Yrjälä K. Detection of methanogenic Archaea in peat: comparison of PCR primers targeting the mcrA gene[J]. Research in Microbiology, 2006, 157(10): 914.

