用于CT心脏分割的几种超像素过分割和块合并的方法比较
张耀楠 吴秋实 何颖 安晓莉


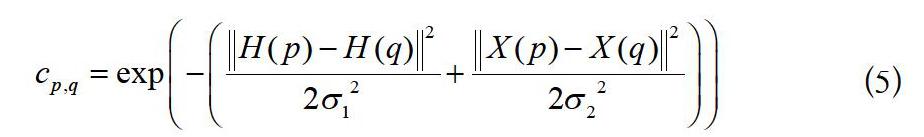
摘要:针对从CT图像中提取心脏结构信息还是一个尚未解决的问题,本文利用超像素思想对CT图像进行分割。本文利用4种方法(N-cut算法、熵率、简单线性迭代、均值漂移)进行超像素过分割,并进行了量化比较。进一步通过动态融合方法和谱聚类方法得到分割结果。在动态融合方法中设计了一种相似性度量的计算方法,并对两种合并方法进行了比较。实验表明本文提出的方法用于CT心脏图像的分割是可行的。在四种超像素过分割方法中,简单线性迭代运行速度较快,在各项评价指标中都比较不错。动态融合方法和谱聚类的合并准确性都较高,但谱聚类的运算速度远快于超像素的動态合并。
关键词:CT;医学图像;Adaboost;图像分割;超像素
中图分类号:R445.1 文献标识码:A 文章编号:1007-9416(2018)10-0000-00
任晓峰等人在2003年最早提出超像素分割的思想[1]。图像中单个像素对人和计算机认识图片都无太大意义,而超像素分割将图像分割成有某种相同图像语义的超像素块,保留了边缘信息,能为后续图像分析减少很大的计算量。已有学者将此思想应用到医学图像分析领域中。比如定位癌症病灶、定位内出血位置、体内异物检查等诸多方面[2][3]。本文将超像素分割应用于CT心脏图像的分割,对一些过分割和块合并的方法进行比较。
超像素分割有很多具体实现的算法,大体可分为两类:第一种是基于图论的分割方法[4],例如N-cut算法[5],熵率(Entropy Rate) 算法[6]。 第二种是基于梯度下降的方法[7],例如均值漂移(Mean Shift)算法[8],简单线性迭代(SLIC, Simple Linear Iterative Clustering) 算法[9]。本文设计超像素动态融合算法实现超像素块的合并,并设计了一种相似性度量的计算方法。还利用谱聚类实现超像素块的分类,最后设计了自动确定最终聚类的个数。
1 方法
基于超像素的图像分割有两大步骤:过分割和块合并。本文通过4种方法进行超像素过分割(N-cut算法、熵率(Entropy Rate)、简单线性迭代(Slic)、均值漂移(Mean Shift)),得到一个过分割的图像。再将过分割的图像进行块合并工作。这部分合并也采用了两种不同的方法:动态融合方法和谱聚类方法。超像素块合并后的图像即为分割图像。关于4种超像素过分割,已有大量文献描述,本文不再重复。针对本文的应用特性,下面对块合并的两种方法进行描述。
1.1动态融合流程
超像素块合并,就是将图像特征接近的超像素块进行合并,成为新的超像素块。本文提出了一种代价函数的计算方法来计算相邻超像素之间的代价函数,并将代价函数最小的超像素块进行合并。本文建立超像素的代价函数矩阵来记录相邻的超像素块之间的代价函数值,融合代价函数值小的超像素块,随后更新该矩阵,并不断重复上述步骤,得到最终结果。
假设图中有个超像素块,则需建立的超像素矩阵。的一个元素代表超像素块与超像素块之间的代价函数,的具体计算公式如下:
(1)
在此公式中,、是常数,需要手动设置。可以看出,上述公式有三部分组成。其分别代表超像素块间的相似度距离、相邻超像素块公共边界与超像素块的相似度距离、以及合并后的超像素块与现有超像素块之间的相似度距离。下面分别介绍这三部分。
代表超像素块与超像素块的相似度距离。本文的研究对象为CT图像,为灰白图像,而且超像素过分割后的每块超像素块相对较小,且形状不规则。因此本文计算相似度距离是利用相邻两超像素块的灰度分布计算的。具体定义为:
(2)
其中表示超像素块的灰度直方图。可以从公式中看出当和的灰度直方图越接近时,的值越小。
经过过分割得到的超像素块还保留了原始图像的边界信息。想要得到好的合并结果,我们需要利用这些边界信息。若两个相邻超像素块可以合并,则表明两个超像素块公共边缘的灰度分布应该和这两个超像素块的灰度分布都相近。我们将两个相邻超像素块的共同边界记为。那么(1)中的计算如公式(3)所示。
(3)
的值越小,代表边界与这两个超像素块越接近,这两块越有可能合并为一个新超像素块。
另外,若两个相邻超像素块可以合并,每一块的超像素与合并后的新超像素块的灰度分布应该接近。将合并后的超像素块记为,按下列公式计算
(4)
对每两个相邻超像素块通过上述公式进行代价函数的计算,不相邻的超像素块的代价函数设为无穷大,并用矩阵记录下来。合并时,将矩阵内代价函数最小的两块超像素合并为一块,并重新更新建立的矩阵,不断重复此过程。直到新的超像素块数达到先前预设的块数。
1.2谱聚类
上面介绍的超像素块合并方法每一次进行合并时,都需要重新建立代价函数矩阵,并且重新判断超像素块之间是否相邻。无论是时间复杂度还是空间复杂度都比较大。这一节我们利用谱聚类的方法来对超像素块进行分类[10-12]。谱聚类是一种基于图论的聚类方法,它能够识别任意形状的样本空间且收敛于全局最有解,其基本思想是利用样本数据的相似矩阵进行特征分解后得到的特征向量进行聚类[21]。
本文中譜聚类的主要步骤为:
(1)设计超像素块之间的维连接矩阵。
(2)根据连接矩阵计算连接矩阵的拉普拉斯矩阵以及度矩阵。
(3)计算的特征值,选取特征值最小的个特征向量,建立的特征矩阵。
(4)对特征矩阵进行k-means聚类。
(5)得到最后的划分结果。
本文利用的图像区域特征为灰度直方图,同时本文又加入了坐标信息。本文所用的连接矩阵中元素的计算公式为:
(5)
其中、分别为灰度直方图和坐标信息的均方差。
1.3融合数目的确定
超像素块合并最终聚类个数需要事先指定。 本文利用Gap Statistic方法对最终的聚类个数进行预测。 Gap statistic方法是通过对平均分布参考数据集的期望值和观测数据集的进行比较,查找下降最快的值为最优聚类数,具体方法描述可参见文献[13]。
2 实验结果
本文利用Microsoft Visual Studio 2013软件平台、Opencv计算机视觉库以及Matlab 2014软件对上文描述的方法进行实验。 图像采集设备为西门子CT,CT图像的大小为512*512。共有42名病人的CT影像,每名病人的数据含有200-300张图像。
2.1四种超像素过分割方法对比
表1是四个超像素过分割算法复杂度的比较。从表中看出,只有简单线性迭代算法的时间复杂度是线性的,其他算法的计算量随像素个数的增加而大幅提高。
下面本文对四种超像素过分割结果进行分析比较。衡量超像素分割效果主要关注超像素边界边缘与目标边缘是否精确贴合,以及超像素形状和大小规则情况。本文以下面几个指标对上述超像素过分割算法进行定性的评价:(1)欠分割错误率,该指标表示超像素区域超出人工分割边界的比例,该值越小,超像素的边缘贴合度越小;(2)边缘召回率,用于度量超像素边缘与原始图像边缘重叠比例,该值越高,超像素边缘贴合度越好;(3)面积方差,度量超像素面积的差异大小;(4)圆度率(CM, Circle Measurement),衡量超像素整体形状逼近园的程度。
这几个指标的实验结果如图1-4所示。熵率算法在边缘处理上表现最好,分割出的边缘与原边缘贴合的越密切, 但是在图像的规则程度上较差。而均值漂移的形状相对紧凑规则,但是在边缘处理上较差。而且上述两种算法在计算量大,运行慢,不能实现连续断层图片的超像素分割的要求。相比之下,简单线性迭代的速度要快的多,而且在上述评价指标中都比较不错,而且分割的超像素块可用。因此,本文选用简单线性迭代算法作为后面超像素合并的预处理算法。
2.2超像素块合并结果比较
超像素谱聚类结果如表2、表3所示。
从表2和3分别表示了动态融合和谱聚类超像素块合并的准确性。可以看出,两者的准确程度都比较高,可以用于心脏的分割。谱聚类的超像素合并的时间比动态合并超像素的时间要少,这是因为动态合并需要不断生成新的相似度距离矩阵,不断的判断不同超像素之间的相邻关系,而谱聚类只需建立一次邻接矩阵,所以谱聚类的运算速度远快于超像素的动态合并。
3 结语
本文利用超像素思想对CT心脏图像进行分割。整个方法有两大步骤:过分割和块合并。本文通过4种方法进行超像素过分割(N-cut算法、熵率、简单线性迭代、均值漂移),并进行了量化比较。块合并也采用了两种不同的方法:动态融合方法和谱聚类方法。在动态融合方法中设计了一种相似性度量的计算方法,利用了谱聚类实现超像素块的分类,并设计了自动确定最终聚类的个数。实验表明本文提出的方法用于CT心脏图像的分割是可行的。在四种超像素过分割方法中,简单线性迭代运行速度较快,在各项评价指标中都比较不错。动态融合方法和谱聚类的合并准确性都较高,但谱聚类的运算速度远快于超像素的动态合并。
4 感谢语
本文实验中的一些医学图像由中国医科大学研究生医工结合创新实践基地提供。
参考文献
[1]Malik J.Learning a classification model for segmentation[J].Proc.int.conf.computer Vision,2003(1):10-17 vol.1.
[2]Achanta R,Shaji A,Smith K, et al. SLIC Superpixels Compared to State-of-the-Art Superpixel Methods[J].IEEE Transactions on Pattern Analysis & Machine Intelligence,2012,34(11):2274.
[3]苏坡.癌症诊疗中的医学图像配准和分割算法研究[D].西北工业大学,2014.
[4]Zhang Z,Xing F & Wang H, et al.(2018). Revisiting Graph Construction for Fast Image Segmentation[J].Pattern Recognition,2018,(78):344-357.
[5]Fu K,Gu Y H,Yang J.Spectral Salient Object Detection[J].Neurocomputing,2017:1-6..
[6]Liu M Y,Tuzel O,Ramalingam S,et al. Entropy rate superpixel segmentation[C]// Computer Vision and Pattern Recognition.IEEE,2011:2097-2104.
[7]Levinshtein A,Stere A, Kutulakos K N,et al. Turbopixels: Fast superpixels using geometric flows[J].IEEE Transactions on Pattern Analysis and Machine Intelligence,2009,31(12): 2290-2297.
[8]Shih HC,Liu ER,Automatic Reference Color Selection for Adaptive Mathematical Morphology and Application in Image Segmentation[J].IEEE Transactions on Image Processing,2016(25):4665-4676.
[9]Achanta R,Shaji A,Smith K,et al.SLIC superpixels[J].Epfl,2010.
[10]Yang Y,Wang Y,Xue X.A novel spectral clustering method with superpixels for image segmentation[J].Optik - International Journal for Light and Electron Optics,2016,127(1):161-167.
[11]Zou X,Xiaodong Y E,Tan Z,et al. Image segmentation method based on improved similarity measure of spectral clustering[J].Computer Engineering & Applications,2017.
[12]Zelnik-Manor L, Perona P. Self-Tuning Spectral Clustering[C]// Advances in Neural Information Processing Systems.2004:1601--1608.
[13]肖 宇,于 劍.Gap statistic与k-means算法.计算机研究与发展,2007,44(Suppl.):176-180.
Comparison of Several Methods of Superpixel-Based over-Segmentation
and Region Merging for Cardiac CT Segmentation
ZHANG Yao-nan1,2,WU Qiu-shi2 ,HE Ying1 ,AN Xiao-li1
(1.College of Electronics and Information Engineering, Siyuan University, Xian Shanxi 710038;
2.Sino-Dutch Biomedical and Information Engineering School,Northeastern University,Shenyang Liaoning 110169)
Abstract: To tackle the unresolved problem of extracting cardiac structure information from CT images, this paper uses superpixel paradigm to segment CT images. In this paper, four methods (N-cut algorithm, entropy rate, simple linear iterative clustering, mean shift) are used to perform superpixel-based over-segmentation and their quantitative comparison is performed. The segmentation results are obtained by further dynamic fusion method and spectral clustering method. A calculation method of similarity measure is designed in the dynamic fusion method, and the two region merging methods are compared. Experiments show that the proposed method is feasible for segmentation of CT cardiac images. Among the four superpixel-based over-segmentation methods, the simple linear iterative clsutering runs faster and is perfoming well in all evaluation indicators. The accuracy of both dynamic fusion method and spectral clustering is good, but the spectral clustering operation speed is much faster than the dynamic fusion.
Key words: CT; medical images; Adaboost; image segmentation; superpixels

