利用GEO数据库寻找结直肠癌肝转移生物标志物
(青岛大学附属医院胃肠外科,山东 青岛 266003)
结直肠癌(CRC)作为最常见的消化道肿瘤之一,近年来在发展中国家的发病率快速增高[1]。我国CRC的发病率、死亡率近几年也在逐步上升,已成为巨大的公共健康问题。为了控制这种增长趋势,通过加强人群筛选来预防和早期发现高危病人显得尤为重要,对于我国东部地区以及60岁以上的高危人群需要更加有效的预防和治疗措施[2]。全世界每年约有120万例新发CRC病人被确诊,其中约50%的病人发生肝转移,未经治疗的肝转移病人12个月内生存率约为零[3]。2005—2014年,美国的CRC的发病率每年下降约2%~3%,可能与新的筛选方案有关,也可能与病人较高的根治性切除率相关[4]。CRC的治疗方法包括手术、放疗、化疗和靶向治疗,当CRC局限于肠道时可通过手术治愈;然而,对于已经扩散的CRC或者术后复发的病人通常是无法治愈的[5]。CRCⅠ期病人的10年生存率为90%,但对于无法手术的Ⅳ期疾病病人,目前生存率只有5%[6]。对于肝转移病人,只有少数早期的病人适合外科手术切除治疗[7]。美国癌症联合委员会将越来越多的分子标志物用于癌症精准的风险分层,从而更好地评估患者的治疗方案和预后情况[8]。因此,开发有效的生物标记物进行特异的靶向治疗是极其必要的。生物标志物能够早期提示CRC肝转移的情况,进而提高CRC的预后预测水平。本研究利用生物信息学分析方法从公共基因芯片数据库(GEO)中获取有关CRC肝转移的芯片,分析不同阶段组织的差异表达基因,从而为CRC肝转移的防治提供新的生物标志物和治疗靶点。现将结果报告如下。
1 资料与方法
1.1 基因芯片数据的获取
在GEO数据库中检索与CRC转移密切相关的AFFY U133 PLUS2.0芯片数据,选取其中样本组织分期比较接近,同时数据量比较大的数据集作为分析数据集。其中GSE81980是早期(Ⅰ/Ⅱ)CRC样本集,主要包含150个样本信息(100个非转移样本和50个转移样本),GSE18105主要是Ⅱ/Ⅲ期肿瘤样本,包含111个样本(67个非转移和44个转移样本)。考虑到两批样本肿瘤时期分布不一样,将两批样本混合后随机拆分为训练集195个样本(75%)和验证集66个样本(25%)。
1.2 方法
1.2.1数据预处理 利用Transcriptome Analysis Console软件对数据芯片中提供的原始数据(CEL)进行Robust multi-chip average (RMA)归一化处理,然后利用R-package Combat去除批次效应。根据AFFY U133 PLUS2.0的基因注释文件对探针进行过滤,去除同时注释到多个基因的探针,筛选平均信号值最高的探针信号值作为基因的表达值。然后筛选Average Exp≥6且Max Exp≥7的基因进行后续分析。
1.2.2生物标志物的筛选 转移组与非转移组间比较采用t检验进行统计学分析,以P<0.05作为筛选条件。对筛选基因进行Lasso回归算法分析,根据Lasso算法的分值进行重要性排序,筛选重要性排序前12个基因作为潜在的生物标志物。
1.2.3模型的建立 运用逻辑回归算法,根据上述12个基因的表达情况进行机器学习分类。其中训练集195个样本,验证集66个样本(表1)。参数设置:最大迭代次数300,采用L2正则化,正则化系数λ设置为0.1。

表1 训练集和验证集模型构建
TP:真阳性, FN:假阴性,FP:假阳性,TN:真阴性。
1.2.4GSEA分析 采用GSEA 3.0版本进行分析。将GSE81980、GSE18105数据集中所有样品纳入GSEA,选择C2 CGP基因集进行分析。
1.2.5预后分析 使用在线Surv Express(http://bioinformatica.mty.itesm.mx:8080/Biomatec/SurvivaX.jsp)对筛选出的12个生物标志物在CRC病人中的表达与预后的关系进行分析。
2 结 果
2.1 显著差异基因筛选
转移组和非转移组通过t检验分析,按照P<0.05作为筛选条件筛选到426个基因。用Lasso回归算法对426个筛选基因进行重要性排序,选重要性排序前12个基因作为潜在的生物标志物,分别为CD163L1、FAM210B、LGR5、LRRC16A、PIK3R3、PLEKHA6、PROSER2、RBBP9、SEMA6D、STOM、THBS1、ZNF544。见表2。
2.2 ROC曲线结果
根据受试者工作特征曲线(ROC曲线),同时兼顾灵敏度和特异度,ROC曲线下面积(AUC)>0.5的情况下,越接近于1,说明诊断效果越好,AUC<0.5时,说明无诊断价值。本研究中训练集AUC=0.87,验证集AUC=0.84(图1),其AUC>0.5,说明两数据集诊断效果非常好。

表2 重要性排序前12个基因
2.3 GSEA分析结果
将所有的样本数据进行GSEA富集分析,选择C2 CGP基因集进行分析。研究结果显示,非转移组394/2737基因集上调,转移组2343/2737基因集上调,其中多个转移相关的基因在转移组中表达上调。筛选GSEA显著性基因组时,多个转移相关基因集上调,以圆圈大小代表基因集的基因数目(图3)。肿瘤肝转移基因集:
ROESSLER_LIVER_CANCER_METASTASIS_DN NOMP值=0.002016129,
SWEET_KRAS_TARGETS_DNP值=0.004032258,
LOCKWOOD_AMPLIFIED_IN_LUNG_CANCERP值=0.0186722,
NIKOLSKY_BREAST_CANCER_15Q26_AMPLICONP值=0.018108651(见图2)。
2.4 预后分析
通过Surv Express网站对与CRC转移相关的CD163L1、FAM210B、LGR5、LRRC16A、PIK3R3、PLEKHA6、PROSER2、RBBP9、SEMA6D、STOM、THBS1、ZNF544等基因在CRC中的表达与预后的关系进行分析。结果表明上述基因高表达的CRC病人预后差。
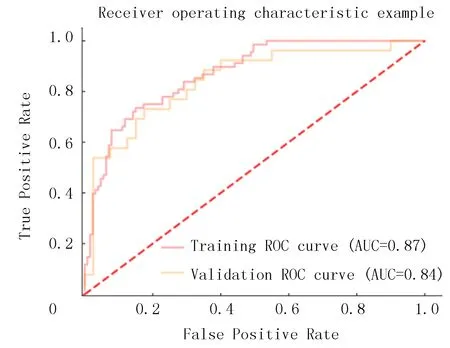
图1 训练集和验证集AUC
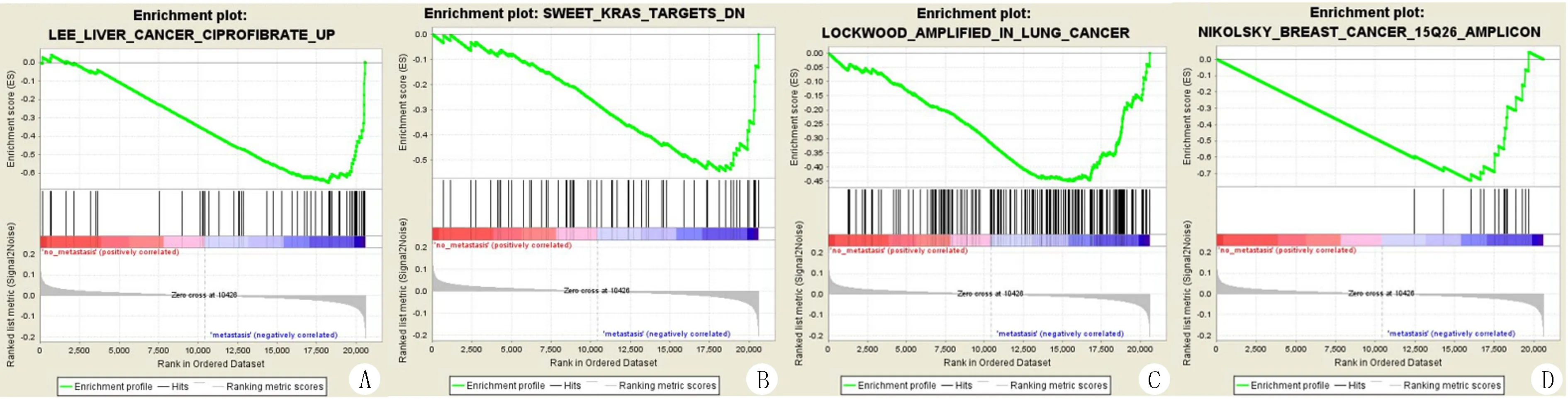
A:Roessler_Liver_Cancer_Metastasis_Dnnom(P=0.002,FDR=1.0,ES=0.512);B:Sweet_Kras_TarGets_Dn(P=0.004,FDR=1.0,ES=0.544);C:Lockwood_Amplified_In_Lung_Cancer(P=0.018,FDR=1.0,ES=0.450);D:Nikolsky_Breast_Cancer_15q26_Amplicon(P=0.018,FDR=1.0,ES=0.743)。富集基因集的显著性以P<0.05判断。FDR:错误发现率。ES:富集分数。
图2高表达的癌组织GSEA
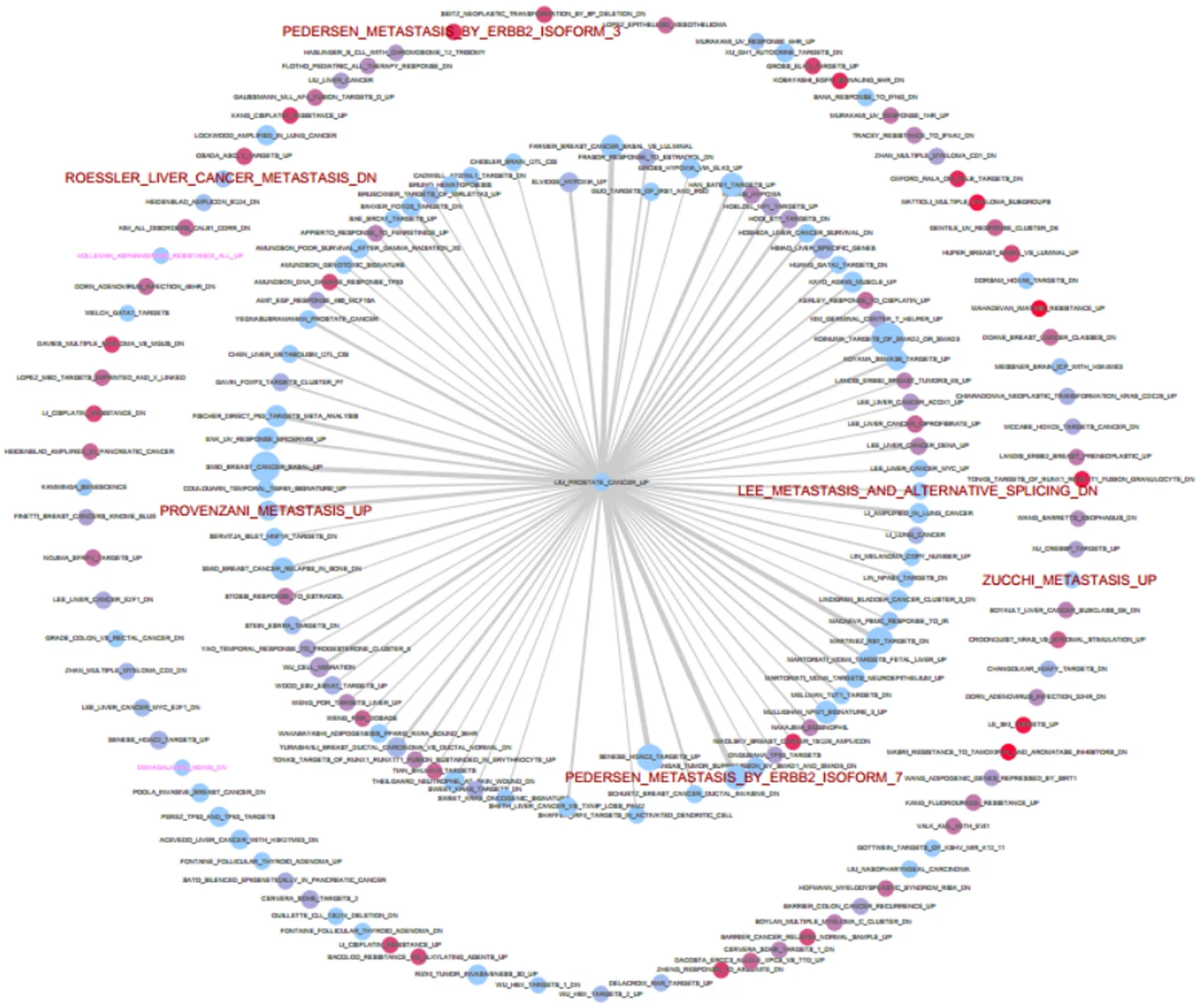
圈的大小代表基因集的基因数目,连线表示两个基因集中共有基因数目的多少,圈的颜色代表富集的ES值,标红的标签为转移相关的基因集,粉色的标签为非转移组富集到的基因集。
图3筛选GSEA显著性P<0.05的genesets进行网路图展示
3 讨 论
近年来,GEO、TCGA、Array Express等众多基因表达数据库相继建立。GEO致力于建立一个基因表达数据仓库和在线资源,支持公共使用和发布基因表达数据,用于检索从任何物种来源的基因表达数据,是生物信息学依赖的重要资源[9-11]。生物信息学是以计算机为工具对生物信息进行储存、检索和分析的科学,研究重点体现在基因组学和蛋白质组学两方面,即从核酸和蛋白质序列出发,分析序列表达的结构功能等生物学信息。随着高通量测序、计算机科学以及生命科学的迅速发展,生物信息学在基因调控网络和蛋白质结构功能研究中的重要性显得日益突出。本研究从GEO数据库中下载了GSE81980、GSE18105两套同一平台上的基因芯片,并用RMA归一化处理,分析平台进行差异基因分析等,挖掘出12个基因,为CRC肝转移的预防、诊断、治疗提供了新的研究方向。但本研究存在一些不足,首先,研究数据是从在线的公共数据库中获取的,RNA检测平台差异是不可避免的因素,可能限制当前结果的准确性;其次,由于RNA检测样品来源的多样性,因此需要在临床上收集更多的样品进行荧光实时定量聚合酶链反应检测。
一般差异分析(GO和Pathway)倾向于比较两组间的基因表达差异,重点是少数几个基因表达的明显上调或下调,这些差异分析往往忽略了一些没有显著差异表达但具有重要生物学意义的基因。由于忽略了这些基因的生物学特性,基因调控网络与有价值基因功能信息之间的关系就不可能被揭示。GSEA不需要指定明确的差异基因阈值。该算法根据实际数据的总体趋势,为当前芯片分析存在的问题提供了合理的解决方案。本研究所有样本数据进行GSEA富集分析,并用C2 KEGG基因集进行进一步分析。489个基因组中有329个在转移表型上调,9个基因集在P<0.05时显著富集,489个基因集中有160个在无转移表型上调。
目前,本研究的相关基因在CRC肝转移中的分子机制及相关信号转导通路具体机制尚不清楚,KATTAN等[12]研究发现CD163L1受miRNA的调控,因此,它可以作为口腔鳞状细胞癌的生物标志物。SUN等[13]采用细胞增殖、集落形成和侵袭分析的方法评估FAM210B在人癌细胞中的作用,证明FAM210B丢失与癌症转移显著相关。LGR5在肺癌、乳腺癌、宫颈癌、胃癌、CRC、胰腺癌中的表达均有报道,LGR5在血管生成和肿瘤预后中起重要作用[13-19]。WEI等[20]研究发现LRRC16A在调节急性呼吸窘迫综合征基线血小板计数中起介导作用。GROSSMANN等[21]发现PIK3R3参与癌症相关信号的传导和细胞生长。LANGEVIN等[22]发现PLEKHA6在鉴定头颈部鳞状细胞癌血液DNA甲基化标记物中起重要作用。HAMADA等[23]发现PROSER2在人胎盘生殖细胞DNA甲基化中起印记基因的作用。SHIELDS等[24]认为RBBP9具有胰腺肿瘤形成所需的丝氨酸水解酶活性。PENG等[25]发现SEMA6D是BMP信号传导的靶标,并且SEMAPHORIN信号传导对房室通道中的间充质细胞生成发挥着重要作用,揭示了一种新型调节房室间隔的BMP-SEMA6D-Rho轴。PAL等[26]认为THBS1在血管生成和肿瘤进展中起重要作用,THBS1是一种肿瘤特异性细胞外基质蛋白,由TGFB1诱导,促进癌细胞的迁移,通过整合素信号刺激MMPS的表达,在口腔鳞状细胞癌的侵袭中起重要作用。WALTON等[27]认为ZNF544参与DNA甲基化的变异,从而引起多动症。研究表明筛选出的12个基因,部分基因在相关癌症转移中起重要作用,但具体分子机制及相关信号转导通路仍需进一步研究。
综上所述,随着微阵列技术的日趋完善,研究人员可以进行转录组研究,寻找感兴趣的标记基因;临床医师可参考基因及蛋白质组学研究成果,制定个体化的治疗方案。目前辅助化疗的分子标志物研究还处于初始阶段,部分成果仅供参考,但一部分成果已经指导临床实践,目前,一线和二线姑息治疗方法有所改善,辅助治疗的生存率有所提高。通过生物标志物来确定治疗方案和新辅助疗法,将为改善病人预后提供机会。选择精准的“个体化”化疗方案,筛选化疗或靶向治疗的获益人群,可使更多CRC肝转移的病人能够通过筛选进行精准治疗,最大限度地避免无效医疗。随着基因组学及蛋白组学的快速发展,CRC肝转移的个体化治疗仍有许多问题有待解决,结合基因的表达情况以选择合理的治疗方式可能是将来精准治疗的方向。

