禾谷镰孢碳源代谢调控因子FgCreA的功能
侯瑞,王晨芳
禾谷镰孢碳源代谢调控因子FgCreA的功能
侯瑞1,王晨芳2
(1贵州大学林学院,贵阳 550025;2西北农林科技大学植物保护学院,陕西杨凌 712100)
【目的】敲除禾谷镰孢()中碳源代谢调控因子FgCreA,并对其营养生长、有性生殖和致病力等方面进行研究,为研究禾谷镰孢中碳源代谢机制提供依据。【方法】根据酵母数据库SGD和NCBI数据库中的序列,利用酿酒酵母中碳源代谢调控因子Mig1确定禾谷镰孢中的碳源调控因子FgCreA;在NCBI数据库中检索其他物种中碳源代谢调控蛋白,使用ClustalW2软件进行多重序列比对,并用MEGA5软件构建系统进化树,同时在InterProScan网站上预测其蛋白结构域;在NCBI数据库中检索与禾谷镰孢碳源吸收相关的结构基因和真菌毒素DON生物合成的相关基因,调取起始密码子上游1 000 bp的片段,预测基因结合位点位置;用Primer5软件设计引物,利用Split-PCR和PEG介导原生质体转化的方法进行基因敲除,并通过PCR和Southern blot验证获得基因敲除突变体。根据突变体营养生长、有性生殖和致病力等方面的变化分析的功能。【结果】通过生物信息学方法,明确禾谷镰孢中只有一个碳源代谢调控因子基因(FGSG_09715),氨基酸序列416 aa,共包含两个保守的C2H2锌指结构区域。FgCreA同源蛋白在各类真菌中均存在一定程度的同源性,具有较高的保守性。禾谷镰孢碳源吸收相关结构基因(、、、、)和真菌毒素DON生物合成相关基因(、、、、、、、、、)启动子区均含有DNA结合位点。采用Split-PCR技术和PEG介导的原生质体转化,通过PCR和Southern blot获得并验证了两个基因敲除突变体。表型观察发现,突变体的生长速度与野生型相比下降90%;分生孢子形态正常,但产孢量与野生型相比降低88%;有性生殖阶段,突变体可以产生正常的子囊壳、子囊和子囊孢子,但需要比野生型长20—28 d;突变体对钠盐较敏感,侵染小麦穗不发病。【结论】获得禾谷镰孢碳代谢调控因子FgCreA敲除突变体,明确FgCreA参与营养生长和有性生殖,并能够影响致病过程。但是否影响相关结构基因的转录水平和DON生物合成仍需进一步验证。
禾谷镰孢;;基因敲除;表型;碳源代谢
0 引言
【研究意义】小麦赤霉病(Fusarium head blight,FHB)近年来已成为世界性病害,造成小麦大面积减产[1]。小麦赤霉病的主要致病菌为禾谷镰孢()[2-4],其不仅可以影响小麦产量,还可产生真菌毒素脱氧雪腐镰刀菌烯醇(deoxynivalenol,DON)和玉米烯酮(zearalenone,ZEA)等[5-6],严重影响小麦的品质。近年来,禾谷镰孢全基因组测序的工作已经完成[7],目前已经开始进行基因组[8]、蛋白质组[9]、转录组[10-11]和代谢组学[12]等方面的研究。碳源代谢作为真菌生长发育重要的代谢途径之一,其代谢机制对于生长发育、次生代谢产物、抗病性等方面都具有重要作用[13]。研究禾谷镰孢碳源代谢调控因子基因的功能,明确其在生长发育阶段和致病力方面的作用,对于研究禾谷镰孢中碳源代谢机制具有至关重要的意义。【前人研究进展】微生物可以利用多种碳源物质,但当生长环境中存在碳源葡萄糖时,真菌会首先使用葡萄糖作为碳源,因此葡萄糖被称为优先碳源。当生长环境中缺少葡萄糖时,其他碳源如半乳糖、蔗糖、麦芽糖和木聚糖等就会被吸收和利用,这些碳源被称为次级碳源[14]。在酵母和丝状真菌中,优先碳源葡萄糖的存在会抑制吸收次级碳源的相关结构基因转录和表达,故葡萄糖抑制也被称为碳代谢抑制(carbon catabolite repression,CCR)[13]。碳源代谢调节机制就是使葡萄糖在生长环境中能够被优先吸收和利用,如果生长环境中没有葡萄糖,酵母和丝状真菌就可以选择性地吸收和利用次级碳源。这种调节机制可以确保许多编码酶类和通透酶类的结构基因能够被激活表达以吸收和利用次级碳源[15]。在酵母和丝状真菌中,碳源代谢受Mig/CreA转录因子调控,多数结构基因的转录可被Mig/CreA转录因子激活。Mig/CreA转录因子含有两个Cys2/Cys2-锌指结构,能够识别目标基因启动子序列中DNA序列5′-SYGGRG-3′(其中,S代表C、G,Y代表C、T,R代表A、G)[16]。酿酒酵母()中,葡萄糖抑制可被碳源代谢调节因子Mig1和Mig2调控,当有葡萄糖存在时,与碳源代谢相关的结构基因,如、、和等,其转录水平都会受到抑制[17-18]。Mig1能够结合碳源代谢相关结构基因的启动子区,与碳源共抑制因子Ssn6和Tup1形成复合体抑制碳源代谢相关基因的转录[19-21]。Mig1的活性受到蛋白激酶Snf1/Snf4的调节,当环境中葡萄糖含量较高时,Mig1蛋白会被去磷酸化且定位于细胞核,抑制其他结构基因的转录。当葡萄糖含量较低时,Mig1蛋白会被迅速磷酸化并转移至细胞质中[22]。然而的缺失,并没有完全消除葡萄糖抑制机制,例如转录水平依然被抑制。现已研究发现,Mig2作为另一碳源代谢因子也参与了调节,起着次要作用[23]。博伊丁假丝酵母()中,葡萄糖能够完全抑制甲醇诱导基因的表达,但基因敲除突变体却能加强甲醇诱导的编码醇氧化酶的表达,不过基因敲除突变体在各种碳源培养基中并没有生长变化[24]。尖镰孢()中,CreA同源蛋白Cre1能够抑制细胞壁降解酶CWDEs和吸收C2关键酶的转录水平[25]。构巢曲霉()中,CCR同样可被转录因子CreA调控。在葡萄糖存在的情况下,编码木聚糖酶、纤维素酶和阿拉伯糖酶等酶类的结构基因会被抑制转录。CreA能够直接结合木聚糖酶编码基因和启动子区来调节其表达[26]。此外,碳源代谢因子还参可与次生代谢产物的合成[27-28]。构巢曲霉中,青霉素合成相关基因能够被CreA抑制[27]。在藤仓赤霉()中,许多赤霉素相关基因中都能找到潜在的CreA结合位点。说明可能参与了额外的激素水平的调节[28]。【本研究切入点】禾谷镰孢中碳源代谢调控因子编码基因为(FGSG_09715)。碳源代谢是微生物生长的重要代谢途径,但在禾谷镰孢中尚未报道过碳源代谢调控因子的功能研究。【拟解决的关键问题】利用同源重组和原生质体转化等手段,获得禾谷镰孢的敲除突变体,鉴定该基因可参与调控禾谷镰孢的营养生长、有性生殖和致病力等方面,为禾谷镰孢中碳源代谢机制的研究提供理论依据。
1 材料与方法
试验于2014—2016年在西北农林科技大学西农普度联合中心完成。
1.1 材料
1.1.1 菌株和试剂 禾谷镰孢野生型PH-1菌株[29]、突变体菌株、质粒pCB1003、载体PFL2和菌株DH10B等均保存于西北农林科技大学西农普度联合中心实验室。Tap酶和限制性内切酶购于Fermentas公司,DNA胶回收试剂盒购于Bio-Teke公司,小量质粒提取试剂盒购于OMEGA公司,DNA poly I klenow试剂盒购于Promega公司,其他常用试剂均为国内采购。
1.1.2 培养基和溶液 马铃薯葡萄糖琼脂培养基(PDA)培养基:PDA粉(国产)38 g·L-1;羟甲基化纤维素(carboxymethylcellulose,CMC)培养基:羟甲基化纤维素(低黏度,sigma公司)15 g·L-1,硝酸铵1g·L-1,磷酸二氢钾1 g·L-1,七水硫酸镁0.5 g·L-1,酵母提取物1g·L-1; YEPD(yeast extract peptone dextrose)液体培养基:酵母提取物3 g·L-1,蛋白栋10 g·L-1,葡萄糖20 g·L-1;胡萝卜培养基:100 g·L-1胡萝卜(用榨汁机榨成汁),琼脂粉10 g。保存于室温。
1×STC缓冲液、PTC缓冲液、TB3液体培养基、Bottom培养基/Top培养基、燕麦培养基、LB液体/固体培养基等具体配方详见文献[29]。1.2 mol·L-1KCl溶液、原生质体缓冲液(酶解液)、CTAB抽提液、southern变性转移液、southern中和液,20×SSC、50% PEG3350等主要溶液配方详见文献[30]。
1.2 氨基酸序列获得与分析、结构域预测及结合位点位置查找
从酵母数据库(SGD: http://www.yeastgenome. org/)中得到酿酒酵母的Mig1蛋白序列,然后利用NCBI数据库进行蛋白序列比对,找到禾谷镰孢中Mig1/CreA蛋白同源序列,之后进行反向查找,最终确定禾谷镰孢中CreA同源蛋白基因号和氨基酸序列。从NCBI数据库或者真菌数据库中获得酿酒酵母、裂殖酵母()、玉米黑粉菌()、粗糙脉孢()、稻瘟菌()、构巢曲霉、米曲霉()、棒曲霉()、禾谷镰孢、拟轮枝镰孢()、尖镰孢等碳源代谢调节因子蛋白序列,利用MEGA软件进行病原真菌碳源代谢调节因子蛋白的系统进化树构建。并用InterProScan(http://www.ebi.ac.uk/interpro/)数据库对其结构域进行预测。通过NCBI数据库查找禾谷镰孢真菌毒素DON生物合成相关基因家族主要基因和碳源吸收相关基因和以及CWDEs基因。下载相关基因启动子区1 000 bp,查找基因结合位点5′-SYGGRG-3′(其中,S代表C、G,Y代表C、T,R代表A、G)。
1.3 重组片段的扩增和转化子的筛选
利用Split-PCR的方法进行基因序列重组,之后进行禾谷镰孢野生型PH-1原生质体转化,用潮霉素()抗性进行初步筛选,提取初步筛选的转化子DNA,利用4对引物进行PCR验证,分别为1:H852-F+H850-R,2:FgCREA-JF+H855-R,3:H856- F+FgCREA-JR以及4:FgCREA-OF+FgCREA-OR,获得PCR验证的敲除转化子。根据目的基因的DNA序列以及敲除后的预设DNA序列选取限制性内切酶Ⅰ进行Southern blot验证。Split-PCR和Southern blot的具体试验方法参照文献[31]。将含有的互补载体转入PCR验证的敲除转化子原生质体中,利用新潮霉素()抗性进行初步筛选,观察表型是否能够恢复(表1)。
1.4 禾谷镰孢相关菌株表型分析
生长速率测定:将活化相同天数的野生型菌株和突变体菌株的菌饼接种至PDA固体培养基中,将菌饼正面朝下接到90 mm培养皿中心位置,各做5个重复。倒置培养皿于25℃恒温培养箱中培养3 d,测量菌落半径并拍照。
菌落边缘菌丝形态的观察:将15 ml 1/2 CM固体培养基倒至装有载玻片的90 mm无菌玻璃皿内。接种小块菌株于凝固载玻片的两端。各做5个重复。25℃恒温培养16—24 h(根据菌株的生长速度决定),使菌丝刚好长到载玻片的边缘位置。用无菌刀片将载玻片周围培养基划掉,将载玻片取出。在显微镜下进行观察,微分干涉照相。

表1 禾谷镰孢FgCREA基因敲除和互补所用引物
产孢量测定:将活化相同天数的野生型菌株和突变体菌株用打孔器在菌落边缘打孔。转移一块菌饼至装有50 ml CMC培养基的150 ml三角瓶中。各做5个重复。在25℃恒温摇床中,175 r/min培养5 d,收集滤液用血球计数板计数CMC滤液中分生孢子的浓度。
分生孢子、分生孢子萌发形态观察:分生孢子观察时,直接吸取菌株CMC培养液,置于显微镜下观察分生孢子形态并进行微分干涉照相。分生孢子萌发观察时,首先收集CMC培养液中的分生孢子,然后转移分生孢子至150 ml三角瓶(内含50 ml YEPD)中。在25℃恒温摇床中,175 r/min培养12 h后用显微镜观察并进行微分干涉照相。
有性生殖检测:将活化相同天数野生型菌株、突变体菌株的菌落边缘菌饼接种至胡萝卜培养基的中心位置,各做5个重复。在25℃恒温培养箱培养7 d后加入0.1% Tween-20湿润气生菌丝,使其贴伏于培养基表面,晾干。在黑光灯﹕黑暗=12 h﹕12 h,25℃条件下继续诱导培养,如果培养皿中有气生菌丝生长出来则要及时压(操作同上一步)。4周后对培养皿进行拍照,在显微镜下观察压开的子囊壳内的子囊及子囊孢子形态。
过氧化氢和氯化钠压力筛选:分别将活化相同天数的野生型菌株和突变体菌株的菌饼接到含0.03% H2O2、0.7 mol·L-1NaCl的PDA平板两侧,各做5个重复。25℃恒温培养6 d后,观察菌落生长情况,并照相。
致病力检测:分别收集野生型菌株和突变体菌株的分生孢子悬浮液,然后将分生孢子悬浮液浓度调制(1.5—2.0)×105个孢子/ml,接种扬花期小麦感病品种Norm,各做15个重复,保湿48 h,两周后观察致病情况。
2 结果
2.1 禾谷镰孢FgCreA序列分析和相关基因启动子区FgCREA结构位点预测
从酵母数据库中获得已经报道的酿酒酵母碳源代谢因子Mig1的氨基酸序列,利用NCBI BLASTP数据库(https://www.ncbi.nlm.nih.gov/)选定生物名称为禾谷镰孢PH-1(taxid:569360),得到一个同源性最高(73%)的基因FGSG_09715(XP_011327987),氨基酸序列416 aa。对预测的禾谷镰孢FgCreA转录因子FGSG_09715基因蛋白序列进行结构域分析(http://www.ebi.ac.uk/interpro/),发现具有两个锌指结构(IPR013087,66-86;IPR013087,94-116)(图1-a)。利用MEGA软件进行碳源代谢调节因子在真菌中的同源性分析。通过比对结果可以看出,碳源代谢调节因子在各类真菌中存在较高程度的同源性(图1-b)。同时,预测了在禾谷镰孢真菌毒素DON生物合成家族和碳源吸收相关结构基因启动子区的DNA结合位点,结果表明相关基因启动子区域均有潜在的结合位点(表2)。
2.2 FgCREA基因敲除转化子与互补菌株的筛选和验证
通过Split-PCR的方法构建的重组片段,然后转入禾谷镰孢野生型菌株PH-1的原生质体中,通过潮霉素()抗性筛选得到转化子。对筛选得到的转化子进行分子水平验证(图2-b),同时利用Southern blot对PCR验证获得的敲除转化子进一步验证,获得2个基因敲除突变体(图2-d)。同时,克隆并构建互补载体Com-CREA,转化到Southern blot验证后的敲除突变体原生质体中,但由于敲除突变体生长速率太慢,得到的原生质体数量太少,目前并没有得到互补菌株。
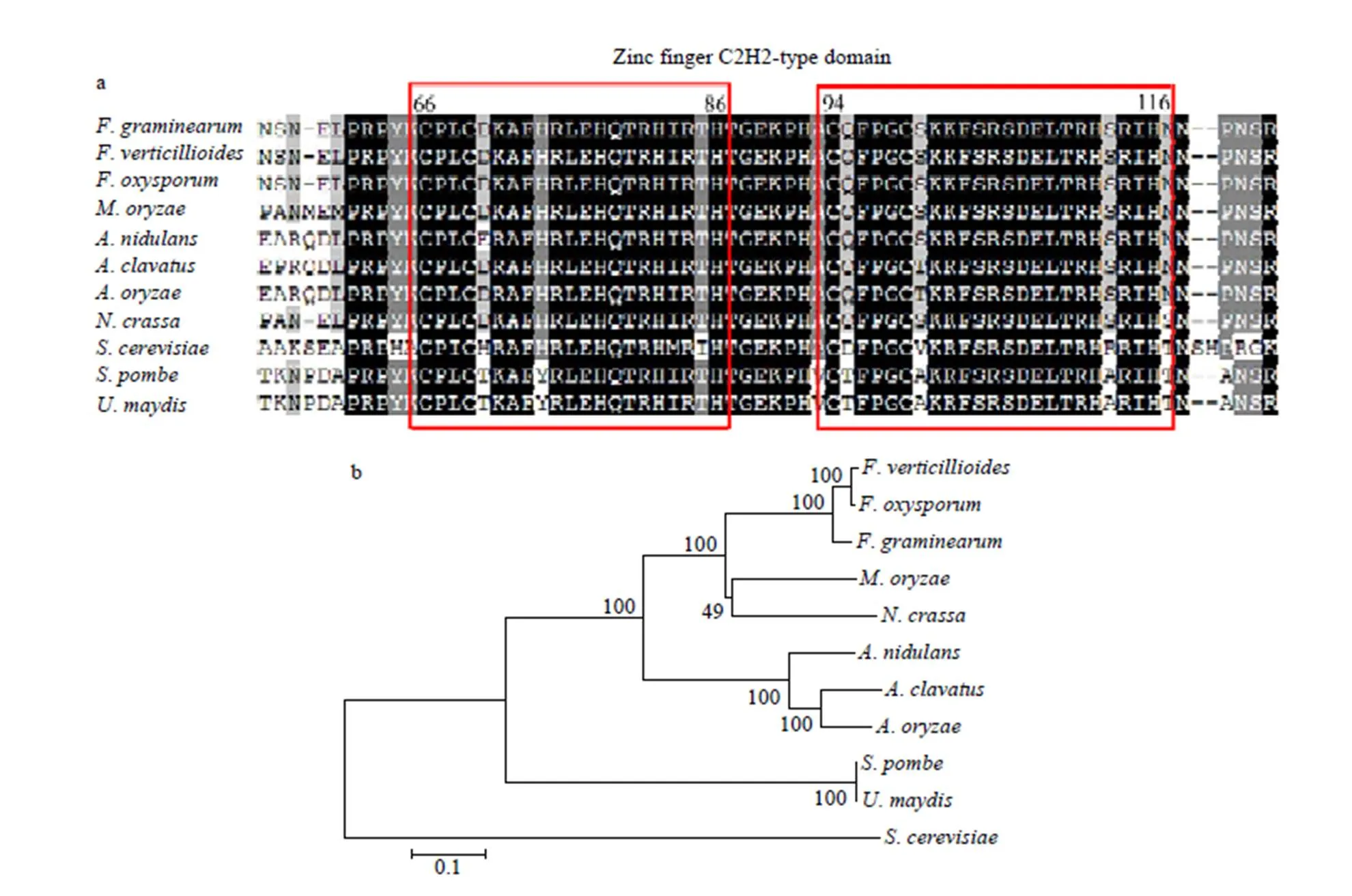
a:禾谷镰孢FgCreA与不同物种中碳源调节因子蛋白的锌指结构示意图The zinc finger structure between FgCreA and CreA homologs from various fungi;b:禾谷镰孢FgCreA与不同物种中碳源调节因子蛋白的进化树分析Phylogenetic analysis of FgCreA and CreA homologs from various fungi
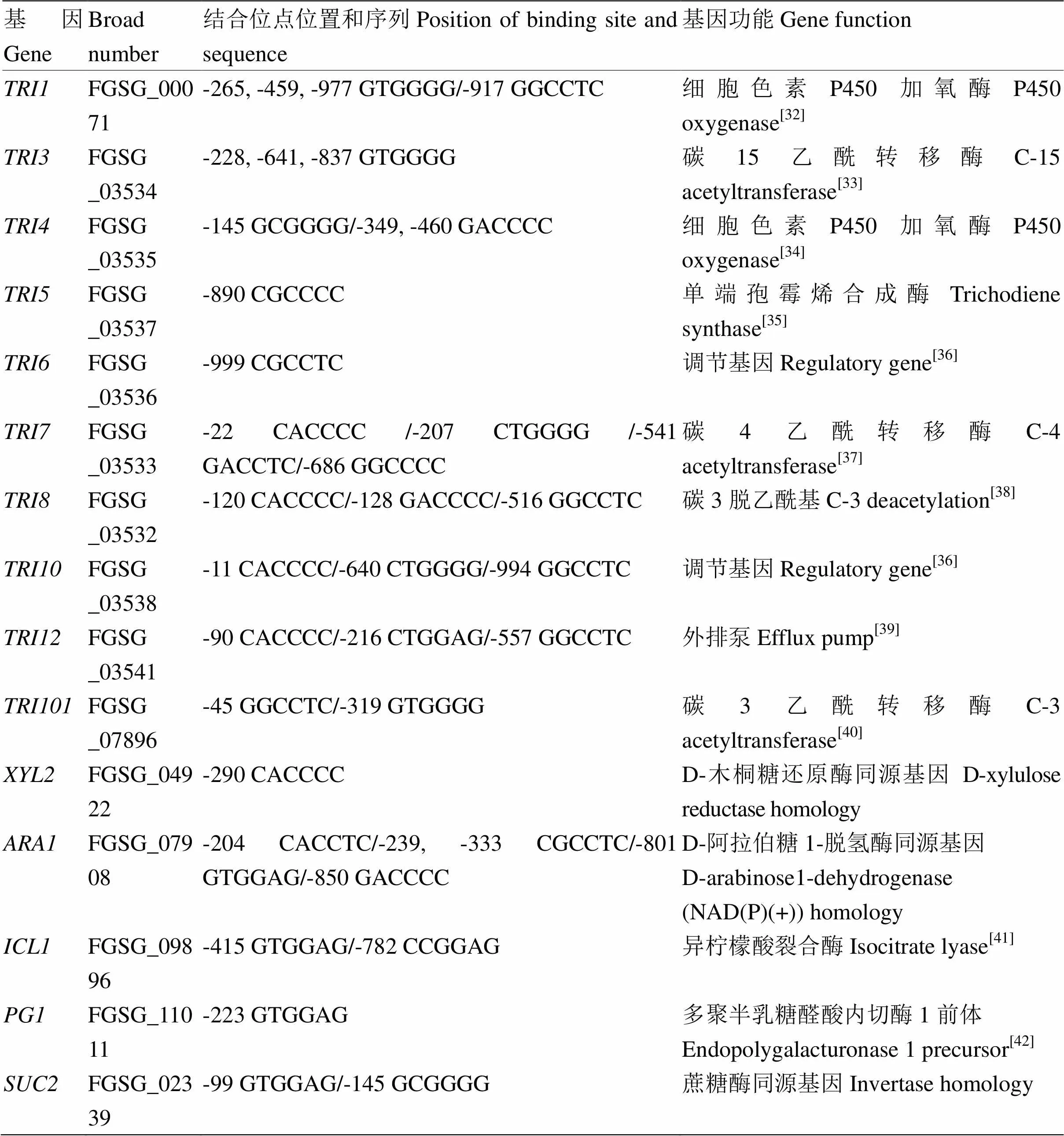
表2 FgCREA在DON生物合成TRI家族主要基因及碳源吸收相关基因启动子区潜在结合位点
结合位点5′-SYGGRG- 3′binding site 5′-SYGGRG- 3′
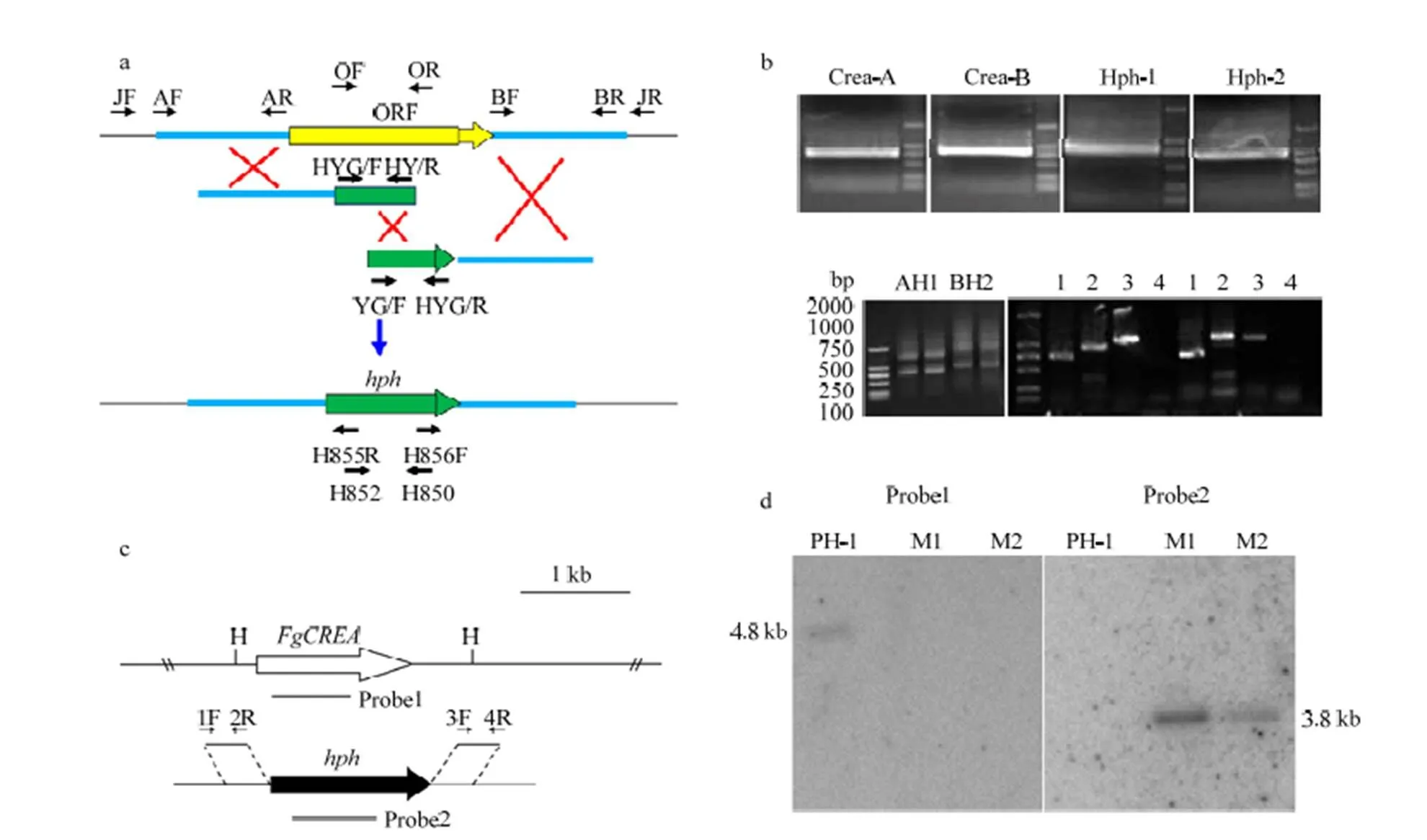
a:FgCREA基因敲除原理Schematic diagrams for FgCREA gene knockout;b:阳性转化子的PCR验证PCR screening for FgCREA mutants;c:FgCREA基因敲除Southern验证原理FgCREA gene knockout construct;d:分子验证转化子的Southern验证Southern blot verification for FgCREA mutants
2.3 FgCREA的缺失对营养生长的影响
基因敲除突变体在PDA培养基上培养3 d后,发现突变体菌落生长速率比野生型PH-1菌株减慢了90%左右(图3-a、表3)。菌落边缘观察发现,突变体在72 h时气生菌丝相比野生型变得致密且分枝变多(图3-b)。结果表明,对菌丝的正常生长是必需的,但应该只是严重影响生长速率,并不影响菌丝的形态。
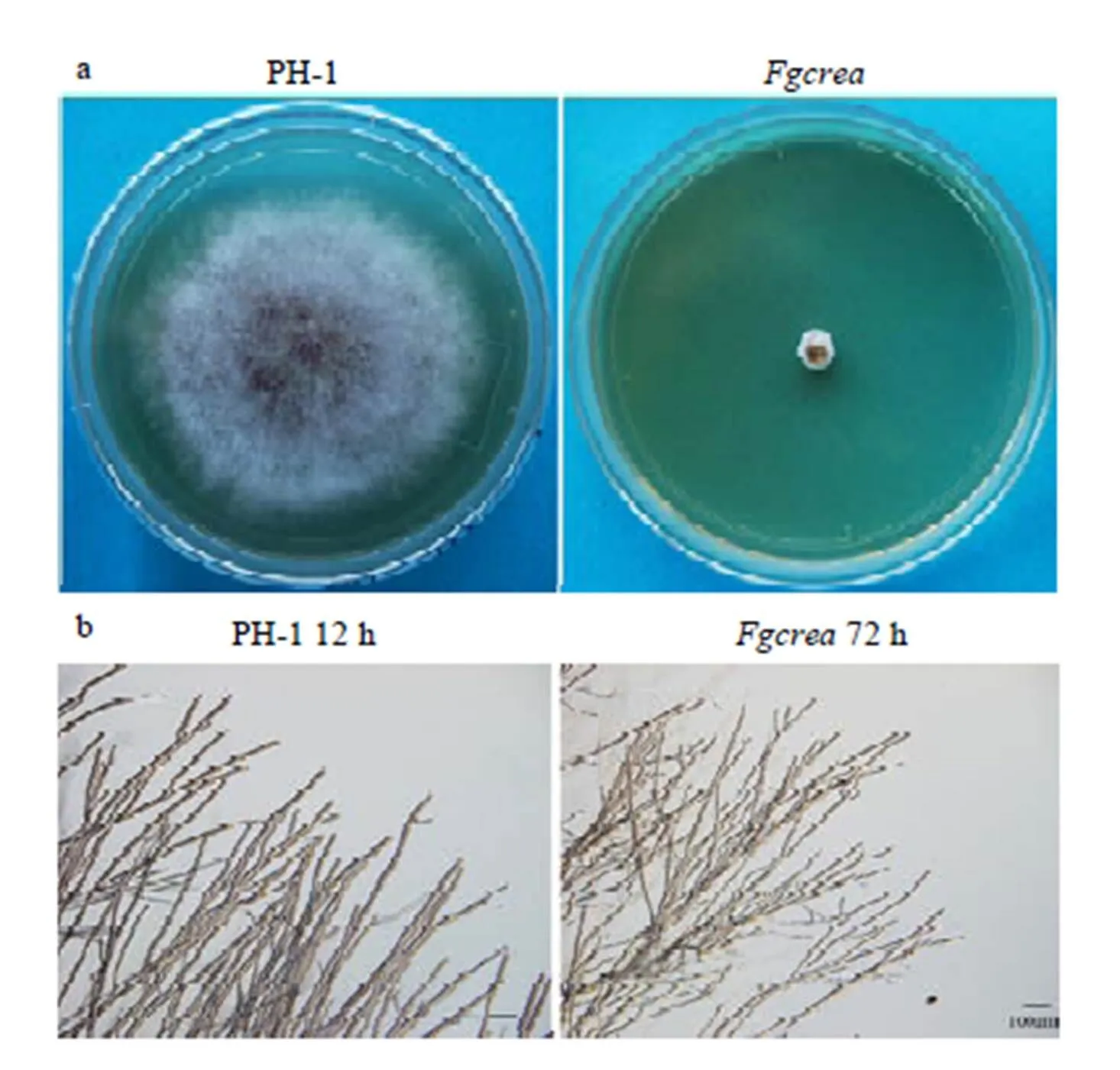
图3 FgCREA基因敲除突变体的菌落形态和菌丝生长
表3 FgCREA基因敲除突变体的生长速率及产孢量
数据为平均数±标准差(=3),数据后不同字母表示在<0.05水平差异显著(LSD法)
Data are average±SD (=3), different letters mean significant difference at<0.05 level (LSD method)
2.4 FgCREA基因缺失对孢子萌发速率的影响
血球计数板统计发现,突变体分生孢子的产量大约为野生型PH-1菌株的12%(表3)。观察突变体分生孢子产生过程,发现的缺失并未使产孢结构受到影响,产孢量下降应该是由于突变体生长缓慢造成的。观察还发现突变体分生孢子形态与野生型PH-1菌株的基本一致。对突变体分生孢子萌发12 h的菌丝形态观察发现,虽然其萌发速率降低,但孢子顶端、基部和中间的细胞也均会形成芽管(图4)。
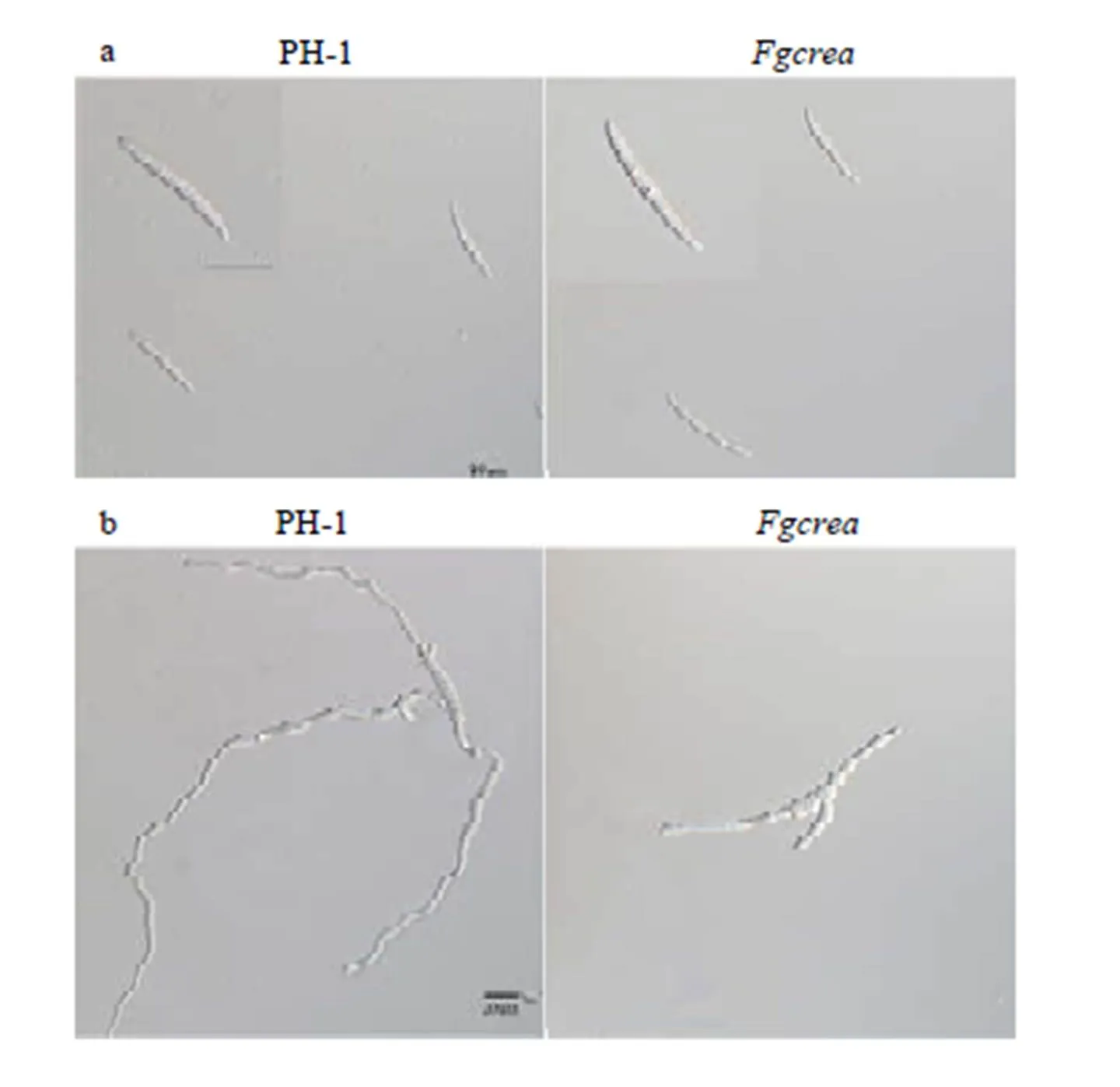
图4 FgCREA基因敲除突变体分生孢子形态(a)和分生孢子萌发形态(b)
2.5 FgCREA基因缺失对有性生殖的影响
将活化好的突变体和野生型PH-1菌株接种于胡萝卜培养基25℃恒温培养10 d后(突变体生长速率过慢),使用0.1%的tween-20处理菌丝,之后以12 h黑光灯﹕12 h黑暗25℃诱导培养28 d,观察并照相。结果发现突变体只能产生较少数量的子囊壳(图5-a)。压开子囊壳,观察子囊以及子囊孢子的形态,发现的缺失并未影响子囊和子囊孢子的形成(图5-b)。
2.6 FgCREA基因缺失对细胞渗透压的影响
通过压力筛选(图6),发现H2O2压力胁迫对于突变体的相对抑制率为1%,说明的缺失对氧化还原通路没有影响。但突变体对0.7 mol·L-1的NaCl表现敏感,几乎停止生长,相对抑制率为95%。
2.7 FgCREA基因缺失对致病力的影响
利用分生孢子悬浮液接种扬花期小麦穗部,发现突变体侵染小麦穗的致病力严重下降,仅在小麦穗接种点发病(图7)。
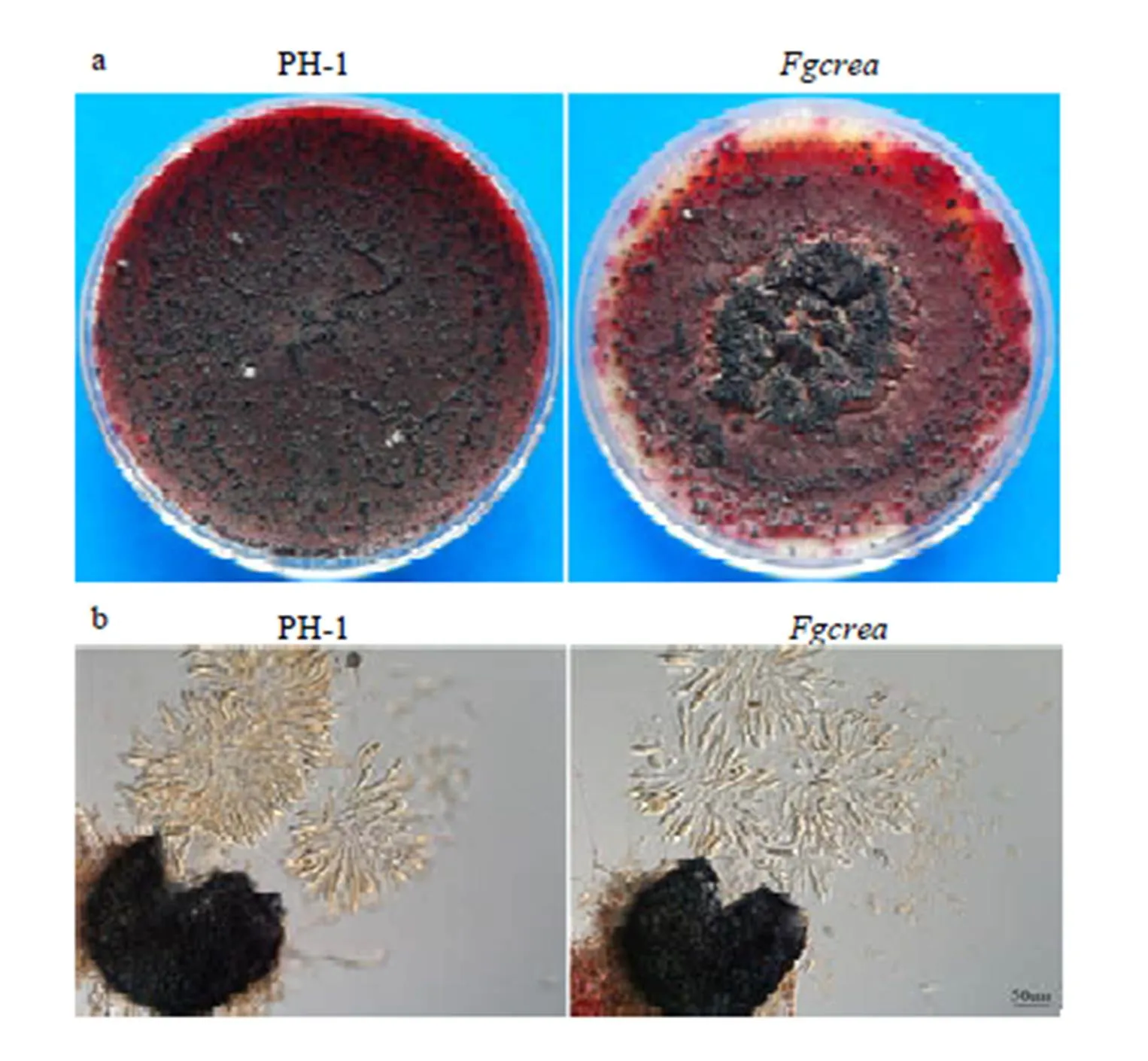
图5 FgCREA基因敲除突变体子囊壳、子囊和子囊孢子形态
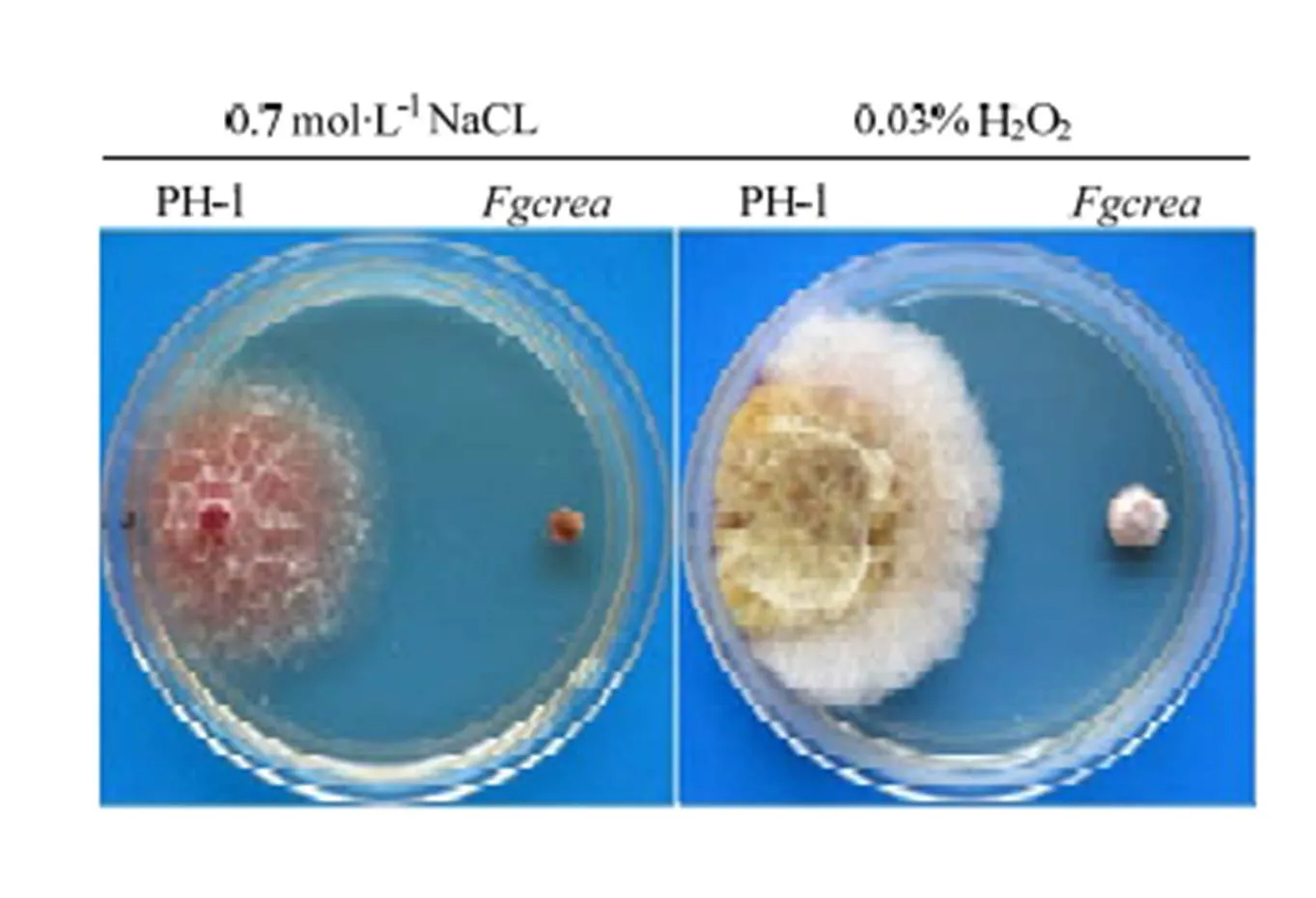
图6 FgCREA基因敲除突变体的压力筛选
3 讨论
利用生物信息学方法,发现禾谷镰孢中碳源代谢调节因子只有一个,即FGSG_09715。与丝状真菌和酵母中碳源代谢调节因子结构类似,均具有两个C2H2锌指结构,能够结合启动子区DNA序列5′-SYGGRG-3′。通过构建进化树,发现同源基因的进化关系与物种进化趋势一致,说明应为酿酒酵母碳源代谢因子的直系同源基因。因此,的功能应与碳源代谢相关。
在细胞壁降解酶CWDEs基因、、,异柠檬酸裂解酶基因,蔗糖酶基因启动子区均有DNA结合位点,这些碳源吸收结构基因在酿酒酵母和尖镰孢中均已报道能够与互作或者可受到基因调控[43]。突变体在生长速率、产孢量、分生孢子萌发速率和有性生殖方面均存在严重缺陷,应该是由于基因缺失导致利用次级碳源的相关结构基因转录水平下降所致[44]。酿酒酵母中,Mig1和Kin2互作[45],Kin2和Ssk2互作[46],而Ssk2蛋白是真菌渗透压调控Hog1信号通路中的MAPKKK(mitogen activated protein kinase kinase kinase)[47]。突变体对渗透压的敏感性表明在禾谷镰孢中,可能也通过与FgSsk2(FGSG_00408)互作而参与调控渗透压信号通路。突变体侵染小麦穗部仅在接种点发病,表明致病力下降的主要原因是受到生长速率和分生孢子萌发率的影响。因为侵染试验中接种的孢子数量是相同的,所以产孢量对致病性的影响并没有作为考虑因素。另外,突变体对渗透压敏感,可能会通过Hog1途径而影响致病力[48]。因此,突变体致病力下降的主要原因应该是生长速率减慢、分生孢子萌发率下降和渗透压敏感。

图7 FgCREA基因敲除突变体对小麦穗部的致病力
禾谷镰孢中葡萄糖不能抑制真菌毒素DON生物合成家族的表达以及DON的生物合成,但蔗糖却能够强烈诱导。在用蔗糖作为唯一碳源的培养基中额外加入葡萄糖,也不能够抑制DON生物合成,说明碳源代谢抑制CCR应该是没有参与DON生物合成系统[49]。但在DON生物合成家族主要基因启动子区均有DNA结合位点,表明FgCreA应该是与DON生物合成相关。研究已发现,酿酒酵母中Mig1蛋白能够和促分裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)通路中Pmk1途径中心激酶Set7互作[50]。敲除禾谷镰孢中的激酶基因(FGSG_09903)可导致DON生物合成量急剧降低且影响病原菌在小麦穗部的定殖和扩展[48,51]。Mig1蛋白同时还可与环腺苷酸单磷酸-蛋白激酶A(cyclic adenosine phosphate-protein kinase A,cAMP-PKA)通路中的cAMP磷酸二酯酶基因互作[52]。敲除可以激活PKA活性并增加DON的合成[53]。禾谷镰孢中是否可与FgSte7和FgPde2蛋白互作,是否可参与调控DON生物合成还需进一步验证。白色念珠菌()中,CaMig1蛋白缺乏潜在的激酶Snf1磷酸化位点[54]。子囊菌红褐肉座菌()中,Cre1虽然具有潜在的激酶Snf1磷酸化位点,但激酶Snf1并不能够磷酸化碳源代谢调节因子Cre1[55],通过生物信息学方法预测到禾谷镰孢中CreA蛋白具有磷酸化位点,接下来将会进一步验证CreA蛋白是否能够被激酶Snf1磷酸化。
4 结论
利用Split-PCR和Southern blot等方法得到禾谷镰孢中碳源代谢调节因子基因缺失突变体。能够影响营养生长、产孢量、孢子萌发速率、有性生殖、致病力和对钠盐的渗透压敏感度,但是否参与了碳源代谢抑制和DON生物合成还需后期验证。
[1] Muriuki J G. Deoxynivalenol and nivalenol in pathogenesis of Fusarium head blight in wheat[D]. USA: University of Minnesota, 2001.
[2] Bai G H, Shaner G. Management and resistance in wheat and barley to Fusarium head blight., 2004, 42(1): 135-161.
[3] Goswami R S, Kistler H C. Heading for disaster:on cereal crops., 2004, 5(6): 515-525.
[4] 王裕中, J D米勒. 中国小麦赤霉病菌优势种—禾谷镰刀菌产毒素能力的研究. 菌物学报, 1994, 13(3): 229-234.
Wang Y Z, Miller J D. Toxin producing potential offrom China., 1994, 13(3): 229-234. (in Chinese)
[5] O’Donnell K, Kistler H C, Tacke B K, Casper H H. Gene genealogies reveal global phylogeographic structure and reproductive isolation among lineages of, the fungus causing wheat scab., 2000, 97(14): 7905-7910.
[6] 王琢, 闫培生. 真菌毒素产生菌的分子鉴定研究进展. 中国农业科技导报, 2010, 12(5): 42-50.
Wang Z, Yan P S. Research progress on molecular identification of mycotoxin-producing fungi., 2010, 12(5): 42-50. (in Chinese)
[7] Cuomo C A, Güldener U, Xu J R, Trail F, Turgeon B G, Di Pietro A, Walton J D, Ma L J, Baker S E, Rep M, Adam G, Antoniw J, Baldwin T, Calvo S, Chang Y L, Decaprio D, Gale L R, Gnerre S, Goswami R S, Hammond-Kosack K, Harris L J, Hilburn K, Kennell J C, Kroken S, Magnuson J K, Mannhaupt G, Mauceli E, Mewes H W, Mitterbauer R, Muehlbauer G, Münsterkötter M, Nelson D, O'donnell K, Ouellet T, Qi W, Quesneville H, Roncero M I, Seong K Y, Tetko I V, Urban M, Waalwijk C, Ward T J, Yao J, Birren B W, Kistler H C.Thegenome reveals a link between localized polymorphism and pathogen specialization., 2007, 317(5843): 1400-1402.
[8] 张大军, 邱德文, 蒋伶活. 禾谷镰刀菌基因组学研究进展. 安徽农业科学, 2009, 37(17): 7892-7894.
Zhang D J, QIu D W, JIang L H. Research progress on the genomics of., 2009, 37(17): 7892-7894. (in Chinese)
[9] Brown D W, Cheung F, Proctor R H, Butchko R A, Zheng L, Lee Y, Utterback T, Smith S, Feldblyum T, Glenn A E, Plattner R D, Kendra D F, Town C D, Whitelaw C A. Comparative analysis of 87,000 expressed sequence tags from the fumonisin-producing fungus., 2005, 42(10): 848-861.
[10] Sikhakolli U R, Lopez-Giraldez F, Li N, Common R, Townsend J P, Trail F. Transcriptome analyses during fruiting body formation inandreflect species life history and ecology., 2012, 49(8): 663-673.
[11] Lysoe E, Seong K Y, Kistler H C. The transcriptome ofduring the infection of wheat., 2011, 24(9): 995-1000.
[12] Kumaraswamy K G, Kushalappa A C, Choo T M, Dion Y, Rioux S. Mass spectrometry based metabolomics to identify potential biomarkers for resistance in barley against fusarium head blight ()., 2011, 37(8): 846-856.
[13] Ruiz B, Chavez A, Forero A, García-Huante Y, Romero A, Sánchez M, Rocha D, Sánchez B, Rodríguez-Sanoja R, Sánchez S, Langley E. Production of microbial secondary metabolites: regulation by the carbon source., 2010, 36(2): 146-167.
[14] Ruijter G J, Visser J. Carbon repression in., 1997, 151(2): 103-114.
[15] Ries L N, Beattie S R, Espeso E A, Cramer R A, Goldman G H. Diverse regulation of the CreA carbon catabolite repressor in., 2016, 203(1): 335-352.
[16] Carlson M. Glucose repression in yeast., 1999, 2(2): 202-207.
[17] Gancedo J M. Yeast carbon catabolite repression., 1998, 62(2): 334-361.
[18] Johnston M. Feasting, fasting, and fermenting: glucose sensing in yeast and other cells., 1999, 15(1): 29-33.
[19] Trumbly R J. Glucose repression in the yeast., 1992, 6(1): 15-21.
[20] Treitel M A, Carlson M. Repression by SSN6-TUP1 is directed by MIG1, a repressor/activator protein., 1995, 92(8): 3132-3136.
[21] Tzamarias D, Struhl K. Distinct TPR motifs of Cyc8 are involved in recruiting the Cyc8-Tup1 corepressor complex to differentially regulated promoters., 1995, 9(7): 821-831.
[22] Treitel M A, Kuchin S, Carlson M. Snf1 protein kinase regulates phosphorylation of the Mig1 repressor in., 1998, 18(11): 6273-6280.
[23] Park S H, Koh S S, Chun J H, Hwang H J, Kang H S. Nrg1 is a transcriptional repressor for glucose repression ofgene expression in., 1999, 19(3): 2044-2050.
[24] Zhai Z, Yurimoto H, Sakai Y. Molecular characterization ofand its role in the regulation of methanol- inducible gene expression., 2012, 29(7): 293-301.
[25] Jonkers W, Rep M. Mutation ofreverts the pathogenicity defects of thedeletion mutant., 2009, 74(5): 1100-1113.
[26] Tamayo E N, Villanueva A, Hasper A A, de Graaff L H, Ramón D, Orejas M.mediates repression of the regulatory genewhich controls the production of xylanolytic enzymes in., 2008, 45(6): 984-993.
[27] Espeso E A, Fernández-Cañón J, Peñalva M A. Carbon regulation of penicillin biosynthesis in: a minor effect of mutations inand., 1995, 126(1): 63-67.
[28] Tudzynski, B, Liu S, Kelly J M. Carbon catabolite repression in plant pathogenic fungi: isolation and characterization of theandgenes., 2000, 184(1): 9-15.
[29] Brown D W, Butchko R A E, Proctor R H. Fusarium genomic resources: Tools to limit crop diseases and mycotoxin contamination., 2006, 162(3): 191-199.
[30] Catlett N L, Lee B N, Yoder O C, Turgeon B G. Split-marker recombination for efficient targeted deletion of fungal genes., 2003, 50: 9-11.
[31] Zhao X H, Xu J R. A highly conserved MAPK-docking site in Mst7 is essential for Pmk1 activation in., 2007, 63(3): 881-894.
[32] McCormick S P, Harris L J, Alexander N J, OUELLET T,SaparnoA, AllardS,DesjardinsA E.encodes a P450 oxygenase., 2004, 70(4): 2044-2051.
[33] Tokai T, Takahashi-Ando N, Izawa M, Kamakura T, Yoshida M, Fujimura M, Kimura M. 4--acetylation and 3--acetylation of trichothecenes by trichothecene 15--acetyltransferase encoded by., 2008, 72(9): 2485-2489.
[34] Menke J, Weber J, Broz K, Kistler H C. Cellular development associated with induced mycotoxin synthesis in the filamentous fungus., 2013, 8(5): e63077.
[35] Boenisch M J, Schafer W.forms mycotoxin producing infection structures on wheat., 2011, 11: 110.
[36] Seong K Y, Pasquali M, Zhou X, Song J, Hilburn K, McCormick S, Dong Y, Xu J R, Kistler H C. Global gene regulation bytranscription factorsandreveals adaptations for toxin biosynthesis., 2009, 72(2): 354-367.
[37] Chandler E A, Simpson D R, Thomsett M A, Nicholson P. Development of PCR assays toandtrichothecene biosynthetic genes, and characterisation of chemotypes of,and., 2003, 62(6): 355-367.
[38] McCormick S P, Alexander N J.encodes a trichothecene C-3 esterase., 2002, 68(6): 2959-2964.
[39] Menke J, Dong Y, Kistler H C.Tri12p influences virulence to wheat and trichothecene accumulation., 2012, 25(11): 1408-1418.
[40] Garvey G S, McCormick S P, Alexander N J, Rayment I. Structural and functional characterization oftrichothecene 15-O-acetyltransferase from., 2009, 18(4): 747-761.
[41] Park Y, Cho Y, Lee Y H, Lee Y W, Rhee S. Crystal structure and functional analysis of isocitrate lyases fromand., 2016, 194(3): 395-403.
[42] AndreLeroux G, Tessier D, Bonnin E. Action pattern ofendopolygalacturonase towards pectin fragments: Comprehension and prediction., 2005, 1749(1): 53-64.
[43] Needham P G, Trumbly R J.characterization of the Mig1 repressor fromreveals evidence for monomeric and higher molecular weight forms., 2006, 23(16): 1151-1166.
[44] Qin S, Ji C, Li Y, Wang Z. Comparative transcriptomic analysis of race 1 and race 4 off. sp.induced with different carbon sources., 2017, 7(7): 2125-2138.
[45] Ptacek J, Devgan G, Michaud G, Zhu H, Zhu X, Fasolo J, Guo H, Jona G, Breitkreutz A, Sopko R, McCartney R R, Schmidt M C, Rachidi N, Lee S J, Mah A S, Meng L, Stark M J, Stern D F, De Virgilio C, Tyers M, Andrews B, Gerstein M, Schweitzer B, Predki P F, Snyder M. Global analysis of protein phosphorylation in yeast., 2005, 438(7068): 679-684.
[46] Sharifpoor S, Dyk D V, Costanzo M, Baryshnikova A, Friesen H, Douglas A C, Youn J Y, VanderSluis B, Myers C L, Papp B, Boone C, Andrews B J. Functional wiring of the yeast kinome revealed by global analysis of genetic network motifs., 2012, 22(4): 791-801.
[47] Zheng D W, Zhang S J, Zhou X Y, Wang C, Xiang P, Zheng Q, Xu J R. The FgHOG1 pathway regulates hyphal growth, stress responses, and plant infection in., 2012, 7(11): e49495.
[48] Wang C, Zhang S, Hou R, Zhao Z, Zheng Q, Xu Q, Zheng D, Wang G, Liu H, Gao X, Ma J W, Kistler H C, Kang Z, Xu J R. Functional analysis of the kinome of the wheat scab fungus., 2011, 7(12): e1002460.
[49] Jiao F, Kawakami A, Nakajima T. Effects of different carbon sources on trichothecene production and Tri gene expression byin liquid culture., 2008, 285(2): 212-219.
[50] Karunanithi S, Cullen P J. The filamentous growth MAPK pathway responds to glucose starvation through the Mig1/2 transcriptional repressors in., 2012, 192(3): 869-887.
[51] Jenczmionka N J, Maier F J, Losch A P, Schäfer W. Mating, conidiation and pathogenicity of, the main causal agent of the head-blight disease of wheat, are regulated by the MAP kinase gpmk1., 2003, 43(2): 87-95.
[52] Balciunas D, Ronne H. Yeast genes: multicopy suppressors of the Gal- phenotype ofcells., 1999, 262(4/5): 589-599.
[53] Jiang C, Zhang C, Wu C, Sun P, Hou R, Liu H, Wang C F, Xu J R.andplay different roles in the regulation of deoxynivalenol (DON) production by cAMP signalling in., 2016, 18(11): 3689-3701.
[54] Zaragoza O, Rodríguez C, Gancedo C. Isolation of thegene fromand effects of its disruption on catabolite repression., 2000, 182(2): 320-326.
[55] Cziferszky A, Seiboth B, Kubicek C P. The Snf1 kinase of the filamentous fungusphosphorylates regulation- relevant serine residues in the yeast carbon catabolite repressor Mig1 but not in the filamentous fungal counterpart Cre1., 2003, 40(2): 166-175.
(责任编辑 岳梅)
The Function of the carbon metabolism regulator FgCreA in
HOU Rui1, WANG ChenFang2
(1College of Forestry, Guizhou University, Guiyang 550025;2College of Plant Protection, Northwest A&F University, Yangling 712100, Shaanxi)
【Objective】The objective of this study is to knock out the FgCreA of, which is the carbon source metabolic regulation factor, to research the vegetative growth, sexual reproduction and pathogenicity of the knock out mutant, and to provide a basis for carbon source metabolic mechanism of. 【Method】 According to the budding yeast carbon source regulation factorMig1gene sequence on SGD and NCBI databases, the carbon source regulation factor FgCreA inwas determined. the sequences of FgCreA orthologs of other species were retrieved from NCBI database. multiple sequence alignment of CreA orthologs was performed with ClustalW2 software, and phylogenetic tree was constructed by MEGA5 software to determine their evolutionary relationships. The protein properties of FgCreA were predicted via InterProScan. The promotor regions of the carbon absorption structure genes and DON biosynthesis genes were obtained from the NCBI database, then the potentialbinding sites in these promotor regions were analyzed. Primer5 software was used to design primers, thegene knockout construct was generated by Split-PCR and then introduced into the wild type strain PH-1 by PEG-mediated protoplast transformation. PCR and Southern blot were used to confirm thegene deletion mutants (). The function ofwas analyzed according to the vegetative growth, sexual reproduction and pathogenicity of themutants. 【Result】There is only one carbon metabolism regulator gene(FGSG_09715) in. The 416-amino acid protein encoded byhas two conserved C2H2 zinc finger regions.FgCreA homologue proteins have a certain degree of homology and are highly conserved in fungi. The promoter regions of the carbon absorption structure genes (,,,,) and DON biosynthesis genes (,,,,,,,,,) containedDNA binding sites. Twogene knockout mutants were obtained and screened by PCR and Southern blot. The growth rate ofmutants was 90% lower than that of wild type. The conidia morphology of mutants was normal, but the conidiation decreased by 88% compared with the wild type. In the sexual reproduction stage,themutants could produce normal perithecia, ascus and ascospore, but it delayed 20-28 d longer than the wild type. Themutants were sensitive to salt stress, and were almost non-pathogenic on wheat.【Conclusion】The carbon metabolic regulator FgCreA plays important roles in vegetative growth and sexual reproduction, and is essential for plant infection in. However, whether FgCreA affects transcription of the carbon absorption structure genes and DON biosynthesis genes remains to be verified.
;; gene knockout; phenotype; carbon metabolism
2017-07-21;
2017-09-05
国家自然科学基金(31271989)、陕西省科技新星项目(2014KJXX-41)、贵州省科技支撑项目(黔科合支撑(2017)2567)、贵州省留学人员科技创新项目(黔人项目资助合同(2016)23号)、贵州大学引进人才项目(贵大人基合字(2015)65号)
侯瑞,E-mail:jiayouhourui123@163.com。
王晨芳,E-mail:wangchenfang@nwsuaf.edu.cn

