玉米扩展蛋白Expansin基因家族定位及基因表达模式分析
姜志磊, 周 琳, 武奉慈, 宋新元
吉林省农业科学院农业生物技术研究所, 吉林省农业生物技术重点实验室, 长春 130033
扩展蛋白(Expansin)是一类能够使植物细胞壁松弛的、非酶类的细胞壁活性蛋白,广泛存在于植物界,参与了植物生长发育过程中的许多环节,如根、茎、叶、花、果实、种子的生长发育等,同时还参与了抗旱、抗病、耐高温等诸多抗逆反应[1~3]。1992年,McQueen-Mason等[4]从黄瓜中纯化出2个能诱导热失活的细胞壁恢复扩张活性的特异性蛋白——扩展蛋白。随后,Shcherban等[5]发现禾本科花粉过敏原蛋白与Expansin具有蛋白质序列的同源性,Cosgrove等[6]经试验证实花粉过敏原蛋白具有使细胞壁松驰的活性。至今,研究人员已发现多种植物扩展蛋白[7~8]。扩展蛋白中与首次从黄瓜中发现的2个Expansin同源关系较为相近的蛋白称为 EXPA (Expansin A),与禾本科花粉过敏原蛋白同源关系较为相近的称为EXPB(Expansin B),另两个家族EXLA(Expansin-like A) 和EXLB(Expansin-like B)是在进行植物基因组分析时发现的,对其他非植物基因组分析时发现的Expansin称为EXLX(Expansin-like X),它们都同时具有Pollen_allerg_1(NCBI登陆号pfam01357)和DPBB_1(NCBI登陆号pfam03330)2个特征结构域[9]。
伴随着各种基因组测序计划的展开、分子结构测定技术的突破和互联网的普及,生物学数据库迅速地出现和成长,而有效地从这些数据库获取潜在的信息成为生物工作者亟待掌握的技能。本文利用PLAZA、NCBI、MaizeGDB、Uniprot、PLEXdb等基因组数据库对玉米扩展蛋白Expansin进行了生物信息学分析,以期为玉米扩展蛋白的深入研究奠定基础,为基因信息的获取方法相关研究提供参考。
1 材料与方法
1.1 玉米Expansin家族基因序列及染色体基因座位的获得
将已知的黄瓜、拟南芥、水稻等的Expansin基因序列在基因组比对网站plaza_v2_5[10](http://bioinformatics.psb.ugent.be/plaza/versions/plaza_v2_5/)进行同源比对,找到其所属的类群,再在这些类群中找到相应玉米基因;继而利用NCBI的保守结构域数据库(Conserved Domain Database,CDD)检测其是否具有Expansin的2个特征结构域Pollen_allerg_1(NCBI登陆号pfam01357)和DPBB_1(NCBI登陆号pfam03330)。获得的所有玉米Expansin基因通过比对maizeGDB[11]玉米B73基因组获得其相应的染色体基因座位信息。并进一步考察Expansin基因的串联重复情况,2个或2个以上Expansin基因在基因组中较短距离重复出现的区段被认为是Expansin基因的串联重复区段。
1.2 玉米Expansin家族系统进化树的构建
利用蛋白质数据库Uniprot 的在线工具Clustal Omega[12](http://www.uniprot.org/help/sequence-alignments)对玉米Expansin蛋白进行多序列比对分析,生成玉米Expansin家族的系统进化树。鉴于玉米中EXLA较少且未发现EXLB,生成进化树时加入了拟南芥(AtEXLA1、AtEXLB1)、水稻(OsEXLA1、OsEXLB1)相关基因,以便与其他物种的Expansin进化树进行比较。
1.3 玉米Expansin基因家族组织特异表达分析
利用基因表达数据库 PLEXdb[13](http://www.plexdb.org)的在线分析工具和其提供的芯片数据ZM37[14]进行玉米Expansin基因表达分析。芯片数据ZM37描述了玉米B73全生育期13类组织60个样品3次重复80 301个探针的表达数据。
2 结果与分析
2.1 玉米Expansin基因家族序列及染色体基因座位
将已知的黄瓜、拟南芥、水稻等的Expansin基因序列在基因组比较网站plaza_v2_5进行同源比对,所有的这些物种中的 Expansin基因都被包括到基因家族HOM000065类群中。基因家族HOM000065有22个物种的916个基因,其中玉米有93个。
利用NCBI的CDD对这93个蛋白质进行分析,其中,88个蛋白质同时具有Pollen_allerg_1和DPBB_1特征结构域。而ZM02G06330、ZM02G22010、ZM05G24920、ZM09G22180只有Pollen_allerg_1结构域,ZM09G28030只有DPBB_1结构域。虽然严格来讲,这5个蛋白质不属于Expansin家族,但鉴于其序列比对的相似性,在接下来的分析中将其保留。
通过比对maizeGDB玉米B73基因组获得的染色体基因座位信息表明,玉米Expansin基因分布于玉米10条染色体中的9条(图1)。其中,22个基因在1号染色体中,23个基因在5号染色体中,19个基因在9号染色体中,4号染色体中只有1个Expansin基因,而8号染色体中没有玉米Expansin基因。此外,19个Expansin基因的串联重复区段包含了60个Expansin基因。另一方面,通过比对NCBI和 Uniprot的蛋白质数据库,多数玉米Expansin具有250~300个氨基酸。
2001年Wu等[15]从玉米根的cDNA文库中克隆并测序了13个Expansin基因,包括5个EXPA(ZmEXPA1~ZmEXPA5)、8个EXPB(ZmEXPB1~ZmEXPB8)。2003年Li等[16]获得4个EXPB基因序列,其中1个是ZmEXPB1,另外3个被命名为ZmEXPB9、ZmEXPB10、ZmEXPB11。将这些基因序列与本研究所获得的93个玉米Expansin基因序列进行同源比对,结果发现Zm-EXPB2和Zm-EXPB8由于序列极为相近,被定位于9号染色体的同一位置ZM09G18160处,并且在NCBI数据库中这2个基因属于同一个EST聚类群(UniGene Zm67304);而Zm-EXPB1和Zm-EXPB9同上所述,被定位于9号染色体的同一位置ZM09G18450处,在NCBI数据库中也属于同一个EST聚类群(UniGene Zm94167),这2个基因是同一基因位点的选择性剪切而产生的不同的mRNA剪接异构体;其他已发现的Expansin基因的对应位置分别为Zm-EXPA1(ZM03G28280)、Zm-EXPA2(ZM05G40190)、Zm-EXPA3(ZM10G13870)、Zm-EXPA4(ZM01G57960)、Zm-EXPA5(ZM06G-26650)、Zm-EXPB3(ZM01G00380)、Zm-EXPB4(ZM07G07620)、Zm-EXPB5(ZM09G18170)、Zm-EXPB6(ZM01G21320)、Zm-EXPB7(ZM01G-21250)、Zm-EXPB10(ZM05G37630)、Zm-EXPB11(ZM09G28320)。
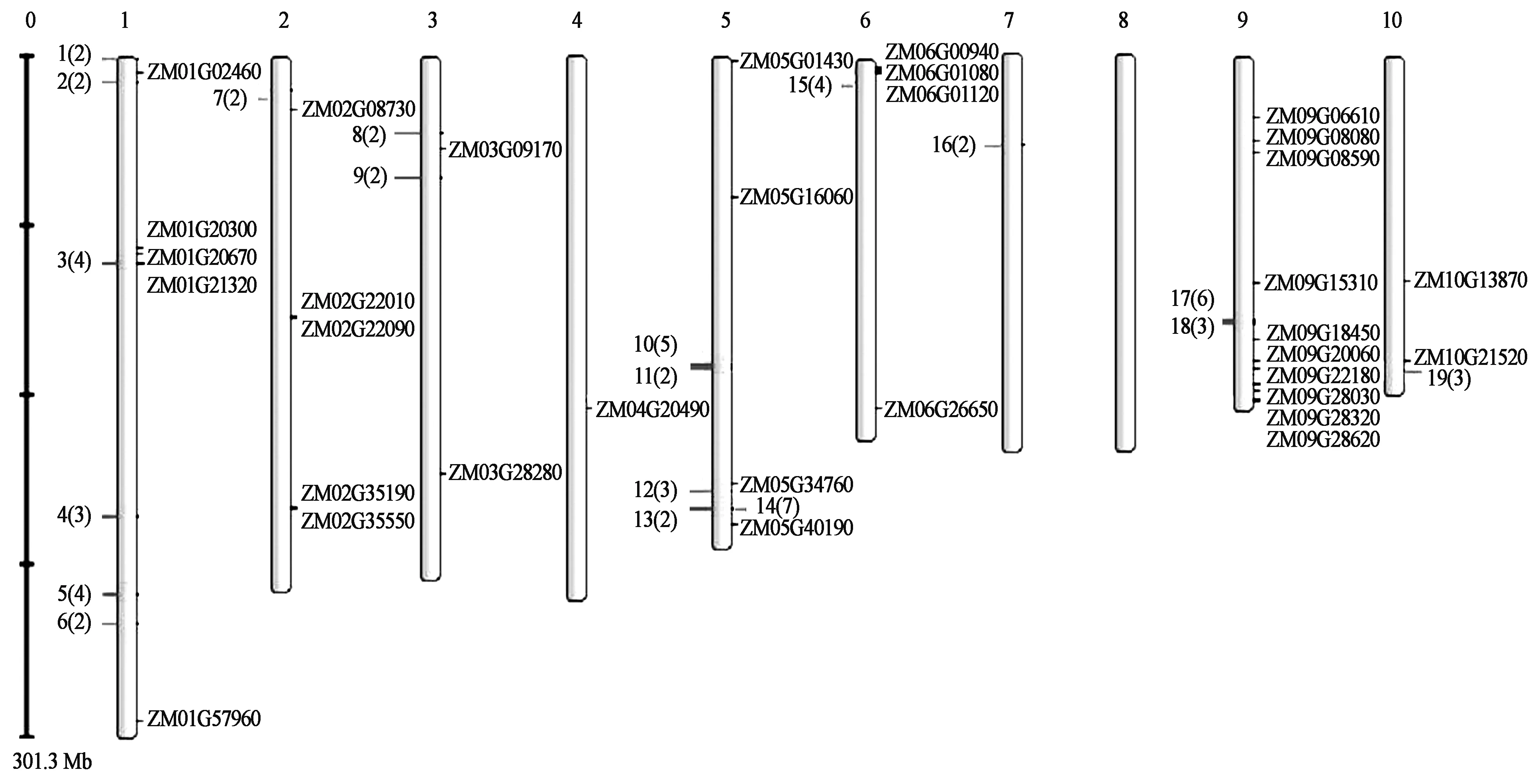
图1 玉米Expansin基因的染色体分布Fig.1 The genome location of Expansin gene in Zea mays.注:染色体上标注的1~19分别为Expansin基因的19个串联重复区段,括号中为这个区段的基因数目。具体基因编号为1(2):ZM01G00370、ZM01G00380;2(2):ZM01G04000、ZM01G04010;3(4):ZM01G21220、ZM01G21240、ZM01G21250、ZM01G21270;4(3):ZM01G35980、ZM01G36000、ZM01G36020;5(4):ZM01G43730、ZM01G43740 、ZM01G43750、ZM01G43770;6(2):ZM01G46530、ZM01G46540;7(2):ZM02G06320、ZM02G06330;8(2):ZM03G08060、ZM03G08080;9(2):ZM03G10740、ZM03G10750;10(5)ZM05G24900、ZM05G24920、ZM05G24930、ZM05G24940、ZM05G24950;11(2):ZM05G25120、ZM05G25140;12(3):ZM05G35620、ZM05G35630、ZM05G35640;13(2):ZM05G37580、ZM05G37590;14(7):ZM05G37620、ZM05G37630、ZM05G37640、ZM05G37650、ZM05G37660、ZM05G37670、ZM05G37680;15(4):ZM06G01240、ZM06G01250、ZM06G01260、ZM06G01270;16(2):ZM07G07610、ZM07G07620;17(6):ZM09G18160、ZM09G18170 、ZM09G18180、ZM09G18190、ZM09G18200、ZM09G18230;18(3):ZM09G18500、ZM09G18510、ZM09G18520;19(3):ZM10G22770、ZM10G22780、ZM10G22790。
2.2 玉米Expansin基因家族的系统进化与分类
对Expansin基因家族进行系统进化与聚类分析,结果发现,40个玉米Expansin基因属于家族EXPA,47个玉米Expansin基因属于家族EXPB(Expansin B),6个玉米Expansin基因属于家族EXLA,未发现玉米具有EXLB基因(图2)。从进化树中可以看出,玉米EXPB与EXLA、EXLB序列同源性较高, EXPA在聚类图的最外围。
玉米串联重复区段的基因具有较强的同源性,聚类分析中往往聚集在相同类群,例如第10区段和第11区段在玉米5号染色体ZM05G24900到ZM05G25140之间就有7个EXPA基因,并在聚类分析被划分在同一类群;在第13区段和第14区段ZM05G37580到ZM05G37680之间集中了9个EXPB基因;第15区段ZM06G01240、ZM06G01250、ZM06G01260、ZM06G01270这4个EXPA基因在6号染色体串联重复(图2)。
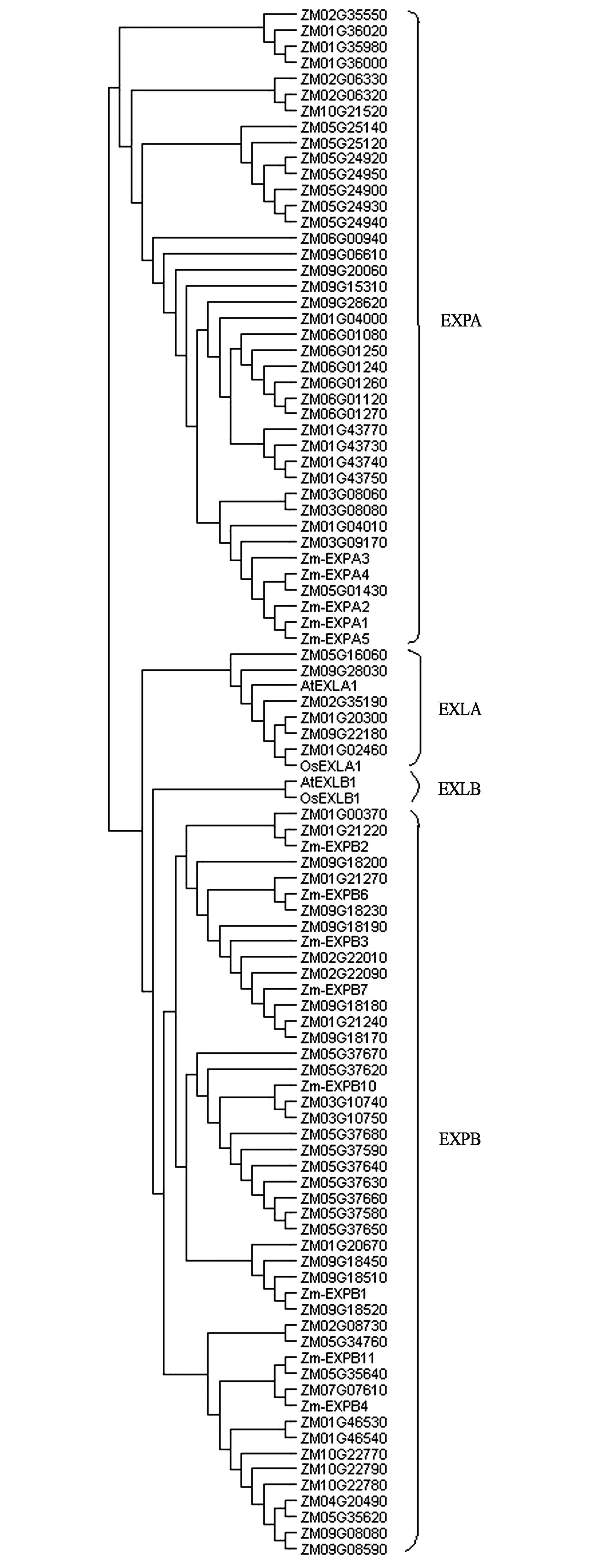
图2 玉米Expansin家族的蛋白进化树Fig.2 The phylogenetic tree for Zea mays Expansin protein.注:93个玉米Expansin和拟南芥(AtEXLA1、AtEXLB1)、水稻(OsEXLA1、OsEXLB1)。
2.3 玉米Expansin基因家族组织表达分析
利用PLEXdb分析工具对玉米Expansin基因表达进行分析。结果表明,不同基因通过PLEXdb分析得到了相同基因芯片ZM37的结果,这是由于Expansin基因的同源性较强,芯片探针不能区分。尽管芯片ZM37含有 13类组织60个样品的表达数据,但是还有8个Expansin基因未检测到表达数据,即其最大值低于芯片检测信号的背景噪音值200[14],这些基因有可能在逆境或胁迫诱导下才能得以表达。
Expansin基因的表达有很强的相关性,同源序列聚类单元中基因的表达模式非常相似,而在不同聚类单元中的某些基因之间的表达模式也非常相近。例如,ZM02G06320、ZM10G21520、ZM02G06330、ZM09G18160在60个样品的平均表达量差异很大,分别为1 401、3 526、6 830、15 801,但它们在60个样品表达比例(波形)极为相似(图3,彩图见封三图版),而在聚类分析中ZM02G06320、ZM10G21520、ZM02G06330聚类在EXPA中的1个小分类单元中,ZM09G18160则属于EXPB基因(图2)。
根据Sekhon等[14]的研究,将不同组织表达量的变异系数CV>0.15的基因归为组织特异性表达基因,玉米Expansin基因有43个基因的表达具有组织特异性。花药中特异性表达的基因最多,有16个EXPB、4个EXPA,并在各自内部聚类关系中非常相近(表1,图2),这些基因在花药中的表达量较高,在其他组织中表达量较低。还有2个EXPA在根中特异表达,2个EXPA在苞叶中特异表达,1个EXLA和2个EXPB在胚芽中特异表达,2个EXPA基因和1个EXPB在胚芽和种子中特异表达,2个EXPB在种子和穗轴中特异表达,其他12个基因在2种或3种组织中特异表达(表1)。
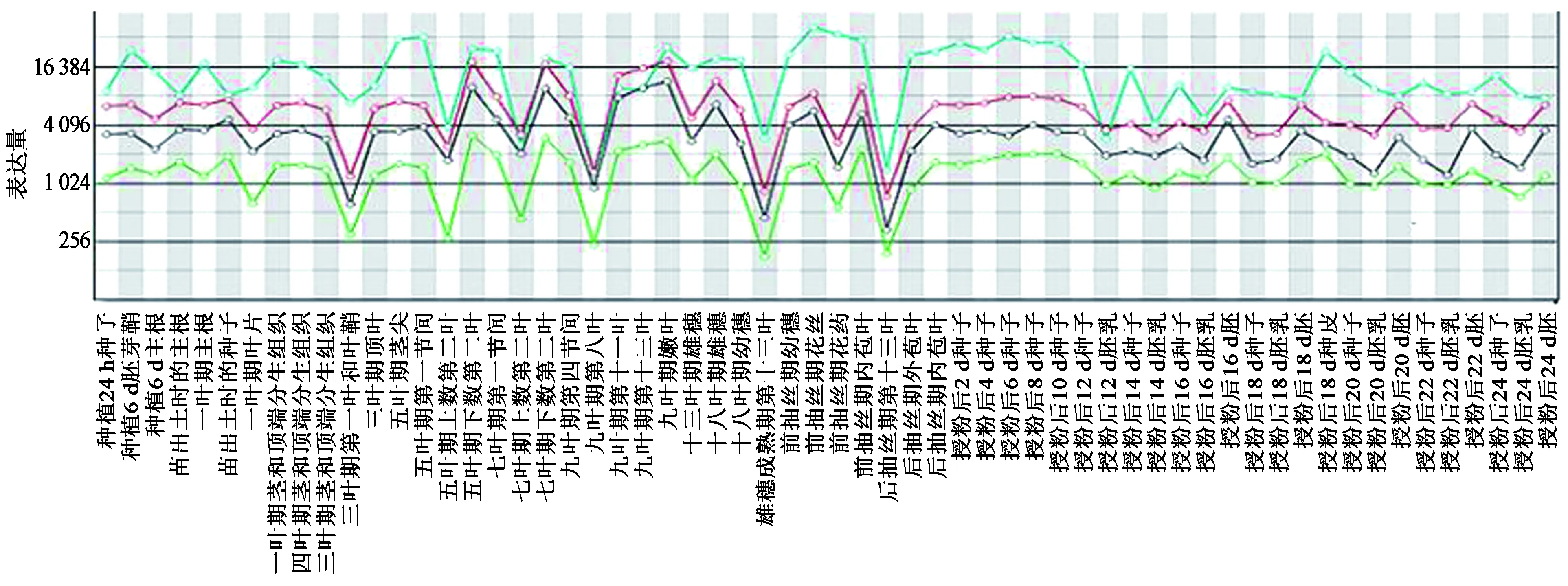
图3 玉米Expansin 基因ZM09G18160(蓝色)、ZM02G06320(红色)、ZM10G21520(绿色)与ZM02G06330(黑色)的表达模式Fig.3 The expression patterns of Expansin gene ZM09G18160(blue),ZM02G06320(red),ZM10G21520(green) and ZM02G06330(black).(彩图见封三图版)
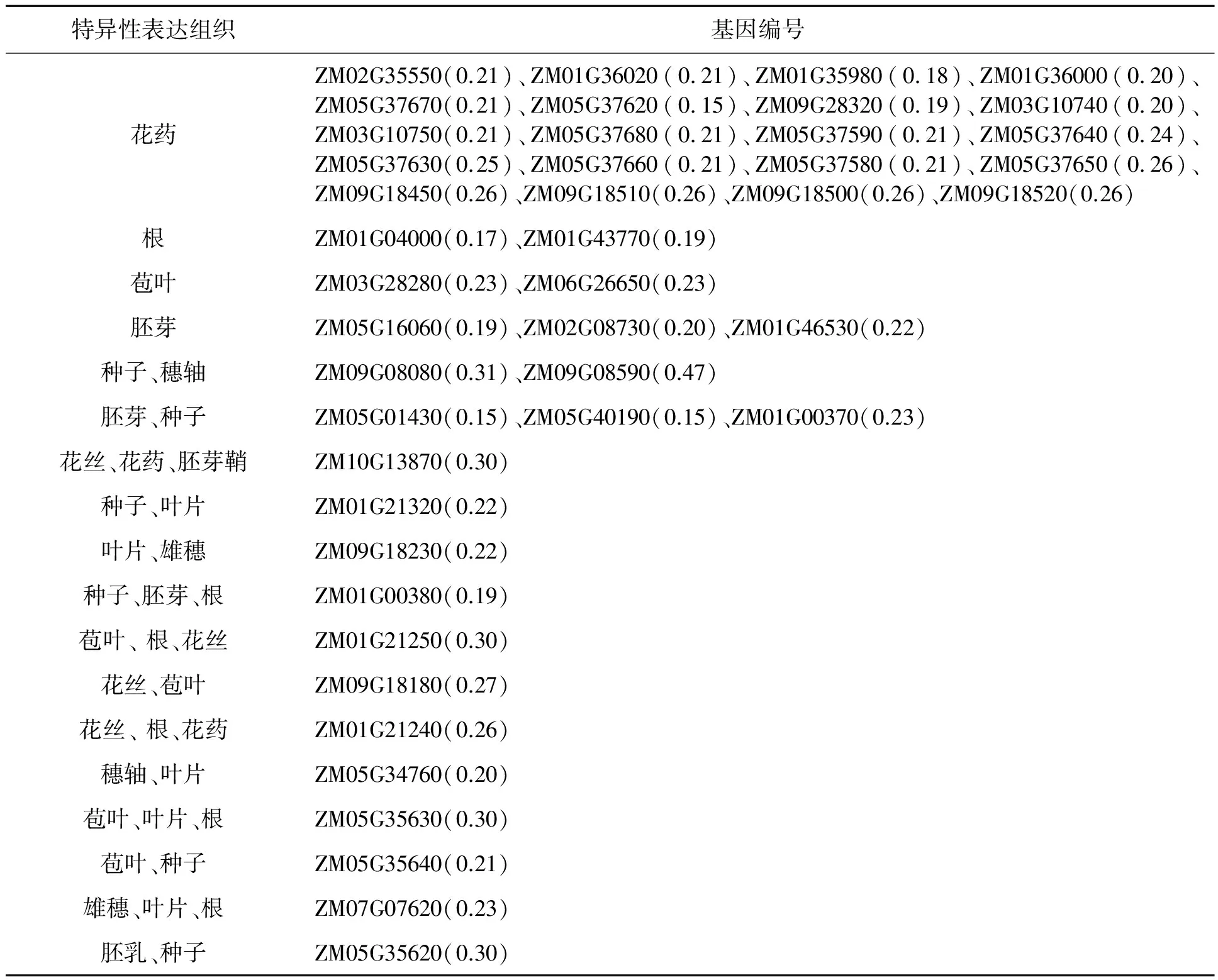
特异性表达组织基因编号花药ZM02G35550(0.21)、ZM01G36020(0.21)、ZM01G35980(0.18)、ZM01G36000(0.20)、ZM05G37670(0.21)、ZM05G37620(0.15)、ZM09G28320(0.19)、ZM03G10740(0.20)、ZM03G10750(0.21)、ZM05G37680(0.21)、ZM05G37590(0.21)、ZM05G37640(0.24)、ZM05G37630(0.25)、ZM05G37660(0.21)、ZM05G37580(0.21)、ZM05G37650(0.26)、ZM09G18450(0.26)、ZM09G18510(0.26)、ZM09G18500(0.26)、ZM09G18520(0.26)根ZM01G04000(0.17)、ZM01G43770(0.19)苞叶ZM03G28280(0.23)、ZM06G26650(0.23)胚芽ZM05G16060(0.19)、ZM02G08730(0.20)、ZM01G46530(0.22)种子、穗轴ZM09G08080(0.31)、ZM09G08590(0.47)胚芽、种子ZM05G01430(0.15)、ZM05G40190(0.15)、ZM01G00370(0.23)花丝、花药、胚芽鞘ZM10G13870(0.30)种子、叶片ZM01G21320(0.22)叶片、雄穗ZM09G18230(0.22)种子、胚芽、根ZM01G00380(0.19)苞叶、根、花丝ZM01G21250(0.30)花丝、苞叶ZM09G18180(0.27)花丝、根、花药ZM01G21240(0.26)穗轴、叶片ZM05G34760(0.20)苞叶、叶片、根ZM05G35630(0.30)苞叶、种子ZM05G35640(0.21)雄穗、叶片、根ZM07G07620(0.23)胚乳、种子ZM05G35620(0.30)
注:括号中数据为此基因在不同组织表达量的变异系数CV。
3 讨论
Expansin基因家族非常庞大,本研究鉴定获得了93个玉米Expansin基因。ZM02G06330、ZM02G22010、ZM05G24920、ZM09G22180只有Pollen_allerg_1结构域,ZM09G28030只有DPBB_1结构域。其余88个基因与 Zhang等[17]利用玉米基因组数据鉴定的88个玉米Expansin基因并非完全相同,本研究鉴定的4个基因(ZM05G-16060、ZM05G24930、ZM05G24940、ZM05G37670)未被其所用方法在基因组中发现,而且其鉴定的基因中有4个是因基因组位点的可变剪切而产生的不同的mRNA剪接异构体。
植物Ⅰ类花粉过敏源被命名为EXPB基因,其通过松弛柱头部位的细胞壁经花粉管进入子房内部[5~6]。本研究发现玉米有16个EXPB基因在花药中特异表达,可能行使相类似的基因功能,但还发现4个EXPA也在花药中特异表达,EXPA在授粉过程中的作用有待进一步研究。
玉米EXPB与EXLA、EXLB序列同源性较高, EXPA在聚类图的最外围,玉米与拟南芥[18]、水稻[18]、葡萄[19]等物种的Expansin序列聚类分析的结果显示其具有很强的相似性,表明Expansin基因非常古老,EXPA、EXPB、EXLA、EXLB在这些物种分化前已经完成各自的分化。玉米Expansin基因数目明显多于拟南芥的36个和水稻的58个[18],这不仅是由于玉米基因组进化过程中经过了多次的全基因组复制[20],而且玉米基因组的19个Expansin基因的串联重复区段包含了60个基因,而拟南芥、水稻分别只有3个和10个Expansin基因的串联重复区段。
Wu等[21]在模拟的干旱胁迫条件下对玉米根的生长研究表明,Zm-EXPA1(ZM03G28280)、Zm-EXPA5(ZM06G26650)、Zm-EXPB8(ZM09G-18160)基因表达在根尖分生区快速升高,进而促进根的伸长,以达到充分利用水资源的目的。然而本研究的基因表达分析表明,Zm-EXPA1(ZM03G28280)、Zm-EXPA5(ZM06G26650)在根中的平均表达量分别为889和386,苞叶中的平均表达量分别为12 538和3 908,苞叶中的表达量远远高于根中的表达量,从统计学上认为这2个基因在苞叶中特异表达(CV=0.23)。Zm-EXPB8(ZM09G18160)在所有组织中的平均表达量为15 801,而特异性非常小(CV=0.08),说明ZM09G18160是一个在多种组织高表达的基因。本研究得到2个基因(ZM01G04000和ZM01G43770)在根中特异表达,其在根中的平均表达量分别为936和1 178,在其他组织中的平均表达量只有108和105(大多数组织的表达量低于芯片检测信号的背景噪音值200,可能根本不表达)。综上所述,在对单个组织的单个基因进行分析时,往往会得到错误或片面的结果,只有综合分析各类信息才能得到全面且正确的结果。
Yan等[22]研究发现AtEXP2参与了拟南芥种子的萌发,赤霉素(gibberellin,GA)可以促进AtEXP2在种子萌发过程中的表达,从而提高拟南芥种子萌发对盐胁迫和渗透胁迫的耐受性。我们发现在玉米胚芽和种子中特异表达的ZM05G01430、ZM05G40190与AtEXP2有较高的序列同源性,从而推测ZM05G01430、ZM05G40190可能与拟南芥AtEXP2具有相似的功能。此项推论有待进一步的实验验证。
本研究鉴定得到多个Expansin基因在多种组织特异表达,大多数基因还未进行过具体研究。EXLA和EXLB与EXPA和EXPB具有一定的同源性,但尚未有EXLA和EXLB基因在相关物种中功能的研究发现。本研究发现EXLA基因ZM05G16060在胚芽中特异表达,这一结果可能为EXLA基因的功能研究找到突破口。
Expansin蛋白同时具有2个高度保守的同源结构域,所有Expansin基因来源于一个共同的祖先基因[17]。Li等[1]推测首先是EXPA/EXPB与EXLA的分离,随后EXPA与EXPB分离,而EXLB是从EXPB的一个亚组起源的。Expansin蛋白结构的保守性决定了蛋白生化性质的单一性,EXPA 和EXPB的功能都要通过其对细胞壁的松弛作用而体现,但每种植物中都存在大量的Expansin基因,甚至同种植物的同一组织中有多个Expansin表达,其协同作用的机制、存在的意义以及必要性等问题都需要深入的研究。随着植物基因组测序的展开、各种数据库的建立,大量扩展蛋白基因被鉴定获得,为我们对其基因家族的演化、表达的时空及组织特异性、不同组织不同功能的关联和协调以及表达调控研究提供了便利。
Expansin蛋白具有使植物细胞壁松弛的作用,在细胞生长中具有重要功能,几乎所有组织都有Expansin基因表达,对其序列、功能及作用模式的深入研究,可以为其应用提供理论依据。比如通过控制Expansin的表达来调控植物的生长发育进而提高作物产量、延长贮藏时间。通过Expansin控制细胞壁的生长或修饰这些植物细胞壁的结构和物理特性,从而改变作物的品质。
本研究鉴定获得了93个玉米Expansin基因,对各种数据库的基因编号进行了相互对照,获得了Expansin的核酸、蛋白质序列和其在染色体中的位置,深入且系统地分析了其分类与进化、组织的特异性表达,为玉米扩展蛋白的深入研究奠定了基础。
本研究所有的数据获得、数据分析、结果和图片制作均由一台连接互联网并安装有Windows操作系统的普通计算机完成,无需专业的编程和生物信息学知识,此方法可以为非生物信息学专业的生物工作者对基因信息的获取提供参考。
[1] Li Y, Jones L, McQueen-Mason S. Expansins and cell growth[J]. Curr. Opin. Plant Biol., 2003, 6(6):603-610.
[2] 赵美荣,李永春,刘 辉,等. 抗旱性不同的小麦扩展蛋白活性及基因表达分析[J].江苏农业科学, 2016, 44(1):108-110.
[3] 王子剑,孙 丹,于 颖,等. 白桦扩展蛋白基因的克隆分析及植物表达载体构建[J].西南林业大学学报, 2016, 36(1):1-8.
[4] McQueen-Mason S, Durachko D M, Cosgrove D J. Two endogenous proteins that induce cell wall expansion in plants[J]. Plant Cell, 1992, 4(11):1425-1433.
[5] Shcherban T Y, Shi J, Durachko D M,etal.. Molecular cloning and sequence analysis of Expansins—a highly conserved, multigene family of proteins that mediate cell wall extension in plants[J]. Proc. Natl. Acad. Sci. USA, 1995, 92(20):9245-9249.
[6] Cosgrove D J, Bedinger P, Durachko D M. Group I allergens of grass pollen as cell wall-loosening agents[J]. Proc. Natl. Acad. Sci. USA, 1997, 94(12):6559-6564.
[7] 赵美荣,李永春,王 玮. 扩展蛋白与植物抗逆性关系研究进展[J].植物生理学报, 2012, 48(7):637-642.
[8] 张 安,曹清河,周志林,等. 植物细胞壁松弛因子——Expansin研究进展[J]. 江苏农业科学, 2013, 41(6):11-13.
[9] Kende H, Bradford K, Brummell D,etal.. Nomenclature for members of the Expansin superfamily of genes and proteins[J]. Plant Mol. Biol., 2004, 55(3):311-314.
[10] Michiel V B, Sebastian P, Elisabeth W,etal.. Dissecting plant genomes with the PLAZA comparative genomics platform[J].Plant Physiol., 2012, 158(2):590-600.
[11] Schaeffer M L, Harper L C, Gardiner J M,etal.. MaizeGDB: Curation and outreach go hand-in-hand[J]. Database, 2011, doi:10.1093/database/bar022.
[12] Sievers F, Wilm A, Dineen D G,etal.. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega[J]. Mol. Syst. Biol., 2011, 7(1):1429-1432.
[13] Dash S, Van Hemert J, Hong L,etal.. PLEXdb: Gene resources for plants and plant pathogens[J]. Nucl. Acids Res., 2012, 40(D1):D1194-D1201.
[14] Sekhon R S, Lin H, Childs K L,etal.. Genome-wide atlas of transcription through maize development[J]. Plant J., 2011, 66(4):553-563.
[15] Wu Y, Meeley R B, Cosgrove D J. Analysis and expression of the α-expansin and β-expansin gene families in maize[J]. Plant Physiol., 2001, 126(1):222-232.
[16] Li L C, Bedinger P A, Volk C,etal.. Purification and characterization of four β-expansins (Zeam 1 isoforms) from maize pollen[J]. Plant Physiol., 2003, 132(4):2073-2085.
[17] Zhang W, Yan H W, Chen W J,etal.. Genome-wide identification and characterization of maize expansin genes expressed in endosperm[J]. Mol. Genet. Genomics, 2014, 289(6):1061-1074.
[18] Sampedro J, Lee Y, Carey R E,etal.. Use of genomic history to improve phylogeny and understanding of births and deaths in a gene family[J]. Plant J., 2005, 44(3):409-419.
[19] Dal Santo S, Vannozzi A, Tornielli G B,etal.. Genome-wide analysis of the expansin gene superfamily reveals grapevine-specific structural and functional characteristics[J]. PLoS ONE, 2013, 8(4):e62206.
[20] Schnable P S, Ware D, Fulton R S,etal.. The B73 maize genome: Complexity, diversity, and dynamics[J]. Science, 2009, 326(5956):1112-1115.
[21] Wu Y, Thome E T, Sharp R E,etal.. Modification of expansin transcript levels in the maize primary root at low water potentials[J]. Plant Physiol., 2001, 126(4):1471-1479.
[22] Yan A, Wu M J, Yan L M,etal..AtEXP2 is involved in seed germination and abiotic stress response inArabidopsis[J]. PLoS ONE, 2014, 9(1): e85208.

