重离子的辐射生物效应及其在生命科学中的应用
贾蓉 苏锋涛 胡步荣
(1. 中国科学院重离子辐射生物医学重点实验室 甘肃省空间辐射生物学重点实验室 中国科学院近代物理研究所,兰州 730000;2. 复旦大学附属肿瘤医院肿瘤研究所,上海 200032;3. 中国科学院大学,北京 100049)
重离子是指重于元素周期表中2号元素氦并被电离的粒子。线性能量传递(Linear energy transfer,LET),又称为传能线密度,是描写射线性质的一种物理量,单位为J/m,常用keV/μm表示,指的是电离粒子在其单位长度径迹上消耗的平均能量,即电离辐射贯穿物质时,因碰撞而发生的能量转移。重离子属于高LET类型的粒子,这种粒子在其穿透的路径上,产生很强的局部电离,与传统的光子辐射(如X、γ射线)相比,会诱导更严重的辐射损伤生物效应。外太空中存在着高能量的质子和重离子,它们对生命体细胞会产生严重的电离作用,导致细胞死亡、染色体畸变和癌症发生,影响着外太空航天员的健康安全[1]。在近层宇宙空间(即距离地球100 km到几百公里的空间)中,由于地球磁场的捕获作用(Van Allen Belt,范艾伦辐射带),这些高能的质子与重离子被屏蔽,因此,生活在地面的人们,处在一个相对安全的环境[2]。随着核科学研究的需要和核技术的发展,人们在地面建立了重离子加速器装置,这种装置的建立,不仅大大促进了核科学的研究与相应技术的发展,也促进了核技术在生命科学基础与应用领域的研究,更好的理解DNA团簇损伤与修复机制及重离子在生命科学中的应用,能为未来研究方向提供方便,促进对重离子辐射危害的评估与防护策略的建立,以及其得到更好的应用。
1 高能重离子诱导的DNA损伤
1.1 高能重离子诱导DNA团簇损伤
电离辐射通过射线的直接作用和间接作用引起DNA分子发生多种类型损伤,包括如图1所示的碱基位点损伤(Apurinic/apyrimidinic sites,APs),损伤碱基(Oxidized purines or pyrimidines),单链断裂(Single-strand breaks,SSBs)或双链断裂(Double-strand breaks,DSBs)[3]。DSBs损伤是辐射引起的各种生物效应中最重要的原初损伤之一,无论是光子(如X或γ射线)还是带电粒子诱导的未修复的或错误修复的DSBs,均会导致细胞毒性、突变和辐射致癌效应[4-5]。一系列的证据揭示高LET辐射诱导细胞在DNA局部一到两个螺旋(约10 nm)之间出现2个或更多个损伤(单一或复合类型损伤)[6],这种密集的损伤类型被称之为DNA团簇损伤(又称之为复杂的或簇集DNA损伤,Clustered DNA damage,图2)。DNA团簇损伤比单个独立的DSBs更难以修复,有的情况下更是不可修复[7-15],这是高LET辐射比低LET辐射有更高的相对生物学效应(如细胞杀伤、染色体畸变、突变和致癌形成)的一个主要原因[16-18]。利用识别DNA损伤不同类型的抗体,不同荧光素结合的二抗,进行免疫荧光染色,激光共聚焦显微镜细胞三维成像,可直观地观察到DNA团簇损伤的不同类型(hOGG1-碱基损伤,XRCC1-单链损伤,53BP1-双链损伤;如图3所示)高度共定位。这些团簇损伤复杂、修复困难,致使细胞周期停止时间长,导致更多的染色体发生畸变(图4)[19]。软琼脂囊球实验表明DNA团簇损伤将会引发更多的细胞发生转化事件[20]。但为什么DNA团簇损伤难以修复以及这些损伤激发的癌症形成事件的机制还不十分清楚,还需要继续深入研究。
David-Cordonnier等的研究显示,在DNA团簇损伤区域内,当有第二个AP位点或单链断裂存在时,第一个AP位点的切开以便于修复的效率显著地降低[23]。一系列研究也显示,不论是利用哺乳动物细胞提取物还是纯化的修复酶,对DNA团簇损伤中AP位点或8-氧鸟嘌呤位点(8-oxoguanine,8-OxoG)的修复,都不起作用[23-25]。在大肠杆菌和哺乳动物细胞中,一些DNA团簇损伤位点的单个损伤类型在试图进行修复时,也可能导致致命的DSBs[3,26-30]。DNA团簇损伤中不同损伤类型的空间分布和在细胞核内(常染色质或异染色质上)损伤的实际位置可能影响细胞DNA团簇损伤的修复能力。用分子动力学模拟的方法,已经能检验带有一个碱基或8-氧鸟嘌呤的DNA团簇损伤分子的构象变化及其能荷特性,其结果显示,含有团簇损伤的DNA分子具有特殊的性能特点:在损伤位点能剧烈地弯曲,8-氧鸟嘌呤和互补链的碱基之间的静电相互作用能削弱或彻底失去[31]。这些构象可能使得修复酶与损伤区域的结合变得困难,也可能由于DNA团簇损伤中多种损伤的致密空间分布使修复酶不能结合于某种损伤类型位点,从而导致修复能力的下降[13]。研究显示,非双链断裂的簇集损伤类型(如单链断裂、碱基损伤),如果没有修复,也会导致基因突变和染色体异常[32]。

图1 电离辐射诱导的DNA损伤类型
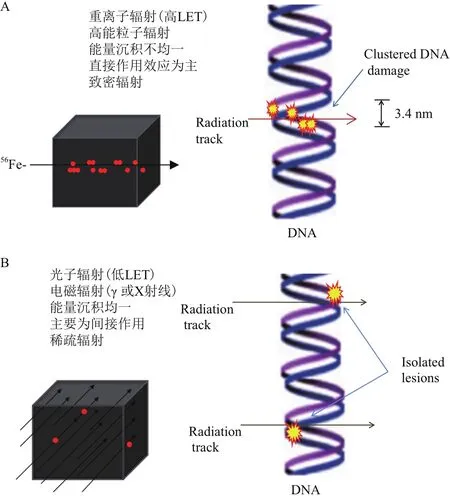
图2 电离辐射诱导的DNA损伤

图3 低LET与高LET辐射诱导的DNA损伤类型与差异

图4 低LET与高LET辐射诱导的染色体畸变类型与差异
1.2 重离子诱导的DNA团簇损伤修复途径
重离子诱导的DNA团簇损伤包括两个基本组成类型:DSBs和非DSBs。DSBs的正确修复对细胞生存和基因组完整性至关重要[33]。在哺乳动物细胞,DSBs主要由两个明确的途径被修复:非同源末端连接修复(Non-homologous end-joining repair,NHEJ)和同源重组修复(Homologous recombination repair,HR)[34](图 5)。这两条途径有着不同的底物需求,按不同的修复动力学进行,并且在细胞周期的不同时期被区别使用[35]。HR和NHEJ被看成是相互竞争的通路,但是最新的证据表明,这两条途径的结合可以增强所有DNA的修复,并且保护基因组的完整性[33,36-37]。在HR修复途径,断裂的基因组以另一条完整的姐妹染色单体作为模板来填补缺口,会出现基因转换[34],因此这一途径被认为是无错修复途径。相比于HR,NHEJ很少利用同源的模板来延伸DNA断端[38],在只有一个染色体拷贝的情况下也能正常进行修复。与高保真的HR修复相反,NHEJ可以是高保真的,也可以带来大量的突变。NHEJ是G1与早S期细胞首选的DSBs修复途径,而对于晚S期与G2期的DSBs修复,HR与NHEJ均有贡献[39]。NHEJ途径缺陷型哺乳动物细胞对X和γ射线极为敏感,DSBs大量积聚[40],表明NHEJ在低LET辐射诱导的DSBs 修复中占支配地位。对α-粒子和高能重离子辐射来说,NHEJ途径缺陷型哺乳动物细胞的相对敏感性程度低于X射线,表明DNA修复的NHEJ途径在高LET辐射诱导的DNA团簇损伤修复中作用受限[7,14-15,40]。研究认为,高LET射线作用于细胞后,在DNA分子中产生一定密度的不均匀能量沉积,其结果是在局部范围内形成的DNA损伤不止一种,而是几种类型损伤的簇集,这种局部多重损伤位点(Multiple damage sites,MDS)及断裂的小片段DNA碎片影响Ku70/80修复蛋白结合到DNA断端上(NHEJ的启动步骤),导致由NHEJ介导的DSBs修复的抑制[41-42],限制了NHEJ途径的进程。

图5 DNA双键断裂修复的两种途径:HR和NHEJ

图6 不同修复通路缺陷性细胞系对不同射线、离子敏感度分析[51]

表1 辐射诱导的染色单体及染色体畸变
研究显示,在整个细胞周期过程中,NHEJ和HR修复途径对X和γ射线诱导的DSBs修复都是需要的,HR与NHEJ在低LET辐射诱导的DSBs修复中相互协作[44-49]。有几篇报道揭示HR修复因子,包括Rad54和Rad51同系物(Rad51D和XRCC3)缺陷型哺乳动物细胞系对高能重离子辐射敏感,出现较高的细胞毒性和遗传突变性(图6)[7,50-51]。进一步,HR缺陷型细胞在S期时表现更为敏感(此时HR是DSB修复的主要途径),这一结果强烈暗示HR在复杂DSBs修复中作用关键。
对于低LET辐射诱发的DSBs而言,NHEJ是主要的修复方式[52],但是能够精确地处理DNA团簇损伤的DSB修复途径还不清楚。有报道指出,在处理HZE粒子诱导的DNA损伤中需要RAD51介导的DNA修复(HR)[53-54]。另有研究显示,范可尼贫血症(Fanconi anemia,FA)蛋白通路可能与HR因子协调作用,并且在高LET的Fe离子辐射诱发的DNA团簇损伤修复中起着非常重要的作用[55]。而且,涉及到多种DNA修复过程的Mre11、WRN和Artemis核酸酶,在由重离子诱导的DNA复杂损伤末端的修复过程中可能均有重要作用[56-58]。尽管Mre11和WRN在响应离子辐射时能结合在DNA损伤位点,但在修复通路选择的机制,与其在响应DNA团簇损伤过程中蛋白质的精确作用这些方面,现在依然有很多不清楚的地方。这就要求将来能确定涉及DNA团簇损伤过程中的多蛋白复合体。
2 重离子辐射在生命科学中的应用
电离辐射能够诱导生命体细胞的DNA损伤,这种损伤能够正确修复的,将幸存下来;无法修复的,细胞死亡或凋亡;不能正确修复的,引起突变,侥幸存活,成为突变株。为了在较短时间内培育出优良高性能突变品种,X或γ射线已经成为开展辐射诱变育种的常规手段,但仍存在突变率低,所产生的变异绝大多数不能稳定遗传等问题。重离子具有高LET,接触受体样品后首先会引起细胞表面穿孔的物理过程[59],甚至可直接穿透植物或微生物,在此过程中伴随着能量的局部沉积,对生物体作用区域更集中,对作用部位细胞的损伤比X或γ射线显著增加,而且集中受损的区域可随离子能级控制,因此,重离子束用于诱变育种,能诱发基因组当代可遗传变异,并具有较高的突变率,在生物品种改良方面具有突出优势,在微生物、植物等生物品种选育中已经取得了良好的效益[59-63]。
低LET的X或γ射线因其有效的细胞杀伤作用,已普遍应用于临床实体肿瘤的治疗。然而,这其中也存在一些问题,如剂量主要损失在人体浅表部位、适型性不好,对肿瘤周围健康组织损伤较大,对乏氧的肿瘤细胞抗拒、治疗周期长、缺乏在线实时观察手段等缺点。高LET的高能重离子诱导的DNA损伤很难修复,因此在杀死肿瘤细胞中是更有效的。更为重要的是,带电粒子的物理特征允许其靶向肿瘤,传递更高剂量和能量的粒子,减少对周围正常组织的损伤。如高LET的碳离子束,已经应用于几个国家的放射治疗中,取得了很好的治疗效果[63]。
2.1 重离子辐射在植物育种中的应用
重离子辐照作为诱变育种的一种新方法,已经被应用到作物育种和模式植物功能基因的研究中。早在1986年,我国余增亮研究员等就发现了重离子注入水稻的诱变效应[64]。先后成功选育出抗病虫S9042、优质D9055 和早籼14、高产中粳63、晚粳M3122、高产早熟晚粳48和晚粳M1148等不同特点的水稻品种[65]。近年来,中国科学院近代物理研究所用重离子辐照植物获得了多个有利突变体。如在小麦中选育出了矮秆、抗逆、高产、优质的春小麦M-920和丰产、高蛋白含量、抗黑穗病的陇辐2号[66-67]。在能源植物方面选育出了丰产、早熟、抗逆、含糖量高的甜高粱突变体“近甜1号”,为我国甜高粱乙醇燃料产业提供新思路[68]。在花卉方面通过重离子辐照选育出了叶片细胞色素定位发生变化的白花紫露草突变体“冬花夏草”[69],并且通过RAPD技术分析,选育出较母本株DNA片段新增的深红色和鲜红色花朵的一串红突变体[70]与花色发生变化的天竺葵突变体[71]。还选育出了花盘增大的万寿菊突变体,花色和花叶都改变的鸡冠花突变体,矮杆、花色变异、花期提前的大丽花芽突变体,并且得到“新兴红”和“新兴白”两个新品系[72]。通过重离子辐照,百脉根形态学突变频率约5.14%,但其DNA多态率仅0.567%,其具体机制还有待研究[73]。
国外重离子辐照植物诱变育种及其机理方面也有很多研究成果,如在水稻中选育出了一个DNA片段缺失的叶绿素缺失突变体CDM[74],OsSPY转录水平明显增加的半矮水稻突变体PKOS1、HyKOS1,14-3-3表达量增加最多的细长型水稻突变体TKOS4[75];在拟南芥中选育出了种皮呈现紫红色斑点的与花青素合成有关的突变体ast[76]。在花卉方面选育出了花色发生变化的菊花突变体[77]。并且通过对小麦的重离子辐照,发现了可能与选择性非同源末端连接相关的两种DNA修复基因,这对重离子辐照获得小麦突变体的机制研究有很大贡献[78]。
2.2 重离子辐射在微生物育种中的应用
近年来重离子在辐照药用微生物诱变育种方面也广受研究者的关注,尤其是我国在这方面有很多突出贡献。微生物优良菌种的选育在微生物药物的研究开发中很关键,利用不同吸收剂量的12C6+离子束辐照斜盖菇属真菌原生质体,选育出了截短侧耳素高产变异株C.pin15I5E和C.pin15II6B,其产量较出发菌株分别提高16.17%和15.47%[79]。重离子诱变处理庆大霉素生产菌绛红色小单孢菌与阿维菌素生产菌阿维链霉菌等,其代谢产物产量也有一定提高[80-81]。并且得到了土曲霉突变株 Z15-7[82],还能通过负调控突变菌株基因缺失导致代谢过程中蛋白表达上调来提高阿维菌素B1a的生产,而且能够稳定遗传[83]。在食品工业方面,重离子辐照也同样获得了高转化率的发酵新菌种,并使之产业化。如重离子辐照黑曲霉、酿酒酵母等,已筛选获得高产稳定菌株[84-85]。利用重离子辐照黑曲霉菌,胡伟等最终成功选育出一株柠檬酸生产菌高产突变菌株H4002,此菌株产柠檬酸能力,是目前国际上的最好水平,在生产中大大提高了经济效益[85-86]。重离子束辐照处理3株乳酸菌菌株,得到了乳酸产量比原菌株显著提高的突变菌株,且遗传稳定性良好[87]。而且还通过辐照筛选出了高产β-葡聚糖的面包酵母突变菌株,经过实验室发酵条件优化,产量较出发菌株YS-00可以提高1.73倍[88]。在微生物能源方面,利用重离子束辐照诱变得到了PHB 高产株菌G15[89],产油微藻突变株 D90G-19[90],两株生长速率明显加快的微拟球藻突变株HP-1和HP-2[91],建立了磺基-磷-香草醛快速筛选高产油脂酵母菌模型,最终筛选出一株突变菌株M16[92];并且利用离子束辐照酪丁酸梭菌ATCC 25755,能显著改善以葡萄糖为底物的丁酸生产率[93]。
2.3 重离子辐射在癌症治疗中的应用
使用带电的或者高LET粒子的放射疗法已经有很久的历史了,如使用质子有将近60年,使用重离子也有将近30年[94-95]。1954年,在美国劳伦斯国家实验室(LBNL)的第一次粒子治疗是粒子疗法的开始。
由于用于重离子治疗必需的加速器非常昂贵,而且在临床方面因相对生物学效应的难题需要不断探索分析,所以与质子治疗设备的发展相比,重离子治疗设备发展得非常慢。在重离子治疗方面,主要的开创性工作是在1977-1992年间,劳伦斯国家实验室对许多病人采用了氦和氖离子治疗[95-96]。1994年在日本千叶市(Chiba)的国立放射学研究所(NIRS),开始了全面用碳离子治疗的临床研究。1977年, 德 国 Darmstadt的 GSI(Gesellschaft für Schwerionenforschung)开始尝试用碳离子进行临床治疗试验,但随后终止了其临床应用。不过,海德堡重离子束治疗中心(HIT)的肿瘤治疗实验在2009年获得成功[97]。2001年,在NIRS支持下的第一个商业性的重离子放射治疗中心,日本兵库县离子束医学中心(HIBMC)建立[98]。位于中国兰州,有着重离子加速器的中国科学院近代物理研究所,于2006年开始开展重离子癌症治疗临床前期研究。目前,全世界开展重离子放射治疗的主要国家及设施如表2所示。全世界利用重离子开展治疗的患者的数量在稳定增长,到目前为止[99],10 000多名患者已经在日本国立放射学研究所获得治疗,超过5 000名患者在海德尔堡重离子束治疗中心获得治疗。中科院近代物理研究所与当地医院合作自1996年即开始国内首例碳离子治疗癌症病例,目前已有103例浅层肿瘤、110例深部肿瘤治疗案例。复旦大学附属肿瘤医院分院——质子重离子医院目前已收治900多病例。这些病例均有很好的治疗效果[100]。
在肿瘤治疗方面,一个关键问题是在尽可能保护正常组织的前提下有效地杀死肿瘤细胞。重离子贯穿靶物质时,通过与靶原子核外电子的碰撞损失其能量,并且这种碰撞的几率随离子能量的降低而增大。因此,离子在接近其射程末端时损失其大部分初始动能,形成一个高剂量的能量损失峰,这就是布拉格峰(Bragg peak,以学者威廉·布拉格名字命名,他测定出α粒子在空气中的射程末端,电离作用增加)。在其射程末端之后,即布拉格峰之后很少有剂量吸收;同时,离子在其入射通道上损失的能量较小,因而形成一个低剂量的坪区。对于常规辐射(X射线、γ射线、电子束和中子束),这就是重离子束特有的倒置深度剂量分布(图7)。常规辐射呈指数衰减型剂量分布,无法控制将其大部分剂量调整到深部肿瘤上,而且损伤总是表皮浅层最大,直至较深处健康组织仍能受到较大伤害。离子束布拉格峰位的深度还可以通过改变入射离子束的能量来调节,因而治疗时布拉格峰位可精确地调整在肿瘤靶区上。同时,为达到对实体瘤的治疗,可以实施不同能量离子束对靶区的照射,从而形成不同布拉格峰在靶区的叠加,即布拉格峰展宽技术[103]。通过设计可以做到展宽区域的物理剂量或生物剂量分布均匀(尽管通过此技术会导致峰坪剂量比减小,但叠加的深度剂量分布仍远好于常规辐射治疗)。

表2 迄今世界主要重离子放射治疗设备[101-102]
重离子的倒转剂量分布,以及在进入组织的入口通道处(即粒子径迹的坪区)剂量低、在束流的末端很少有剂量吸收的特性,对于处于深部的、具有辐射抗性的肿瘤细胞来说,这是一个最佳的杀灭方式(图7)。另一方面,DNA是电离辐射使细胞失活的主要靶点。前面已分析过,重离子比低LET辐射能诱导更多的DNA团簇损伤。在低剂量的X射线下,主要产生如单链破坏的独立损伤。对于这种损伤,甚至是DNA双链损伤,细胞都有非常有效的修复系统,DNA都能被高保真的修复。但是如果损伤部位发生了更复杂的DNA损伤(团簇损伤),就不太可能修复了。DNA团簇损伤是致密电离辐射的相对生物学效应高的主要原因[104]。通常肿瘤细胞,特别是乏氧的肿瘤细胞是非常抗辐射的,但是由于重离子辐射诱导的DNA团簇损伤难修复,氧增比(Oxygen enhancement ratio,OER)依赖性低[105],所以在重离子下就变得非常敏感,对常规射线治疗抗拒的肿瘤乏氧细胞、肿瘤干细胞杀伤效果好[106]。而且利用重离子的散射特性,还可建立实时在线的治疗观察手段。重离子治疗特别适宜治疗外科手术无法实施、化疗、常规放疗无效或易复发的难治病例[107]。因此,以碳离子为代表的重离子射线,正成为肿瘤放射治疗发展的新方向,被认为是最有前景的放疗手段。2000年9月第145次与2013年10月第472次的“香山科学会议”均认为:“尽管道路曲折,但重离子束治癌最科学、最先进,前景好”。

图7 不同类型射线的深度剂量分布
3 结语
重离子因其致密的电离辐射特性,诱导细胞产生复杂的DNA团簇损伤并难以正确修复,易产生突变株,使其在辐射生物突变育种方面,具有明显的优势,这对创新型农业与食品工业的发展将有很大的帮助。未来除了继续根据需要,选取不同品种的植物种子和微生物菌株,进行重离子的单次或累进多次辐射,优化选育条件,筛选突变株并改善生产工艺,实现高产与稳产之外,对突变选育的品种,应加强突变株与对照组的基因组、蛋白质组与代谢组学等大数据分析,为物种的发育进化研究提供基础数据。对突变株中与高产稳产相关的关键基因进行深入研究,将其与遗传工程技术(如最新的CRISPR技术[108-111])相结合,实现对更多的品种进行定点定向突变,使育种工作变得更省时、低成本和低工作量。
重离子诱导的DNA团簇损伤难以修复并且具有倒转的剂量分布特性,利用碳离子束治疗癌症被认为是最有前景的放疗手段。未来重离子治癌技术的提高主要包括:(1)加强硬件设施的建设以便降低成本,诸如建造重离子加速器的材料技术性能提高;加速器的紧凑小型化、低成本、产业化等。(2)肿瘤位置的精准成像与精确定位研究;不同肿瘤类型细胞的重离子杀伤效应与辐照剂量的相关性研究等;依据相关研究结果,开展重离子放射治疗程序软件的自主编制与优化,以提高粒子的精确靶向性和高效性。
在碳离子诱导生物效应的机理认识、增强碳离子辐射增敏效果等生物学基础研究方面也有需要提高的地方。虽然低LET电离辐射产生的简单DSBs损伤HR修复机制目前已经较清楚,但是对于重离子引起的多损伤类型的DNA团簇损伤,HR通路的作用和机理还有待进一步阐释,如在碳离子辐射诱导复杂DNA团簇损伤修复中,复杂的多类型损伤末端如何剪切;不同损伤如何协同修复;除了已知HR修复蛋白分子外,是否还有新的蛋白分子参与及参与程度与重要性如何;它们之间的相互作用关系又是怎样的;它们如何影响团簇损伤Rad51-ssDNA核蛋白丝修复复合体的形成。我们初步的研究显示,组蛋白赖氨酸甲基转移酶SET8响应辐射应激,其表达抑制,增加了包括重(碳)离子在内的细胞辐射敏感性。SET8调控组蛋白单甲基化水平,这些结果暗示染色体重塑对DNA的损伤修复也起着重要作用,那么染色体重塑因子对重离子DNA团簇损伤修复影响的程度与具体机理又如何。
揭示以上重离子诱导DNA团簇损伤修复新机制,不仅有助于提高对重离子辐射生物效应的认识,丰富辐射生物学基础知识,而且有助于更好地认识和评估空间重离子对航天员的辐射危害。依赖对DNA团簇损伤修复新机制的研究突破,研发相关抑制剂,与碳离子束联用,建立重离子放疗肿瘤细胞增敏新方法,可实现既能靶向放射增敏肿瘤细胞又能同时更好地降低对正常组织的损伤,促进重离子治癌技术的发展与应用,以及为太空航天员的辐射防护策略制定提供理论依据。
[1]王同权, 沈永平, 王尚武, 等. 空间辐射环境中的辐射效应[J].国防科技大学学报, 1999, 21(4):36-39.
[2]葛本伟, 郭世先, 葛淼. 地球磁场与人体健康[J]. 国外医学(医学地理分册), 2005, 26(1):39-42.
[3]Blaisdell JO, Harrison L, Wallace SS. Base excision repair processing of radiation-induced clustered DNA lesions[J].Radiation Protection Dosimetry, 2001, 97(1):25-31.
[4]Goodhead DT, Thacker J, Cox R. Effects of radiations of different qualities on cells:molecular mechanisms of damage and repair[J]. Int J Radiat Biol, 1993, 63(5):543-556.
[5]Dianov GL, O’Neill P, Goodhead DT. Securing genome stability by orchestrating DNA repair:removal of radiation-induced clustered lesions in DNA[J]. Bioessays, 2001, 23(8):745-749.
[6]Brenner DJ, Ward JF. Constraints on energy deposition and target size of multiply damaged sites associated with DNA double-strand breaks[J]. Int J Radiat Biol, 1992, 61(6):737-748.
[7]Okayasu R, Okada M, Okabe A, et al. Repair of DNA damage induced by accelerated heavy ions in mammalian cells proficient and deficient in the non-homologous end-joining pathway[J].Radiation Research, 2006, 165(1):59-67.
[8]Hamada N, Imaoka T, Masunaga S, et al. Recent advances in the biology of heavy-ion cancer therapy[J]. J Radiat Res, 2010, 51(4):365-383.
[9]Heilmann J, Rink H, Taucher-scholz G, et al. DNA strand break induction and rejoining and cellular recovery in mammalian cells after heavy-ion irradiation[J]. Radiat Res, 1993, 135(1):46-55.
[10]Tokuyama Y, Furusawa Y, Ideh, et al. Role of isolated and clustered DNA damage and the post-irradiating repair process in the effects of heavy ion beam irradiation[J]. J Radiat Res, 2015, 56(3):446-455.
[11] Werner E, Wang Y, Doetsch PW. A single exposure to low-or high-LET radiation induces persistent genomic damage in mouse epithelial cells in vitro and in lung tissue[J]. Radiation Research, 2017.Doi:10. 1667/RR14685. 1.
[12]Asaithamby A, Uematsu N, Chatterjee A, et al. Repair of HZE-particle-induced DNA double-strand breaks in normal human fibroblasts[J]. Radiat Res, 2008, 169(4):437-446.
[13]Lorat Y, Timm S, Jakob B, et al. Clustered double-strand breaks in heterochromatin perturb DNA repair after high linear energy transfer irradiation[J]. Radiother Oncol, 2016, 121(1):154-161.
[14]Taucher-scholz G, Heilmann J, Kraft G. Induction and rejoining of DNA double-strand breaks in CHO cells after heavy ion irradiation[J]. Adv Space Res, 1996, 18(1-2):83-92.
[15] Tucker JD, Marples B, Ramsey MJ, et al. Persistence of chromosome aberrations in mice acutely exposed to56Fe+26ions[J]. Radiation Research, 2004, 161(6):648-655.
[16] Oike T, Niimi A, Okonogi N, et al. Visualization of complex DNA double-strand breaks in a tumor treated with carbon ion radiotherapy[J]. Scientific Reports, 2016, 6:22275. Doi:10. 1038/srep22275.
[17]Ritter S, Durante M. Durante M:Heavy-ion induced chromosomal aberrations:a review[J]. Mutation Research, 2010, 701(1):38-46.
[18]Schipler A, Mladenova V, Soni A, et al. Chromosome thripsis by DNA double strand break clusters causes enhanced cell lethality,chromosomal translocations and 53BP1-recruitment[J]. Nucleic Acids Research, 2016, 44(16):7673-7690.
[19]Asaithamby A, Hu B, Chen DJ. Unrepaired clustered DNA lesions induce chromosome breakage in human cells[J]. Proc Natl Acad Sci of the USA, 2011, 108(20):8293-8298.
[20]Sutherland BM, Cuomo NC, Bennett PV. Induction of anchorageindependent growth in primary human cells exposed to protons or HZE ions separately or in dual exposures[J]. Radiation Research, 2005, 164(4):493-496.
[21]Urushibara A. JAEA R&D Review, 2007, 71. (http://jolisfukyu.tokai-sc. jaea. go. jp/fukyu/mirai-en/2007/index_set. html)
[22]Shikazono N. JAEA R&D Review, 2008, 71(http://jolisfukyu.tokai-sc. jaea. go. jp/fukyu/mirai-en/2008/index_set. html)
[23]David-cordonnier MH, Cunniffe SM, Hickson ID, et al. Efficiency of incision of an AP site within clustered DNA damage by the major human AP endonuclease[J]. Biochemistry, 2002, 41(2):634-642.
[24]Gulston M, Fulford J, Jenner T, et al. Clustered DNA damage induced by gamma radiation in human fibroblasts(HF19),hamster(V79-4)cells and plasmid DNA is revealed as Fpg and Nth sensitive sites[J]. Nucleic Acids Research, 2002, 30(15):3464-3472.
[25]Lomax ME, Salje H, Cunniffe S, et al. 8-OxoA inhibits the incision of an AP site by the DNA glycosylases Fpg, Nth and the AP endonuclease HAP1[J]. Radiation Research, 2005, 163(1):79-84.
[26]Gulston M, Delara C, Jenner T, et al. Processing of clustered DNA damage generates additional double-strand breaks in mammalian cells post-irradiation[J]. Nucleic Acids Research, 2004, 32(4):1602-1609.
[27]Blaisdell JO, Wallace SS. Abortive base-excision repair of radiation-induced clustered DNA lesions in Escherichia coli[J].Proc Natl Acad Sci USA, 2001, 98(13):7426-7430.
[28]D’Souza DI, Harrison L. Repair of clustered uracil DNA damages in Escherichia coli[J]. Nucleic Acids Research, 2003, 31(15):4573-4581.
[29]Harrison L, Brame KL, Geltz LE, et al. Closely opposed apurinic/apyrimidinic sites are converted to double strand breaks in Escherichia coli even in the absence of exonuclease III, endonuclease IV, nucleotide excision repair and AP lyase cleavage[J]. DNA Repair, 2006, 5(3):324-335.
[30]Malyarchuk S, Castore R, Harrison L. DNA repair of clustered lesions in mammalian cells:involvement of non-homologous endjoining[J]. Nucleic Acids Research, 2008, 36(15):4872-4882.
[31]Fujimoto H, Pinak M, Nemoto T, et al. Molecular dynamics simulation of clustered DNA damage sites containing 8-oxoguanine and abasic site[J]. J Comput Chem, 2005, 26(8):788-798.
[32]Sedelnikova OA, Redon CE, Dickey JS, et al. Role of oxidatively induced DNA lesions in human pathogenesis[J]. Mutation Research, 2010, 704(1-3):152-159.
[33]Haines JW, Coster M, Bouffler SD. Impairment of the nonhomologous end joining and homologous recombination pathways of DNA double strand break repair:Impact on spontaneous and radiation-induced mammary and intestinal tumour risk in Apc min/+ mice[J]. DNA Repair, 2015, 35:19-26.
[34] Jackson SP. Sensing and repairing DNA double-strand breaks[J]. Carcinogenesis, 2002, 23(5):687-696.
[35]Wu X, Petrini JH, Heine WF, et al. Independence of R/M/N focus formation and the presence of intact BRCA1[J]. Science, 2000,289(5476):11.
[36]Cousineau I, Abaji C, Belmaaza A. BRCA1 regulates RAD51function in response to DNA damage and suppresses spontaneous sister chromatid replication slippage:implications for sister chromatid cohesion, genome stability, and carcinogenesis[J].Cancer Res, 2005, 65(24):11384-11391.
[37]Fang S, Weissman AM. A field guide to ubiquitylation[J]. Cell Mol Life Sci, 2004, 61(13):1546-1561.
[38]Jeggo PA. Identification of genes involved in repair of DNA doublestrand breaks in mammalian cells[J]. Radiat Res, 1998, 150(5):S80-91.
[39]Rothkamm K, Kruger I, Thompson LH, et al. Pathways of DNA double-strand break repair during the mammalian cell cycle[J].Mol Cell Biol, 2003, 23(16):5706-5715.
[40] Nagasawa H, Little JB, Inkret WC, et al. Response of X-ray-sensitive CHO mutant cells(xrs-6c)to radiation. II. Relationship between cell survival and the induction of chromosomal damage with low doses of alpha particles[J]. Radiat Res, 1991, 126(3):280-288.
[41]Wang HY, Wang X, Zhang PY, et al. The Ku-dependent nonhomologous end-joining but not other repair pathway is inhibited by high linear energy transfer ionizing radiation[J]. DNA Repair,2008, 7(5):725-733.
[42]Wang JH, Pluth JM, Cooper PK, et al. Artemis deficiency confers a DNA double-strand break repair defect and Artemis phosphorylation status is altered by DNA damage and cell cycle progression[J]. DNA Repair, 2005, 4(5):556-570.
[43]Symington LS, Gautier J. Double-strand break end resection and repair pathway choice[J]. Annual Review of Genetics, 2011,45:247-271.
[44]Couedel C, Mills KD, Barchi M, et al. Collaboration of homologous recombination and nonhomologous end-joining factors for the survival and integrity of mice and cells[J]. Genes Dev, 2004, 18(11):1293-1304.
[45]Kass EM, Jasin M. Collaboration and competition between DNA double-strand break repair pathways[J]. Febs Letters, 2010, 584(17):3703-3708.
[46]Shrivastav M, De haro LP, Nickoloff JA. Regulation of DNA doublestrand break repair pathway choice[J]. Cell Res, 2008, 18(1):134-147.
[47]You ZS, Bailis JM. CtIP coordinates DNA repair and cell cycle checkpoints[J]. Trends Cell Biol, 2010, 20(7):402-409.
[48]Allen C, Kurimasa A, Brenneman MA, et al. DNA-dependent protein kinase suppresses double-strand break-induced and spontaneous homologous recombination[J]. Proc Natl Acad Sci USA, 2002, 99(6):3758-3763.
[49]Pierce AJ, Hu P, Han MG, et al. Ku DNA end-binding protein modulates homologous repair of double-strand breaks in mammalian cells[J]. Genes Dev, 2001, 15(24):3237-3242.
[50] Zafar F, Seidler SB, Kronenberg A, et al. Homologous recombination contributes to the repair of DNA double-strand breaks induced by high-energy iron ions[J]. Radiat Res, 2010, 173(1):27-39.
[51]Gerelchuluun A, Manabe E, Ishikawa T, et al. The major DNA repair pathway after both proton and carbon-Ion radiation is NHEJ,but the HR pathway is more relevant in carbon ions[J]. Radiat Res, 2015, 183(3):345-356.
[52]Hammet A, Pike BL, Mcnees CJ, et al. FHA domains as phosphothreonine binding modules in cell signaling[J]. Iubmb Life,2003, 55(1):23-27.
[53]Durant ST, Nickoloff JA. Good timing in the cell cycle for precise DNA repair by BRCA1[J]. Cell Cycle, 2005, 4(9):1216-1222.
[54]Deng CX. BRCA1:cell cycle checkpoint, genetic instability, DNA damage response and cancer evolution[J]. Nucleic Acids Res,2006, 34(5):1416-1426.
[55]Yan ZJ, Guo R, Paramasivam M, et al. A ubiquitin-binding protein,FAAP20, links RNF8-mediated ubiquitination to the Fanconi anemia DNA repair network[J]. Mol Cell, 2012, 47(1):61-75.
[56]Cao L, Xu X, Bunting SF, et al. A selective requirement for 53BP1 in the biological response to genomic instability induced by Brca1 deficiency[J]. Mol Cell, 2009, 35(4):534-541.
[57]Wang B, Matsuoka S, Carpenter PB, et al. 53BP1, a mediator of the DNA damage checkpoint[J]. Science, 2002, 298(5597):1435-1438.
[58]Schultz LB, Chehab NH, MalikzaYA, et al. p53 binding protein 1(53BP1)is an early participant in the cellular response to DNA double-strand breaks[J]. J Cell Biol, 2000, 151(7):1381-1390.
[59]李仁民, 王菊芳, 李文建. 重离子束在微生物诱变育种及生物能源开发中的应用[J]. 原子核物理评论, 2007, 24(3):234-237.
[60]Okamura M, Yasuno N, Ohtsuka M, et al. Wide variety of flower-color and -shape mutants regenerated from leaf cultures irradiatedwith ion beams[J]. Nucl Instrum Methods B, 2003,206:574-578.
[61]Yamaguchi H, Nagatomi S, Morishita T, et al. Mutation induced with ion beam irradiation in rose[J]. Nucl Instrum Methods B,2003, 206:561-564.
[62]Kikuchi S, Saito Y, Ryuto H, et al. Effects of heavy-ion beams on chromosomes of common wheat, Triticum aestivum[J]. Mutat Res, 2009, 669(1-2):63-66.
[63]Li Q, Wei ZQ, Li WJ. Calculation of depth-dose distribution of intermediate energy heavy-ion beams[J]. Chinese Science Bulletin, 2002, 47(20):1708-1710.
[64]余增亮, 何建军, 邓建国, 等. 离子注入水稻诱变育种机理初探[J]. 安徽农业科学, 1989(1):12-16.
[65]郭高, 钱坤. 安徽省农科院育成世界上第一个离子束小麦新品种[J]. 安徽农业 . l998, 25(5):4.
[66]卫增泉, 颉红梅, 梁剑平, 等. 重离子束在诱变育种和分子改造中的应用[J]. 原子核物理评论, 2003, 20(1):38-41.
[67] 赵连芝, 壬勇, 甄东牛, 等. 春小麦突变新品种-“陇辐2号”[J].核农学报, 2005, 19(1):80.
[68]董喜存, 李文建, 何金玉, 等. 碳离子束辐照对甜高粱主要性状的影响[J]. 核技术, 2009, 32(2):146-149.
[69]He JY. Pigment analysis of a color-leaf mutant in Wandering Jew(Tradescantia fluminensis)irradiated by carbon ions[J].Nuclear Science and Techniques, 2011, 22(2):77-83.
[70] Wu DL, Hou SW, Qian PP, et al. Flower color chimera and abnormal leaf mutants induced by12C6+heavy ions in Salvia splendens Ker-Gawl[J]. Sci Hortic, 2009, 121(4):462-467.
[71]Yu LX, Li WJ, Du Y, et al. Flower color mutants induced by carbon ion beam irradiation of geranium(Pelargonium× hortorum,Bailey)[J]. Nuclear Science & Techniques, 2016, 27(5):112-120.
[72]周利斌, 李文建, 曲颖, 等. 重离子束辐照育种研究进展及发展趋势[J]. 原子核物理评论, 2008, 25(2):165-170.
[73]Luo SW, Zhou B, Li WJ, et al. Mutagenic effects of carbon ion beam irradiations on dry Lotus japonicus seeds[J]. Nuclear Instruments& Methods in Physics Research Section B-beam Interactions with Materials and Atoms, 2016, 383(15):123-128.
[74]Abe T, Matsuyana T, Shigeko S, et al. Chlorophyll-deficient mutants of rice demonstrated the deletion of a DNA fragment by heavy-ion irradiation[J]. J Radiat Res, 2002, 43(4):S157-S161.
[75]Phanchaisri B, Samsang N, Yu LD, et al. Expression of OsSPY and 14-3-3 genes involved in plant height variations of ion-beaminduced KDML 105 rice mutants[J]. Mut Res, 2012, 734(1-2):56-61.
[76]Tanaka A, Tano S, Chantes T, et al. A new Arabidopsis mutant induced by ion beams affects flavonoid synthesis with spotted pigmentation in testa[J]. Genes Genet Syst, 1997, 3(3):141-148.
[77]Okamura M, Hase Y, Furusawa Y, et al. Tissue-dependent somaclonal mutation frequencies and spectra enhanced by ion beam irradiation in chrysanthemum[J]. Euphytica, 2015, 202(3):333-343.
[78]Ishii K, Kazama Y, Morita R, et al. Linear Energy Transfer-Dependent Change in Rice Gene Expression Profile after Heavy-Ion Beam Irradiation[J]. PLoS One, 2016, 11(7):e0160061.
[79]都雯玥. 重离子辐照并筛选截短侧耳素高产菌株的研究[D].兰州:兰州理工大学, 2016:1-59.
[80]颉红梅, 卫增泉, 李文建.7MeV/u O6+离子对庆大霉素生产菌诱变的初步研究[J]. 辐射研究与辐射工艺学报, 1995, 13(2):99-101.
[81]王曙阳, 薄永恒, 王丽华, 等.12C6+离子辐照对阿维链霉菌代谢效应研究[J]. 原子核物理评论, 2013, 30(2):195-200.
[82]Li SW, Li M, Song HP, et al. Induction of a high-yield lovastatin mutant of Aspergillus terreus by12C6+heavy-ion beam irradiation and the influence of culture conditions on lovastatin production under submerged fermentation[J]. Applied Biochemistry and Biotechnology, 2011, 165(3):913-925.
[83]Wang SY, Bo YH, Zhou X, et al. Significance of heavy-ion beam irradiation-induced avermectin b1a production by engineered streptomyces avermitilis[J]. Biomed Res Int, 2017, 2017 :5373262. Doi:10. 1155/2017/5373262.
[84]胡伟, 陈积红, 张珍, 等. 重离子辐照柠檬酸菌株的诱变选育[J]. 辐射研究与辐射工艺学报, 2012, 30(1):53-57.
[85]严亚平, 王菊芳, 陆栋, 等. C离子束诱变产生甜高粱汁酒精酵母高产菌株的研究[J]. 原子核物理评论, 2009, 26(3):269-273.
[86]Hu W, Chen JH, Li WJ, et al. Mutant breeding of Aspergillus nigerirradiated by12C6+for hyper citric acid[J]. Nuclear Science and Techniques, 2014, 25(2):1-4.
[87]王雨辰, 王曙阳, 董妙音, 等. 重离子束辐照选育高产植物乳酸菌[J]. 辐射研究与辐射工艺学报, 2017, 35(1):50-56.
[88]李垄清, 马良, 李文建.12C6+辐照环境下酵母高产β-葡聚糖菌株筛选及条件优化研究[J]. 食品科技, 2017, 42(3):2-6.
[89]薛林贵, 赵旭, 常思静, 等.80MeV/u C-12离子诱变选育 PHB高产菌株[J]. 核技术, 2010, 33(4):284-288.
[90]Hu GR, Fan Y, Zhang L, et al. Enhanced lipid productivity and photosynthesis effi ciency in a Desmodesmus sp. mutant induced by heavy carbon ions[J]. PLoS One, 2013, 8(4):e60700.
[91]王芝瑶, 马玉彬, 牟润芝, 等. 重离子诱变创制高产油微拟球藻新品种[J]. 生物工程学报, 2013, 29(1):119-122.
[92]Wang JF, Li RM, Lu D, et al. A quick isolation method for mutants with high lipid yield in oleaginous yeast[J]. World Journal of Microbiology and Biotechnology, 2009, 25(5):921-925.
[93]Zhou X, Lu XH, Li XH, et al. Radiation induces acid tolerance of Clostridium tyrobutyricum and enhances bioproduction of butyric acid through a metabolic switch[J]. Biotechnology for Biofuels,2014, 7:22.
[94]Suit H, Urie M. Proton beams in radiation therapy[J]. Journal of the National Cancer Institute, 1992, 84(3):155-164.
[95]Linstadt DE, Castro JR, Phillips TL. Neon ion radiotherapy:results of the phase I/II clinical trial[J]. International Journal of Radiation Oncology Biology Physics, 1991, 20(4):761-769.
[96]Castro JR. Results of Heavy-Ion Radiotherapy[J]. Radiat Environ Bioph, 1995, 34(1):45-48.
[97]Combs SE, Jakel O, Haberer T, et al. Particle therapy at the heidelberg ion therapy center(HIT)- integrated research-driven university-hospital-based radiation oncology service in Heidelberg,Germany[J]. Radiother Oncol, 2010, 95(1):41-44.
[98] Sawada K, Sawada J, Sakata T, et al. Performance test of electron cyclotron resonance ion sources for the Hyogo Ion Beam Medical Center[J]. Rev Sci Instrum, 2000, 71(2):987-989.
[99]辻进博彦. 日本重离子治疗临床研究[C]. 兰州:第二届全国重离子治疗高端论坛, 2016. 12. 27.
[100] 王小虎. 国产碳离子治癌设备应用进展[C]. 兰州:第二届全国重离子治疗高端论坛, 2016. 12. 27.
[101] Tsujii H, Kamada T. A review of update clinical results of carbon ion radiotherapy[J]. Jpn J Clin Oncol, 2012, 42(8):670-685.
[102] Kitagawa A, Fujita T, Muramatsu M, et al. Review on heavy ion radiotherapy facilities and related ion sources(invited)[J].Rev Sci Instrum, 2010, 81(2):02B909.
[103] Sunada S, Cartwright IM, Hirakawa H, et al. Investigation of the relative biological effectiveness and uniform isobiological killing effects of irradiation with a clinical carbon SOBP beam on DNA repair deficient CHO cells[J]. Oncol Lett, 2017, 13(6):4911-4916.
[104] Hada M, Sutherland BM. Spectrum of complex DNA damages depends on the incident radiation[J]. Radiation Research,2006, 165(2):223-230.
[105] Sunada S, Hirakawa H, Fujimori A, et al. Oxygen Enhancement ratio in radiation-induced initial DSBs by an optimized flow cytometry-based gamma-H2AX analysis in A549 human cancer cells[J]. Radiat Res, 2017, 188(5):591-594.
[106] Cui X, Oonishi K, Tsujii H, et al. Effects of carbon ion beam on putative colon cancer stem cells and its comparison with X-rays[J]. Cancer Research, 2011, 71(10):3676-3687.
[107] Osama M, Brock JS, Janapriya S, et al. Carbon ion radiotherapy:a review of clinical experiences and preclinical research, with an emphasis on DNA damage/repair[J]. Cancers(Basel),2017, 9(6). pii:E66. Doi:10. 3390/cancers9060066.
[108] Kuscu C, Parlak M, Tufan T, et al. CRISPR-STOP:gene silencing through base-editing-induced nonsense mutations[J].Nat Methods, 2017, 14(7):710-712.
[109] Sattar MN, Iqbal Z, Tahir MN, et al. CRISPR/Cas9:A practical approach in date palm genome editing[J]. Front Plant Sci,2017, 8:1469.
[110] Huang JJ, Wang YF, Zhao JG. CRISPR editing in biological and biomedical investigation[J]. J Cell Physiol, 2017. doi:10.1002/jcp. 26141.
[111] Donohoue PD, Barrangou R, May AP. Advances in industrial biotechnology using CRISPR-Cas systems[J]. Trends Biotechnol, 2017, pii:S0167-7799(17)30187-7.

