基于PCR的转基因家蚕快速识别方法
赵天福 杜文华 辛 佳 付巧娟 赵 兵 朱 勇
(1.西南大学纺织服装学院, 重庆 400716;2.重庆市生物质纤维材料与现代纺织工程技术研究中心, 重庆 400716;3.西南大学生物技术学院, 重庆 400716)
基于PCR的转基因家蚕快速识别方法
赵天福1,2杜文华3辛 佳3付巧娟3赵 兵1,2朱 勇3
(1.西南大学纺织服装学院, 重庆 400716;2.重庆市生物质纤维材料与现代纺织工程技术研究中心, 重庆 400716;3.西南大学生物技术学院, 重庆 400716)
在转基因家蚕的制作方法中,基于piggyBac转座子的显微注射法是当前最常用和最有效的方法。通过piggyBac转座子方法获得的家蚕,由于其以随机方式切出和转座的特征,所获得家蚕的转入位置往往带有随机性,不可人为预测和控制。利用转基因插入位置的随机性,配合转入的目的基因序列识别,建立了家蚕指纹识别方法。该方法快捷方便,可用于转基因家蚕品种繁育过程中的品系识别和转基因家蚕知识产权的举证手段。
转基因;家蚕; PCR;识别
在转基因家蚕的制作方法中,基于piggyBac转座子的显微注射法是当前最常用和最有效的方法[1]。随着转基因技术的成熟,利用该技术制作的各种转基因蚕品种日益丰富,利用这些转基因蚕生产具有特殊性能的创新蚕丝、或具有各种功能的有用蛋白质成为蚕业科学研究的新热点。通过piggyBac转座子方法获得的家蚕,由于piggyBac转座子以随机方式转座的特征,所获得家蚕的转入位置往往不可人为预测和控制,也带有随机性[2]。基于piggyBac转座子改良丝蛋白基因生产特殊蚕丝或有用蛋白的转基因家蚕,外观和习性与未转基因的蚕几乎无异,在繁育过程中难以区分,一旦混杂,难以判别。尽管有荧光标记等标志基因可以识别,但标记基因种类有限,无法在不同的转基因蚕中都使用不同的标志基因。
本研究利用piggyBac转座子的随机转座特征,将转入位置的核酸序列信息和转入基因的序列信息作为该转基因品系蚕的指纹特征,用聚合酶链式反应(PCR)和测序技术相结合,建立一种快速高效的转基因蚕指纹识别方法。本方法可用于转基因蚕繁育中的不明品系鉴定,也可用于转基因家蚕育种材料的知识产权举证。
1 材料和方法
1.1 材料
供试蚕品种为西南大学保存的转蜘蛛丝蛋白基因家蚕品系SSMS1218。该材料遗传背景清楚,已经3年以上继代,遗传稳定[3]。用桑叶饲养,上蔟羽化后取蚕蛾用于实验。
1.2 方法
1.2.1 基因组DNA的抽提
蚕蛾基因组的抽提参照Sambrook[4]的方法进行。蚕蛾用DNA抽提缓冲液磨碎后,离心取上清液。用碱性酚及氯仿抽提后,用乙醇沉淀回收DNA。再经无DNA酶的RNA酶处理,以去除混入的少量RNA。
1.2.2 基因组的酶切及自身环化
用限制性内切酶HaeIII处理基因组DNA。步骤按照试剂说明书进行。每样品处理约40μg DNA。通过琼脂糖凝胶电泳检测处理效果,处理至整个泳道呈较均匀拖带,没有大分子量核酸为止。经乙醇沉淀处理后用适量灭菌纯水溶解DNA。用Takara Ligation kit于16℃连接过夜,使酶切后的DNA片段自身环化。经乙醇沉淀,回收环化DNA。
1.2.3 PCR反应
分别以上述1.2.1得到的纯化DNA或1.2.2得到的环化DNA为模板,进行转入基因序列PCR或末端反向PCR。所用引物序列及反应条件如表1。引物位置及目标序列位置示意如图1。
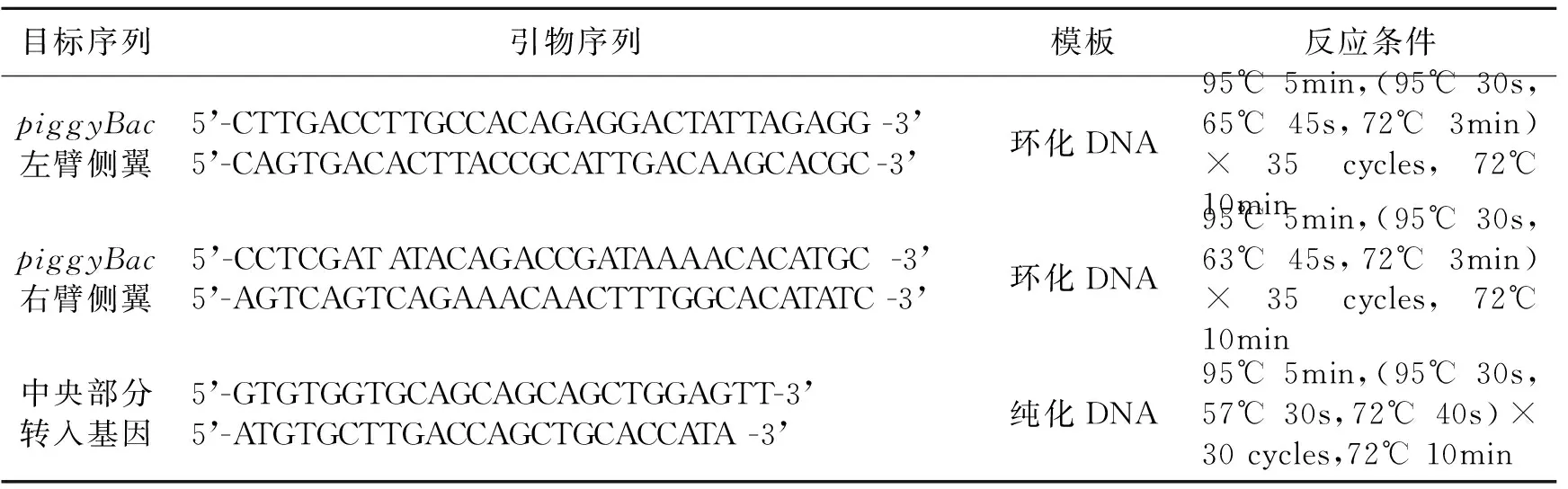
表1 PCR反应引物及反应条件

图1 PCR反应引物及扩增目的片段位置示意图
1.2.4 克隆和测序
PCR产物条带单一清晰的可用原引物直接测序。部分测序困难的样品,克隆到T载体后,用载体通用引物测序。测序利用Takara公司的测序服务。piggyBac两臂侧翼序列测出后需要经过适当处理加以拼接,具体方法是:利用blast软件找出已知的piggyBac末端序列和特异识别的TTAA序列,与TTAA相邻的即为侧翼序列。由于侧翼序列中的HaeIII位点远近不一,通常只标识其中最近的10-20个碱基做为标志,更长序列可能通过家蚕基因组数据库间接获得。中央部分的插入基因测序结果直接去除引物序列即为目标序列。
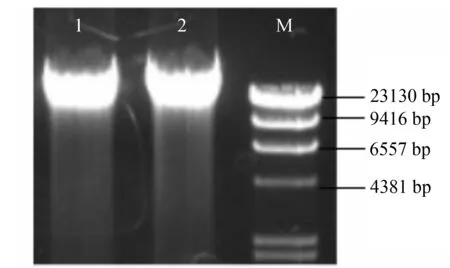
1:转基因家蚕;2:对照(非转基因家蚕);M:分子量标记 图2 基因组DNA的电泳
2 结果与分析
2.1 基因组DNA的抽提质量
本文采用通用的方法抽提了转基因家蚕和非转基因家蚕的基因组DNA,电泳图上并无差异(图2),可见一般的核酸抽提方法可以用于转基因家蚕蛾基因组的抽提。
2.2 测序结果
2.2.1piggyBac左臂和右臂侧翼序列
经重复3次以上测序确定piggyBac左臂侧翼的家蚕基因组序列,最近的15个碱基从5′到3′的顺序为: 5′-AGCTAGCTCTCTTGGTTAA-3′。其中TTAA为已在其他文献中报告过的piggyBac转座子特异识别序列,TTAA的下游方向进入插入的转座子序列。用同样的方法确定piggyBac右臂侧翼的家蚕基因组序列的最近15个碱基,从5′到3′的顺序为:5′-TTAATATACGATCGGAGTA-3′。 TTAA的上游方向为插入的转座子序列。
综合测得序列的信息,通过与家蚕基因组数据库进行比对,定位插入位点位于家蚕第11号染色体。
2.2.2 中央部分插入的转入目标基因序列
以纯化未酶切的转基因蚕蛾基因组为模板扩增的片段,测序后去除载体及引物序列,即为目标核心序列,全长747bp,其中的5′部分为GCCGGACAAGGATTA,3′部分为ATCGGCCAAGTTAAT,均为从5′到3′的顺序。所有序列测序波形清楚无误。
3 讨论
利用家蚕作为生物反应器开展有用物质合成和生产的研究是当前蚕业科学研究的热门领域之一。上世纪末到本世纪初,piggyBac转座子为载体的显微注射转基因蚕制作技术日渐成熟。到今天利用该技术制作转基因家蚕,以生产具有特殊性能的新型蚕丝,或者生产有用蛋白物质的研究方兴未艾。由于piggyBac转座子近乎随机插入的特点,插入位置随机,不可人为预测和控制,目前为止的研究报告中尚未发现有将外源基因插入到家蚕基因组中同一位置的报告。
随机插入的特性也带来了插入位置的难以再现性。本研究将这个“难以再现”的位置特征作为转基因家蚕的指纹特征加以利用,建立了一种利用PCR技术快速识别特定转基因家蚕品系的方法。本方法从磨蛾抽提DNA开始,到测序结果完成最快在1 d之内可以完成,时间快捷。可以利用已产卵雌蚕蛾或已交雄蛾为材料,可与制种过程同步进行,具有较好的实用性。实验中采用了反向PCR的识别插入片段侧翼序列,可用于探究未知的插入位置。在对插入位置已经清楚的转基因家蚕末端序列进行识别时,可以参考家蚕基因组数据库或先期的测试数据,进行普通的PCR,可进一步缩短实验时间。进一步的研究还可以考虑引物及酶的预混,PCR反应条件的优化,模板的微量化等,进一步提高识别工作效率。
[1] TAMURA T, THIBERT C,ROYER C, et al. Germline transfo rmation of the silkworm Bombyx mori L. using a piggyBac transposon-derived vector[J]. Nat Biotechnol. 2000, 18(1):81-4.
[2] 徐汉福,幸俊逸,王职峰,等. 外源piggyBac转座元件在转基因家蚕中的整合位点分析[J]. 蚕学通讯,2010,30(01):1-7.
[3] 杜文华. 棒络新妇蜘蛛拖牵丝蛋白基因编码序列的克隆及重组表达[D].西南大学,2012.
[4] SAMBROOK J, FRISCH E F, MANIATIS T. (1989) Molecular Cloning. A Laboratory Manual. Cold Spring Harbor Press, Cold Spring Harbor, NY.
重庆市蚕桑产业发展补助资金项目“转蜘蛛丝蛋白基因家蚕的快速指纹识别研究项目”。
赵天福(1968-),副教授,从事纤维材料与工程、蚕丝生物学研究。
朱勇,教授。E-mail:zhuy@swu.edu.cn
——大蚕蛾

