基于ITS序列对野生大型真菌进行分子鉴定及系统地位研究
李秋玲 ,吴智艳 ,乔 洁 ,黄 岩 ,侯晓强
(1.廊坊师范学院,河北廊坊 065000;2.河北省高校食药用菌资源开发应用技术研发中心,河北廊坊 065000;3.廊坊市食用菌技术重点实验室,河北廊坊 065000)
基于ITS序列对野生大型真菌进行分子鉴定及系统地位研究
李秋玲1,2,3,吴智艳1,2,3,乔 洁1,2,3,黄 岩1,侯晓强1,2,3
(1.廊坊师范学院,河北廊坊 065000;2.河北省高校食药用菌资源开发应用技术研发中心,河北廊坊 065000;3.廊坊市食用菌技术重点实验室,河北廊坊 065000)
针对目前我国食用菌存在资源广、利用少的问题,对采集于河北省廊坊市自然公园的7株大型野生真菌进行鉴定,为其开发利用与驯化栽培奠定基础。提取子实体基因组DNA,利用PCR技术对其rDNA的ITS区段进行扩增并测序,与GenBank数据库中序列进行Blast比对。采用邻接法对7株菌株进行系统发育分析。鉴定结果:ZR-1和ZR-7为大肥蘑菇(Agaricus bitorquis)、ZR-3属于伞菌属(Agaricus julius),ZR-4属于黏滑菇属(Hebeloma sp.),ZR-5属于伞菌属(Agaricus sp.),ZR-8为罕见类群中国产地菇状马蒂奥洛菌(网孢地菇)(Mattirolomyces terfezioides≡Terfezia terfezioides),TYG-1为野生金针菇(Flammulina velutipes)。这些结果为野生资源利用奠定了基础。
野生大型真菌;鉴定;ITS;系统地位
0 引言
野生大型真菌种质资源的收集与鉴定是一项长期的基础性研究工作。传统的鉴定分类方法主要依据子实体形态、颜色、气味和孢子特征等,一般直接观察形态,或借助显微镜观察菌丝和孢子形态[1]。大型真菌的形态特征不仅受到子实体的生长环境、生长时期等客观因素的影响,而且形态性状的选择和判断也受主观因素的影响,因此具有一定的局限性和主观性,并且同一个属中不同种的菌丝特征和孢子形态虽有略微差异,即使在显微镜下观察,也会因为其相似性而造成误判,如蘑菇属的不同种之间,其菌丝无锁状联合现象[2],所以难以分辨。要将一株大型真菌准确鉴定到某一个种的水平比较困难,特别是同一个属中,有些种菌丝和孢子形态特征极为相近,这就很难准确区分鉴定到种一级水平[3]。
随着分子生物学技术的发展,利用分子生物学方法,通过DNA序列比较分析,同时借助国际核酸数据库的大量信息资源,有助于准确鉴定野生大型真菌,并且分子生物学技术鉴别种质资源具有快速、准确等优点。rDNA上的ITS(Internal Transcribed Spacer)序列长度一般在650~750bp之间,是中度保守序列,表现为种内相对一致,种间差异较明显,这些特征使ITS广泛应用于真菌物种的分子鉴定及遗传多样性研究[4]。
虽然我国野生大型真菌资源丰富,但开发利用得非常少。因此,结合分子生物学技术,采用一种准确、快速、有效的方法对野生大型真菌资源进行鉴定势在必行。通过对采自廊坊自然公园的7株野生大型真菌子实体进行基因组DNA提取,进一步根据ITS序列进行核酸序列数据库GenBank同源性检索比对,构建系统发育树,以期为野生大型真菌物种鉴定提供分子依据,为野生菌的开发和利用奠定基础。
1 材料与方法
1.1 菌种来源
7株野生大型真菌采集自廊坊自然公园,其中5株为新鲜子实体(图1),2株为保存于廊坊师范学院生命科学学院实验室标本。
1.2 试剂
化学试剂均为分析纯,购自国药集团化学试剂北京有限公司和OXIOD公司;分子生物学试剂,引物合成及序列测定由上海生工生物工程技术服务有限公司完成。

图1 5株野生大型真菌的子实体图片
1.3 DNA的提取及检测
取少量子实体于1.5 mL的离心管中,DNA的提取反应程序参照许峰等[5]的方法。提取的基因组DNA经0.8%的琼脂糖凝胶电泳检测。取DNA样品5μL进行凝胶电泳20 min,EB染色,凝胶成像系统观察基因组DNA的提取质量并拍照记录。
1.4 ITS-PCR扩增
ITS1(5’-TCCGTAGGTGAACCTGCGG-3’)和ITS4(5’-TCCTCCGCTTATTGATATGC-3’)作为引物,以提取的基因组DNA作为模板进行PCR扩增。扩增条件:95℃5min(预变性);95℃30s,58℃1min,72℃ 30 s,30 个循环;72℃10 min;4℃保存。反应体系为50 μL。PCR反应完成后进行电泳检测。
1.5 ITS基因测序及分析
PCR产物切胶回收后送北京生工测序部测序。将菌株的ITS序列与GenBank核酸序列数据库进行比较,并用MEGA4.0.2软件构建系统发育树[6]。用Alignment程序对序列进行多重对位排列,采用Neighbor-Joining法构建系统发育树,重复抽样1000次分析系统树各分枝的置信度。
2 结果与分析
2.1 基因组提取
取5μL提取的基因组DNA样品,经0.8%琼脂糖凝胶电泳检测,结果如图2所示,基因组DNA样品条带明亮清晰,无拖尾现象,说明提取的基因组DNA可用于后续实验。
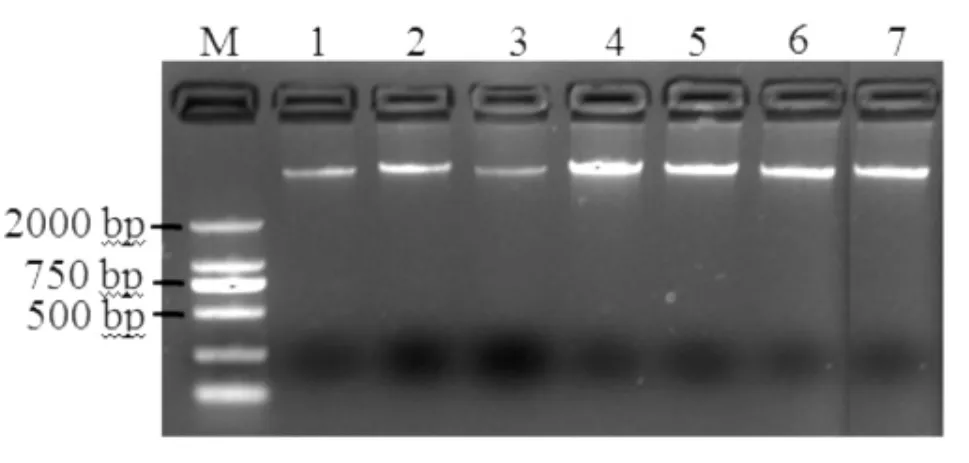
图2 基因组的电泳图
2.2 ITS扩增结果
以提取的基因组DNA为模板,选取2个通用引物进行PCR扩增ITS序列,结果如图3所示,所得产物片段大小700 bp左右,与预期结果相一致,条带单一,表明扩增结果较好,可用于后续测序分析。

图3 ITS片段扩增结果
2.3 系统发育树的构建
将7个样品测得的ITS序列与通过BLAST检索比对,从NCBI的GenBank数据库获得30个与供试菌株具有较高相似度的物种(表1)。
对7个样品ITS序列及相似序列构建系统发育树(图4)。结果表明30个ITS序列聚为4类,分别为伞菌属(Agaricus)、黏滑菇属(Hebeloma)、地菇属(Terfezia)和金针菇属(Flammulina)。ZR-1、ZR-3、ZR-5和 ZR-7是伞菌属(Agaricus),其中 ZR-1和ZR-7与Agaricus bitorquis聚在一起,鉴定为大肥蘑菇,ZR-3与 Agaricus julius聚在一起,ZR-5与 A-garicus sp.聚在一起,鉴定为拟双环林地蘑菇。ZR-4与Hebeloma sp.聚在一起,ZR-8 与Mattirolomyces ter-fezioides ≡ Terfezia terfezioides聚在一起,鉴定为罕见类群中国产地菇状马蒂奥洛菌(网孢地菇),TYG-1与Flammulina velutipes聚在一起,鉴定为野生金针菇。

表1 与供试7个未知真菌ITS序列最相似的物种
3 讨论
利用ITS序列对大型真菌包括食药用菌、外生菌根菌进行分类鉴定和分子系统发育关系的研究越来越受到人们的关注[7-9]。研究者对东亚鹅膏菌[10]、日本鹅膏菌[11]及各种鹅膏菌标本[12-13]的系统发育和一些鹅膏菌[14]的纯培养菌种进行了分子鉴定,这些研究从分子水平对鹅膏菌属大型真菌的自然分布群体、属内系统发育关系等进行了系统研究。另外,Masaki等[15],采用ITS-RFLP方法通过限制性内切酶酶切研究了不同香菇菌株间的亲缘关系。杨永彬等[16]对福建13个香菇主要栽培品种进行了ITS系统发育分析。黄晨阳等[17]分析了侧耳属16个种不同菌株间ITS序列。可见,ITS分析用于研究不同品种的亲缘关系也日趋成熟。本研究利用大型真菌rDNA ITS区段的保守性,同时借助GenBank数据库的大量信息,在分子水平上对采自廊坊自然公园的7株真菌的ITS序列进行了BLAST比对,结果表明,这几株野生大型真菌与GenBank数据库中相关序列同源性达到99%。

图4 根据ITS基因序列构建的系统发育树
野生食药用菌种质资源的收集与鉴定是其开发利用的前提。田慧敏等[18]利用形态学及ITS序列鉴定了赤峰地区5个未知真菌。笔者采集的7株野生大型真菌中,有1株属于黏滑菇属(Hebeloma sp.),4株属于伞菌属(Agaricus)。伞菌属的食用菌是当前全球性食用菌产业中栽培面积最大、产量最高的食用菌品种。伞菌属大型真菌大多数品种可以食用或药用,但真正广泛栽培的仅有双孢蘑菇(A.bisporus)和大肥菇(A.bitorquis)[19]。因此,野生伞菌属资源的开发利用还有很大空间。
ITS序列是位于rDNA编码基因18S、5.8S和28S之间的小基因片段[20],由于ITS区在进化中承受的自然选择压力较小,能容忍更多的变异,因此在大多数的真核生物中表现出了较为广泛的序列多态性[21]。虽然本研究中7株大型真菌序列与数据库中同源性高达99%,但仍然有单核苷酸突变,将来可进一步研究其单核苷酸突变与性状的关系。本研究根据ITS基因序列构建系统发育树,伞菌属的4种大型真菌亲缘关系较近,与黏滑菇属的大型真菌亲缘关系较远。黄龙花等[22]对20株灵芝的ITS序列进行了测定分析,将形态学特征难以区分的灵芝属的菌株进行了有效分类。戴璐等[23]同样用ITS序列测定与分析对秦岭冷水沟的10株羊肚菌进行了鉴定。Ro等[24]对12株不同的杏鲍菇进行了ITS序列系统发育分析。因此,ITS序列分析在已知的不同品种的亲缘关系研究方面有着重要的地位。
4 结论
本研究中的野生大型真菌是在廊坊自然公园人员稀少处获得的,对廊坊地区野生资源的收集有一定的意义。因目测无法辨别其遗传地位,因此利用ITS序列分析方法,对采集的菌株生物学地位未知的野生大型真菌进行了序列测定和系统发育分析,为这7种野生菌的开发利用奠定了基础。根据ITS序列分析的分子生物学方法,对形态学上不易辨别的野生大型真菌进行遗传学鉴定,这表明利用分子生物学方法与形态学方法相结合确定物种的生物学遗传地位,是可行和有效的。
[1]冯晓宇,邹莉,王志英,等.内蒙古野生银白离褶伞子实体组织分离试验[J].中国食用菌,2010,29(3):13-14.
[2]杨新美.食用菌研究法[M].北京:中国农业大学出版社,1998.
[3]张国广,邹金美,郑琳,等.一株野生大型真菌的ITS分析及生物学特征研究 [J].云南民族大学学报 (自然科学版),2010,17(6):395-399.
[4]李晶,林雄杰,林占熺.一株野生大型真菌的形态鉴定及ITS序列分析[R].福建:第九届全国药用真菌学术会议暨药用真菌专业组织成立三十周年庆,2009.
[5]许峰,刘宇,王守现,等.一种适于PCR反应的快速提取食用菌基因组 DNA 提取方法[J].生物技术,2011,21(2):43-44.
[6] Tamura K,Dudley J,Nei M,et al.MEGA:Molecular Evolutionary Genetics Analysis (MEGA)Software Version 4.0 [J].Mol.Niol.Evol.,2007,24(8):1596-1599.
[7] Avin FA,Bhassu S,Shin TY,Sabaratnam V.Molecular classification and phylogenetic relationships of selected edible Basidiomycetes species [J].Mol Biol Rep,2012,39 (7):7355-7364.
[8] Bunyard B A,Chaichuchote S,NichoIson M S,et a1.Ribosomal DNA analysis for resolution of genotypic classes of Pleurotus[J].Mycol Res,1996,100(2):143-150.
[9]林晓民,李振岐,王少先.真菌rDNA的特点及在外生菌根菌鉴定中的应用[J].西北农业学报,2005,14(2):120-125.
[10] Zhang L F,Yang J B,Yang Z L,et a1.Molecular phylogeny of eastern Asian species of Amanita (Agaricales,Basidiomycota):taxonomic and biogeographic implications [J].Fungal Diversity,2004,17(1):219-238.
[11] Oda T,Tanaka C,Tsuda M.Molecular phylogeny of Japanese Amanita species based on nucleotide sequences of the internal transcribed spacer region of nuclear ribosomal DNA[J].Mycoscience,1999,40(1):57-64.
[12] Wei B M,Yang Z L,Oberwinkler F.Molecular phylogenetic studies in the genus Amanita[J].Canada Journal of Botany,
1998,76(7):1170-1179.
[13] Drehmel D,Moncalvo J M,Vilgalys R.Molecular phylogeny of Amanita based on large-subunit ribosomal DNA sequences:implications for taxonomy and character evolution [J].Mycologia,1999,91(4):610-618.
[14] Zhang P,Chen Z H,Hu J S,et a1.Production and characterization of Amanita toxins from a pure culture of Amanita exitialis[J].FEMS Microbiol Lett,2005,252(2):223-228.
[15] Masaki F,Tadashi S,Yoko O.Genetic analysis of nuclear ribosomal DNA of Lentinula edodes [J].Mycoscience,2006,47(6):347-350.
[16]杨永彬,兰家细,谢福泉,等.基于香菇菌株rDNA-ITS序列的系统发育分析[J].微生物学杂志,2010,30(4):27-29.
[17]黄晨阳,陈强,高山,等.侧耳属主要种类ITS序列分析[J].菌物学报,2010,29(3):365-372.
[18]田慧敏,刘铁志,文静,等.基于形态学及ITS序列鉴定赤峰地区五个未知真菌[J].食用菌学报,2017,24(1):21-26.
[19]吕彩莲,赵磊,陈有君,等.野蘑菇菌丝生长培养基的优化筛选[J].农产品加工,2011,(6):36-41.
[20]陈剑山,郑服丛.ITS序列分析在真菌分类鉴定中的应用[J].安徽农业科学,2007,35(13):3785-3786.
[21] Wang P,Liu Y,Yin Y G,et al.Diversity of microorganisms isolated from the soil sample surround Chroogomphus rutilus in the Beijing region [J].Int.J.Biol.Sci.,2011,7(2):209-220.
[22]黄龙花,杨小兵,张智,等.基于ITS序列分析鉴定灵芝属菌种[J].中国食用菌,2010,29(1):55-57.
[23]戴璐,李峻志,李安利,等.基于ITS序列分析对秦岭冷水沟羊肚菌的鉴定[J].中国食用菌 2010,29(6):45-46.
[24] Ro HS,Kim SS,Ryu J S,etal.Comparative studies on the diversity of the edible mushroom Pleurotus eryngii:ITS sequence analysis,RAPD fingerprinting,and physiological characteristics[J].Mycol Res,2007,(111):710-715.
Molecular Identification and Analysis of Phylogenetic Position for
Seven Wild Fruitingbody-forming Fungi
LI Qiu-ling1,2,3,WU Zhi-yan1,2,3,QIAO Jie1,2,3,HUANG Yan1,HOU Xiao-qiang1,2,3(1.Langfang Teachers University,Langfang 065000,China;2.Edible and Medicinal Fungi Research Center of Hebei Universities,Langfang 065000,China;3.Langfang Key Laboratory of Edible Fungi Technology,Langfang 065000,China)
For the case of less domestication of the wild mushrooms in contrast with their abundant distribution at present time,this study identified seven wild mushrooms collected from Natural Park in Langfang city,Hebei province to lay the foundation for the exploitation,domestication,and cultivation of wild mushrooms.The Genomic DNA was extracted from fruiting body.Based on PCR,ITS regions of rDNA of the macrofungi were amplified using the special pairs of primers ITS1/ITS4 and sequenced.Further phylogenetic analysis based on the ITS sequence was conducted to confirm the genetic position of these fungi.The results showed that about 700 bp length fragment was amplified.Then the phylogenetic trees were constructed by neighbor-joining method.Phylogenetic analysis indicated that ZR-1 and ZR-7 areAgaricus bitorquis,ZR-3 is Agaricus Julius,ZR-4 is Hebeloma sp.,ZR-5 is Agaricus sp.,ZR-8 is Mattirolomycesterfezioides ≡ Terfeziaterfezioides,and TYG-1 is Flammulina velutipes.These results established an identification method for the wild fruitingbody forming fungi and laid the foundation of the application of these fungi.
wild macrofungi;identification;ITS;phylogenetic position
S184
A
1674-3229(2017)04-0053-05
2017-09-28
河北省高校食药用菌应用技术研发中心(YF201411);国家自然科学基金项目(31402056);河北省高等学校青年拔尖人才计划项目(BJ2014048)资助
李秋玲(1979-),女,博士,廊坊师范学院生命科学学院副教授,研究方向:分子生物学。
吴智艳(1962-),女,廊坊师范学院生命科学学院教授,研究方向:微生物学。

