斑海豹线粒体基因组序列分析及比较研究
高祥刚,鹿志创,田甲申,韩家波,赫崇波,宋新然
( 1.辽宁省海洋水产科学研究院,辽宁省海洋水产分子生物学重点实验室,辽宁 大连 116023; 2. 大连圣亚旅游控股股份有限公司,辽宁 大连 116023 )
斑海豹线粒体基因组序列分析及比较研究
高祥刚1,鹿志创1,田甲申1,韩家波1,赫崇波1,宋新然2
( 1.辽宁省海洋水产科学研究院,辽宁省海洋水产分子生物学重点实验室,辽宁 大连 116023; 2. 大连圣亚旅游控股股份有限公司,辽宁 大连 116023 )
本研究采用LA-PCR扩增和引物步移相结合的方法对我国辽东湾斑海豹的线粒体基因组全序列进行了测定,并完成了基因组成和系统发生学分析。辽东湾斑海豹线粒体基因组长为16 754 bp,其基因构成与其他海洋哺乳动物基本一致,包括37个基因(2个rRNA基因、22个tRNA基因和13个蛋白质编码基因)和1个控制区。在其37个基因中,ND6、tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer(UCN)、tRNAGlu、tRNAPro位于L链上,其余均位于H链上。对我国辽东湾、韩国和美国阿拉斯加斑海豹线粒体基因组进行了比较分析,结果显示,三者线粒体基因组长度略有不同,差异主要体现在以重复片段形式存在的控制区序列中。值得注意的是,在编码区中,3个目标群体的线粒体ND5基因核苷酸差异最为显著,共检测到了18个突变位点,造成4处氨基酸序列变异。斑海豹线粒体基因组蛋白编码基因的变异分析及海豹科系统进化分析结果显示,我国辽东湾和韩国斑海豹的序列同源性显著高于美国阿拉斯加斑海豹,说明辽东湾和韩国斑海豹很可能来自同一个种群。
斑海豹;线粒体基因组;结构比较;系统进化
线粒体DNA(mtDNA)是动物细胞核外唯一的遗传物质,与核DNA相比,具有母系遗传、分子结构简单、进化速度快、无组内特异性等特点。目前mtDNA已成为研究物种起源与系统发生、近缘物种和种内群体间遗传分化、物种鉴定、遗传多样性分析等的有力工具[1]。
海豹是对鳍足亚目种海豹科动物的统称,现存的海豹种类较多,目前共有10属19种。斑海豹(Phocalargha)属食肉目、犬形亚目、海豹科、斑海豹属,主要分布于北太平洋的北部和西部海域及其沿岸和岛屿。斑海豹在我国主要分布于渤海和黄海海域,偶见于南海。是唯一能在我国海域繁殖的鳍脚类动物,属于国家二类重点保护野生动物,也是黄、渤海生态系统健康状况的重要指示生物,因而受到国内外学者的普遍重视[2-3]。
笔者通过LA-PCR扩增和引物步移相结合的方法对我国辽东湾斑海豹的线粒体基因组全序列进行了测定,初步分析了斑海豹线粒体基因组序列的基本特征,探讨了我国、韩国、美国阿拉斯加斑海豹线粒体基因组基因排列、蛋白质编码基因和多态位点等。同时利用线粒体基因组蛋白编码序列,分析了海豹科的系统分类关系。本研究对于海豹的遗传分化与系统发生研究、物种鉴定、遗传多样性的保护等诸多方面提供宝贵的遗传信息。
1 材料与方法
1.1 试验材料和基因组DNA提取
解剖盘锦双台河口救助的当年死亡斑海豹幼崽的肝脏,清洗后立即保存于-20 ℃冰柜中。采用常规酚氯仿法提取全基因组DNA[4],溶于TE缓冲液中,4 ℃保存备用。
1.2 PCR扩增及序列测定
参考韩国、美国阿拉斯加斑海豹(GenBank号分别为:FJ895151、NC_008430)线粒体基因组序列,应用FastPCR v3.6软件设计引物COⅠ_F 和COⅠ_R。利用课题组已设计引物[5-6]COⅠ_F、COⅠ_R、L16274_F 、H34_R,形成引物组合(表1)L16274_F + COⅠ_R 、COⅠ_F + ND4L_R、 ND4L_F + H34_R进行辽东湾斑海豹的线粒体基因组Long-PCR扩增。反应条件和反应体系见表2。测序采用引物步移法进行双向测通。因为PCR产物直接测序法不能准确读出测序引物后面约50 bp长度的序列,所以在设计扩增片段的引物时,尽可能保证相邻扩增片段之间包含80 bp以上的序列重叠,以保证测序后序列的准确性及正确拼接。由于在控制区测序时出现套峰现象,此段区域采用克隆后测序的方法。

表1 用于扩增辽东湾斑海豹线粒体基因组的引物
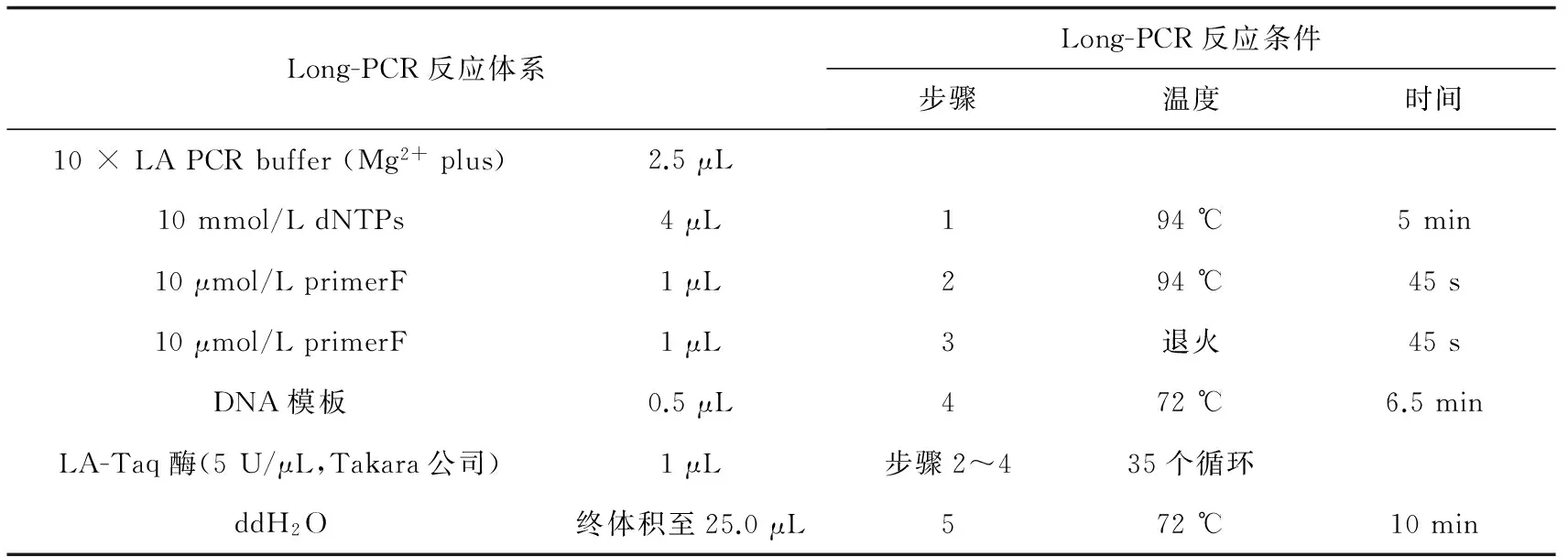
表2 辽东湾斑海豹线粒体基因组扩增的反应条件和反应体系
1.3 序列分析、基因预测
首先使用Chromas 2.4.1软件打开和核实基因测序得到的峰图,并用CONSED手工检查以避免错配[7]。利用DOGMA软件对13个蛋白质编码基因和2个核糖体RNA 的位置进行预测[8],与其他海豹属物种的线粒体基因组比对后确定精确的起始位置。利用tRNAscan-SE 1.21软件对转运RNA及它们的二级结构特征进行预测[9]。
1.4 斑海豹线粒体基因组的比较分析
将所测得的辽东湾斑海豹mtDNA序列,应用Clustal 1.83和Mega 5.0生物信息学软件,分别与韩国、美国阿拉斯加斑海豹线粒体基因组全序列进行比较分析,探讨三者在蛋白编码基因的氨基酸和核苷酸序列、tRNAs、rRNAs上的差异,以及在非编码区的变异情况。
1.5 海豹科线粒体基因组的系统进化
对NCBI上已公布的海豹科的15个种、17条线粒体基因组DNA序列的13个蛋白编码基因的氨基酸序列和核苷酸序列采用邻接法分别构建系统关系树。
2 结 果
2.1 辽东湾斑海豹线粒体基因组的组成
将所测得的辽东湾斑海豹线粒体基因组全序列拼接完整后提交至GenBank(NCBI登录号:KT818831),其长度为16 754 bp,呈现典型的海洋哺乳动物线粒体基因组的特点。即斑海豹线粒体基因组为环状,由37个基因组成,其中的 13个蛋白质编码基因包括:1个细胞色素b基因(CytB)、2个ATP酶亚基的基因(ATPase6, ATPase8)、3个细胞色素氧化酶亚基的基因(COⅠ,COⅡ,CO Ⅲ)和7个呼吸链NADH脱氢酶亚基的基因(ND1-6,ND4L);2个rRNA基因分别为1个12S rRNA和 1个16S rRNA;另外还有22个tRNA基因。
在13个蛋白质编码基因中,12个是由H 链编码,仅ND6由L链编码;在22个tRNA中,tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer(UCN)、tRNAGlu、tRNAPro的编码基因位于L链上,其余则位于H 链上。
2.2 与其他斑海豹的比较
将所测得的中国辽东湾斑海豹线粒体基因组与韩国、美国阿拉斯加斑海豹线粒体基因组进行比较分析发现,三者线粒体基因组长度略有不同,分别为16 754、16 728、16 701 bp,主要的变化出现在控制区(OH),OH长度分别为1316、1290、1264 bp。
对上述3个线粒体基因组的37个基因分别进行了比较研究,在rRNA中,发现5个单核苷酸变异;在tRNA中,发现阿拉斯加斑海豹的tRNALeu出现1个碱基缺失,其他的tRNAPhe、tRNAAla、tRNATyr、tRNAThr等各有1个单核苷酸变异,除此之外的17个tRNA完全一致;13个蛋白质编码基因中,均发现有核苷酸变异,其中8个基因(ATPase6、COⅠ、COⅡ、CytB、ND1、ND3、ND4、 ND4L)相对比较保守,其氨基酸序列在3个基因组中完全一致,5个基因出现氨基酸序列变化(表3),其中,ND5的核苷酸突变程度最大,总长1821 bp的核苷酸中有18个突变位点,变异率为0.99%,出现4个氨基酸序列突变。
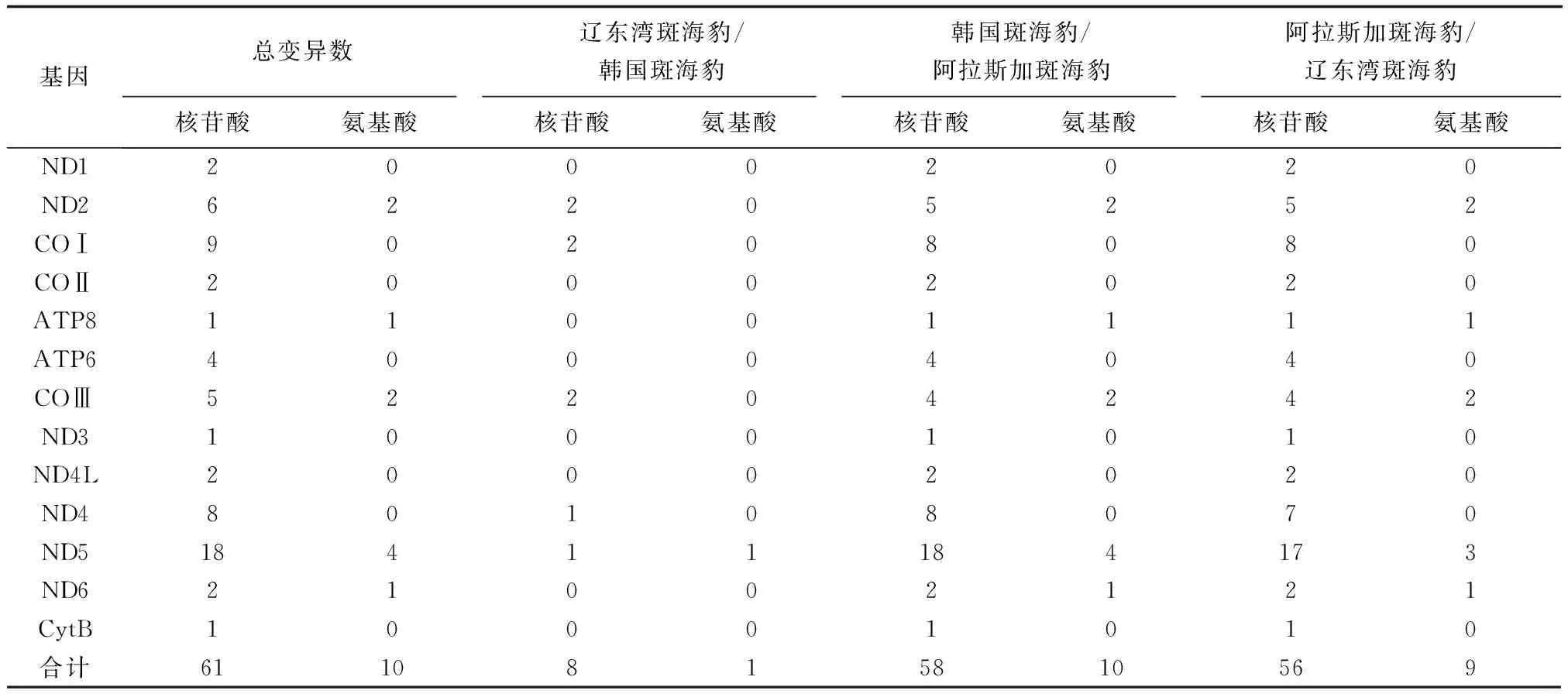
表3 斑海豹3个线粒体基因组蛋白编码基因的突变比较分析
2.3 海豹科线粒体基因组的系统进化
以海洋哺乳动物的鲸目白鳍豚(Lipotesvexillifer,登录号:NC_007629)、和海牛目儒艮(Dugongdugon,登录号: NC_003314)作外群,根据鳍足亚目海豹科的2个亚科17条mtDNA序列,用邻接法构建13个编码蛋白的氨基酸序列系统关系树(图1),与最大似然法和最小进化法构建的系统树基本一致。结果显示辽东湾斑海豹首先和韩国斑海豹聚为一支,然后与阿拉斯加斑海豹聚为一支,3个斑海豹再与海豹亚科的其他海豹聚为一大支。僧海豹亚科的5个物种序列单独聚为一大支。根据13个编码蛋白的核苷酸与氨基酸序列构建的系统关系树结果基本一致,并且均显示辽东湾斑海豹与韩国斑海豹的关系较阿拉斯加斑海豹更近。

图1 基于13种线粒体蛋白质的氨基酸序列的邻接法进化树中国辽东湾斑海豹(KT818831)、韩国斑海豹(FJ895151)、美国阿拉斯加斑海豹(NC_008430)、港海豹(NC_001325)、环斑海豹(NC_008433)、贝加尔环海豹(NC_008432)、里海环斑海豹(NC_008431)、灰海豹(NC_001602)、鞍纹海豹(NC_008429)、带纹海豹(NC_008428)、冠海豹(NC_008427)、髯海豹(NC_008426)、夏威夷僧海豹(NC_008421)、食蟹海豹(NC_008423)、韦德尔海豹(NC_008424)、豹形海豹(NC_008422)、南象海豹(NC_008422)、白鳍豚(NC_007629)和儒艮(NC_003314).
3 讨 论
3.1 斑海豹线粒体控制区异质性现象
异质性是指在有些脊椎动物,如中华鲟(Acipensersinensis)、弓鳍鱼(Amiacalva),体内存在多种重复序列数目不同的线粒体基因组。至于控制区异质型和多态性水平高的原因,有学者指出线粒体基因异质性中的重复序列可能形成发卡结构,发卡结构引起高频率回复突变,这也许是导致异质性形成的原因,当然也不排除父本mtDNA的渗漏造成异质性的可能性。同时异质性也是物种进化的原因,线粒体中的串联重复、存在的大量插入与缺失等是物种进化的特征[10-12]。
而本文通过比较分析我国辽东湾、韩国、美国阿拉斯加斑海豹线粒体基因组控制区发现,三者主要变化出现在控制区CSB-Ⅲ上游约500 bp序列中,其上有大量重复片段,且重复片段长度有差异,这些重复片段主要为:ACGTAC、GCACAC、ACGT、ACGC、AC、GC。Bahn等[13-14]在比较韩国与日本斑海豹的控制区57个单倍型时,未发现完全一致的序列。孙凡越[15]也对7头辽东湾斑海豹控制区序列进行了克隆测序,得到了146个单倍型,其中异质型数目占所测序列总数的92%,并发现序列差异的原因主要是存在6种重复单元(GTACACAC、GTACAC、GTAC、GCACACAC、GCACAC和GCAC),而各单倍型的重复单元及重复次数均不相等,由此造成了辽东湾斑海豹不同个体之间出现长度异质性。这一结论与本试验的研究结果较类似,说明我国辽东湾斑海豹的线粒体DNA异质性程度比较高,因此,OH重复单元的特征以及线粒体ND5基因的序列多态性,都有可能成为区分斑海豹个体和不同种群的重要分子标记。
3.2 我国辽东湾斑海豹与韩国、阿拉斯加斑海豹的亲缘关系
本文在比较分析斑海豹线粒体基因组蛋白编码基因的突变时,发现在5个突变蛋白质编码基因中,共有10个位点出现氨基酸变化,总突变核苷酸位点数为61个,由表3可见,辽东湾和韩国斑海豹的蛋白编码基因中,只出现1个氨基酸突变,证明这两个个体的序列比较保守;相对应的阿拉斯加与辽东湾、阿拉斯加与韩国的突变氨基酸位点数分别为6个、10个,突变核苷酸位点数分别为56个、58个。通过对斑海豹3个线粒体基因组蛋白编码基因的突变比较分析(表3)及系统进化分析(图1)可以看出,辽东湾和韩国斑海豹的序列同源性显著高于阿拉斯加与辽东湾、阿拉斯加与韩国,说明辽东湾和韩国斑海豹很可能来自同一个斑海豹种群。
北太平洋的北部和西部海域及其沿岸和岛屿都是斑海豹在世界上的主要分布区[16],美国阿拉斯加海域与中国、韩国沿海相距较远,而从韩家波等[17]的卫星信标跟踪斑海豹研究可以看出,我国辽东湾的斑海豹可以到达韩国西海岸的白翎岛,说明我国海域与韩国西海岸的斑海豹有交流,这进一步表明了我国辽东湾与韩国斑海豹较美国阿拉斯加斑海豹的亲缘关系更近。
[1] 邢晶晶. 分子遗传标记及其技术在水产生物中的研究与应用[J]. 水产学杂志, 2002, 14(2):61-70.
[2] Shaughnessy P D, Fay R H. A review of the taxonomy and nomenclature of North Pacific harbour seals[J]. J Zool, 1997,182(3):385-419.
[3] Won C, Yoo B H. Abundance, seasonal haul-out patterns and conservation of spotted sealPhocalarghaalong the coast of Bak-ryoung Island[J]. Oryx,2004,38(1):109-112.
[4] Sambrook J,Fritsch E F,Maniatis T. Molecular Cloning[M]. New York:Cold Spring Harbor Laboratory Press,1989.
[5] 韩家波,赫崇波,王强,等. 辽东湾斑海豹线粒体ND4、tRNA Arg、ND4L和ND3基因序列分析[J]. 水产科学, 2006,25(10):500-504.
[6] 韩家波,赫崇波,王效敏,等. 辽东湾斑海豹线粒体苏氨酸和脯氨酸及控制区部分序列分析[J]. 水产科学,2007,26(2):74-78.
[7] Gordon D, Abajian C, Green P. Consed: a graphical tool for sequence finishing [J]. Genome Res, 1998, 8(3):195-202.
[8] Wyman S K, Jansen R K, Boore J L. Automatic annotation of organellar genomes with DOGMA[J]. Bioinformatics, 2004, 20(17):3252-3255.
[9] Lowe T M, Eddy S R. tRNAscan-SE: a program for improved detection of transfer RNA genes in genomic sequence [J]. Nucleic Acids Res, 1997, 25(5): 955-964.
[10] 苏瑛. 动物mtDNA控制区及保守与异质[J]. 四川动物, 2005, 24(4):669-672.
[11] 王巍, 朱华, 胡红霞,等. 五种鲟鱼线粒体控制区异质性和系统发育分析[J]. 动物学研究, 2009, 30(5): 487-496.
[12] 肖海霞, 帕热哈提江·吾甫尔, 玉山江,等. 线粒体DNA在动物起源进化和遗传多样性中的应用[J]. 兽医导刊, 2015(12):39-40.
[13] Bahn S Y, Choi E H, Jang K H, et al. Complete mitochondrial genome of the Korean spotted sealPhocalargha(Mammalia, Pinnipedia, Phocidae): genetic differences betweenP.larghaandPhocavitulina[J]. Mitochondrial DNA, 2009, 20(5/6):105-106.
[14] Mizuno A W, Onuma M, Takahashi M, et al. Population genetic structure of the spotted sealPhocalarghaalong the Coast of Hokkaido, based on mitochondrial DNA sequences[J]. Zool Sci, 2003, 20(6): 783-788.
[15] 孙凡越. 辽东湾斑海豹种群遗传特征的研究 [D]. 大连:辽宁师范大学,2009.
[16] Rugh D J, Shelden K E W, Withrow D E. Spotted seals,Phocalargha, in Alaska[J]. Marine Fisheries Review, 1997, 59(1):1-18.
[17] 韩家波,鹿志创,田甲申,等. 基于卫星信标跟踪的斑海豹放流效果研究[J]. 兽类学报, 2013, 33(4): 300-307.
AnalysisandComparisonofCompleteMitochondrialGenomeinSpottedSealPhocalargha
GAO Xianggang1, LU Zhichuang1, TIAN Jiashen1, HAN Jiabo1, HE Chongbo1, SONG Xinran2
( 1. Key Lab of Marine Fishery Molecular Biology of Liaoning Province, Liaoning Ocean and Fisheries Science Research Institute, Dalian 116023, China; 2. Dalian Sun Asia Tourism Holding Co, Ltd, Dalian 116023, China )
The complete sequence of the mitochondrial genome of the spotted seals (Phocalargha) in the Liaodong Gulf has been determined by long and accurate polymerase chain reaction (LA-PCR) and primer walking technology. The genome is 16 754 bp in size, containing 2 ribosomal RNA genes, 13 protein-coding genes, 22 transfer RNA (tRNA) genes and one control region, demonstrating a structure basically in accordance with other marine mammals. The ND6, tRNAAla, tRNAAsn, tRNACys, tRNATyr, tRNASer(UCN), tRNAGluand tRNAProgenes were located on the light strand (L strand), and the remainders lied on the heavy strand (H strand). The comparative analysis of the seal mitochondrial genome among Liaodong, Korean and Alaskan populations revealed the existence of length differences and that the major variations exhibited in the form of repeat motifs were located in the control region. Notably, in the coding region of the three targeted populations, ND5 showed the maximal mutation degree in total of 18 mutant bases was detected, which resulted in four amino acid changes. The variant and phylogenetic analysis of protein-coding genes indicated that the Liaodong seal shared higher homology with Korean seal than with Alaskan seal. It is inferred that Liaodong seal and Korean seal probably belong to one population.
Phocalargha; mitochondrial genome; comparative structure;phylogeny
10.16378/j.cnki.1003-1111.2017.04.018
Q786
A
1003-1111(2017)04-0509-05
2016-07-01;
2016-08-16.
辽宁省海洋与渔业科研项目(201417);香港海洋公园保育基金资助项目(MM04-1516).
高祥刚(1980-),男,副研究员;研究方向:动物分子遗传学. E-mail:xiangganggao@163.com. 通讯作者: 赫崇波(1961-),男,研究员,博士;研究方向:动物分子遗传学. E-mail:chongbohe@163.com.

