一种基于本体语义驱动的开放生物医学数据集成方法
王 凯,刘玉文,2
(1.蚌埠医学院 卫生管理系,安徽 蚌埠 233030;2.中国科学技术大学 计算机科学与技术学院,安徽 合肥 230027)
一种基于本体语义驱动的开放生物医学数据集成方法
王 凯1,刘玉文1,2
(1.蚌埠医学院 卫生管理系,安徽 蚌埠 233030;2.中国科学技术大学 计算机科学与技术学院,安徽 合肥 230027)
生物医学研究通常需要结合大量异构数据,数据间的语义鸿沟限制了生物医学领域知识大规模融合与开发。语义Web使用机器可读的数据格式,为数据语义集成提供了可行的技术支持。本文提出了一种面向语义Web的开放式异构生物医学数据语义转换和集成方法,建立基于XML(Extensible Markup Language)数据实体与语义本体概念关系之间的映射关系集,包含不同类型的映射关系以及复杂数据变换模式,自动生成具备语义逻辑关系一致的映射数据集,实现多个异构数据源数据之间的互联与集成。试验结果表明,基于本体语义驱动的开放生物医学数据集成方法可以进一步提高计算机的异构数据理解能力,证明转换和集成异构生物医学数据信息是切实可行的。
语义本体;生物医学数据;映射;转换与集成
生物医学数据的异构性和分散性使得数据的检索和管理异常困难,主要存在领域数据资源的信息难以挖掘、异构数据类型和字段无法语义解释以及资源访问和查询错误率较高等问题。生物医学数据集成的目的是将重要的生物学数据最终能够应用到临床诊断活动中,并为诊疗工作提供必要的决策支持。因此,迫切需要找到能够识别异构数据资源的集成方法,消除语义鸿沟。目前使用较为广泛的数据语义转换方法是面向数据仓库以及联机分析处理(Online analytical processing ,OLAP)的XML数据和关系数据库处理。文献[1]提出了一种将XML元素转换成RDF(Resource Description Frame)语句的方法,实现数据格式的语义变化,但该方法无法实现XML的属性关系映射。文献[2]以XSD( XML Schemas Definition )和owl(Ontology Web Language)之间的映射关系为基础,通过应用相同的规则,构建RDF与 XML实例之间的关系映射。文献[3]提出基于XPath的数据转换机制,将XML格式数据转换成RDF格式。在关系数据库转换方面,W3C (World Wide Web)提出关系数据库到RDF的规范化变换图,改变数据格式。上述转换方法,由于没有考虑到数据的潜在语义信息,缺乏对数据核心语义信息的保留,导致转换后的数据语义信息流失率较高。
本文提出一种面向异构数据源的开放生物医学数据集成方法。通过与关系数据库、XML文档以及电子病历等数据载体的协同操作,产生基于领域本体的数据描述集,实现数据转换。数据集成过程由领域语义驱动,通过定义数据模式与本体间的映射,获取满足逻辑一致性的数据信息。结合动态集成机制采用多源数据集,创建原始数据语义库,用于合并包含在不同资源中的同一实体数据。本文的目标是转换和集成异构生物医学数据;构建面向领域知识驱动的映射规则。
1 生物医学数据集的半自动化建模方法
图1是单个输入数据资源的数据集成与转换框架。使用XML和关系数据库作为输入数据模型,通过定义基于数据输入约束规则和OWL本体之间的映射关系,建立统一的标识规则,确定属性与本体类的实例,该规则允许合并同一类的不同个体。此外,利用数据检测技术以及自动推理技术,检查OWL本体的一致性,避免创建逻辑不一致的内容。通常情况下,该方法可以扩展到任何包含实体、属性和关系的输入数据模型,输出RDF或OWL格式数据实例,如图1所示。
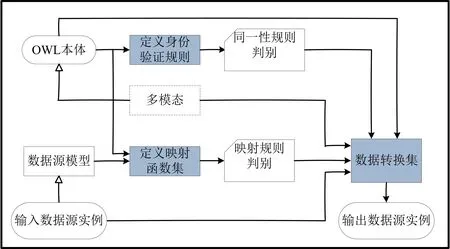
图1 数据集成与转换框架
2.1生物医学数据表示
病人电子健康记录存储了病人在医疗诊断和健康保健等过程中的大量有效信息。相关专家研究解决了电子病历的标准化和规范化问题,采用双建模层次构建信息模型,用于信息表示和规范化说明,制定了基于双模型架构的openEHR[4]以及ISO EN13606[5]等标准格式。标准格式结构的电子病历提供了通用化的信息表示模型,规范了数据的表达形式,有利于统一化信息的语义标准。电子病历数据的提取通常表示为一组XML文件,其内容应满足规定的约束条件。跨语义数据融合需要将数据转换成具有语义识别能力的语义格式。XML技术和关系数据库提供了定义数据集结构的基本模式和结构框架。本文基于XML schemas和关系数据模式定义数据的通用语义处理方法。
开放数据集中的数据在数据粒度、数值范围、规模以及来源等方面存在巨大差异,同时伴随着信息动态增长,数据差异呈现动态变化。万维网联盟开发了一系列用于数据交换的语义Web标准(如RDF),用于语义表示的形式化工具(如OWL本体语言),数据查询结构(如SPARQL)以及用于存储数据的语义机制(如RDF的存储架构triplestores)。自动描述逻辑推理机(如Hermit或 Pellet)可用于检查语义Web数据的一致性以及语义信息推理。开放数据集[6](Open data set)是基于语义Web数据标准下的语义信息存储、发布和共享的语义格式数据共享集合。开放数据集应满足四个基本要求:(1)基于URI的实体命名规则;(2)基于HTTP URI的数据查询格式;(3)面向语义Web标准的数据检索形式(如RDF和SPARQL);(4)面向数据发现的URIs链接。
2.2数据转换及映射规则
数据转换规则的核心是如何确定输入数据集的内容转化为语义格式,主要涉及两个方面:(1)输入数据是否按语义模式转化为语义格式;(2)输出数据集是否存在数据冗余。本节定义了两种主要类型的规则,即映射规则和同一性规则。
映射规则的定义将用图2所示的例子说明。该例采用基于orthoxml的标准化输入模式(图2左上)表示同源基因信息,同源领域知识模型用本体表示(图2右上)。用方框表示输入数据模式的实体,用@表示属性,用箭头表示关系。本体中的类使用圆角框表示,数据的属性使用实心箭头链接。利用映射规则建立实体、属性与本体类之间的语义关系以及数值类型属性和对象之间的语义关系。类和对象的属性通过虚线相连,表示从xml架构到本体的映射。本体包含一系列的前缀,其中 ro表示关系本体,ncbi表示NCBI 分类,cdao表示数据对比分析本体以及sio表示语义集成本体。
本方法需要转换实体、属性和关系,映射规则允许实现三个层次的一致性。为此,定义了三种基本映射规则:
实体映射规则。它是指将输入实体映射到OWL本体中的类。允许在OWL本体中创建个体实例。若S表示标准输入模式实体,T表示目标本体的类,则实体映射函数entity_rule(S,T)表示对任何实例 ,存在一个符合一致性约束的个体t与之对应。如图2中的实体映射规则实现基于XML架构的元素基因和本体中的基因类的映射链接。实体映射规则允许使用条件语句,进行补充定义,只将某些满足特定属性值的实例进行转换。若A1是与S相关联的属性, C1在A1条件下的布尔变量,则entity_rule(S,T,C1)表示对任何实例 ,总存在C1不为假的条件下的一致性约束的个体t∈T。
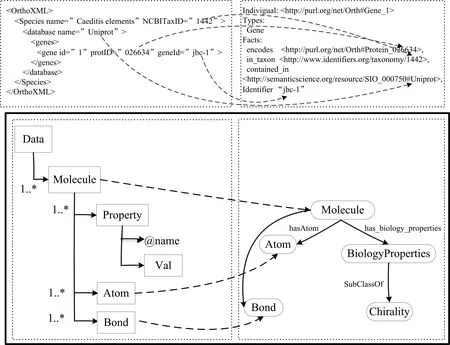
图2 OrthoXML与本体实例映射关系图
属性映射规则。它是指将实体中的属性映射到OWL本体类中的数值型属性。允许在本体中指定数值型属性的数值。设S是输入模式的一个实体,T是一个本体类,属性A1、A2是与S和T分别关联的数值型属性,则该映射函数attribute_rule((S,A1),(T,A2))表示对实体S中任何与A1相关联的实例,通过映射,总能在本体的类T中找到与数值型属性A2相关联的一致性个体T,且A1和A2具有相同的属性值。如图2中的属性映射规则表示OrthoXML中元基因属性id与本体基因类中数值型属性Identifier映射链接。
关系映射规则。它是指将两个实体的关联关系映射到OWL本体中两个类之间的对象属性关系。若实体S1和S2通过关系R1相关联,本体类T1和T2通过对象属性R2相关联,则该映射函数relation_rule((S1,R1,S2),(T1,R2,T2))是对任意给定的S1、S2的关联关系R1,实体映射函数entity_rule(S1,T1)和entity_rule(S2,T2),均存在一个对象属性R2,使得类T1、T2分别与其构成关联关系。如图2中的关系映射规则将XML模式中的物种与基因之间的层次关系映射到本体中的对象属性关系in_taxon RO。
2.3同一性规则判别
同一性规则定义的对象是数据类型属性以及对象属性,在本体中实现对个体的区分。目的是为了防止重复内容的创建以及支持面向多数据源的数据整合。同一性规则能够区别不同的URI实体。
若IR是数据类型属性集或本体类C的对象属性集, identity_rule(C,IR)表示在C中的所有实例与IR中的元素具有相同的值。利用数值型属性identifier以及对象属性定义如下同一性规则
(1)检索和执行基本实体规则。为本体中的所有类生成一组新的实例集I。
(2)每一个转换模态组代表了一组新添加的实例集,需要添加到I上,通过检查他们的定义,区别每组模态信息。
(3)对于集合的每个实例,执行如下过程:模式内剩余的其他说明语句也将被执行以添加附加语义内容;检索和执行基本属性规则,对实例的数值型属性赋值以及将对象属性实例化;检查同一性规则,如果实例是唯一的,则将其添加到输出数据集;否则,将其合并或链接到一个等效的元素上。

表1 蛋白质的模式化定义
2.4数据集成
面向异构资源的数据集成模型以相同的OWL本体作为数据驱动,采用上述数据转换规则处理不同来源的异构数据。集成核心内容是将XML模式数据映射到OWL本体,OWL本体可能包含一系列相关联的本体转换模态,以支持集成过程。使用数据集成模型有利于重用不同资源的转换规则,降低输入数据的结构异质性。表2显示了使用的OWL本体中定义蛋白质的模式用例,这种模式不仅降低用户在构建数据模型时对本体结构认知所产生的数据偏差,而且只需做少量修改就能实现以不同方式存储关系蛋白CDS转录数据,而不需要提前在输入模式时加以定义。表2显示了在处理与直接蛋白质转录没有关系的数据资源时,如何从变量 protein中设置参数变量 cds。

表2 蛋白质的修正模式化定义(不含CDs)
通过对每个输入资源进行数据变换来实现数据集成,利用映射规则生成OWL本体,并且在转换过程中应用同一性规则限制数据冗余,同时合并数据实例,确定来自不同数据源的实例是否对应于同一个实例域,合并具有相同URI的数据实例。
数据集成模型所处理的核心内容描述如下:
◆命名冲突:不同的输入模式数据可能使用不同的术语表达相同的数据元素(即实体、属性与关系)[7]。从不同的XML资源到OWL本体的映射解决了输出本体中通用词汇的集成问题。
◆数据冗余:多个数据输入资源实例可能描述同一个实体域,并被映射到OWL本体的同一类中[8]。同一性规则能够检测上述冗余情况,通过实体合并或链接到相应的OWL数据,以减少数据规模。
◆数据不一致:数据缺失会引起数据的不一致问题。对于给定的实体,相比于OWL本体,XML数据模式可能存储较少的属性和关系信息。在数据映射时,XML数据可能只对部分OWL本体实例产生语义关系,会导致数据部分缺失,带来OWL知识库的不一致。本模型采取的处理方法是:当检测到这种情况时,将不转换相应的源数据,从而防止不一致的发生。为降低该方法所带来的数据信息缺失量,将未参与映射的剩余本体实例数据添加到映射结果集。
◆资源之间的差异:由于不同的数据资源可能生成同一个OWL实例,其共同的属性或关系数值可能不同。这可能是在使用同一性规则时,未将来自不同资源的信息实体区别标注,导致属性间存在假性关联。在这种情况下,增加关系映射后验条件判断,若该实体的存在会引起知识库的不一致性,则它们被认为是不同的个体,分别生成各自的本体实例。
3 实验设计
在本节中,首先将描述实现转换方法的整体实验设计。其次将介绍如何将该模型用于不同的生物医学场景。
3.1实验用例
通过从生物医学领域选取典型数据电子病历,分析本模型所提出的方法在数据映射、转换以及集成等环节的数据整合效果。
电子病历数据涉及医疗系统的数字化信息,本实验用例选取超过2000名结直肠癌患者的电子病历数据,采用去隐私化技术隐去敏感字段,将数据转化成XML语义格式。使用自动推理方法确定每个病人的风险水平。采用领域本体技术将XML转换成openEHR格式数据,其中组织病理学报告的模式化定义如表3所示。这一模式定义了基于领域本体的组织病理报告数据类,包含一个结果集(hasfinding)记录、发现腺瘤总数以及腺瘤的大小。

表3 组织病理学报告的模式化定义
3.2设计内容
通过MySQL数据库,将XML Schema和ADL作为输入数据模式。输出数据集使用OWL或RDF格式,用户可以定义输入模式以及OWL本体之间的映射关系集。为此,在其他转换过程中允许创建映射上载和重用。一旦映射被定义,可以顺序执行,从而产生相应的RDF或OWL格式的数据内容。应用映射规则的数据源生成的语义内容,通过一致性规则约束保证不产生多余的数据信息;通过自动推理模块,以确保转化的内容具有逻辑一致性。采用OWLAPI[9]和Jena API来处理和生成的RDF和OWL数据,使用Hermit推理机[10]作为语义数据推理工具。
图3显示了映射接口的核心部分,包含三个主要部分。左侧使用分层关系表示数据输入模式。右侧对应OWL本体。图的下部是一个文本框,包含定义的映射规则,如第三行定义了从实体molecule类的coorddimension属性到本体Molecule类的数据类型属性coord_dimension的映射关系。
图4是将XML输入模式的实体映射到转换模式的定义系统截图。图的左边是输入模式openEHR原型,被映射到以本体形式表示的转换模式组织病理学报告中。图中可以看到,该映射与原型模式的各变量的特定元素产生关联关系。

图3 映射接口关系图
4 总结与分析
本模型相比较于rdb-owl的手工定义映射模型,不会受限于关联格式输入数据,且处理复杂的本体或异构源数据的集成能力较强。比较于Karma的半自动database-ontology数据集成模型,对先前映射过程的知识基础依赖度较小,适合处理规模较大的领域知识集。以数据仓库为导向的集成方法,集成数据语义链接功能,通过定义数据转换规则与映射规则,允许定义外部数据集。与bio-rdf模型所不同的是,本模型的语料库包含来自多个数据源的数据,语义信息更加丰富,集成后的数据信息可操作性较强。将减少关系数据或XML数据源的约束条件,只需要定义映射的主要规则,通过数据语义转换,实现半自动化数据集成,并通过同一性规则检查,降低数据冗余度,提高映射集数据质量与可靠性。

图4 XML输入模式的实体映射到转换模式的定义系统截图
生物医学数据集的开放性以及语义格式的可用性,将有利于生物医学数据的互操作。本文提出了一种基于本体的异构数据源转换与集成模型。较其他方法有以下改进:采用基于数据仓库的数据转换方法。首先,面向语义Web的生物医学数据需要开发程序具备兼容bio rdf或EBI的RDF平台的数据接口,数据语义仓库能够满足语义资源池的基本条件,即包含LOD的可用性资源DF和OWL。其次,在生成OWL知识库的同时,需要使用OWL DL的推理机制,实现数据的一致性处理以及降低数据冗余度,使获得的数据集链接能够使用较小的系统开销,完成外部资源的数据融合。第三,为解决OBDA方法不便于应用于本体与XML模式映射的问题,使用数据语义仓库能够丰富的数据语义表示,提高数据映射的逻辑准确性。
[1] Galperin M Y, Rigden D J, Fernández-Suárez XM. Nucleic Acids Research Database Issue and Molecular Biology Database Collection[J]. Nucleic Acids Res, 2015:112-120.
[2] Tapuria A, Kalra D, Kobayashi S. Contribution of Clinical Archetypes, and the Challenges, towards Achieving Semantic Interoperability for EHRs[J]. Healthcare Informatics Research, 2013, 19: 286-293.
[3] Jupp S, Malone J, Bolleman J, et al. The EBI RDF platform: linked open data for the life sciences[J].Bioinformatics, 2014, 30:1338-1345.
[4] Wang Y, Tao J, et al. Information retrieval and data mining based on open network knowledge[J].Journal of Computer Research and Development, 2014, 52: 456-474.
[5] Evangelista A T, Hassanien A E .Dimensionality reduction of medical big data using neural-fuzzy classifier[J].Soft Computer, 2014, 19: 1115-1122.
[6] Abello A, Romero O, Bach Pedersen T, Berlanga R, Nebot V, Aramburu MJ, Simitsis A. Using Semantic Web technologies for exploratory OLAP: a survey[J]. IEEE Transactions on Knowledge and Data Engineering, 2015(2): 571-585.
[7] 李勇,张志刚.基于本体语义检索技术研究[J].计算机工程与科学,2015(4): 17-19.
[8] 刘宇鹏,李生,赵铁军.基于WordNet 词义消歧的系统融合[J].自动化学报,2014(11): 1575-1580.
[10] Martínez-Costa C, Schulz S. Ontology content patterns as bridge for the semantic representation of clinical information[J]. Applied clinical informatics, 2014: 660-668.
(责任编辑:熊文涛)
AnIntegrationMethodofOpenBiomedicalDrivenbyDataSemanticOntology
Wang Kai1, Liu Yuwen1,2
(1.DepartmentofHealthManagement,BengbuMedicalCollege,Bengbu,Anhui233030,China;2.SchoolofComputerScienceandTechnology,UniversityofScienceandTechnologyofChina,Hefei,Anhui230027,China)
Biomedical research usually requires a large number of heterogeneous data. The semantic gap between data limits the large-scale integration and development of biomedical knowledge. Semantic Web provides a feasible technical support for data semantic integration using the machine-readable data format. This paper presents a method for Semantic Web oriented open semantic heterogeneous biomedical data conversion and integration. In this approach, the mapping relationship between XML data entity and the concept of ontology based semantic set is established to obtain the mapping relationship between different types and complicated data transformation model. The semantic logical relation mapping data consistent set is automatic generated automatically to achieve interoperability between data from heterogeneous data sources and integration. Experimental results show that the integrated method of open biomedical data ontology driven by the heterogeneous data for computer to further improve the understanding. It is verified to be feasible for the transformation and integration of heterogeneous biomedical data.
semantic ontology; biomedical data; mapping; transformation and integration
TP391
A
2095-4824(2017)06-0078-07
2017-02-25
安徽省高校自然科学一般项目(KJ2015B023by);蚌埠医学院自然科学重点项目(Byky1411ZD)
王 凯(1985- ),男,安徽蚌埠人,蚌埠医学院卫生管理系讲师,硕士。
刘玉文(1982- ),男,安徽凤阳人,蚌埠医学院卫生管理系讲师,中国科技大学计算机科学与技术学院访问学者,硕士。

