血型分子分型技术的进展和应用
赵桐茂
血型分子分型技术的进展和应用
赵桐茂
血型基因分型 血型基因组学 分子生物学技术
1900年人类红细胞血型被发现,开启了免疫学的一个分支免疫血液学,使用经典的血清学方法检测血型。20世纪下半期,随着基因研究和分子生物学技术的快速发展,出现了分子免疫血液学,在分子水平研究血型抗原和血型基因的分子结构。21世纪初整个生物医学领域进入了后基因组时代,一门新的血型基因组学(Blood Group Genomics)学科应运而生,建立了以DNA为基础的血型分子分型技术。虽然红细胞凝集技术可以很好地检测常见血型抗原,但是对于鉴定比较复杂的抗原变异体,单独使用血清学方法能力有限,为此需要分子生物学技术作为一个辅助工具。本综述介绍血型分子分型技术最新进展及其在临床输血中的应用,其中包括一些分子遗传学和血型基因多态性的基础知识。
1 分子遗传学基础
1.1 DNA 细胞是具有生命功能的基本单位。真核细胞含有细胞核和细胞器,细胞核内的DNA与组蛋白结合成染色体。DNA是脱氧核糖核酸的简称,是染色体的主要化学成分,同时也是组成遗传密码的物质。DNA的化学成份是磷酸、脱氧核糖和硷基。1个磷酸分子、1个脱氧核糖和1个硷基组成1个单核苷酸,单核苷酸是构建DNA分子的基本单位。总共有腺嘌呤(A)、鸟嘌呤(G)、胞嘧啶(C)和胸腺嘧啶(T)等4种碱基。核苷酸通过三磷酸根互相连接形成单股多聚核苷酸链。单股DNA的一端称为5'端,另一端称为3'端,两端以外有不同的DNA序列。DNA分子是由2条单核苷酸链以互补配对原则所构成的双螺旋结构,1条单链的5'端对应另一条单链的3'端。2条DNA链中的对应碱基A-T以双氢键形式连接,C-G以三氢键形式连接。
1.2 基因结构 人类基因一般可以分为4个区域:①转录区:该区域包含外显子与内含子,其两侧被称为侧翼序列,分别用5'UTR 和3'UTR表示;②前导区:位于基因编码区上游,相当于RNA的5'末端非编码区(非翻译区);③尾部区:位于RNA的3'编码区下游,相当于末端非编码区;④调控区:位于基因编码区域两侧,含有启动子和增强子等基因调控序列(图1)。 启动子包括某些保守序列,能促进转录过程。真核基因转录起始点的上游或下游一般都有增强子,不能启动基因转录,但有增强转录的功能。不同基因的大小差异甚大,比如LW血型基因跨度仅2 600碱(bp),而编码S抗原的GYPB基因全长58 000bp,该基因含有较长的内含子序列,实际编码序列仅为276bp,仅占全部序列的0.5%。基因中的编码片段又称为外显子,其DNA序列决定编码产生的氨基酸多肽序列。外显子被非编码的内含子序列隔离开。外显子和内含子接头区都有一段高度保守序列,内含子5'端大多数是gt开始,3'端大多是ag结束,称为gt-ag法则,是普遍存在于真核基因中RNA剪接的识别信号。
1.3 从基因到蛋白质 从基因到蛋白质遗传信息传递的第1步是DNA被转录为mRNA。基因转录在细胞核内进行,其中转移RNA(tRNA)的合成发生在核仁,mRNA的的合成在核质中进行。转录过程首先是以DNA的1条链为模板,按照碱基互补配对原则转录成前mRNA。然后通过剪接加工,内含子片段被去除,外显子片段连接在一起产生mRNA。mRNA只含有外显子顺序,保留了编码序列的连续性(图1)。
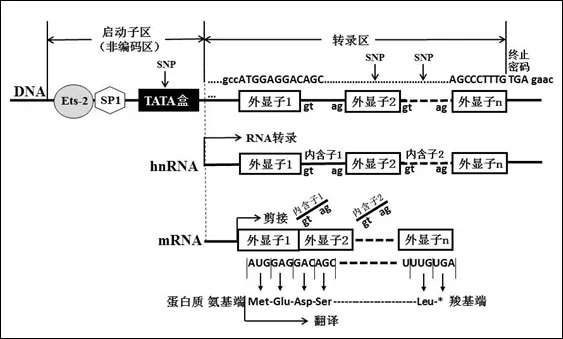
图1 基因结构和基因转录示意图
启动子区属于非编码区,含有多种控制基因转录的转录因子,如Ets-2、SP1、TATA盒等。异质核RNA(hnRNA)又称为前mRNA,是RNA转录产物。一旦开始转录,hnRNA被剪接以去除内含子侧翼部分gt ... ag的非编码序列,留下只有外显子的编码序列,因此mRNA核糖核酸序列中含有正确的阅读框。从转录起始位点开始,三联密码子通过核糖体机制翻译成多肽。按照惯例第1个核苷酸(+1)密码子AUG翻译成第1个氨基酸甲硫氨酸(Met),但是基因最终产物可以在细胞表达过程中被切割,因此成熟蛋白质的第一氨基酸可以不是甲硫氨酸。在转录中遇到UGA,UAG,UAA等终止密码子时转录将终止,不编码任何氨基酸,一般用*号表示。SNP的位置可以在启动子区、外显子和内含子等区域,可以有1个或多个。
1.4 基因表达调控 基因表达受到转录起始点上游的启动子DNA序列的调控。启动子可以延伸至上游1千多个碱基,其他影响转录的调节因子序列可以在更远的位置上。位于启动子区域的TATA盒存在于大多数基因中,其功能是将RNA聚合酶定位于适当位置以起始转录。启动子区域的突变有可能导致失去起始转录能力,结果无转录产物。如Duffy血型基因启动子TATA盒内序列突变,导致不能产生Duffy血型抗原蛋白。
1.5 等位基因和SNP 在同1个遗传位点上,由于基因突变产生的变异体被称为等位基因,由此产生遗传多态性。单核苷酸取代产生的多态性简称SNP(Single Nucleotide Polymorphism,SNP),是最常见的基因突变。如果单核苷酸取代没有改变所编码的氨基酸,称为沉默取代;改变所编码的氨基酸称为错义取代;如果产生终止信号,称为无义取代。单核苷酸的缺失或插入也可以产生新的DNA序列,或产生终止密码(图2)。

图2 产生单核苷酸多态性示意图
1.6 其他基因突变 由于DNA分子中碱基对的改变、DNA片段增添或缺失、基因转换和基因重组等机制引起的基因结构的改变,可以统称为基因突变,一些常见的基因变异机制见图3。基因突变通常发生在DNA复制时期,即细胞分裂间期,包括有丝分裂间期和减数分裂间期。在进化中,一些基因序列由于突变而不能产生相应的蛋白质,这些无功能的基因被称为假基因。
2 血型多态性分子基础 目前被国际输血协会(ISBT)认可的红细胞血型抗原总数为346个,其中308个分属36个血型系统,其余38个抗原尚未被归类。几乎所有具有临床意义的血型系统的分子基础已被阐明,并已开发出相应的分子检测技术[1]。根据美国国家生物技术中心的血型基因突变数据库(Blood Group antigen gene Mutation Database,BGMUT)统计,至2017年4月已检测出45个血型基因,共含有1 802个等位基因[2]。
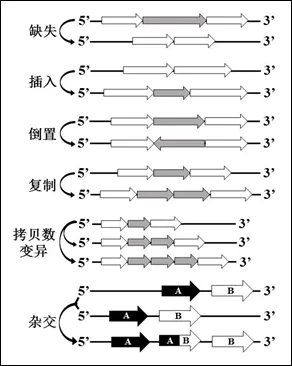
图3 常见基因变异示意图
2.1 多态性产生机制 36个血型系统遗传多态性的产生机制大致可以分为10种类型[3],在表1中用数字1~10表示。数字1代表最常见的单核苷酸取代产生的 SNP,可以发生在单个外显子、多个外显子、内含子以及基因调节区域。外显子中发生的SNP,可以产生同义突变、错义突变和无义突变等3种结果。如编码红细胞Lua和Lub、Aua和Aub、K1和K2、Jka和Jkb、Dia和Dib,以及Doa和Dob等所谓对偶抗原的等位基因,都是由于单个碱基取代而产生。数字2 代表缺失,最典型的例子是白种人中RhD阴性个体缺失整个RHD基因。数字3表示存在插入1个或多个核苷酸碱基。如黑人中,RHD基因内含子3和外显子4之间有1段37bp的插入片段,导致产生RhD阴性表型。数字4代表基因重复作用,如C4A和C4B基因之间的不等交换,使1条单体型带有重复的C4A基因片段。数字5代表基因重排,如Gerbich血型基因含有的4个外显子,通过重排产生新的基因型和新的表型。数字6表示内含子SNP造成信息RNA的GT和AG剪接位点突变,不能产生正常的多肽分子。在MMS和Rh血型系统中常见的基因重组和杂交基因用数字7表示。数字8表示基因转换作用;数字9代表不等重组作用;由于转录产物不同产生的多态性用数字10表示。

表1 红细胞血型基因多态性机制
2.2 无效型表型的遗传型 无效型个体的红细胞表面不表达相应抗原,如果接受输血通常会产生抗体。无效型个体的群体频率较低,为满足这类群体的输血,需要预先筛选无效型供者。由于相应抗体来源一般有限,所以多采用血型分子分型方法。产生无效型的机制包括:①转录突变。如在表型Fy(a-b-)的黑人中,由于红细胞转录因子GATA中-46位置上T>C突变,改变增强子GATA结合位点,结果不能产生Duffy 糖蛋白;②表型S-s-、Gy(a-)、Dr(a-)等缺失型的产生,是由于转录过程中剪接位点核苷酸突变,使部分或全部外显子被跳过;③由于碱基缺失、插入或取代作用,导致阅读框架移位而产生终止信号,不能产生完整的蛋白质。如ABO血型中的O基因,由于外显子6中261位置上单核苷酸缺失,导致产生终止信号,不能产生完整的ABO转移酶;④表型Rhnull、Ko、McLeod是由于核苷酸错意突变而改变了氨基酸序列;⑤ 表型Co(a-b-)是由于错意突变而降低蛋白质的表达;⑥Rh血型中的RhD缺失型是由于基因交换或基因转换而产生;⑦Kell和Ge抗原弱表达涉及蛋白相互作用,他们分别缺少Kx和4.1蛋白;⑧ 由于修饰基因的作用,产生Lu(a-b-)、Jk(a-b-)等表型。
3 血型分子分型技术
3.1 聚合酶链反应 聚合酶链反应 (Polymerase Chain Reaction,PCR)是在体外大量扩增DNA片段的快速方法。PCR需要下列材料:①耐热的DNA聚合酶,在高温时不被破坏。最常用的是Taq聚合酶;②纯化的DNA样板;③4种三磷酸碱基脱氧核苷酸 (dATP、dCTP、dGTP、dTTP),它们是合成DNA的原料;④人工合成的寡核苷酸PCR引物。PCR扩增需要使用1对引物,它们连接在被复制的DNA的两侧,与其方向相反的1股DNA杂交,因此2股DNA可以同时被复制。在5'端的引物,被称为正向引物,或编码引物;在3'端的引物,被称为反向引物,或非编码引物。引物序列必须与它们的靶序列完全互补(图4)。
PCR反应一般需要4个步骤:①DNA变性。DNA被加热到94℃ ~ 96℃,变性产生单股DNA样板;②引物退火。根据引物的变性温度Tm,降低反应温度,使引物与其序列匹配的DNA特异性地结合;③DNA的延伸。在72℃左右合成新的DNA;④然后循环重复①~③程序,一般进行30~40个循环,可以将1个DNA片段扩增为上百万个拷贝。

图4 寡核苷酸引物设计示意图
3.2 PCR-SSP技术 采用序列特异性引物(Sequence Specific Primer,SSP)进行PCR扩增。SSP的3’端具有独一无二的序列,在退火时只能与某特定等位基因结合,因此能够特异性地扩增该基因片段。检测PCR扩增产物的方法包括:直接凝胶电泳、限制性内切酶水解后凝胶电泳、酶联免疫吸附等。使用实时 (real time)PCR扩增仪,结合使用荧光标记的探针,可以实时记录PCR反应过程中荧光强度的变化,根据熔解温度曲线可以检测特定的等位基因,此法又称为TaqMan分析,具有敏感度高、可自动记录分析结果等特点。目前已报告的血型基因分型方法多以PCR-SSP为基础,它属于低至中等通量检测技术。根据PCR引物的设计、反应温度、聚合酶的性质、产品的检测方法、扩增仪的类型,有多种检测方法(表2)[4-7]。
3.3 PCR-SSOP和基因芯片技术 基因芯片又称为DNA 芯片、寡核苷酸阵列、杂交阵列等,是基于核酸链之间分子杂交的一种技术。该方法的第1步是使用PCR扩增某段待检基因片段,然后通过与序列特异性寡核苷酸探针(Sequence Specific Oligonucleotide Probes,SSOP)的杂交来鉴定相应基因,这个技术又称为PCR-SSOP。杂交反应可以在尼龙薄膜、玻璃或塑料片等固体支持物上进行。探针用荧光标记,通过化学显色或荧光信号检测显示出杂交结果。在芯片表面能够固定数千个寡核苷酸探针,对多个待测基因进行检测。在Luminex 流式荧光技术中,SSOP探针和数百种微球偶联,1次反应可同时鉴定100多个等位基因。
3.4 DNA测序技术 测序技术可以提供更精确的DNA碱基序列信息,基因检测的金标准。在以上介绍的PCR-SSP和PCR-SSOP技术中,需要知道突变基因的DNA序列,才可以制备相应的引物或探针,因此不适合检测未知DNA序列的基因突变,或是筛选基因突变。而测序技术有可能发现新的等位基因。测定DNA序列可以使用两种检材:①如果以RNA为检材,首先需要通过反转录制备互补cDNA,再以cDNA为模板通过PCR扩增,然后对扩增产物测序;②如果以DNA为检材,可以直接通过PCR扩增特定基因片段,然后对扩增产物测序,可以检测出包括编码区和非编码区的整个基因序列,通过和基因参考品序列的比对可以发现基因突变。

表2 以PCR为基础的血型基因检测技术
3.4.1 PCR-SBT技术 为了取得足够数量的基因拷贝用于测定序列,受检基因组DNA样品一般先用PCR扩增,然后对扩增的DNA模板测序,被称为PCR-SBT (Sequence Based Typing,SBT)技术。由于PCR扩增产物含有2条链DNA,测序结果是2条链序列的加合结果,一般需要计算机软件协助分析。该技术的缺点是可能产生模棱两可的分型结果。
3.4.2 一代测序技术 一代测序方法包括Sanger末端终止测序法和焦磷酸测序法。改良的Sanger测序方法使用荧光染料标记的A,C,T,G 碱基的双脱氧核苷三磷酸,使用Taq酶在高温做PCR循环反应,或是使用Bst酶在低温做PCR循环反应,然后通过凝胶电泳检测DNA序列。焦磷酸测序法是通过依赖核苷酸掺入的焦焦磷酸盐释放来测定DNA序列。
3.4.3 二代测序技术 二代测序又称为下一代测序(Next generation sequencing,NGS),该技术采用微乳液PCR或桥式PCR等方法获得测序模板,DNA片段的PCR扩增产物聚集在微珠的表面,不需要电泳之类的物理分离方法来区分碱基序列。其原理是通过捕捉新合成的末端标记,边合成边确定DNA序列,是一种高通量测序技术,允许检测单独1条单体型的DNA序列,可以避免模棱两可的分型结果。虽然已经成功地用于某些血型基因分型,但是目前的NGS技术还不能准确区分诸如RHD / RHCE或GYPA / GYPB等基因的单倍型。
3.4.4 PCR-基因克隆测序技术 使用PCR扩增和分子克隆技术,将待测基因克隆到载体后再测序,每个克隆只含有1条单体型的基因片段。此法优点是不存在模棱两可的测序结果,缺点是比较费时费力,因此一般只用于鉴定新发现的等位基因。
3.5 高通量血型基因分型 高通量血型基因分型常用于大规模筛查携带罕见基因或抗原阴性血液。根据不同的工作原理,目前已经开发出多种高通量检测技术,表3只是介绍其中的一部分。这些技术的共同点是都需要专门的检测仪器,都检测血型基因SNP突变位点,除TaqMan技术外都采用多重PCR同时检测多种血型基因。美国Immucor公司的HEA BeadChip是首个获得美国食品暨药品管理局(FDA)批准的非ABO/非RhD血型基因分型诊断试剂盒,该技术使用PCR扩增产物和携带约4 000个荧光探针的BeadChip基因芯片杂交,然后通过荧光显微镜检测[8]。ID CORE XT平台使用流式微阵列系统,是一个高度自动化的测试平台,允许在小型或更大型样品批次上进行测试,实施相对容易。特别是Luminex平台已经在许多免疫血液学实验室中使用,除检测红细胞血型基因外,还用于检测HPA和HLA系统的基因[9]。
Thermo Fisher公司的SNaPshot技术,用户可以自行设计PCR引物,检测特定的SNP,不受厂商产品的限制[10]。Agena的HemoID使用基质辅助激光解吸/离子化飞行时间质谱仪(MALDI-TOF MS),这是一种敏感的分析方法,具有高通量和高重复性等优点,但是使用的仪器价格昂贵[11]。MRC-Holland的SALSA平台,使用多重连接依赖性探针扩增(multiplex ligation-dependent probe amplification,MLPA)。MLPA是一种易于应用的方法,只需要热循环仪和毛细管电泳设备,允许在单管反应中同时检测50多个多态性[12]。Thermo Fisher公司的QuantStudio平台,使用TaqMan基因分型技术,可以根据已知血型抗原SNP设计引物,筛查罕见的表型和罕见的抗原组合[13]。
4 血型分子分型的应用 红细胞血型抗原是能够刺激免疫系统产生抗体的红细胞表面遗传标记,其化学成分可以是蛋白质或碳水化合物多糖。编码血型系统的1个遗传位点上,可以有1对或多个,乃至数百个等位基因,因此血型抗原通常表现出多种变异体。有时使用血型血清学方法难以区分和鉴定这些变异体,如在临床输血上有重要意义的Rh弱D抗原,已检测出的变异体有130余种[2],没有相应的特异性抗体来区分它们,必须使用分子分型办法来鉴定。另外,为了在血库中保存稀有血型供者,包括罕见的抗原阴性(无效型表型)供者,需要预先在大量供者群体中进行筛查,由于相应抗体试剂来源的限制,也需要采用分子分型方法。血型分子分型的临床应用大致可以归纳为患者和供者两方面,临床方面主要解决ABO/Rh变异体和ABO和Rh血型以外的相容型输血问题,供者方面主要筛查罕见血型供者(表4)[14]。
4.1 疑难血型鉴定 鉴定红细胞直接抗人球蛋白试验阳性和具有多凝集作用红细胞的血型,通常都会遇到麻烦,在这种情况下可以使用基因分型技术鉴定血型。对于新近输血患者或多次输血患者,由于外周血中含有供者的血液细胞,使用经典的红细胞凝集试验不能正确鉴定血型,也可以使用基因分型方法鉴定[15]。
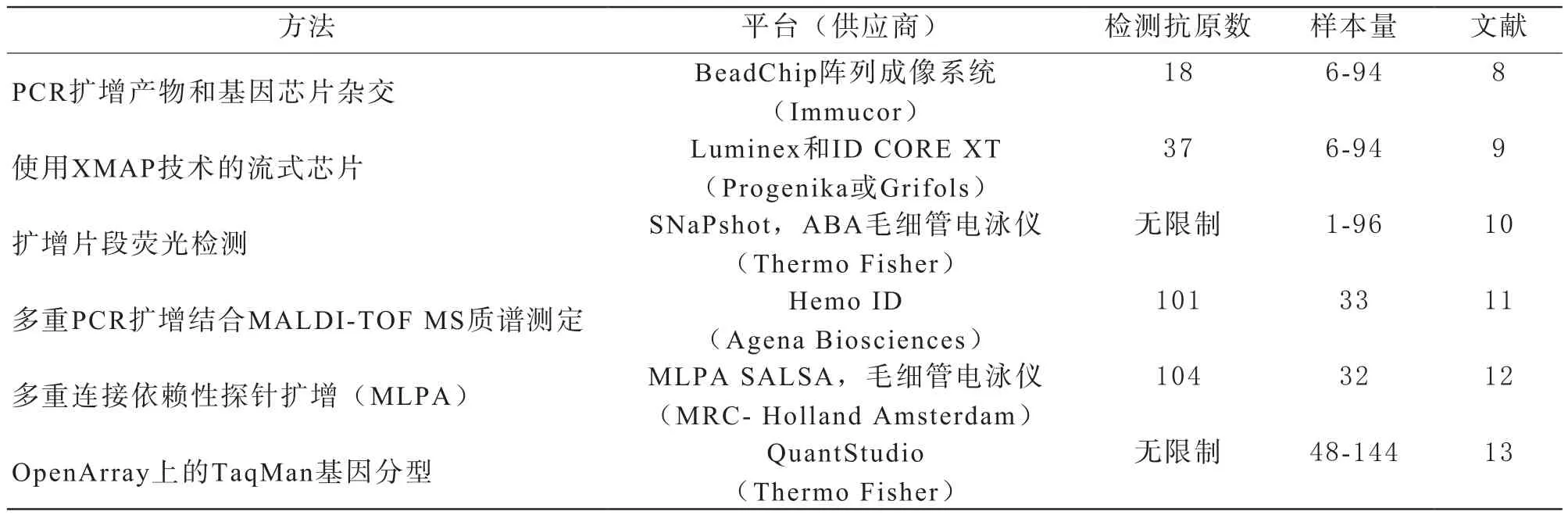
表3 高通量血型基因分型技术

表4 血型分子分型技术在预测供者血型和鉴定患者血型中的应用
4.2 筛查罕见血型和无效型供者 筛选罕见血型供者需要大量标准抗血清,在缺少相应抗血清的情况下,可以使用DNA分型技术进行筛选。无效型个体比较少见,通常是在患者已经产生抗体后才被发现,这些抗体一般对应高频率抗原。现在可以在输血前对患者做无效型基因检测,确认患者是否是无效型,以避免输血后产生抗体[16,17]。
4.3 无创伤胎儿血型鉴定 胎儿血型鉴定对新生儿溶血病的产前诊断有重要意义。过去在产前抽取羊水,再从中取得胎儿细胞做血型鉴定,是一种有危险性的临床手术,不易被产妇接受。近年来发现在妊娠期以及产后3 d内,可以从母亲血浆或血清中提取游离的胎儿DNA,使用高敏感度的实时PCR扩增等技术,可以鉴定胎儿的血型。这个无创伤胎儿血型产前鉴定技术,已成功地鉴定了胎儿ABO、RhD、Rh CE、Kell和Duffy等血型基因[18,19]。在使用基因分型技术鉴定胎儿RHD基因时应该注意,由于不同种族的RHD基因结构不尽相同,相应的基因分型方法也有所不同。如在白种人中,RhD阴性个体主要是由于缺失整个RHD基因。而在东方人中,RhD阴性个体除缺失整个RHD基因外,也可以是缺失RHD基因中的某些外显子,或RHD基因中的点突变等多种机制。
4.4 分子分型预测血型的局限性 虽然基于DNA的分子分型对预测血型有很大的价值,但是也有其局限性[5]。这主要涉及到检测技术和受检样品等两方面因素:①血型分子分型的前提是已知各等位基因的DNA序列,已知血型基因型和血型表型之间的相互关系。由于不同种族血型变异体遗传背景不尽相同,对于未知群体的分子分型应谨慎行事;②DNA分析一般检测特定位置的SNP,但是如果其它位置的SNP可以导致编码抗原不表达,在该SNP纯合子时将产生无效型表型,这时的基因分型结果可能被错误鉴定为抗原阳性。对于供者来说,假阳性结果意味着将错失有价值的抗原阴性血液,但不会危及受血者。但是对于患者,将无效型表型误定为抗原阳性,接受阳性血液输注可能产生抗体。无效表型的确认需要借助血清学方法,使用相应抗体做血细胞凝集试验,和/或使用交叉配型方法检测抗体/抗原的不相容性;③在1个位点上有多个等位基因的情况下,虽然红细胞表达相应抗原,但是DNA分析可能无法检测出探针/引物结合点,或限制酶切割点发生改变的等位基因;④在ABO、Rh等血型系统中,大量的等位基因编码同一种表型,不可能检测出所有的等位基因。对于某些杂交等位基因,特别是MNS和RH系统的基因,可能会产生假阳性或假阴性结果;⑤在杂合子个体中,某一个等位基因可能被优先扩增,因而漏检另外一个等位基因;⑥由于输血或干细胞移植产生血型嵌合体的情况,和天然产生的嵌合体难以区别。
1 Storry JR,Castilho L,Chen Q,et al. International society of blood transfusion working party on red cell immunogenetics and terminology:report of the Seoul and London meetings [S]. ISBT Science Series,2016,11:118-122.
2 T h e B l o o d G r o u p A n t i g e n G e n e M u t a t i o n Databasehttps://www.ncbi.nlm.nih.gov/projects/gv/mhc/xslcgi.cgi?cmd=bgmut/home [EB/OL],[2017-05-26] .
3 Denomme GA,Molecular basis of blood group expression [J]. TransfusApherSci,2011,44(1):53-63.
4 Monteiro F,Tavares G,Ferreira M,et al.Technologies involved in molecular blood group genotyping [S]. ISBT Science Series,2011,6:1-6.
5 Reid ME,Denomme GA. DNA-based methods in the immunohematology reference laboratory [J].TransfusApherSci,2011,44(1):65-72.
6 Jungbauer C,Hobel CM,Schwartz DW,et al.Highthroughput multiplex PCR genotyping for 35 red blood cell antigens in blood donors [J]. Vox Sang,2012,102(3):234-242.
7 St-Louis M,Molecular blood grouping of donors [J].TransfusApherSci,2014,50(2):175-182.
8 Paccapelo C,Truglio F,Villa MA,et al. HEA BeadChip™ technology in immunohematology [J].Immunohematology,2015,31(2):81-90.
9 Goldman M,Nogués N,Castilho LM. An overview of the Progenika ID CORE XT:an automated genotyping platform based on a fluidic microarray system [J].Immunohematology,2015,31(2):62-68.
10 Latini FRM,Castilho LM. An overview of the use of SNaPshot for genotyping blood group antigens [J].Immunohematology,2015,31(2):53-57.
1 1 McBean RS,Hyland CA,Flower RL. Blood group genotyping:the power and limitations of the Hemo ID Panel and MassARRAY platform [J].Immunohematology,2015,31(2):75-80.
12 Veldhuisen B,van der Schoot CE,de Haas M.Multiplex ligation-dependent probe amplification(MLPA)assay for blood group genotyping,copy number quantification,and analysis of RH variants [J].Immunohematology,2015,31(2):58-61.
13 Denomme GA,Schanen MJ. Mass-scale donor red cell genotyping using real-time array technology [J].Immunohematology,2015,31(2):69-74.
14 Keller MA,The role of red cell genotyping in transfusion medicine [J]. Immunohematology,2015,31(2):49-53.
15 Johnsen JM. Using red blood cell genomics in transfusion medicine.Hematology Am SocHematolEduc Program. 2015 [S],2015:168-176.
16 Sandler SG,Horn T,Keller J,et al. A model for integrating molecular-based testing in transfusion services[J].Blood Transfus,2015,14(6):566-572.
1 7 Flegel WA,Gottschall JL,Denomme GA,Implementing mass-scale red cell genotyping at a blood center [J]. Transfusion,2015,55(11):2610-2615.
18 Wenqian S,Shihang Z,Linnan S,et al. Noninvasive fetal ABO genotyping in maternal plasma using realtime PCR [J]. ClinChem Lab Med,2015,53(12):1943-1950.
19 Picchiassi E,Di Renzo GC,Tarquini F,et al. Noninvasive prenatal RHD genotyping using cell-free fetal DNA from maternal plasma:an Italian experience [J].Transfus Med Hemother,2015,42(1):22-28.
R457.1+1 R392.11
A
1671-2587(2017)05-0530-07
10.3969/j.issn.1671-2587.2017.05.039
美国国立卫生研究院 (NIH)
赵桐茂 (1943–),男,吉林省长春市人,研究员,主要从事分子免疫血液学和分子免疫遗传学工作,(Tel)13585621829(E-mail)tomhla@163.com。
2016-05-28)
(本文编辑:姚萍)

