基因组选择技术及其在猪育种中的应用探讨
叶 健,郑恩琴,胡晓湘,王爱国,吴珍芳,2,蔡更元,2*
(1.华南农业大学动物科学学院,国家生猪种业工程技术研究中心,广东 广州 510642;2. 广东温氏食品集团股份有限公司,广东 新兴 527400;3. 中国农业大学生物学院,北京 100193;4.中国农业大学动物科技学院,北京 100193)
基因组选择技术及其在猪育种中的应用探讨
叶 健1,郑恩琴1,胡晓湘3,王爱国4,吴珍芳1,2,蔡更元1,2*
(1.华南农业大学动物科学学院,国家生猪种业工程技术研究中心,广东 广州 510642;2. 广东温氏食品集团股份有限公司,广东 新兴 527400;3. 中国农业大学生物学院,北京 100193;4.中国农业大学动物科技学院,北京 100193)
基因组选择(Genomic Selection,GS)技术是利用覆盖全基因组与性状相连锁的标记信息,通过标记效应的求解和加和,得到个体基因组估计育种值(GEBV),从而达到对畜禽个体进行准确选择的目的.该技术率先在奶牛育种中得到广泛应用.在猪育种中,以杜洛克猪为代表,基因组选择技术的应用可以达到早期选择和提高选择准确性的效果,然而对于母系猪(以繁殖性状选择为主),并没有经济有效的利用方案.本文首先对基因组选择应用过程中关键问题进行讨论;其次简要介绍了基因组选择技术在父系猪中的应用情况;最后围绕我国母系猪育种的现状,探讨基因组选择技术在母系猪中如何应用.
基因组选择;奶牛;父系猪;母系猪
在奶牛的育种中,基因组选择(Genomic Selection,GS)能够实现早期选择,节省公牛后裔测定的成本,缩短世代间隔,且能够通过扩大或者共享参考群提高性状选择的准确性[1],因此基因组选择在奶牛育种中一经推出就引起极大的反响,并取得了非常好的效果.猪的个体价值不及奶牛,且缩短世代间隔非常有限,因此基因组选择在猪育种中的成本问题非常突出.然而,基因组选择在低遗传力性状(繁殖性状)、测定成本较高(如饲料转化率)以及特定性状(公猪膻味)上的选择有着非常大的优势.
近些年来,分型技术和计算方法的发展为基因组选择技术更加广泛的应用提供了基础.通过简化基因组或低覆盖度全基因组测序能够大大降低动物个体基因分型的成本,且对不同种群和群体进行分型,标记密度明显优于SNP芯片.测序数据对基因组上结构变异等信息的挖掘更有潜力.目前,国内已经在奶牛、猪等主要农业动物育种中启动了相关科研项目,在"十三五"规划中,基因组选择技术也被列为主要发展的育种技术之一,如何利用好该技术,已然成为农业动物育种领域能否赶超发达国家的标志之一.本文结合近些年来国内外基因组选择技术的发展,首先分析基因组选择在猪育种上应用的关键技术问题(如分型方法、统计分析模型以及育种方案),并介绍了基因组选择在父系猪育种上的应用情况;此外,围绕母系猪如何育种、如何提高繁殖性能水平这一问题,对繁殖性状当前面临的困境加以解析,同时重点综述了当前基因组选择在繁殖性状上的研究和应用情况,以期为繁殖性能的提高提供思路.
1 实现猪基因组选择的关键点
奶牛育种中,由于节省了后裔测定的成本,其基因分型成本基本可以忽略不计.然而在猪育种中,由于产品价值以及世代间隔的差异,分型成本相对较高,这限制了基因组选择的应用.此外,由于猪为多品系选育,各品系主选性状有所差异,基于此,基因组选择参考群的组成可能会有所不同,如父系与母系猪.因此,合适的选择方案与育种值计算方法也会对猪基因组选择效果产生影响.
综上可知,只有针对不同的育种目标,选用性价比较高的分型技术和适用的计算方法,再结合有效的育种方案(包括选育流程、参考群组成等),猪基因组选择才会在猪育种中发挥其应有的效力.
1.1 分型技术的选择
1.1.1 SNP芯片 DNA芯片技术萌发于20世纪末,它能够通过光学手段,检测到微点阵上异源双链结合荧光强度的变化[2],从而达到对特定位点基因型识别的目的.
利用SNP芯片分型技术能够简单快捷地获得覆盖全基因组的多态性位点.根据畜禽品种和芯片用途的差异,基因芯片多种多样.以奶牛为例,截至2016年4月,美国奶牛育种协会使用的芯片多达21种[3].在猪中,主要有PorcineSNP60 v2(64 232 SNPs)[4]和Porcine SNP80 BeadChip(70 231 SNPs)[5]2款芯片,在国内,也有公司开发了猪55 K的SNP芯片.
用SNP芯片进行基因分型,存在以下优点:一是方便快捷,DNA样本的收集无需从组织样品中提取,可直接利用采血卡,几滴血便可达到应用目的,样品的储存也相对简单;二是实验周期短,从采样到计算出育种值估计结果,周期较短.但也有明显的缺点:首先是由于SNP芯片的价格与其定制量和标记密度息息相关,此类公司一般会一次性定制数万张,并且标记密度在60 K左右,这数万张芯片的位点完全一样,并且会兼顾好几个品种,所以特定品种使用时,其标记密度一般在40 K左右,标记密度限制了其应用准确性的提高;其次是由于SNP芯片位点的开发是基于某个小群体重测序或其他研究的结果,因此在其他群体中使用时,不会发现新的或待选群体特异性的标记,对其使用效果也会有所影响.但随着使用量的增大,以上缺点可能会有所缓解,一方面价格会继续下降,此外可通过定制芯片来解决特定群体适用性的问题.
1.1.2 简化基因组技术 简化基因组(Genotyping by-Sequencing,GBS)测序技术的实验流程首先是利用限制性核酸内切酶打断基因组;再在酶切片段两端加上带有标记信息的接头序列;连接好接头序列后,需要对连接产物片段进行选择扩增;最后对目的片段进行测序.
GBS技术的实验方案最早是Elshire等[6]提出,在国内,由胡晓湘老师课题组开发并优化了一套利用EcoR1和Msp1进行双酶切,适用于猪基因组选择的分型方法[7],并与温氏集团合作,应用于猪的基因组选择研究.
GBS技术与SNP芯片相比,有其突出的优点.首先其弥补了针对特定群体适用性的问题,由于该技术产生的大量标记是基于目标群体,即使参考群是来自不同品系,也能保证有足够的标记可以利用;其次是灵活多变,该技术可以灵活更换酶切组合或者测序深度,达到特定的实验目的和成本控制;再者是成本优势明显[8],该技术的主要成本来自建库和测序,随着建库的不断优化和测序通量的不断提升,成本会进一步降低;最后是该技术集应用与研究于一身,通过测序,不仅能够得到足量的SNP标记,还能得到基因组结构变异的信息,有进一步研究的价值.当然,在目前看来,该技术也有其无法逃避的缺点.首先是实验过程相对于芯片技术,较为繁琐,由于建库过程是手动完成,其建库的好坏直接关系到数据质量,且建库时间成本也较高;其次基础投入大,除专利成本外,引进该技术除了必要的基础设备(如计算服务器),还需要有相关数据分析背景的人员,一般企业难以承受;再者是分型标记的质量不及SNP芯片,SNP芯片位点的检出率在99%左右,GBS技术一般在90%左右,SNP的检出率可通过增大测序乘数来提高[9],不过成本也会随之上升;最后是育种公司使用心态的问题,由于SNP芯片简单快速,检出率高,且应用较为普遍,导致育种公司对新技术较为排斥.然而,这些不足会随着组织方式的变化而改变,如成立专门化公司,聘请熟练的建库和数据分析人员,会让实验周期和数据质量都会有所保证;另外通过提高测序乘数,标记质量会有所上升,大量的标记也会极大缓解SNP检出率的问题.对猪育种而言,值得注意的是,地方品种的利用还存在巨大潜力,GBS技术比SNP芯片技术具有很大的先天优势.
1.1.3 全基因组重测序 全基因组重测序就是利用测序的方法,获得个体完整的基因组,再与参考基因组进行比对,得到覆盖全基因组的标记信息,可见此种方法预期可以获得最高的准确性.但由于目前全基因组重测序的成本较高,一般通过降低测序乘数和基因组覆盖度来降低个体分型成本,这种方法又称之为低覆盖度重测序.
低覆盖度重测序一般通过2种方式来提高标记的准确性和在基因组上的覆盖度.一是对核心个体(如公猪站内的公猪)进行高覆盖度的重测序,用于对其他个体进行填充;二是增加低覆盖度重测序个体的数量.早在2013年,Hickly[10]提出GS2.0的理念,该理念提出对百万动物进行全基因组测序.通过这种方法,有望能够发现大量与性状相连锁的数量性状基因座(QTL),甚至是数量性状致因突变(QTN),并能够大幅度提高基因组选择的准确性.该方法应用的核心在于2个方面:一是基因型填充的准确性,包括低覆盖度个体间的相互填充和低覆盖度向高覆盖度的填充;二是分型方案的确定,需要根据实验目标和群体构成,确定需要测序的个体和测序深度.
这种基于全基因组重测序基因组选择的方法,随着测序价格的不断降低,分型成本也会不断下降.该方法目前还处于科研阶段,不过基于测序的基因组选择方案在不远的将来会给猪品种遗传改良带来福音.
1.2 基因组育种值估计模型
1.2.1 GBLUP方法和一步法 基因组选择中的GBLUP方法和一步法与传统BLUP方法模型和计算方法一致,差别在于GBLUP方法利用全基因组标记构建亲缘关系矩阵,为G矩阵,而传统BLUP方法是利用系谱构建个体亲缘关系矩阵,为A矩阵.一步法则是把通过基因分型信息构建的G矩阵与通过系谱记录构建的A矩阵合并到了一个模型中,用H矩阵来表示个体间的亲缘关系.构建方法:
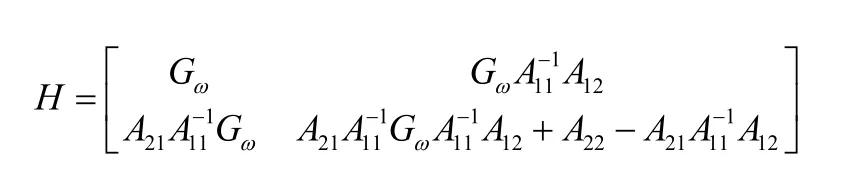
GBLUP方法和一步法都可用以下模型来表示:
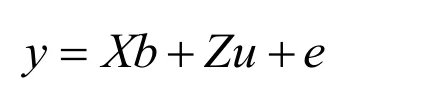
其中,y是反应变量;b是固定效应向量;u是育种值向量;X和 Z为关联矩阵;e为随机残差向量.对模型效应的求解,都可根据以下混合模型方程组的求解方法:

其中 ,

其中,G-1为基因组关系矩阵G的逆矩阵,一步法中只需将G-1替换为H-1矩阵即可.
为计算方便,Christensen等[11]和Agiular等[12]推导了该矩阵逆矩阵的构建方法:
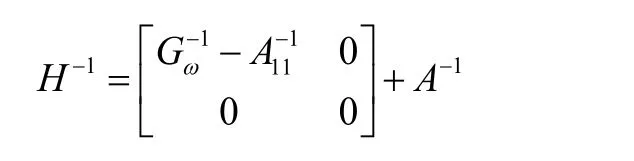
由于GBLUP方法和一步法构建的亲缘关系矩阵更加准确,所以其育种值估计的准确性更高.
1.2.2 贝叶斯方法 在基因组选择计算过程中,标记数往往会远远大于表型记录的数目,直接对标记效应求解,会出现过度拟合的问题,所以在计算过程中,需要限定所有的标记符合某一分布,会极大地简化运算过程.GBLUP方法的基本假设是标记效应都符合某一正态分布,然而对于某些受少数大效应基因控制的性状,该方法基本假设可能存在偏差.这种基于对标记效应假设的不同,衍生了很多计算方 法, 如 BayesA、BayesB、Bayes Lasso、Bayes cpi等[13].针对特定性状,基于不同假设的计算结果也会有所差异.
在某些特定的性状上,贝叶斯方法用于基因组选择的准确性可能会优于GBLUP方法.但是在猪基因组选择的应用上还比较少,首先是由于其运算过程对计算资源的要求较高,计算时间也较长;其次是由于猪育种为多品系选育,每个品系目标性状也有所差异,这就需要针对每一品系中的每一性状对标记效应单独求解,工作量巨大.最后,基于大数据的机器学习方法(以神经网络为代表)在育种领域的应用潜力还有待研究.
1.3 基因组选择育种方案 基因组选择在猪育种中应用,理论上所有后代,无论公母都应该进行基因分型,这也是为什么许多专家学者经常用全同胞选择来举例基因组选择优势的原因.然而,在应用过程中,基因组选择技术与传统育种是相互配合的,分型个体如用于早期选择,可能会优先经历初选,再对剩下的个体全部基因分型;如用于终测选留,可能在保育后才会去选择分型的个体,这样能够节省一部分分型成本.根据目标性状的不同,如主选生长性状的品系,分型个体会以公猪为主;主选繁殖性状的品系,则会以母猪为主.由此可见,当前猪基因组选择技术的应用还存在分型个体的选择,或者可以说是育种规划的问题.
Lillehammer等[14]比较了不同分型方案下,基因组选择应用于猪繁殖性状选择的差异.该实验分别比较了传统BLUP选择、后裔测定以及对不同比例子代公母猪进行基因分型,所产生的遗传进展、近交系数以及公母猪选择准确性的差异.实验结果显示出,每窝测3头公猪,并对所有母猪进行基因分型,遗传进展最大,达到了0.42,而传统BLUP方法只有0.22,遗传进展增加近1倍,同时公母猪对繁殖性状的选择准确性分别达到0.56和0.72;当只对母猪进行基因分型,而不测公猪时,选择准确性稍有下降,但是遗传进展降低到了0.37;当对后代50%母猪测序而不测公猪时,遗传进展为0.33,公母猪繁殖性状选择准确性分别为0.51和0.60,每窝加测2头公猪并不会提高选择准确性.由此可以看出,只对后代50%的母猪进行分型,性价比较高.当然,当个体分型成本足够低时,测得越多,遗传进展会越大.Lopez等[15]利用ZPLAN+比较了后裔测定、基因组选择、基因组选择与常规育种结合以及基因组选择与后裔测定结合4种育种方案遗传进展以及成本的差异,结果显示只用基因组选择的性价比最高.
总之,针对不同群体不同育种目标,差异化的基因组选择育种方案,性价比不尽相同.此外,根据以上研究可见:一是在个体分型成本足够低时,对所有后代进行分析,无论是对于何种性状,选择效果最好;二是基因组选择可能会对常规育种性能测定产生深远影响,该技术会极大地降低性能测定的费用.
2 基因组选择在父系猪中应用情况
目前瘦肉型商品猪生产主要依赖于杜X长X大(或杜X大X长)三元杂交体系.在我国,杜X长X大三元杂交猪更是占据90%以上的市场份额.在该杂交体系中,杜洛克猪对商品猪遗传上的贡献率为50%,大白猪与长白猪则各占25%.此外,又由于杜洛克猪作为终端父本,利用价值最大.因此,基因组选择优先是在杜洛克猪中加以应用,并以生长性状(如日龄、背膘、日增重、眼肌面积和饲料转化率等)为主要目标性状.
Tage等[16]利用1 375头杜洛克猪建立参考群,通过GBLUP和Bayesian Lasso方法,预测了536头猪日增重和饲料转化率的选择准确性.结果表明日增重的准确性介于0.50~0.58,饲料转化率的准确性介于0.39~0.45;Do等[17]利用968头杜洛克猪组建的参考群,评估了304头猪平均日采食量、剩余饲料残余和日增重的选择准确性.结果显示3种性状的选择准确性分别介于0.506~0.532、0.276~0.357和0.308~0.362.世界知名猪育种公司Pig Improvement Company(PIC),自2002年开始,利用1 000个SNP标记用于育种,2008年则增加到7 000个SNP标记,直至2009年60K SNP芯片的开发,基因组选择可利用标记数越来越多.公司报告显示,从2014年起,基因组选择技术提高了35%的遗传进展[18].此外,TopigsNorsvin自全面实施基因组选择技术后,遗传进展也提高了30%.
可以看出,基因组选择在父系猪中有一定的应用价值.但是,结合以上研究和温氏杜洛克猪基因组选择研究(会议报告)经验,基因组选择与常规BLUP方法相比,在达100 kg体重日龄和背膘上,选择准确性的提高非常有限(约10%).此外,PIC和TopigsNorsvin所报道的遗传进展提高,比较模糊,并没有说明是基于什么性状的基因组选择,可能包括了繁殖性状.从这个角度看,基因组选择用于选择准确性本来就较高的生长性状(日龄和背膘)的选择,性价比并不高.但基因组选择在常规育种进展较慢和测定成本较高的性状上,如饲料转化率和肌间脂肪含量等,能够显著加快遗传进展和减少测定成本.在我国,很多育种企业的选择指数仅包括达100 kg(或115 kg)体重日龄和100 kg体重背膘厚,并没有测定饲料转化率等测定成本较高性状的能力,基因组选择技术的应用效果也会大打折扣.
3 基因组选择在繁殖性状改良上应用探讨
3.1 猪繁殖性状改良现状 在当前繁育体系下,猪繁殖性状的育种目标最终是为了提高二元母猪每头母猪每年提供的断奶仔猪数(PSY).二元母猪繁殖效率的提升,主要包括2个方面:一是不断提高核心群的遗传潜力;二是通过优-优组合来尽可能提高杂交优势的利用.对总产仔数和活仔数(或健仔数)的选择以及对遗传缺陷个体的淘汰,是主要的育种方法.
近年来,一方面我国核心种猪群繁殖性状的表型数据不断积累,遗传进展有所加快;另一方面随着养猪产业规模化提升,生产管理水平得到提高,我国整体PSY水平有所提升.但是对于核心群育种,也存在一些问题:一是群体遗传水平没有得到实质性的提高.通过遗传评估可以发现,经常存在表型得到提升,而总产仔数EBV上下波动或者基本没有进展的情况;二是性状收集不细致,缺少新性状的加入,如仔猪均匀度等;三是生产管理水平有待进一步优化,如母猪发情鉴定以及妊娠期体况的控制等.

表1 繁殖性状部分性状基因组选择准确性
我国种猪繁殖性能的提升需要从2个方面考虑:一是重点研究影响母猪繁殖性能发挥的因素或开发相应的指标,如诱导发情措施和不同阶段最佳体况的控制等,制定一套最佳的生产管理流程,减少人为的干预;二是坚持持续的选育,不仅要加强当前主选性状表型的准确性,还要主动尝试开发适合自身的新性状.当然,基因组选择技术的逐渐应用,也为繁殖性状的提升提供了一个高效的手段.
3.2 基因组选择在猪繁殖性状上研究进展 在母系猪中,基因组选择能够通过实现早期选择和提高繁殖性状的选择准确性来达到加快遗传进展的目的[19].通过模拟数据显示,基因组选择能够增加遗传进展23%~91%[14],国际大型猪育种公司PIC和TopigsNorsvin在2012年都在猪育种中正式应用[20-21],更多的研究情况如表1所示.
3.3 我国基因组选择在猪繁殖性状上改良的应用前景 基因组选择技术无疑能够提高繁殖性状的选择准确性,但是在我国,如何有效与当前传统育种方案相结合,是一个需要讨论的问题.需要从下面几个方面入手:首先,基因组选择技术是建立在传统育种的基础上.无论是表型数据还是系谱数据的收集,数据的准确性必须要得到保证.其次,加强数据收集过程中固定效应的收集,如母猪配种人员、配种栏舍以及任何异常情况等,都应有所记录.一是从生产的角度,能够最优化管理;二是能够优化育种值估计模型,提高模型的统计效力.再者,适时增加其他目标性状.可以预见的是,母猪自主性和饲料转化效率的提升将会是种猪企业竞争的着力点,利用基因组选择可以实现早期选择,将初选的个体进行饲料转化率的测定,能够提升育种核心群种猪的质量.另外,仔猪均匀度也是一个比较有前景的性状.最后,基因组选择技术应用的合作模式的探讨.国内育种企业中,将基因组选择应用于生产,温氏集团无疑是排头兵,该技术的应用需要大量的资金投入,中小育种企业应寻求与科研院校或大企业合作.此外,从大处着眼,专业化是未来的发展方向,绝大多数育种企业将会被淘汰,中小育种企业应结合自己的品种(或品系)特点,专注于自身差异化产品的建设.
4 小 结
基因组选择技术在动物育种中的应用,将会越来越普遍,但是每个物种有其自身的特点.在奶牛育种中的成功应用,有许多值得借鉴的地方,但是在猪育种上,有更多的品系和性状等诸多因素影响着基因组选择技术的应用与普及.基因组选择在母系猪中如何应用,还值得进一步研究.
[1] Meuwissen T, Hayes B, Goddard M. Accelerating improvement of livestock with genomic selection[J]. Annu Rev Anim Biosci, 2013, 1: 221‐237.
[2] Schena M, Shalon D, Davis R W, et al.Quantitative monitoring of gene expression patterns with a complementary DNA microarray[J]. Science, 1995,270(5235): 467‐470.
[3] Wiggans G R, Cole J B, Hubbard S M, et al. Genomic selection in dairy cattle: The USDA Experience[J]. Annu Rev Anim Biosci, 2017, 5(1):309‐327.
[4] Ramos A M, Crooijmans R P M A, Affara N A, et al.Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technology[J]. PLoS One, 2009, 4(8):e6524.
[5] Geneseek. Porcine SNP80 BeadChip[J/EB]. http://genomics.neogen.com/pdf/slicks/ag284_ggp_porcine.pdf.2017‐07‐18.
[6] Elshire R J, Glaubitz J C, Sun Q, et al. A Robust, Simple Genotyping‐by‐Sequencing (GBS) Approach for High Diversity Species[J]. PLoS One, 2011, 6(5):e19379.
[7] Tan C, Hu X X. Genotyping‐By‐Sequencing for Genomic Evaluation in Pigs[C]. San Diego C A: PAG XXIV, 2016.
[8] Bajgain P, Rouse M N, Anderson J A. Comparing genotyping‐by‐sequencing and single nucleotide polymorphism chip genotyping for quantitative trait loci mapping in wheat[J]. Crop Sci, 2016, 56(1):1‐17.
[9] Gorjanc G, Cleveland M A, Houston R D, et al. Potential of genotyping‐by‐sequencing for genomic selection in livestock populations[J]. Genet Sel Evol, 2015, 47(1):12.
[10] Hickey J M. Sequencing millions of animals for genomic selection 2.0[J]. J Anim Breeding Genet, 2013,130(5):331.
[11] Aguilar I, Misztal I, Johnson D L, et al. Hot topic: a unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final score[J]. J Dairy Sci, 2010, 93(2):743.
[12] Christensen O F, Lund M S. Genomic prediction when some animals are not genotyped[J]. Genet Sel Evol, 2010,42(1):1‐8.
[13] 王重龙, 丁向东, 刘剑锋, 等. 基因组育种值估计的贝叶斯方法 [J]. 遗传 , 2014, 36(2):111‐118.
[14] Lillehammer M, Meuwissen T H, Sonesson A K. Genomic selection for maternal traits in pigs[J]. J Anim Sci, 2011,89(12):3908‐3916.
[15] Lopez B M, Kang H S, Kim T H, et al. Optimization of swine breeding programs using genomic selection with ZPLAN+[J]. Asian‐Austral J Anim Sic, 2016, 29(5):640‐645.
[16] Tage O, Christensen O F, Mark H, et al. Deregressed EBV as the response variable yield more reliable genomic predictions than traditional EBV in pure‐bred pigs[J].Genet Sel Evol, 2011, 43(1):38.
[17] Do D N, Janss L L, Jensen J, et al. SNP annotation‐based whole genomic prediction and selection: an application to feed efficiency and its component traits in pigs[J]. J Anim Sci, 2015, 93(5):2056‐2063.
[18] Van Eenennaam A L, Weigel K A, Young A E, et al.Applied animal genomics: Results from the field[J]. Ann Rev Anim Biosci, 2014, 2(2):105‐139.
[19] Ibañezescriche N, Gonzalezrecio O. Promises, pitfalls and challenges of genomic selection in breeding programs[J].Span J Agric Res, 2011, 9(2):253‐266.
[20] McLaren D. Implementation of Genomic Selection in Commercial Pig Breeding[R]. https://connect.innovateuk.org/documents/3085453/6368149/Dave+McLaren+‐+Imp lementation+of+Genomic+Selection+in+Commercial+Pi g+Breeding.pdf/ad2d23f3‐f801‐43fd‐b02a‐15eed6322aa2,2012.
[21] Andersen‐Ranberg I, Grindflek E. Implementation of genomic selection in Norsvin genetic program; genetic gain in production and maternal traits in Norsvin Landrace[R].Vancouver Canada: World Congress on Genetics Applied to Livestock Production, 2014.
[22] Uimari P, Sevon‐Aimonen M L, Serenius T. Reliability of Genomic Selection of Reproduction Traits in Finnish Yorkshire Pig Breed[R]. Vancouver Canada: World Congress on Genetic Applied to Livestock Production,2014.
[23] Hidalgo A M, Bastiaansen J W, Lopes M S, et al. Accuracy of predicted genomic breeding values in purebred and crossbred pigs[J]. G3 (Bethesda, Md.), 2015, 5(8):1575‐1583.
The Genomic Selection and Its Application in Pig Breeding
YE Jian1, ZHENG En‐qin1, HU Xiao‐xiang3, WANG Ai‐guo4, WU Zhen‐fang1,2, CAI Geng‐yuan1,2*
(1.National Engineering Research Center For Breeding Swine Industry, College of Animal Science, South China Agricultural University, Guangdong Guangzhou 510642, China; 2. Guangdong Wen′s Food Group Co.Ltd. Yunfu,Guangdong Xinxing 527400, China; 3.College of Biological Sciences, China Agricultural University, Beijing 100193;4. College of Animal Science and Technology, China Agricultural University, Beijing 100193, China)
The Genomic Selection (GS) technology takes the advantage of the marker information, which cover the genome and connect to the economic traits, to calculate the total effects and selection individuals on the basis of the genomic estimated breeding value (GEBV). GS was first widespread used in dairy cattle, while in pigs, the benefit of early selection and increasing in the selection accuracy were first emerged in Duroc pigs. However, in maternal pigs, the genetic breeding program is more complex. To find out the problem, this paper firstly discussed the key issues in the application of genomic selection and then introduced the application in Duroc pigs. Finally, how to combine the genomic seletion to the present pig breeding program in maternal pigs was mainly discussed.
Genomic Selection; Dairy Cattle;Terminal pigs; Maternal pigs
S813
A
10.19556/j.0258-7033.2017-11-005
2017-08-18;
2017-09-22
种猪全基因组选择技术研发与应用(2015B020231010);广东省现代农业产业技术体系生猪创新团队(2016LM1101、2016LM1104)
叶健(1992-),男,安徽人,博士研究生,主要从事猪遗传育种研究,E‐mail:jye1992@126.com
*通讯作者:蔡更元,E‐mail: cgy0415@163.com

