基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析
沈 朕,关洪斌,郑风荣,胡发文,郭 文,王 波*
(1.山东大学(威海)海洋学院,山东威海264209;2.国家海洋局第一海洋研究所,山东青岛266061;3.山东省海洋生物研究院,山东青岛266104)
基于cytb和D-loop的4个大泷六线鱼群体遗传多样性分析
沈 朕1,2,关洪斌1,郑风荣2,胡发文3,郭 文3,王 波2*
(1.山东大学(威海)海洋学院,山东威海264209;2.国家海洋局第一海洋研究所,山东青岛266061;3.山东省海洋生物研究院,山东青岛266104)
利用线粒体DNA的细胞色素b(cytb)和控制区(D-loop)的部分序列来研究大泷六线鱼4个群体(包括野生和养殖群体)的遗传多样性。PCR扩增后分别得到365 bp(cytb)和387 bp(D-loop)的碱基序列。Mega计算结果显示cytb中A+T的含量(52.6%)高于G+C的含量(47.3%);D-loop中A+T的含量(69.3%)同样高于G+C的含量(30.7%)。4个群体平均的变异位点、单倍型数、单倍型多样性、核苷酸多样性及平均核苷酸差异数,cytb基因分别为10,7.75,0.739,0.007 3和2.656;D-loop区分别为18.25,12.75,0.846,0.012 1和4.673。4个群体的遗传多样性由高到低分别为舟山、大连、琅琊台、即墨养殖群体,养殖群体的多样性低于野生群体。基于野生群体cytb和D-loop的分子变异分析(AMOVA)得出的Fst分别为0.318 6和0.271 4,显示了变异主要发生在群体内部。基于Kimura 2-parameter模型构建的NJ树结果表明3个野生群体间没有显著的分化。线粒体cytb基因和D-loop区均可作为检测大泷六线鱼遗传多样性的有效标记。
大泷六线鱼;野生和养殖群体;线粒体DNA cytb基因和D-loop;遗传多样性
大泷六线鱼(Hexagrammos otakii)为六线鱼科(Hexagrammidae)六线鱼属(Hexagrammos)的鱼类,分布于朝鲜、日本以及中国东海、黄海、渤海等海域,系冷温性近海底层鱼类[1]。大泷六线鱼味道鲜美、营养价值和经济价值高,受广大消费者和养殖者的青睐[2]。随着市场对大泷六线鱼的需求不断增大,沿海过度捕捞使得大泷六线鱼的资源衰退日渐严重。我国对于大泷六线鱼的研究从20世纪80年代开始至今,大都围绕着其生物学指标、营养成分组成和人工繁育方面来进行[3],关于大泷六线鱼遗传多样性的研究在国内外却很少见,目前国外只有Habib等人基于COI,COIII-ND3-ND4L和cytb对黄海和日本海附近群体遗传结构进行研究[4],国内的研究有刘奇[5]和李莹[6]等人关于大泷六线鱼遗传多样性的研究,但取样地都集中在黄、渤海海域,且只使用了D-loop序列进行遗传多样性与遗传结构的分析,并未使用cytb或COI基因进行对比分析。
遗传多样性的研究可以为了解物种的进化历史以及进化和发展的潜力提供可行性资料,进而根据其现状制定合理有效的保护方案[7]。线粒体DNA(mt DNA)具有母系遗传、拷贝数多、编码效率高、进化速度快等特点,已经广泛应用于海洋鱼类的遗传研究中[8-9]。细胞色素b(cytb)和控制区(D-loop)是研究应用比较广的序列,进化速度相对较快,适用于群体水平的遗传多样性分析[10],已经被用于多种鱼类的多样性分析,如短吻鲟(Acipenser brevirostrum)[11]、鲚鱼(Coilia)[12]、罗非鱼(Oreochromis mossambicus)[13]、云南倒刺鲃(Spinibarbus yunnanensis)[14]、大鳍鳠(Mystus macropterus Bleeker)[15]、翘嘴鲌(Culter alburnus)[16]等。我们利用线粒体cytb基因和D-loop控制区部分序列分析中国大泷六线鱼的4个群体的遗传多样性,对比野生与养殖群体的遗传多样性差异,研究了3个地理群体间的遗传结构和变异,旨在了解这几个大泷六线鱼群体的遗传背景,为加强其渔业资源的保护和开发,提供理论依据。
1 材料与方法
1.1 样本采集
实验所采用的野生大泷六线鱼分别采自大连(122°51'E,39°25'N)30尾,琅琊台(119°58'E,35°30'N)30尾和舟山(122°37'E,30°03'N)30尾,养殖群体为大连♀鱼与大连及即墨鳌山海域♂杂交子代(样品采自即墨山东省海洋生物研究院养殖场)30尾,采样点见图1。选取体长在15 cm左右的健康个体,取尾鳍和肌肉组织存放于体积分数为95%的乙醇中,-20℃保存备用。

图1 大泷六线鱼群体的采样地点(○)Fig.1 Map of Hexagrammos otakii sampling sites(○)
1.2 DNA的提取和PCR的扩增
每个样本取尾鳍或肌肉组织约30 mg(即墨养殖群体的样本取肌肉和尾鳍共30 mg,其余群体只取尾鳍),采用海洋动物组织基因组DNA提取试剂盒(北京天根生化科技有限公司)进行提取,并溶于200μL TE中,于-20℃保存。用于扩增线粒体cytb基因的引物[6]:上游引物(5'-AAC CAC CGT TGT TAT TCA ACT-3'),下游引物(5'-CTC AGA ATG ACA TTT GTC CTC A-3')。用于扩增D-loop控制区的引物序列[2]:上游引物(5'-TAA CTC CCA CCC CTA ACT CC-3'),下游引物(5'-CCA TTA ACT TAT GTA AGC GTC G-3')。PCR反应程序为94℃预变性5 min,35个循环(94℃变性30 s,退火30 s,72℃延伸1 min),最后72℃延伸10 min。扩增cytb基因的引物退火温度为52℃,D-loop引物的退火温度为56℃。所有的PCR反应体系均为50μL:DNA模板2μL(约20 ng/μL),上下游引物各2μL(10μmol/L),25μL 2×Premix Taq(大连宝生物有限公司),dd H2O补齐至50μL。取5μL PCR扩增产物经质量分数为1%的琼脂糖凝胶电泳检测后,将含有目的条带的PCR产物送北京美吉桑格生物医药科技有限公司进行测序。
1.3 数据分析
测序结果经Dnastar软件包(DNASTAR Inc.,Madison,USA)校对后并截取有效片段。采用mega 6.06统计变异位点,碱基组成以及群体间的遗传距离进化树。使用DNASP5.10软件计算序列的单倍型数(h)、单倍型多样性(Hd)、核苷酸多样性(π)、平均核苷酸差异数(k)。用Arlequin 3.11软件采用分子变异分析方法(AMOVA)计算群体间遗传分化指数Fst并用排列测验法(permutation test)检测显著性。
2 实验结果
2.1 mtDNA cytb序列分析结果
2.1.1 cytb碱基序列组成分析
PCR扩增后,将测序结果与GenBank中注册的大泷六线鱼的mtDNA cytb基因序列进行比对,确定所得片段序列为目的片段。经Dnastar比对后截取得到365 bp的有效序列。各群体的碱基组成见表1。T,C,A,G碱基的平均含量分别为29.4%,30.0%,23.2%,17.3%。A+T的含量(52.6%)高于G+C的含量(47.3%),符合动物细胞色素b的特征[17],并与其它鱼类的碱基组成偏向性相似。

表1 各群体cytb与D-loop基因片段碱基组成(%)Table 1 Base composition of cytb and D-loop partial sequence(%)in different populations
2.1.2 遗传多样性
cytb序列计算得出的各群体内多样性信息见表2。舟山群体呈现出的多样性最高,变异位点21个,简约信息位点17个,单倍型10个,平均核苷酸差异数7.080,核苷酸多样性0.019 40,均明显高于其它3个群体。大连群体多样性次之,琅琊台与即墨养殖群体多样性均较差,养殖群体多样性(变异位点4个,简约信息位点3个,单倍型5个,平均核苷酸差异数0.862,核苷酸多样性0.002 36)低于琅琊台(变异位点6个,简约信息位点4个,单倍型8个,平均核苷酸差异数1.301,核苷酸多样性0.003 56)。大连、琅琊台、舟山、即墨cytb序列中19种单倍型中有6种是共享单倍型(表3),占总数的31.6%,剩下13种单倍型为个体特有,其中7种为舟山群体所独有,明显高于其它2个野生群体,与大连(3个)和琅琊台(4个)之间共享的单倍型要少于大连与琅琊台之间的(7个)。Hap1和Hap4在4个群体中均有出现且为多个个体所共享,可能是最原始的单倍型。养殖群体与大连有较多的共享单倍型(4个),与亲本来源相符(表3)。
表4中列出4个群体间Kimura 2-paramter遗传距离和遗传分化指数Fst,其中舟山和大连、琅琊台的遗传距离最高,为0.018 0;琅琊台与大连的遗传距离最低,为0.004 0。3个野生群体中,大连和舟山的遗传分化指数最高(0.356 6),大连与琅琊台分化指数最低(-0.011 1)。3个野生群体分子变异分析(AMOVA)见表5,群体间变异占31.86%,变异大部分来自于群体内部,3个野生群体间没有显著的遗传分化。

表2 各群体cytb基因遗传多样性参数Table 2 Genetic diversity parameters of cytb gene in different populations

表3 大泷六线鱼cytb单倍型在各群体中的分布Table 3 The distribution of cytb haplotypes indifferent populations of Hexagrammos otakii

表4 基于cytb基因得出的野生群体间遗传距离(对角线下方)与遗传分化指数(对角线上方)Table 4 Pairwise genetic distance(below diagonal)and F st(above diagonal)among wild populations based on cytb

表5 基于cytb得出的遗传差异的分子方差分析Table 5 Analysis of molecular variance(AMOVA)among the populations based on cytb
2.1.3 分子系统树
选择单鳍多线鱼(Pleurogrammus monopterygius)线粒体cytb序列作为外群(GenBank登录号:AB087414.1),基于Kimura 2-paramter构建NJ分子系统进化树(图2),Bootstrap检验次数为1 000次。3个野生群体之间没有明显的分界点,没有明显构成一个单独分支的群体。

图2 大泷六线鱼线粒体cytb序列NJ系统树Fig.2 Neighbor-joining tree based on mitochondrial cytb sequence of Pleurogrammus monopterygius
2.2 mtDNA D-loop序列分析结果
2.2.1 D-loop碱基序列组成分析
PCR扩增后,将测序结果与GenBank中注册的大泷六线鱼的D-loop基因序列进行比对,确定所得片段序列为目的片段。经Dnastar比对后截取得到387 bp的有效序列,有效片段各群体碱基含量见表1。T,C,A,G的平均含量33.6%,15.7%,35.7%,15.0%。其中A+T的含量(69.3%)显著高于G+C的含量(30.7%),表现出十分明显的碱基偏向性,与脊椎动物D-loop的特征相符[6]。
2.2.2 遗传多样性
各群体通过D-loop序列得出的遗传多样见表6。舟山群体多样性最高(变异位点33个,简约信息位点27个,单倍型16个,平均核苷酸差异数12,核苷酸多样性0.031 35),大连次之,即墨和琅琊台的多样性最差,与cytb序列的分析结果相同。相较于cytb中的结果,D-loop中琅琊台群体的多样性(变异位点11个,简约信息位点9个,单倍型12个,平均核苷酸差异数2.108,核苷酸多样性0.005 46)明显高于即墨群体(变异位点10个,简约信息位点5个,单倍型7个,平均核苷酸差异数1.366,核苷酸多样性0.003 54)。D-loop序列中38种单倍型中有6种是共享单倍型(表7),占总数的15.8%,其中2种为4个群体共享,剩下32种单倍型为群体特有,其中13个为舟山群体所独占,高于其他2个群体,而且舟山群体与大连(3个)和琅琊台间(2个)共享单倍型要少于大连和琅琊台之间的(5个)。Hap1,Hap8被较多个体共享,可能是最原始的单倍型。即墨与大连群体亲缘关系较近(共享单倍型4个)。D-loop单倍型中得到的结果与cytb中的基本一致。
基于Kimura 2-paramter计算的3个野生群体间的遗传距离见表8,大连与舟山遗传距离最高(0.028 0),大连和琅琊台的遗传距离最低(0.006 0)。琅琊台与舟山遗传分化指数最高(0.316 6,63),大连与琅琊台遗传分化指数最低(-0.004 9,92)。分子变异分析(AMOVA)件表9,群体间变异占27.14%,变异大部分来自于群体内部,3个野生群体间没有显著的遗传分化,这与cytb中所得到的结果基本一致。

表6 各群体D-loop遗传多样性参数Table 6 Genetic diversity parameters of D-loop in different populations

表7 大泷六线鱼D-loop单倍型在各群体中的分布Table 7 The distribution of D-loop haplotypes in H.otakii

表8 基于D-loop基因得出的野生群体间遗传距离(对角线下方)及遗传分化指数(对角线上方)Table 8 Airwise genetic distance(below diagonal)and F st(above diagonal)based on wild population D-loop

表9 基于D-loop得出的遗传差异的分子方差分析Table 9 Analysis of molecular variance(AMOVA)among the populations based on D-loop
2.2.3 分子系统树
外群选择单鳍多线鱼(P.monopterygius)线粒体D-loop序列作为外群(Genbank登录号:FJ858209.1),基于Kimura 2-paramter构建NJ分子系统进化树(图3),Bootstrap检验次数为1 000次。与线粒体cytb序列所得出的进化树基本一致,3个野生群体间没有明显的单独构成分支的群体。
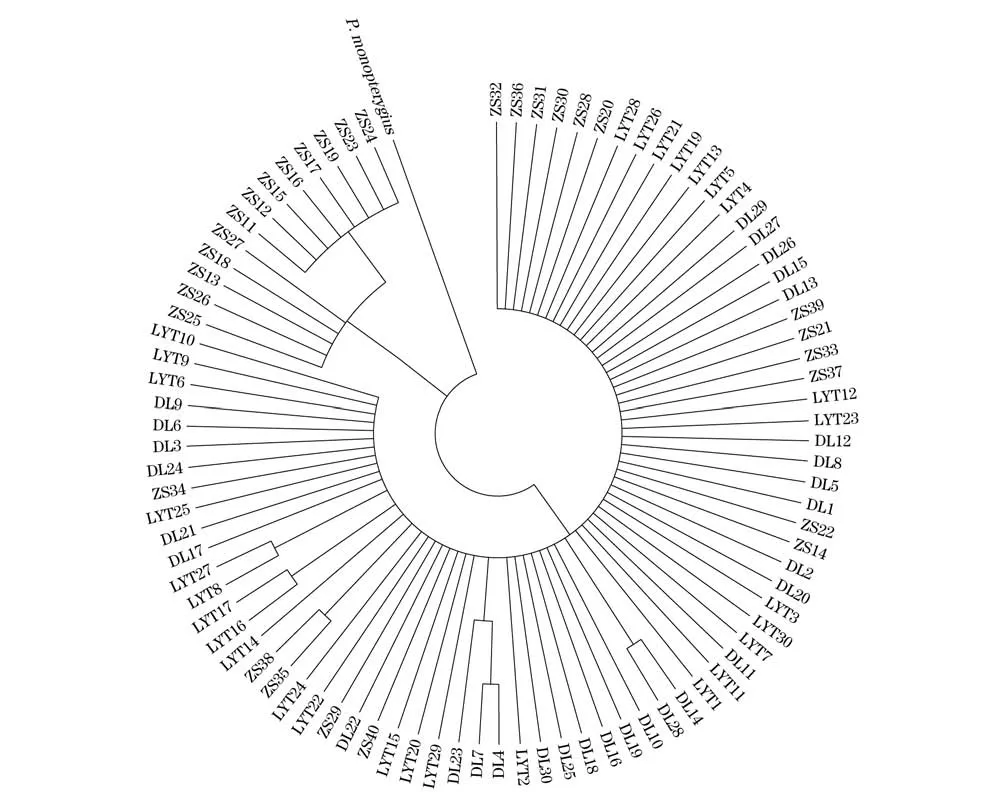
图3 大泷六线鱼线粒体D-loop序列NJ系统树Fig.3 Neighbor-joining tree based on mitochondrial D-loop sequence of Hexagrammos otakii
3 讨 论
我们选取的中国大泷六线鱼主要分布在黄海、渤海、东海海域,研究了4个群体共120尾鱼的cytb序列(365 bp)、D-loop序列(387 bp)的遗传多样性。4个群体中,大连、琅琊台、舟山均为野生群体,即墨为养殖群体。大泷六线鱼的生存与适应环境的能力与其物种内部的遗传多样性有着十分紧密的关系,丰富的多样性是应对复杂环境的保证,也是物种进化的前提;相反的,相对贫乏的多样性则不利于物种长期生存,同时也会降低其进化的潜能。
3.1 大泷六线鱼遗传多样性和遗传分化分析
研究中cytb序列和D-loop序列在4个群体62尾个体检测的单倍型数分别为19个和38个,单倍型多样性指数分别为0.739和0.846。3个野生群体的cytb和D-loop变异位点数最高的均为舟山群体,分别占野生群体总变异位点的58.3%和52.4%,变异位点大多来自舟山野生群体,可见舟山群体与其它2个野生群体间亲缘关系较远。2种序列分析的多样性数据显示,舟山群体核苷酸多样性(分别为0.019 40,0.031 35)最高,大连(0.003 80,0.008 35)、琅琊台(0.003 56,0.005 46)均远低于舟山,即墨群体(0.002 36,0.003 54)作为养殖群体,其单倍型多样性、核苷酸多样性及平均核苷酸差异数均低于3个野生群体。
尽管是野生亲本的杂交子一代,但由于养殖过程中亲本数量较少,近交的概率增加,使得养殖群体遗传多样性要比野生群体低[19]。舟山附近海域岛礁众多,占全国岛屿总数的25.7%,大泷六线鱼作为恋礁鱼类,在岛礁密集的海域分布广,数量多。相对于黄、渤海只有近岸的群体,舟山群体亲本数量大,基因交流频繁,累积的遗传多样性相对较高。在养殖过程选育优良性状的同时,应该保持亲本的数量以及亲本的多样性。只有在优选优育的同时最大程度地保留群体多样性,才能提高养殖群体对于环境及疾病的应对能力和遗传改良能力。
单倍型与核苷酸多样性相反,单倍型多样性高而核苷酸多样性低,这与彭士明研究的野生银鲳鱼遗传多样性的结果[20]相同。可能是由于种群在扩张期是由一个小而有效的群体快速成长而来的,庞大的种群数量、环境的不均一性以及适应种群快速增长的生活习性导致在进化期间通过变异获得较多的单倍型多样性,但是却未能积累核苷酸的多样性[21]。这从侧面说明,我国近海多岩礁海区适合大泷六线鱼的生长,是其维持较高的遗传多样性基础。这种现象大多出现在一些起源于上新世或更新世早期出现的群体[2,22],如鮡科(Sisoridae)[23]、鲤科(Cyprinidae)[24]、翘嘴鲌(Culter erythropterus)[16]等鱼类。
Fst是测量群体间遗传分化的重要参数,Fst值越大表明2种群的分化程度越高[25]。我们研究的3个野生地理群体间的cytb和D-loop的Fst值分别为-0.011 1~0.356 6,-0.004 9~0.316 6,并没有出现十分显著的遗传分化,大连与琅琊台群体分化程度很小,而舟山群体则与其余2个群体的分化程度较大。遗传距离结果与遗传分化结果相符,大连与琅琊台群体的遗传距离较近(cytb为0.004 9,D-loop为0.006 0),舟山与其他2个群体较远(cytb为0.018 0,D-loop为0.027 0~0.028 0)。根据统计资料显示,鱼类在属、种、种群三个水平的分类中,以遗传距离作为判断的标准分别为0.90,0.30和0.05[8,19]。3个群体间的遗传距离均未超过0.05,可见这3个群体间遗传分化远未达到种群的分化标准。AMOVA分析结果显示,群体内的变异占较大比例(cytb为31.86%,D-loop为27.14%)遗传变异主要来自于群体内部,群体间的分化并不显著。
3.2 野生群体地理分化分析
基于cytb和D-loop序列所构建的NJ树显示,野生群体之间并没有明显汇聚成单独一支的群体,90尾个体无序的分布在不同分支中,这个结果与之前的遗传距离和遗传分化所得到的结果相一致。大连和琅琊台群体的遗传距离较近可能因为同属黄、渤海海域,地理位置相近,群体间的杂交及遗传漂变所致的;舟山群体与其他2个群体地理距离较远,基因交流的频率降低,遗传距离相对较远。
大泷六线鱼是底栖鱼类,栖息于岩石或珊瑚礁区域,每年10-11月会在岩石上产卵。每年的秋、冬季胶东半岛流出渤海的沿岸流与黄海暖流的西支会在成山头附近汇合,并形成一股向南流动的黄海沿岸流。大泷六线鱼的幼苗在表层水面度过几个月的浮游期[26],这股寒流会携带同处于黄、渤海海域的大连与琅琊台群体的部分幼苗,漂流至东海海域,与舟山当地的群体形成基因交流。这个推断解释了为什么地理位置相距较远的群体间并没有形成显著的遗传分化,Xiao等[27]和Han等[28]也得出同样的结论。他们研究的小黄鱼[27]和黄姑鱼[28]取样地也在相距较远的黄海和东海海域,因为中国近海的海流给2个海域的野生群体提供了基因交流的通道,群体间并没有形成显著的遗传分化。
4 结 语
与其他野生鱼类,如银鲳鱼(0.000 7)[29]、大口黑鲈(0.002 8)[30]、沙鳅(0.003 65)[31]相比,3个野生群体的大泷六线鱼均具有较高的核苷酸多样性,遗传多样性也较为丰富,说明我国大泷六线鱼的渔业资源具有很大的开发潜力。近年来,因为沿海经济的发展,沿岸过度捕捞以及以陆源为主的污染成为渔业资源的重要威胁。大泷六线鱼自然繁殖能力较弱,且比较依赖海底环境,势必会受到较大的影响,因此进行渔业资源的保护是非常必要的。由于大泷六线鱼的繁殖习性、其栖息海域的洋流等因素、其遗传结构十分复杂,因此进行今后应该开展整个中国海域、日本及朝鲜等主要栖息地的大泷六线鱼遗传结构、亲缘关系及遗传多样性的研究,并结合mt DNA和其他分子标记技术,更好地揭示群体多样性和遗传结构的关系。
[1] NISHIKAWA S,SAKAMOTO K.The karyotype of a hexagrammid,Hexagrammos otakii(Pisces,Scorpaeniformes)[J].Chromosome Information,1982,33:16-17.
[2] GRANT W S,BOWEN B W.Population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J].Journal of Heredity,1998,89:415-426.
[3] PAN L,HU F W,GAO F X,et al.Study of artificial breeding and seedling culture technique for greenling Hexagrammos otakii[J].Marine Science,2012,36(12):39-44.潘雷,胡发文,高凤祥,等.大泷六线鱼人工繁殖及育苗技术初步研究[J].海洋科学,2012,36(12):39-44.
[4] HABIB K A,JEONG D,MYOUNG J G,et al.Population genetic structure and demographic history of the fat greenling Hexagrammos otakii[J].Genes&Genomics,2011,33(4):413-423.
[5] LIU Q.Studies of biology characteristics and genetic diversity on populations of Hexagrammos otakii[D].Qingdao:Ocean University of China,2010.刘奇.大泷六线鱼(Hexagrammos otakii)生物学特征与遗传多样性研究[D].青岛:中国海洋大学,2010.
[6] LI Y,WANG W,MENG F P,et al.Genetic diversity analysis between different stocks of Hexagrammos otakii based on analysis of mitochondrial DNA control region partial sequence[J].Marine Science,2012,36(8):40-46.李莹,王伟,孟凡平,等.利用线粒体DNA控制区部分序列分析不同地理群体大泷六线鱼遗传多样性[J].海洋科学,2012,36(8):40-46.
[7] HU J,HOU X Y,YIN S W,et al.Genetic diversity of different geographical populations of Cheicinus undulatus revealed by microsatellite analysis[J].Advance in Marine Science,2013,31(4):538-545.胡静,候新远,尹绍武,等.波纹唇鱼(Cheilinus undulatus)不同地理种群遗传多样性的微卫星分析[J].海洋科学进展,2013,31(4):538-545.
[8] LYNCH M,CREASE T J.The analysis of population survey data on DNA sequence variation[J].Molecular Biology and Evolution,1990,7(4):377-394.
[9] SHAKLEE J B,TAMARU C S,WAPLES R S.Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J].Pacific Science,1982,36(2):141-157.
[10] YU X R,QIU X M,LIU X Y,et al.Application of research on marine animals'population genetic structure using mitochondrial DNA polymorphism[J].Biotechnology Bulletin,2011,231(10):49-54.于旭蓉,仇雪梅,柳晓瑜,等.线粒体DNA多态性在海洋动物群体遗传结构研究中的应用[J].生物技术通报,2011,231(10):49-54.
[11] WANG W,ZHU H,HU H X.Genetic diversity of the reserved broodstocks in four species of sturgeon[J].Chinese Journal of Zoology,2012,47(1):105-111.王巍,朱华,胡红霞.4种鲟鱼养殖亲鱼群体遗传多样性分析[J].动物学杂志,2012,47(1):105-111.
[12] YANG Q L.Phylogenetic analysis of genus coilia in china and molecular phylogeography of C.nasus and C.mystys[J].Qingdao:Ocean University of China,2012.杨巧莉.中国鲚属鱼类进化关系及刀鲚、凤鲚的分子系统地理学研究[D].青岛:中国海洋大学,2012.
[13] JIE X Y,LI S F.Comparison of base sequence diversity of Cytb and D-loop gene of Niletilapia[J].Genomics and Applied Biology,2014,33(5):982-985.颉晓勇,李思发.罗非鱼选育群体cytb与D-loop序列变异信息对比分析[J].基因组学与应用生物学,2014,33(5):982-985.
[14] ZHENG B R,ZHANG Y P,XIAO H.Genetic diversity of Spinibarbus yunnanensis Tsu based on mitochondrial D-loop gene[J].Reservoir Fisheries,2002,22(3):15-16.郑冰蓉,张亚平,肖蘅.云南倒刺鲃mt DNA D-loop区序列的遗传多样性研究[J].水利渔业,2002,22(3):15-16.
[15] ZHOU L.Studies on genetic diversity and structure of mt DNA control region of mystus macropterus bleeker[D].Chongqing:Southwest University,2008.周丽.大鳍鳠(Mystus macropterus Bleeker)遗传多样性及其线粒体控制区结构的研究[D].重庆:西南大学,2008.
[16] WANG W.Study on population genetic diversity of culter alburnus and phylogeny of cultrinae[D].Shanghai:East China Normal University,2007.王伟.翘嘴鲌(Culter alburnus)群体遗传多样性及鲌亚科鱼类系统发生的研究[D].上海:华东师范大学,2007.
[17] QI D L,CHAO Y,YANG C,et al.Cloning of mitochondrial cytb gene of hucho bleekeri and its phylogenetic relationship in subfamily salmoninae[J].Sichuan Journal of Zoology,2009(6):805-809.祁得林,晁燕,杨成,等.川陕哲罗鲑Cytb基因克隆及其在鲑亚科中的系统发育关系[J].四川动物,2009(6):805-809.
[18] HUANG X Z,QIAN G Y,LI C Y.The genetic diversity of mitochondrial DNA D-loop region of three geographical populations in Trionyx sinensis[J].Journal of Fisheries of China,2012,36(1):17-24.黄雪贞,钱国英,李彩燕.中华鳖3个地理群体线粒体基因D-loop区遗传多样性分析[J].水产学报,2012,36(1):17-24.
[19] LOU J F,LEI S Y,ZHU J Q,et al.AFLP analysis of genetic diversity in two cultured communities of Psedosciaena crocea[J].Advance in Marine Science,2015,33(3):361-366.娄剑锋,雷世勇,竺俊全,等.岱衢洋与官井洋大黄鱼养殖群体遗传多样性的AFLP分析[J].海洋科学进展,2015,33(3):361-366.
[20] PENG S M,SHI Z H,HOU J L.Comparative analysis on the genetic diversity of cultured and wild silver pomfret populations based on mtD-loop and COI gene[J].Journal of Fishieries of China,2010,34(1):19-25.彭士明,施兆鸿,侯俊利.基于线粒体D-loop区与COI基因序列比较分析养殖与野生银鲳群体遗传多样性[J].水产学报,2010,34(1):19-25.
[21] KAVIARASU M,SUBHA B.Genetic structure of locally threatened cyprinid,Osteochilus melanopleurus,in Peninsular Malaysia River systems inferred from mitochondrial DNA control region[J].Biochemical Systematics and Ecology,2015,61:336-343.
[22] LI L.Study on morphology and genetics of pholis fangi and P.nebulosa[D].Qingdao:Ocean University of China,2013.李琳.方氏云鳚和云鳚的形态学与遗传学研究[D].青岛:中国海洋大学,2013.
[23] MA X H.The phylogeny,biogeograph and adaptive evolution of Chinese Sisoridae fishes[D].Chongqing:Southwest University,2015.马秀慧.中国鮡科鱼类系统发育、生物地理及高原适应进化研究[D].重庆:西南大学,2015.
[24] XU W.Studies on molecular phylogeny of Rhynchocypris fishes(Teleiostei:Cyprinidae)and phylogeography of cold-adapting freshwater fishes in the eastern China[D].Shanghai:Fudan University,2013.许旺.鲤科大吻鱥属鱼类的分子系统发育关系与我国东部地区冷水性淡水鱼类的亲缘生物地理[D].上海:复旦大学,2013.
[25] WRIGHT S.Evolution and the genetics of populations,Volume 4:Variability within and among natural populations[M].Chicago:Univesity of Chicago Press,1978.
[26] KANAMOTO Z.On the ecology of Hexagrammid Fish:Iv.Mode of the distribution of Agrammus Agrammus(Temminck Et Schlegel)and Hexagrammos Otakii Jordan et Starks and composition,abundance and food ttems of reef fish around the several reefs[J].Japanese Journal of Ecology,1979,29:171-183.
[27] XIAO Y,ZHANG Y,GAO T,et al.Genetic diversity in the mtDNA control region and population structure in the small yellow croaker Larimichthys polyactis[J].Environmental Biology of Fishes,2009,85(4):303-314.
[28] HAN Z Q,GAO T X,YANAGIMOTO T,et al.Genetic population structure of Nibea albiflora in Yellow Sea and East China Sea[J].Fisheries Science,2008,74(3):544-552.
[29] PENG S M,SHI Z H,CHEN C,et al.Genetic diversity analysis of silver pomfret(Pampus argenteus)in the East China Sea based on mtDNA D-loop sequence[J].Marine Science,2010,34(2):28-32.彭士明,施兆鸿,陈超,等.根据mtDNA D-loop序列分析东海银鲳群体遗传多样性[J].海洋科学,2010,34(2):28-32.
[30] LI S J,BAI J J,YE X.et al.Approach on the taxonomic status and genetic variation of largemouth bass(Micropterus salmodies)cultured in China based on mitochondrial D-loop gene[J].Marine Fisheries,2008,30(4):291-296.李胜杰,白俊杰,叶星,等.基于线粒体D-loop区探讨我国养殖大口黑鲈的分类地位和遗传变异[J].海洋渔业,2008,30(4):291-296.
[31] LIU H Y,CHEN D Q,LIU S P,et al.Genetic diversity of Botia superciliaris in the upper Yangtze River[J].Freshwater Fisheries,2009,39(3):8-13.刘红艳,陈大庆,刘绍平,等.长江上游中华沙鳅遗传多样性研究[J].淡水渔业,2009,39(3):8-13.
Analysis of Genetic Diversity Between Four Populations of Hexagrammos otakii Based on Partial mtDNA cytb and D-loop
SHEN Zhen1,2,GUAN Hong-bin1,ZHENG Feng-rong2,HU Fa-wen3,GUO Wen3,WANG Bo2
(1.Marine College of Shandong University(Weihai),Weihai 264209,China;2.The First Institute of Oceanography,SOA,Qingdao 266061,China;3.Marine Biology Institute of Shandong Province,Qingdao 266104,China)
Nucleotide sequences of mitochondrial DNA cytb and D-loop from 4 different populations of Hexagrammos otakii were amplified by using PCR technique,365 bp and 387 bp nucleotide sequences were obtained to study the genetic diversity of Hexagrammos otakii.The results showed that the content of A+T(52.6%)was higher than G+C(47.3%)in cytb,and it was also higher in D-loop(content of A+T and G+C was 69.3%,30.7%respectively)through Mega calculation,the total variable sites,number of haplotypes(h),haplotype diversity(Hd),nucleotide diversity(π)and mean pairwise nucleotide differences(k)of 4 populations were 10,7.75,0.739,0.007 3,2.656 based on cytb and 18.25,12.75,0.846,0.012 1,4.673 based on D-loop,respectively.Diversities from high to low were Zhoushan,Dalian,Langyatai and Jimo(cultured population).Diversity in cultured population was lower than that in wild population.Fstvalues were 0.318 6 and 0.271 4 based on cytb and D-loop of wild populations through analysis of molecular variance(AMOVA)respectively,demonstrating that the variation is mainly within the population.The NJ molecular phylogenetic trees based on Kimura 2-parameter model showed that three different geographic stocks have no apparent geographic subdivision.In short,it suggested that the mitochondrial DNA cytb and D-loop may serve as an effective marker for analysis of genetic diversity of Hexagrammos otakii populations.
Hexagrammos otakii;wild and cultured geographic stocks;mitochondrial DNA cytb and D-loop gene;genetic diversity
August 30,2016
Q344
A
1671-6647(2017)04-0524-11
10.3969/j.issn.1671-6647.2017.04.009
2016-08-30
海洋公益性行业科研专项——黄、渤海重要经济生物产卵场修复与重建技术集成与示范(201405010);国家高技术研究发展计划项目——重要鲆鲽鱼类良种培育(2012AA10A408)
沈 朕(1990-),男,山东青岛人,硕士研究生,主要从事基因工程方面研究.E-mail:397027997@qq.com
*通讯作者:王 波(1963-),男,山东蓬莱人,研究员,硕士,主要从事海水养殖技术方面研究.E-mail:ousun@fio.org.cn
(高 峻 编辑)

