基于转录组信息的黑果枸杞MYB转录因子家族分析
严莉,王翠平,陈建伟,乔改霞,李健
基于转录组信息的黑果枸杞MYB转录因子家族分析
严莉1,2,王翠平2,陈建伟2,乔改霞2,李健2
(1宁夏大学生命科学学院,银川 750021;2宁夏林业研究院种苗生物工程国家重点实验室,银川 750004)
MYB基因家族是植物中最大的一类转录因子家族,广泛参与植物的生长发育和代谢调控过程。目前仍没有针对枸杞等木本作物MYB转录因子家族的系统分析。基于转录组数据鉴定分析黑果枸杞MYB基因家族,可为该类基因生物学功能和代谢调控机制的研究提供参考。基于黑果枸杞转录组测序(RNA-Seq)数据,利用NR、NT、Swiss-Prot和PFAM 4个数据库和NCBI网站,对黑果枸杞MYB基因进行筛选注释;利用Web Logo3、Prot Comp 9.0、MEGA5.0软件进行保守结构域、亚细胞定位和系统进化等生物信息学分析。基于转录组数据分析MYB基因在黑果枸杞果实不同发育期的差异表达模式,并采用荧光定量PCR方法进行验证。基于转录组测序数据,注释得到83个黑果枸杞MYB类转录因子基因,根据结构特性将其分为4大类:R2R3-MYB、1R-MYB、3R-MYB和4R-MYB。结构域分析表明:R2R3-MYB类转录因子的R2 MYB基序中包含3个极度保守的色氨酸残基,R3 MYB基序中的第一个色氨酸残基被疏水氨基酸替代。比较分析黑果枸杞MYB家族和拟南芥MYB家族共同构建的进化树发现,黑果枸杞的MYB家族在进化上包括3个大类,6个亚类。亚细胞定位预测结果显示,44个MYB转录因子定位于细胞质中,37个定位于细胞核中。基于转录组数据的MYB差异表达模式分析表明,黑果枸杞MYB基因可能参与了果实不同发育时期花青素变化的调控;基于荧光定量PCR的差异表达数据进一步验证了部分MYB转录因子在果实不同发育时期的花青素合成中可能起到调控作用。黑果枸杞MYB基因家族注释得到83个黑果枸杞MYB类转录因子基因,为进一步研究MYB家族的基因结构和生物学功能奠定了基础。
黑果枸杞;转录组测序;MYB家族;分层聚类分析;表达模式
0 引言
【研究意义】MYB转录因子是植物中最大的转录因子家族之一,广泛参与植物的生理生化过程,包括植物表皮细胞分化、气孔发育、类黄酮生物合成、非生物胁迫及病原体抗性等[1]。黑果枸杞(Murr)是茄科枸杞属植物,花青素含量丰富[2]。花青素属类黄酮类化合物,是构成植物花或果实颜色的主要水溶性色素之一[3-5]。对黑果枸杞MYB转录因子家族进行分析鉴定,可为黑果枸杞MYB基因的功能研究奠定基础,为研究MYB基因对黑果枸杞类黄酮或花青素的代谢调控提供重要借鉴。【前人研究进展】MYB基因家族是最大的植物基因家族之一,其成员都具有高度保守的DNA结合结构域,即MYB结构域[6-8]。通常根据其所含高度保守DNA结合结构域的重复数量将MYB类转录因子分为1R-MYB(1R-MYB和MYB-Related)、R2R3-MYB、3R-MYB和4R-MYB[9]。自PAZ-Ares等[10]从玉米中分离出第一个植物MYB转录因子(Clorless1)后,学者先后从金鱼草[11]、棉花[12]、大豆[13]、拟南芥[14]、苹果[15]和白菜[16]等植物中鉴定得到功能各异的MYB转录因子。其中,R2R3-MYB类转录因子广泛存在于陆生植物中[17],并有学者在多种植物中发现其参与了植物的次生代谢过程,如拟南芥可上调查尔酮合成酶和黄酮醇合成酶基因的表达,从而提高类黄酮化合物的积累[18],通过正向调控的表达提高植物中花青素的含量[19];红沙梨中和通过与形成三元复合体调控花青素的生物合成[20];金鱼草中可抑制植物次生代谢途径中重要可溶性酚酸的合成[21];可与共同瞬时表达来诱导小麦高原115白色胚芽鞘中花青素的合成[22]。此外,大量研究结果表明R2R3-MYB类转录因子可响应非生物胁迫如干旱、低温、盐及紫外辐射等,Zhang等[23]通过克隆小麦并构建过表达拟南芥植株,发现其可提高植株在发芽期和幼苗期的耐旱性[23];Zhu等[24]分别构建过表达和RNA干扰的水稻植株,发现过表达植株较野生型植株具有更强的耐盐性,而RNA干扰的水稻植株耐盐性较野生型植株明显减弱,这与YANG等[25]对的研究结果一致;CHENG等[26]发现过表达的马铃薯植株对盐、紫外及干旱胁迫的耐性显著提高。另外,R2R3-MYB转录因子还参与植物生殖发育过程调控,可通过调节植物绒毡层的发育来调控花粉发育[27]。关于其他3类MYB转录因子功能的研究多集中在胁迫响应方面,番茄中1R-MYB蛋白可通过调节气孔开闭来适应高盐环境[28];水稻和小麦既可调控细胞周期,也可响应干旱胁迫[29];(1R-MYB)可提高小麦对条纹绣菌的抗性[30];拟南芥(3R-MYB)可与GSK3样激酶BIN2共同作用来抑制油菜素类固醇信号通路中的表达[31]。【本研究切入点】目前对枸杞属植物MYB转录因子家族基因的功能研究很少,仅有一篇外文文献报道了在黑果枸杞果实发育过程中MYB转录因子的表达升高,未有学者对枸杞属植物的MYB转录因子家族进行过系统分析,因此,有必要对枸杞属植物黑果枸杞的MYB家族进行全面分析和鉴定。【拟解决的关键问题】基于不同发育时期黑果枸杞果实转录组测序数据,筛选并鉴定出黑果枸杞MYB转录因子家族成员,全面分析其基因结构、亚细胞定位、蛋白保守结构域及系统发育等信息,同时研究MYB家族成员在果实发育不同时期的差异表达模式,为后续研究黑果枸杞中MYB转录因子的功能提供一定基础。
1 材料与方法
试验于2016年在宁夏林业研究院种苗生物工程国家重点实验室进行。
1.1 材料
黑果枸杞(Murr)为宁夏黑果枸杞,树龄为3年,常规栽培管理。于2016年7月12日选取生长健康、长势良好的黑果枸杞植株(组培苗扦插繁育)做标记,于开花后25 d开始采摘青果期果实,40 d后采摘转色期果实,58 d后采摘成熟期果实(果实大小、饱满度一致),3棵植株的果实为一个重复,各采收3个重复,材料采集后立即置于液氮中速冻,存于-80℃冰箱备用。
1.2 方法
1.2.1 果实总RNA提取、文库构建及转录组测序 用植物总RNA提取试剂盒(RNA Prep Pure Plant Kit,天根生化科技有限公司,北京,中国)分别提取黑果枸杞不同发育时期果实的总RNA,并用NanoDrop- 2000(Thermo Scientific,上海,中国)分光光度计检测RNA纯度(OD260/280比值),通过Qubit(恒斐生物科技有限公司,上海,中国)对RNA浓度进行精确定量,后用Agilent 2100(Stratagene,LaJolla,CA,USA)精确检测RNA的完整性,最后用1%的琼脂糖凝胶电泳,样品检测合格后送到北京诺禾致源科技股份有限公司进行转录组测序,后续分析基于此测序结果数据。
1.2.2 黑果枸杞和拟南芥MYB转录因子基因的筛选 基于转录组测序数据,在NR、NT、Swiss-Prot和PFAM 4个数据库进行注释,初步注释到137个MYB转录因子相关基因,将这些基因编码蛋白逐个进行NCBI Blast和SMART预测,去除重复序列及冗余转录本后,最终得到83个具有MYB转录因子保守结构域的蛋白序列。根据Dubos等[9]对拟南芥MYB基因家族的分类方法(依据MYB转录因子所含DNA结合结构域的数目分类),分别将候选序列归类到1R-MYB(1R-MYB和MYB-Related)、R2R3-MYB、3R-MYB和4R-MYB类转录因子中。拟南芥蛋白序列从TAIR (http://www.arabidopsis.org/)数据库中下载获得。
1.2.3 黑果枸杞MYB转录因子家族保守域、亚细胞定位及进化关系分析 利用序列分析软件clustal X 2.0对32个黑果枸杞R2R3-MYB类转录因子进行多序列比对,去除保守结构域之外的区域,通过在线软件Web Logo3[32](http://weblogo.Threeplusone. com/)对R2R3-MYB类转录因子蛋白的DNA结合结构域进行基序分析;利用在线软件Prot Comp 9.0(http://linux. softberry.com)对83个候选MYB基因家族各成员的氨基酸序列进行亚细胞定位预测分析;利用MEGA5.0软件对83个黑果枸杞MYB转录因子蛋白和部分功能明确的拟南芥MYB家族蛋白(共33个,包括R2R3-MYB 27个,1R-MYB 6个)进行序列比对,并用邻位(Neighbor-Joining)算法构建系统进化树,进行Bootstrap测试,重复设置为1 000。

图1 黑果枸杞3个发育时期果实(青果、紫果和黑果)
1.2.4 黑果枸杞MYB类转录因子基因的表达模式分析与验证 黑果枸杞果实转录组测序文库中各基因片段表达量的计算使用RPKM(Reads per kb per million)法[33]。采用Heml软件对83个MYB转录因子在果实3个发育期的表达数据进行分层聚类分析。以黑果枸杞3个发育期果实为材料,共选取12个在果实转色期上、下调明显的MYB转录因子进行实时荧光定量PCR。各样品RNA 采用第1链cDNA合成试剂盒(TaKaRa,大连,中国)反转录为cDNA,选用枸杞作内参,其GenBank登录号为HQ415754,采用Primer Premier 5.0设计荧光定量引物(序列见表1),反应体系为:模板cDNA 2 μL(10 ng·μL-1),正、反向引物各1 μL(10 μmol·L-1),SYBR®Green Realtime PCR Master Mix(QPK-201, TOYOBO,大阪,日本)10 μL,ddH2O 6 μL。PCR反应程序为:95℃ 2 min,95℃ 30 s,55℃ 30 s,72℃ 30 s,40个循环,每个样品做3个重复。所用仪器为美国安捷伦MX3000P(Stratagene,LaJolla,CA,USA)实时荧光定量PCR仪,相对表达量的计算采用2-ΔΔCT方法[34]。

表1 实时荧光定量PCR所用到的引物
2 结果
2.1 黑果枸杞MYB类转录因子基因的筛选与蛋白序列的分类分析
基于转录组测序数据,在NR、NT、Swiss-Prot和PFAM 4个数据库进行注释,初步注释到137个MYB转录因子相关基因,通过NCBI Blast和SMART预测,去除重复序列及冗余转录本后,最终得到83个黑果枸杞MYB转录因子。
对所得83条MYB转录因子蛋白序列进行批量SMART预测,并根据Dubos等[9]对拟南芥MYB基因家族的分类方法,按照每个MYB转录因子所包含的DNA结合结构域类型分别将其归类到1R-MYB、R2R3-MYB、3R-MYB和4R-MYB类转录因子类别中,最终鉴定出黑果枸杞R2R3-MYB类转录因子32个,1R-MYB类转录因子45个,3R-MYB类转录因子4个和4R-MYB类转录因子2个。
2.2 黑果枸杞R2R3-MYB类蛋白保守基序分析
利用clustal X 2.0对所得32个黑果枸杞R2R3- MYB类转录因子进行多序列比对,去除DNA结合结构域之外的不保守区域,对其DNA结合结构域进行基序分析。从图2黑果枸杞R2R3-MYB类转录因子的R2 MYB基序图可知其约含52个氨基酸残基,包括3个极度保守的色氨酸残基(W),由R3 MYB基序图可知其所含3个极度保守色氨酸残基(W)中的第一个色氨酸残基(图2箭头处)被苯丙氨酸(F)、异亮氨酸(I)以及亮氨酸(L)所替代,其他两个色氨酸残基也高度保守。此外,在R2和R3 MYB结构域中还存在其他保守氨基酸残基,如结构域起始的赖氨酸(K)、甘氨酸(G)和精氨酸(R)残基、R2结构域中的赖氨酸(K)、丝氨酸(S)、甘氨酸(G)、半胱氨酸(C)、精氨酸(R)和亮氨酸(L),以及R3结构域中的亮氨酸(L)、甘氨酸(G)、脯氨酸(P)、苏氨酸(T)、精氨酸(R)、天冬氨酸(D)和天冬酰胺(N)残基。这些保守的氨基酸残基可能与色氨酸残基共同维持黑果枸杞R2R3-MYB类转录因子的螺旋-转角-螺旋结构,进而发挥特定功能。
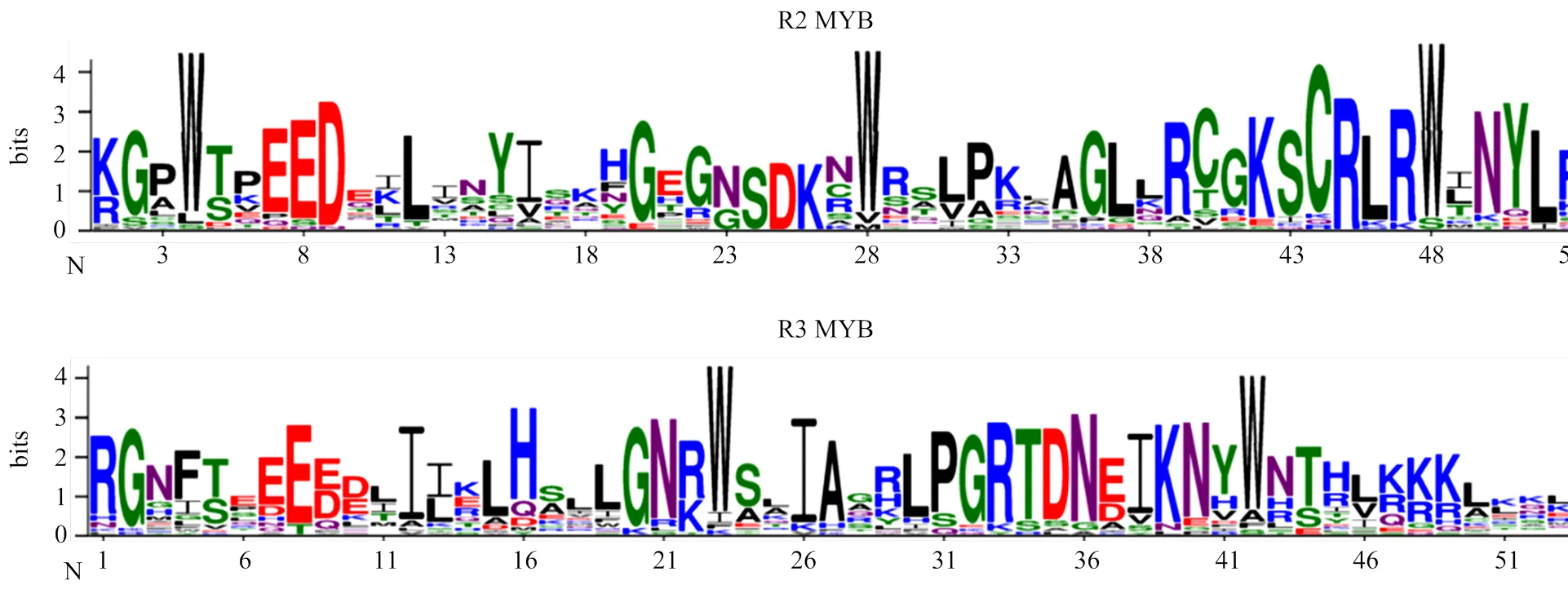
图2 黑果枸杞R2R3-MYB类转录因子DNA结合结构域
2.3 黑果枸杞MYB类转录因子的亚细胞定位分析
通过在线软件Prot Comp 9.0(http://linux1. Softberry.com)对83个黑果枸杞MYB基因家族各成员的氨基酸序列进行亚细胞定位预测,结果如图3所示,共有44个候选MYB基因位于细胞质中,其中1R-MYB类转录因子多达34个,占候选基因总数的77.27%,这些MYB类转录因子可能参与细胞质基因的转录调控;37个位于细胞核中,其中R2R3-MYB类转录因子多达24个,占候选基因总数的64.9%,R2R3-MYB类转录因子多数通过调控细胞核基因的转录来发挥功能;3R-MYB类转录因子基因和4R-MYB类转录因子基因分别定位于细胞核和细胞质中,其可能分别参与细胞质基因和细胞核基因的转录调控,由于本研究得到的3R-MYB和4R-MYB类转录因子数量少,因此还需更多后续研究来探究其在细胞中的分布规律;另外通过亚细胞定位预测,发现和位于细胞质膜,其可能参与细胞质膜某些功能基因的转录调控。
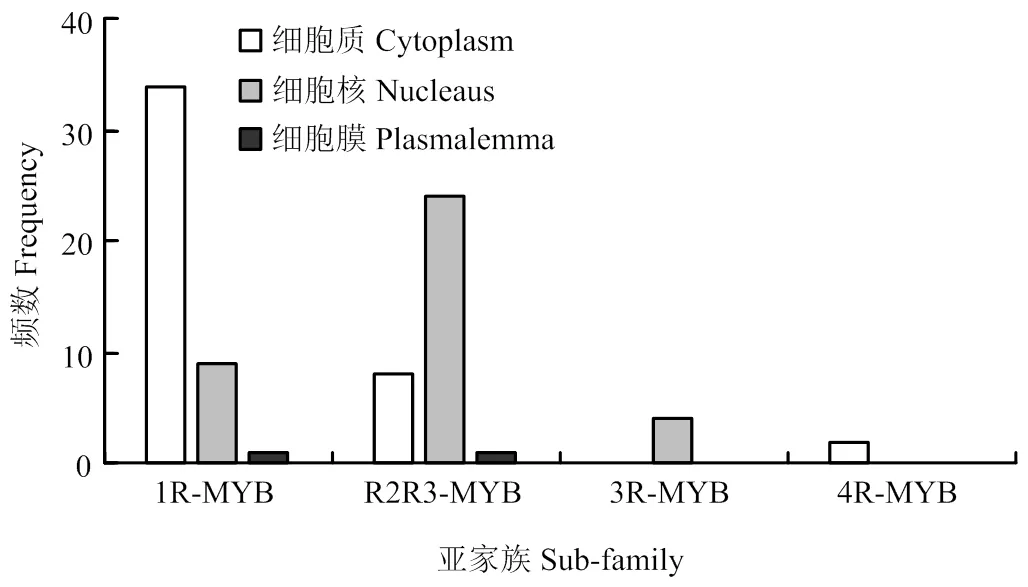
图3 黑果枸杞MYB基因家族的亚细胞定位预测
2.4 黑果枸杞与拟南芥MYB类转录因子的进化关系与功能预测分析
利用MEGA5.0软件构建黑果枸杞和拟南芥MYB蛋白家族的系统进化树,根据进化树(图4)的拓扑结构将两个物种的MYB家族蛋白进行聚类分析,共分为3大类(I、Ⅱ、Ш),其中第一大类又分为两个亚类,包含24个黑果枸杞R2R3-MYB类转录因子蛋白,占黑果枸杞总R2R3-MYB类转录因子的72.7%,且有18个R2R3-MYB类转录因子蛋白位于同一个进化亚枝,这与其N端MYB保守结构域预测结果一致。此外,由于完整的序列比对不仅包括保守的N端,也与各序列的C端密切相关,因此还有9个R2R3-MYB类转录因子蛋白分散位于第Ⅱ、Ш大类。由图可得,LrMYB3与AtMYB51相邻,LrMYB42与AtMYB37、AtMYB38和AtMYB87相邻且位于同一进化亚枝上,LrAN2、LrMYB81、LrMYB93和LrMYB96与拟南芥AtMYB61进化关系较近,推测他们可能具有相似的功能。
1R-MYB类转录因子蛋白分散位于3大类MYB家族蛋白中,尤其在Ⅱ、Ш大类中居多,占黑果枸杞总1R-MYB类转录因子的73.3%,可能由于1R-MYB类转录因子包括1R-MYB和其他MYB相关蛋白,结构多样导致其在进化树上分布范围最广,其中LrMYB16与拟南芥AtMYB0、AtMYB72和AtMYB123位于同一进化亚枝。3R-MYB类转录因子蛋白包括LrMYB17、LrMYB18、LrMYB19和LrMYB70,其中LrMYB17和LrMYB18相邻且与AtMYB91位于同一进化亚枝,LrMYB70与AtMYB62相邻;4R-MYB类转录因子蛋白包括LrMYB12和LrMYB32,其中LrMYB12与AtMYB105相邻,而LrMYB32与AtMYB24进化关系较近。
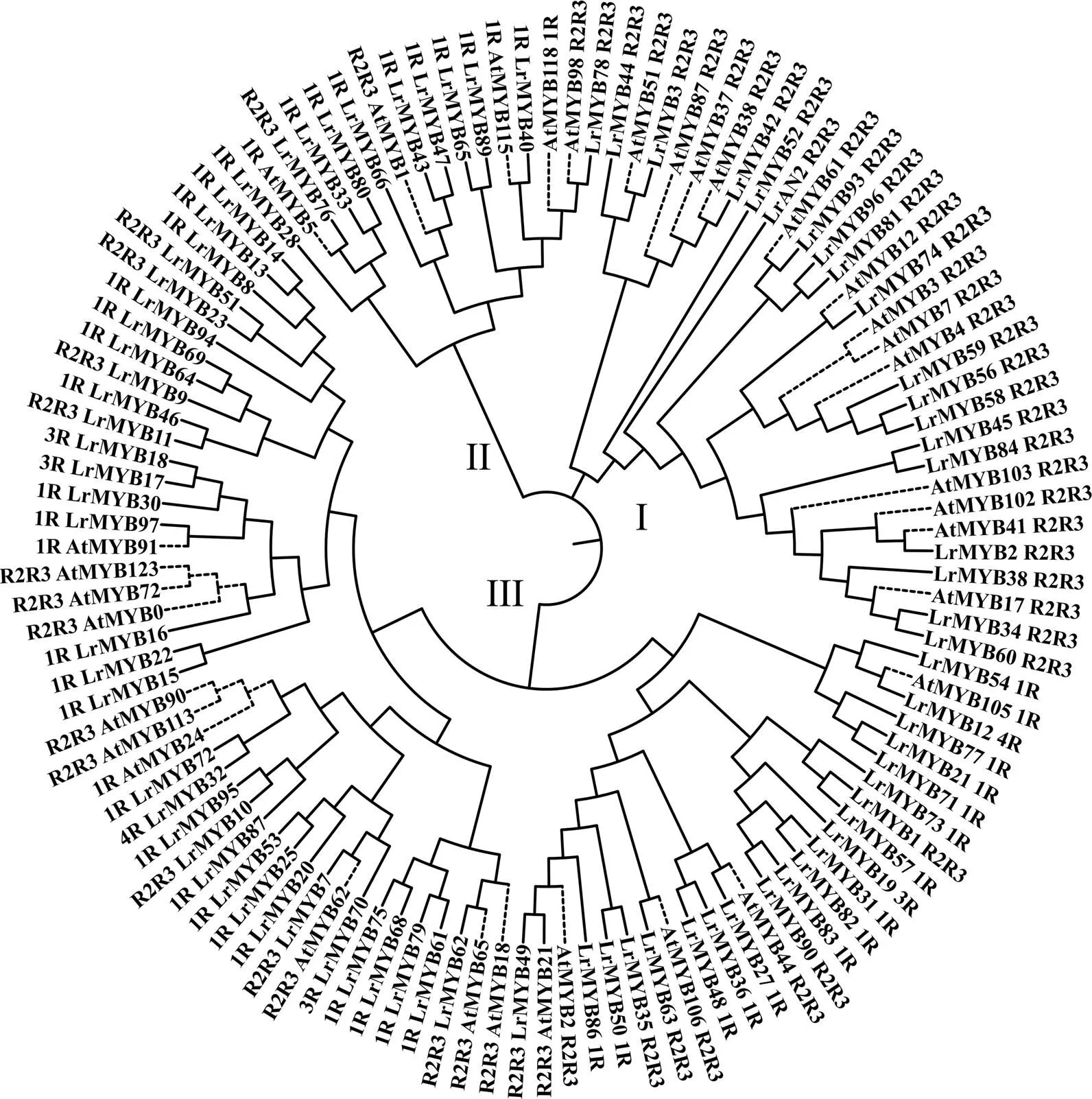
图4 黑果枸杞和拟南芥MYB类蛋白家族的进化分析
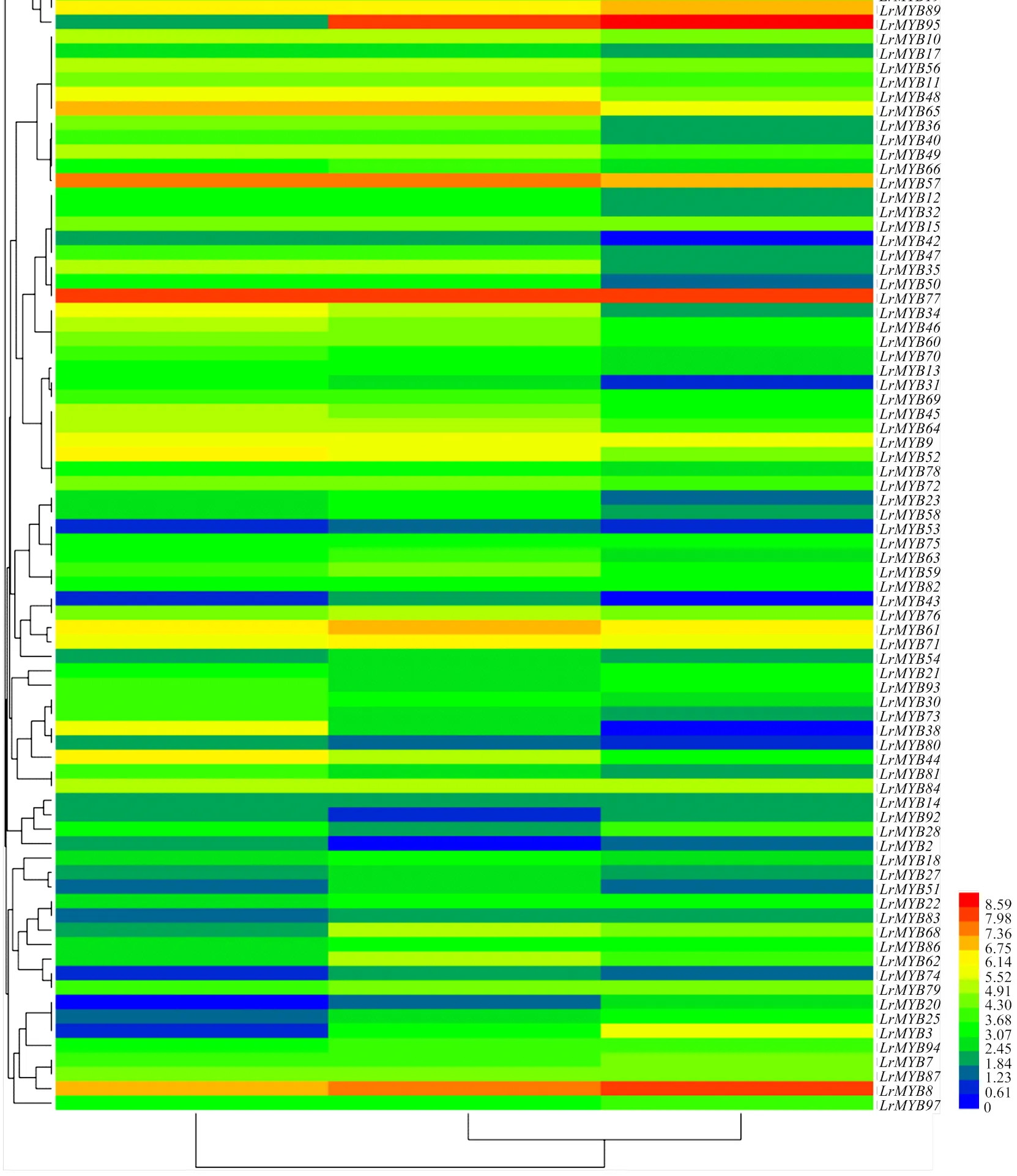
此图基于黑果枸杞果实转录组测序数据的计算得出,每种颜色代表相应表达量的数值,数值越大,表达量越高
2.5 黑果枸杞MYB转录因子基因在果实不同发育期的表达模式分析
基因的表达模式与基因的功能关系密切,据此从黑果枸杞果实转录组测序结果的基因表达数据库中提取83个MYB家族基因在果实3个发育期(青果期、转色期、成熟期)的表达数据,采用Heml软件对这些基因的差异表达进行分层聚类分析(图5)。结果发现,随着果实的发育成熟,黑果枸杞各MYB基因的表达模式表现出差异化,共有16个MYB转录因子基因随果实的发育成熟表达上调,其中和上调表达较明显;共有32个MYB转录因子基因在果实发育的青色期到转色期表达上调,而在果实发育的转色期到成熟期表达下调;共有28个MYB转录因子基因随果实的发育成熟表达下调,其中和的下调表达较明显;共有7个MYB转录因子基因的表达在果实发育的青色期到转色期及转色期到成熟期表现为先降低后升高的趋势。选取从果实青色期到转色期表达上调的MYB转录因子基因7()和表达下调的MYB转录因子基因5个(和),采用荧光定量PCR,在黑果枸杞3个发育时期果实中进行差异表达分析(图6),结果表明所选12个黑果枸杞MYB类转录因子的表达变化随果实发育各不相同,其中的表达在果实青色期到转色期明显升高;而和的表达在果实青色期到转色期明显降低,但其表达趋势与转录组数据所得的表达模式相符合。
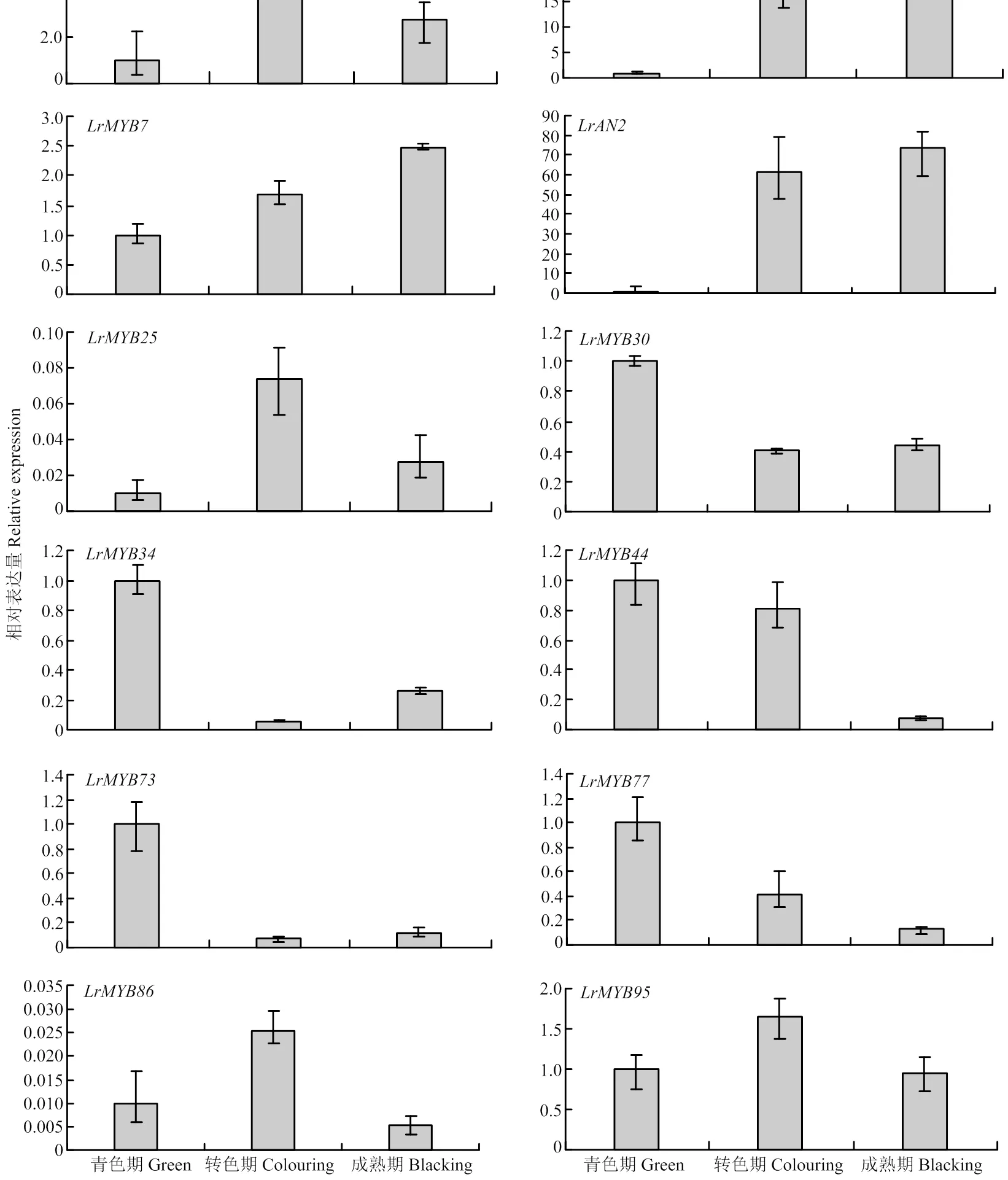
图6 黑果枸杞MYB转录因子基因的实时荧光定量PCR
3 讨论
由于枸杞全基因组序列还未释放,在很大程度上限制了控制其重要性状基因的研究。
通过对黑果枸杞R2R3-MYB转录因子的保守结构域进行基序分析,得出其R2 MYB基序中包含3个极度保守的色氨酸残基,且这3个色氨酸残基之间的间隔分别为23和19个氨基酸残基。其中前两个色氨酸残基之间的间隔比杜海[35]描述的植物中R2R3- MYB类转录因子保守结构域基序中色氨酸残基的间隔多4个氨基酸残基,说明在黑果枸杞R2R3-MYB类转录因子的R2 MYB基序中发生氨基酸残基的插入,该区域可能是黑果枸杞R2R3-MYB类转录因子分子进化和功能分化的活跃区。而R3 MYB基序中的第一个色氨酸残基被疏水氨基酸(苯丙氨酸、异亮氨酸和亮氨酸)所代替,其他两个色氨酸残基仍存在,且相互间隔18个氨基酸残基。这与成舒飞[36]和Matus[37]等在大豆和水稻中对R3 MYB基序的分析结果一致,暗示MYB基因家族的N端在不同科植物间的高度保守性。本研究对黑果枸杞R2R3-MYB转录因子的保守结构域进行基序分析,为研究枸杞属及茄科植物的R2R3-MYB类转录因子的结构和功能进化提供基础。
基因表达模式与基因功能关系密切,因此可通过研究黑果枸杞果实成熟过程中基因表达的变化来发现花青素合成过程中的功能基因。MYB转录因子被证实可作为调控基因参与植物花青素的生物合成代谢,Gulati等[38]发现可能通过调节结构基因的表达来调节植株花青素含量的变化;Sornkanok等[39]通过克隆鉴定苹果中R2R3类MYB转录因子,认为其可作为一种调控蛋白调节植物花青素的生物合成过程。本文基于转录组数据,分析MYB转录因子在黑果枸杞果实不同发育期的差异表达模式,从中筛选出16个随果实发育表达上调的MYB转录因子和28个表达下调的MYB转录因子,说明它们可能在在黑果枸杞果实花青素的生物合成中起着正向或负向调节作用,同时发现随果实转色变化的表达趋势与Zeng等[1]在黑果枸杞果实发育5个时期的表达趋势一致,为研究黑果枸杞中参与花青素生物合成的MYB转录因子的分离和功能研究提供基础。
拟南芥中MYB转录因子的功能研究已比较透彻,、和参与调控拟南芥中吲哚硫代葡萄糖苷的生物合成[40],参与植物种皮和毛状体发育[41],、、和参与了植物花青素的合成调控途径[42-44]。有学者认为可根据进化树中位于同一进化枝中的拟南芥蛋白功能预测研究物种蛋白的功能,Matus等[38]通过构建拟南芥与葡萄、水稻的R2R3-MYB类转录因子的系统进化树,将位于相邻位置上的葡萄、水稻和拟南芥R2R3-MYB转录因子列为具有相同或相似结构的转录因子,从而推测其功能的一致性。本研究用所得83个黑果枸杞MYB类转录因子编码蛋白与33个功能明确的拟南芥MYB类转录因子编码蛋白作系统进化树,根据与33个拟南芥MYB类转录因子编码蛋白进化关系的远近推测黑果枸杞MYB转录因子的功能,如LrMYB3与AtMYB51相邻,根据拟南芥AtMYB51的功能推测LrMYB3可能参与了吲哚硫代葡萄糖苷的生物合成[40];推测LrMYB32可能与AtMYB24功能一致,参与了雄蕊的发育[45];LrMYB7与拟南芥AtMYB90和AtMYB113进化关系较近,可能参与花青素代谢调控[43],这与根据果实不同发育时期的差异表达模式推测得到的LrMYB7的功能具有一定的相关性,证明了相邻或较近进化关系上的蛋白有可能具有相同或相似的功能,为研究MYB类转录因子的功能提供一种思路,同时也说明了MYB转录因子在植物中的保守性。
4 结论
本研究基于黑果枸杞不同发育时期果实的转录组测序数据,筛选注释出83个黑果枸杞MYB转录因子基因;通过分析黑果枸杞MYB基因家族的保守结构域、亚细胞定位、进化关系和随果实发育的表达模式,较为系统地鉴定了黑果枸杞MYB基因家族。MYB基因家族结构保守,家族成员在果实发育不同时期呈现不同的表达模式。
[1] ZENG S, WU M, ZOU C, LIU X, SHEN X, HAYWARD A, LIU C, WANG Y. Comparative analysis of anthocyanin biosynthesis during fruit development in twospecies., 2014, 150(4): 505-516.
[2] 郝媛媛, 颉耀文, 张文培, 宁宝山, 路新军. 荒漠黑果枸杞研究进展. 草业科学, 2016, 33(9): 1835-1845.
HAO Y Y, JI Y W, ZHANG W P, YU B S, LU X J. The research progress on desert., 2016, 33(9): 1835-1845. (in Chinese)
[3] 陈海魁, 蒲凌奎, 曹君迈, 任贤.黑果枸杞的研究现状及其开发利用. 黑龙江农业科学, 2008(5): 155-157.
CHEN H K, PU L K, CAO J M, REN X. Current research state and exploitation ofMurr, 2008(5): 155-157. (in Chinese)
[4] 双全, 张海霞, 卢宇, 刘燕. 野生黑果枸杞化学成分及抗氧化活性研究. 食品工业科技, 2017, 38(4): 94-100.
SHUANG Q, ZHANG H X, LU Y, LIU Y. The research on chemical component and antioxidant activity of wild, 2017, 38(4): 94-100. (in Chinese)
[5] 夏园园, 莫仁楠, 曲玮, 柳文媛. 黑果枸杞化学成分研究进展. 药学进展, 2015, 32(5): 351-356.
XIA Y Y, MO R N, QU W, Liu W Y. Research progress in chemical constituents ofMurr., 2015, 32(5): 351-356. (in Chinese)
[6] AMBAWAT S, SHARMA P,YADAV N R, YADAV R C. MYB transcription factor genes as regulators for plant responses: an overview., 2013, 19(3): 307-321.
[7] LAI B, DU L N, LIU R, Hu B, SU W B, QIN Y H, ZHAO J T, WANG H C, HU G B. Twotranscription factors interacting withandduring anthocyanin accumulation., 2016, 7(212): 166.
[8] SALIH H, GONG W F, HE S P, Sun G F, SUN J L, DU X M. Genome-wide characterization and expression analysis of MYB transcription factors in., 2016, 17(1): 129.
[9] DUBOS C, STRACKE R, GROTEWOLD E, WEISSHAAR B, MARTIN C, LEPINIEC L. MYB transcription factors in., 2010, 15(10): 573-581.
[10] PAZ-ARES J, GHOSAL D, WIENAND U, PETERSON PA, SAEDLER H. The regulatorylocus of Zea mays encodes a protein with homology to MYB-related proto-oncogene products and with structural similarities to transcriptional activators., 1988, 6(12): 3553-3558.
[11] WAITES R, SELVADURAI H R, OLIVER I R, HUDSON A. Thegene encodes a MYB transcription factor involved in growth and dorsoventrality of lateral organs in., 1998, 93(5): 779-789.
[12] LOGUERICO L L, ZHANG J Q, WILKINS T A. Differential regulation of six novel MYB-domain genes defines two distinct expression patterns in allotetraploid cotton (L.)., 1999, 261(4): 660-671.
[13] MIYAKE K, ITO T, SENDA M, ISHIKAWA R, HARADA T, NIIZEKI M, AKADA S. Isolation of a subfamily of genes for R2R3-MYB transcription factors showing up-regulated expression under nitrogen nutrient-limited conditions., 2003, 53(1): 237-245.
[14] CHEN Y, YANG X, HE K, LIU M H, LI J G, GAO Z F, LIN Z Q, ZHANG Y F, WANG X X, QIU X M, SHEN Y P, ZHANG L, DENG X H, LUO J C, DENG X W, CHEN Z L, GU H Y, QU L J. The MYB transcription factor superfamily of: Expression analysis and phylogenetic comparison with the rice MYB family., 2006, 60(1): 107-124.
[15] TAKOS A M, JAFFE F W, JACOB S R, BOGS J, ROBINSON S P, WALKER A R. Light-induced expression of a MYB gene regulates anthocyanin biosynthesis in red apples., 2006, 142(3): 1216-1232.
[16] 向珣, 曹家树, 叶纨芝, 崔辉梅, 俞建浓. 白菜相关MYB家族新基因的克隆与特征分析. 遗传, 2007, 29(5): 621-628.
XIANG X, CAO J S, YE W Z, CUI H M, YU J N. Molecular cloning and characterization of, a novel member of the MYB family involved in OguCMS in., 2007, 29(5): 621-628. (in Chinese)
[17] 朱婷婷, 梁东, 夏惠. R2R3-MYB调控果实花色苷合成的研究进展. 基因组学与应用生物学, 2016, 35(4): 985-991.
ZHU T T, LIANG D, XIA H. Progress in the study of the regulation of anthocyanin synthesis by R2R3-MYB in fruits., 2016, 35(4): 985-991. (in Chinese)
[18] MEHRTENS F, KRANZ H, BEDNA P, WEISSHAAR B. The Arabidopsis transcription factor MYB12 is a flavonol-specific regulator of phenylpropanoid biosynthesis., 2005, 138(2): 1083-1096.
[19] LOTKOWSKA M E. Functional analysis of MYB112 transcription factor in the model plant., 2014.
[20] FENG S Q, SUN S S, CHEN X L, WU S J, WANG D Y, CHEN X S. PyMYB10 and PyMYB10.1 interact with bHLH to enhance anthocyanin accumulation in pears., 2015, 10(11): e0142112.
[21] 李莹. 金鱼草中R2R3-MYB基因的克隆及其功能研究[D]. 北京: 中国科学院大学, 2015.
LI Y. Cloning and functional analysis of the R2R3-MYB transcription factorsin[D]Beijing:, 2015. (in Chinese)
[22] 叶广继, 张波, 陈文杰, 刘宝龙, 张华刚. 紫粒小麦高原115中R2R3-MYB转录因子的克隆及功能分析. 分子植物育种, 2016, 8(8): 1940-1947.
YE G J, ZHANG B, CHEN W J, LIU B L, ZHANG H G. Cloning and functional analysis of the R2R3-MYB transcription factorsin the purple wheat variety Gaoyuan 115., 2016, 8(8): 1940-1947. (in Chinese)
[23] ZHANG L, ZHAO G, XIA C, JIA J, LIU X, KONG X. A wheat R2R3-MYB gene,, improves drought stress tolerance in transgenic., 2012, 63(63): 5873-5885.
[24] ZHU N, CHENG S, LIU X, DU H, DAI M, ZHOU D X, YANG W, ZHAO Y. The R2R3-type MYB genehas a function in coordinating plant growth and salt stress tolerance in rice., 2015, 10(236): 146-156.
[25] YANG A, DAI X, ZHANG W H. A R2R3-type MYB gene,, is involved in salt, cold, and dehydration tolerance in rice., 2012, 63(7): 2541-2556.
[26] CHENG Y J, KIM M D, DENG X P, KWAK S S, CHEN W. Enhanced salt stress tolerance in transgenic potato plants expressing, a sweet potato transcription factor., 2013, 23(12): 1737-1746.
[27] HIGGINSON T, LI S F, PARISH R W.regulates tapetum and trichome development in., 2003, 35(2): 177-192.
[28] CAMPOS J F, CARA B, MARTIN F, PINEDA B, EGEA I, FLORES F B, FERNANDEZ G N, CAPEL J, MORENO V, ANGOSTO T, LOZANO R, BOLARIN M C. The tomato mutant(altered response to salt stress 1) identifies an R1-type MYB transcription factor involved in stomatal closure under salt acclimation., 2016, 14(6): 1345-1356.
[29] BALDONI E, GENGA A, COMINELLI E. Plant MYB transcription factors: Their role in drought response mechanisms., 2015, 16(7): 15811-15851.
[30] ZHANG Z J, CHEN J M, SU Y Y, LIU H M, CHEN Y G, LUO P G, DU X G, WANG D, ZHANG H Y. TaLHY, a 1R-MYB transcription factor, plays an important role in disease resistance against stripe rust fungus and ear heading in wheat., 2015, 10(5): e0127723.
[31] YE H, YIN Y. MYBL2 is a substrate of GSK3-like kinase BIN2 and acts as a corepressor of BES1 in brassinosteroid signaling pathway in., 2012, 109(49): 20142-20147.
[32] CROOKS G E, HON G, CHANDONIA J M, BRENNER S E. Web Logo: a sequence logo generator., 2004, 14(6): 1188-1190.
[33] FENG C, CHEN M, XU C J, BAI L, YIN X R, LI X, ALLAN A C, FERGUSON L B, CHEN K S. Transcriptomic analysis of Chinese bayberry () fruit development and ripening using RNA-Seq., 2012, 13(1): 13-19.
[34] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT-method., 2001, 25(4): 402-408.
[35] 杜海. 植物MYB转录因子家族的分子进化机制及调控类黄酮生物合成MYB基因的鉴定[D]. 四川: 四川农业大学, 2013.
DU H. Molecular evolution of MYB transcription factor gene family in plants and characterization of the MYB genes involved in flavonoid biosynthesis. Sichuan: Sichuan Agricultural University, 2013. (in Chinese)
[36] 成舒飞, 端木慧子, 陈超, 刘艾林, 肖佳雷, 朱延明. 大豆MYB转录因子的全基因组鉴定及生物信息学分析. 大豆科学, 2016, 35(1): 52-57.
CHENG S F, DUAN MU H Z, CHEN C, LIU A L, XIAO J L, ZHU Y M. Whole genome identification of soybean MYB transcription factors and bioinformatics analysis., 2016, 35(1): 52-57. (in Chinese)
[37] MATUS J T, AQUEA F, JOHNSON P. Analysis of the grape MYB R2R3 subfamily reveals expanded wine quality-related clades and conserved gene structure organization acrossandgenomes., 2008, 8(1): 83.
[38] GULATI S, PADGETT W J.MYB75/PAP1 transcription factor induces anthocyanin production in transgenic tomato plants., 2008, 35(7): 606-618.
[39] VIMOLMANGKANG S, HAN Y P, WEI G C, KORBAN S. An apple MYB transcription factor,, is involved in regulation of anthocyanin biosynthesis and flower development., 2013, 13(1): 176.
[40] FRERIGMANN H, GLAWISCHNIG E, GIGOLASHVILI T. The role of,and., 2004, 3(6): 587-591.
[41] Li S F, MILLIKEN O N, PHAM H, SEYIT R, NAPOLI. Thetranscription factor regulates mucilage synthesis, seed coat development, and trichome morphogenesis., 2009, 21(1): 72-89.
[42] ZULUAGA D, GONZALI S, LORETI E, PUCCIARIELLO C, DEGLINNOCENTI E, GUIDI L, ALPI A, PERATA P.MYB75/PAP1 transcription factor induces anthocyanin production in transgenic tomato plants., 2008, 35(7): 606-618.
[43] VELTEN J, CAKIR C, CAZZONELLI C I. A spontaneous dominant-negative mutation within a 35S:transgene inhibits flower pigment production in tobacco., 2010, 5(3): e9917.
[44] HEPPEL S C, JAFFE F W, TAKOS A M, SCHELLMANN S, RAUSCH T, AMANDA W. Identification of key amino acids for the evolution of promoter target specificity of anthocyanin and proanthocyanidin regulating MYB factors., 2013, 82(5): 457-471.
[45] YANG X Y, LI J G, PEI M, L G J, P M, GU H, CHEN Z L, QU L G. Over-expression of a flower-specific transcription factor genecauses aberrant anther development., 2007, 26(2): 219-228.
(责任编辑 赵伶俐)
Analysis of MYB Transcription Factor Family Based on Transcriptome Sequencing inMurr
YAN Li1, 2, WANG CuiPing2, CHEN JianWei2, QIAO GaiXia2, LI Jian2
(1College of Life Science, Ningxia University, Yinchuan 750021;2State Key Laboratory of Seedling Bioengineering, Ningxia Forestry Institute, Yinchuan 750004)
MYB is one of the most common transcription factor families in plants. It is widely involved in plant growth, and metabolic regulations. So far, there is no systematic analysis of the MYB transcription factor family of tree crops. Analysis of MYB family based on the transcriptome data inMurr. was conducted in this study, which laid a foundation for the research on biological function, and mechanism of metabolic regulations of MYB genes.Based on the transcriptome sequencing (RNA-Seq) data, the NR, NT, Swiss-Prot, PFAM and NCBI sites were used at the same time to screen and classify the MYB genes ofMurrThe Web Logo3, Prot Comp 9.0, and MEGA5.0 were also applied to conservative structure prediction, subcellular localization, and phylogenetic analysis. The expression pattern of MYB genes related to fruit development was obtained and Real-time fluorescence quantitative PCR was used to detect the specific expression of those genes.Based on the transcriptome sequencing (RNA-Seq) data, 83 transcription factors of MYB family were annotated, selected, and divided into four categories (R2R3-MYB, 1R-MYB, 3R-MYB and 4R-MYB) according to their structural characteristics. The R2 MYB motif of the R2R3-MYB transcription factor contains three highly conserved tryptophan residues, and the first tryptophan residue in the R3 MYB motif is replaced by some hydrophobic amino acids. The phylogenetic trees of MYB family ofMurrandwere constructed, which showed that the MYB family ofMurrcontained three major branches, and six evolutionary branches. The result of the subcellular localization demonstrated that 44 MYB transcription factors were located in the cytoplasm, and 37 MYB transcription factors were located in the nucleus. The analysis of differential expression of MYB genes ofMurr. based on transcriptome sequencing (RNA-Seq) showed that MYB genes might be involved in the regulation of anthocyanin in three fruit development periods. Additionally, differential expression data based on fluorescence quantitative PCR confirmed that some MYB transcription factors might play a role in the regulation of anthocyanin synthesis in different fruit development periods ofMurr..83 transcription factors of MYB family were annotated ofMurrThe findings have laid a foundation for further studies of the structures and biological functions of MYB family.
; RNA-Seq; MYB family; hierarchical clustering analysis; expression pattern
2017-05-27;接受日期:2017-08-22
国家自然科学基金(31660045)、国家国际科技合作专项(2015DFA90900)、宁夏自然科学基金(NZ16215)
严莉,E-mail:18295085526@163.com。通信作者王翠平,E-mail:wangcuipingcas@163.com。通信作者李健,E-mail:lijian0630@aliyun.com

