普通小麦-十倍体长穗偃麦草衍生新品种抗赤霉病基因的分子鉴别
刘新伦,王超,牛丽华,刘志立,张录德,陈春环,张荣琦,张宏, 王长有,王亚娟,田增荣,吉万全
普通小麦-十倍体长穗偃麦草衍生新品种抗赤霉病基因的分子鉴别
刘新伦1,王超1,牛丽华1,刘志立2,张录德3,陈春环1,张荣琦1,张宏1, 王长有1,王亚娟1,田增荣1,吉万全1
(1西北农林科技大学农学院/旱区作物逆境生物学国家重点实验室,陕西杨凌712100;2陕西省城固县种子管理站,陕西汉中 723200;3陕西省扶贫开发办公室,西安 710006)
赤霉病(Fusarium head blight,FHB)是世界范围内严重危害小麦生产的病害之一。十倍体长穗偃麦草()具有优良的赤霉病抗性,普通小麦-十倍体长穗偃麦草衍生新品种西农509、西农511和西农529在田间展现出较强的赤霉病抗性。本文旨在对这3个品种的赤霉病抗性基因进行分子鉴别,为它们在小麦赤霉病抗性遗传改良中的应用提供理论依据。通过赤霉病菌()人工接种鉴定,明确3个小麦新品种对赤霉病的抗性水平。利用偃麦草E组染色体(臂)第1—7同源群的特异引物对3个普通小麦-十倍体长穗偃麦草衍生新品种及其主要亲本小偃693、小偃597和十倍体长穗偃麦草进行分子鉴定,确定其长穗偃麦草的遗传区段。利用与长穗偃麦草7EL染色体上抗赤霉病基因紧密连锁的标记对实验材料进行分析,明确该抗性基因与的关系。鉴定结果表明,西农509、西农529和西农511的赤霉病抗性与中抗对照品种扬麦158的抗性水平相当,表现为中抗。105个长穗偃麦草E基因组特异标记中有7个在3个新品种中均能扩增出长穗偃麦草的特异条带,其中5个标记定位于7EL染色体臂上,2个标记定位于7ES染色体臂上。利用97个定位于7E染色体的特异标记进一步对小偃693、小偃597和3个新品种的遗传片段进行鉴别,结果表明20个标记能在5个长穗偃麦草衍生品种(系)扩增出稳定的十倍体长穗偃麦草的特异条带,其中包括与紧密连锁的和等6个标记(7EL 149.00—7EL 153.77),即表明该区段源自于十倍体长穗偃麦草,跨距约89 cM。然而,已报道的7EL染色体臂末端与两侧紧密连锁的分子标记(7EL 153.77)(7EL 154.70)、(7EL 156.27)、(7EL 158.02)、(7EL 158.97)和(7EL 160.00)等在3个新品种及小偃597中均没检出长穗偃麦草的特异条带。普通小麦-十倍体长穗偃麦草衍生新品种西农509、西农511和西农529具有较好的赤霉病抗性,携带来自于十倍体长穗偃麦草7E染色体的遗传区段,然而该抗赤霉病基因不同于。
普通小麦;十倍体长穗偃麦;赤霉病;;分子标记
0 引言
【研究意义】小麦赤霉病(Fusarium head blight,Fhb)是由禾谷镰孢()引起的一种小麦重要穗部病害,主要发生于潮湿及半潮湿地区[1]。在小麦赤霉病大流行年份,可以造成70%,甚至是100%的产量损失[2]。小麦赤霉病的发生不仅造成严重的产量损失,而且感病的小麦籽粒还含有各种毒素(尤其是DON毒素),严重威胁人和动物[3-4]的食品安全。近年来,随着全球气候变暖以及耕作制度的变化,小麦赤霉病逐渐由偶发病害上升为常发病害,发生面积也不断扩大。在中国,赤霉病已由常发的长江中下游麦区扩展到黄淮麦区和华北麦区[5-6]。目前还没有彻底防治小麦赤霉病的化学药剂,最为经济环保的控制小麦赤霉病危害的途径就是培育和选用抗赤霉病品种。对普通小麦-长穗偃麦草()衍生小麦新品种进行赤霉病抗性鉴定和抗性基因的分子鉴别,可为深入研究和应用长穗偃麦草的赤霉病抗性基因提供理论依据,对于小麦赤霉病抗性育种具有重要意义。【前人研究进展】赤霉病抗源相对较少,国内外学者通过大量的基因资源研究,迄今从普通小麦及其近缘种属中仅鉴定出7个赤霉病抗性主效基因,分别是来自苏麦3号的[7]和[8]、大赖草的[9]、望水白的[10]和[11]、披碱草属的[12]和十倍体长穗偃麦草的[13]。中国工作者经多年努力,培育出了扬麦系列、宁麦系列和鄂麦系列等优良抗性品种,但大面积生产中缺乏抗赤霉病的高产小麦品种[14]。纵观国内数十年的小麦抗赤霉病育种,其抗源仍然主要局限于苏麦3号、望水白和它们的衍生系。赤霉病抗源基础狭窄是小麦育种工作取得突破性进展的瓶颈,发掘新的小麦赤霉病抗源成为提高小麦赤霉病抗性的关键[15]。由此可见,鉴定和利用小麦近缘种属的赤霉病抗性基因尤为重要[16]。长穗偃麦草是小麦的近缘植物,其E基因组含有许多优异的抗性基因,如抗旱、抗寒能力很强,对白粉病[17]和锈病完全免疫(7E染色体)[18],高抗条纹花叶病和小麦黄矮病(2E、7E染色体)[19-20],对赤霉病表现较强的耐病性(1E、7E染色体)[18,21-23],且又能与小麦进行远缘杂交,实现抗病基因的遗传转移。因此,长穗偃麦草是小麦抗病育种的重要抗源亲本之一[24]。八倍体小偃麦小偃693和小偃7430染色体总数均为56条,但是它们分别含有8对和6对长穗偃麦草染色体,还分别含有4条和2条小麦-长穗偃麦草易位染色体[25]。小偃7430与普通小麦杂交、回交选育而成了许多抗白粉病和条锈病、秆锈病的小偃麦衍生新品种(系)[26-28]。然而,目前关于小偃693衍生后代抗病性的研究鲜有报道。小偃597是利用小偃693与普通小麦杂交、回交选育成功的含有十倍体长穗偃麦草遗传物质的普通小麦品种,对赤霉病表现抗性[29]。研究表明,长穗偃麦草中已发现3个赤霉病抗性主效基因,分别定位于二倍体长穗偃麦草1E染色体[30-32]和7E染色体[22,32],十倍体长穗偃麦草7E染色体[13,18,23]。其中十倍体长穗偃麦草7E染色体长臂上赤霉病抗性基因被正式命名为[13],可解释抗赤霉病表型变异的30.46%[18]。Guo等[13]将定位在7EL染色体末端1.7 cM范围内,且两侧紧密连锁的分子标记分别为和;该抗病区间对应于水稻、短柄草和高粱的基因组大小分别为18.8、37.1和27.0 kb,并开发出与紧密连锁的特异分子标记,如等,这些分子标记可用于该基因的分子标记辅助选择。【本研究切入点】西农509、西农511和西农529是由西北农林科技大学农学院培育出的含有十倍体长穗偃麦草遗传物质的高产、优质、抗病的普通小麦新品种[33-34],田间赤霉病表现轻[6,33-34]。这些品种已在黄淮冬麦区南片的河南省(南部稻茬麦区除外)、安徽省沿淮及淮北地区、江苏省淮北地区和陕西省关中地区示范推广种植。2014—2016年在河南南阳和驻马店、江苏省徐州和安徽淮北生态区域等赤霉病重发区,这些品种赤霉病轻度发生,发病率仅为1%—3%,属于中抗或中感抗性水平[6,35-37]。然而,目前它们赤霉病的抗性遗传基础并不清楚。【拟解决的关键问题】对3个衍生新品种进行赤霉病抗性进行人工接种鉴定,然后用偃麦草E组染色体特异标记对西农509、西农511和西农529及其主要亲本小偃693、小偃597和十倍体长穗偃麦草进行分子鉴定,初步确定其赤霉病抗性基因的来源,在此基础上利用长穗偃麦草7EL染色体上的特异分子标记及与抗性基因紧密连锁的分子标记对实验材料进一步进行分子鉴别,为这些抗赤霉病的长穗偃麦草衍生新品种的推广和利用提供理论依据。
1 材料与方法
1.1 试验材料
普通小麦中国春(),十倍体长穗偃麦草;普通小麦-十倍体长穗偃麦草远缘杂交后代材料和品种:八倍体小偃麦小偃693(染色体组成为AABBDDEE)和普通小麦品种小偃597;新培育的普通小麦-十倍体长穗偃麦草衍生品种西农509、西农511和西农529。赤霉病抗性鉴定的对照品种苏麦3号、扬麦158、宁麦9号和安农8455。小偃693是本研究中3个普通小麦-长穗偃麦草衍生新品种的间接亲本,普通小麦品种小偃597是西农529和陕麦159的直接亲本,是西农511的间接亲本(图1)。
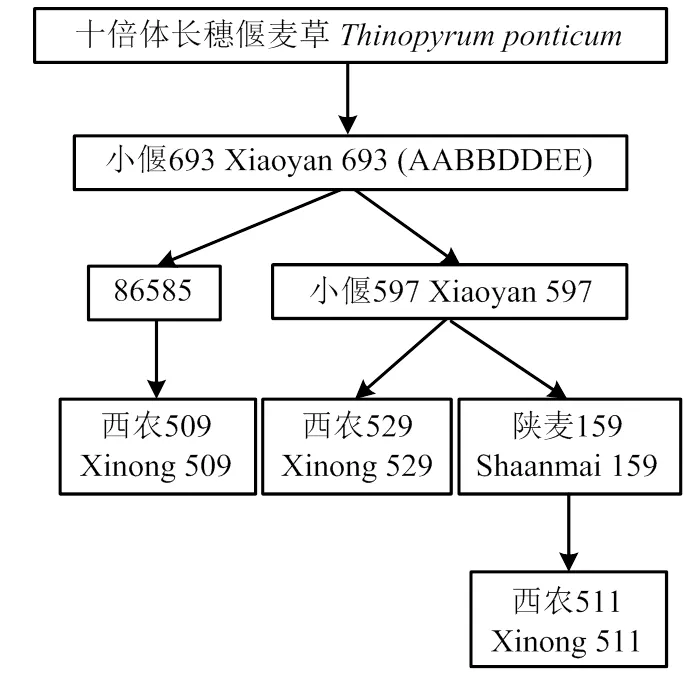
图1 普通小麦-长穗偃麦草衍生小麦新品种长穗偃麦草遗传基因溯源
1.2 赤霉病人工接种鉴定
抗赤霉病人工接种鉴定由江苏省农业科学院植物保护研究所在南京鉴定完成,赤霉菌菌株为F0301、F0609、F0980和F1126,鉴定方法和调查记载标准参见“小麦抗病虫性评价技术规范”系列标准(NY/T 1443—2007)[38]。苏麦3号、扬麦158和宁麦9号为抗病对照品种,安农8455为感病对照品种。抗性分级标准:抗(R),平均严重度<1.5(苏麦3号的平均反应级+标准差);中抗(MR),1.5≤平均严重度<2.5(扬麦158的平均反应级+标准差);中感(MS),2.5≤平均严重度<3.5,感(S),平均严重度≥3.5。
1.3 抗赤霉病基因的分子鉴别
小麦幼叶基因组DNA采用微量CTAB法 提取,干燥后溶于TE缓冲液。由于长穗偃麦草中已发现的赤霉病抗性主效基因均被定位于E基因组[13,18,21-23,30-32],本研究选用长穗偃麦草E组染色体特异分子标记[22,39-47]及7EL上连锁的分子标记[13,18,48-49]对参试材料进行长穗偃麦草遗传物质和的检测(附表1)。引物由生物工程(上海)有限公司合成。PCR扩增反应在GeneAmp 9700型热循环仪(ABI,USA)上进行,反应混合液总体积为10 μL,其中包括0.25 U酶、1.0 μL 10×Buffer、1.5 mmol·L-1MgCl2、0.2 mmol·L-1dNTPs、0.5 μmol·L-1primer(上下游引物)、50—100 ng模板DNA。扩增程序:94℃ 2 min;94℃ 1 min,50—60℃ 1 min,72℃ 1 min,35个循环;72℃ 5 min。PCR产物用8.0%的非变性聚丙烯酰胺凝胶上电泳检测,银染后照相观察并记录。
2 结果
2.1 3个普通小麦-长穗偃麦草衍生新品种的赤霉病抗性表现
江苏省农业科学院植物保护研究所先后对西农509(陕麦509)(2008—2010)、西农529(2013—2015)和西农511(2015—2016)进行了赤霉病抗性人工接种鉴定。由表1调查结果可以看出,西农509、西农529和西农511的赤霉病鉴定结果均与中抗对照品种扬麦158的抗性水平相当,表现为中抗。
2.2 普通小麦-长穗偃麦草衍生新品种外源遗传物质的分子标记检测
用105个长穗偃麦草E基因组的特异标记(其中1E、2E、3E、4E、5E、6E和7E染色体上特异标记分别有29、12、10、5、14、10和25个)分别对中国春、十倍体长穗偃麦草、西农509、西农511和西农529进行分子检测。结果显示,71个标记在中国春和十倍体长穗偃麦草间有多态性,但只有(图2-A)、、、、(图2-B)和等7个标记在3个普通小麦-十倍体长穗偃麦草衍生新品种中均扩增出长穗草的特异产物,其中前5个标记都位于7E长臂,和则位于7E短臂。E基因组分子标记分析结果表明,西农509、西农511和西农529携带来自于十倍体长穗偃麦草7E染色体的遗传物质。
2.3 普通小麦-长穗偃麦草衍生新品种及其亲本的分子检测
用定位于7E染色体长、短臂上的75和22个分子标记(包括DArT标记、SSR标记、EST标记)[13,36,43,45,48-49]分别对中国春、十倍体长穗偃麦草、3个普通小麦-长穗偃麦草衍生新品种及它们重要的亲本小偃693和小偃597进行分子标记分析。结果表明,7ES 64.63到7EL 153.27长约89 cM之间共有20对引物在小偃693、小偃597和3个新品种中均能扩增出稳定的长穗偃麦草的特异条带,其中12个引物定位于7E长臂,8个定位于7E短臂(表2)。由此进一步推断西农509、西农511和西农529这3个普通小麦-长穗偃麦草衍生新品种含有十倍体长穗偃麦草7E染色体跨着丝粒区域的中段遗传物质。
2.4 普通小麦-长穗偃麦草衍生新品种抗赤霉病基因的分子检测
用定位于7EL 140.03到160.00处的与紧密连锁的40个系列标记、7个系列标记、4个SSR标记(和)[13,36,43,45]分别对中国春、十倍体长穗偃麦草、3个普通小麦-长穗偃麦草衍生新品种以及赤霉病抗性基因的供体亲本小偃693和小偃597进行基因标记分型分析。结果表明(表2),(图2-C)(7EL 149.51)、(图2-D)、(图2-E)、和(7EL 153.27)(图2-A)等6个标记在3个新品种及小偃693和小偃597两亲本中具有十倍体长穗偃麦草的特异性条带;(7EL 153.77)、(7EL 154.70)、(7EL 156.27)(图2-F)和(7EL 158.02)(图2-G)4个标记能在八倍体小偃麦小偃693中扩增产生十倍体长穗偃麦草的特异性条带,而在小偃597和3个小麦新品种中没有十倍体长穗偃麦草的特异条带;(7EL 158.97)和(7EL 160.00)(图2-H)在小偃693、小偃597和3个小麦新品种中均没有十倍体穗偃麦草的特异条带。
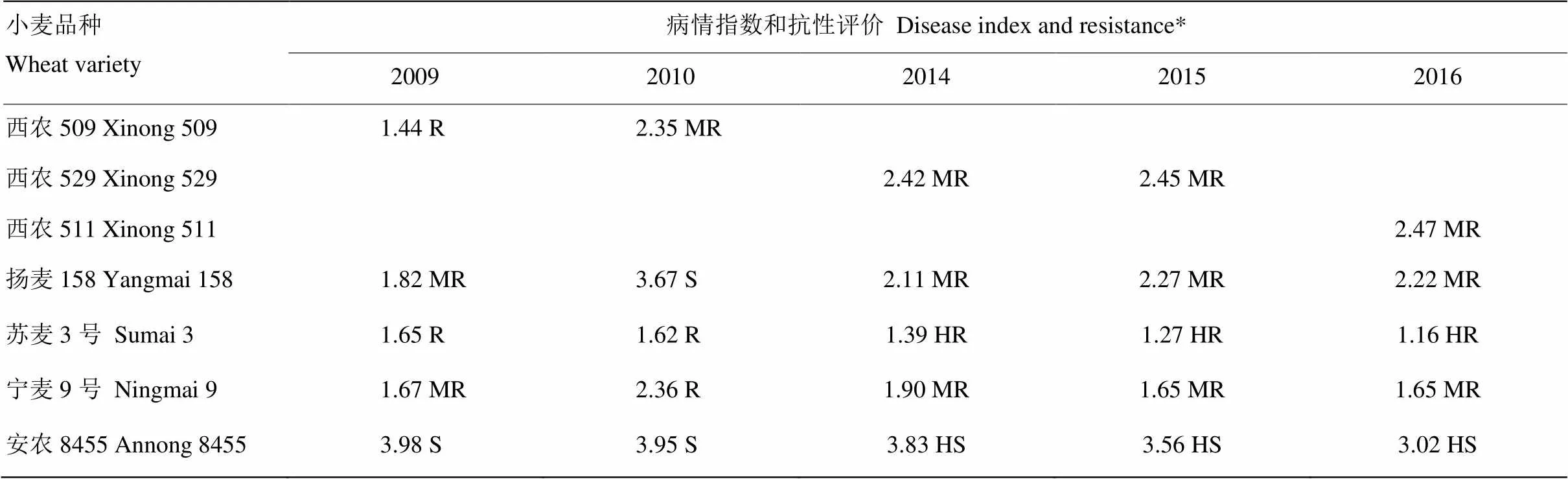
表1 3个普通小麦-长穗偃麦草衍生小麦新品种的赤霉病病情指数和抗性评价
*本试验数据来源于小麦国家区域试验赤霉病抗性鉴定结果 The data of this table were derived from the results of wheat regional resistance test for Fusarium head blight。R:抗病resistant;MR:中抗moderately resistant;S:感病Susceptible;HS:高感Highly susceptible

表2 7E染色体上部分分子标记对3个小麦新品种及其主要亲本的扩增片段大小
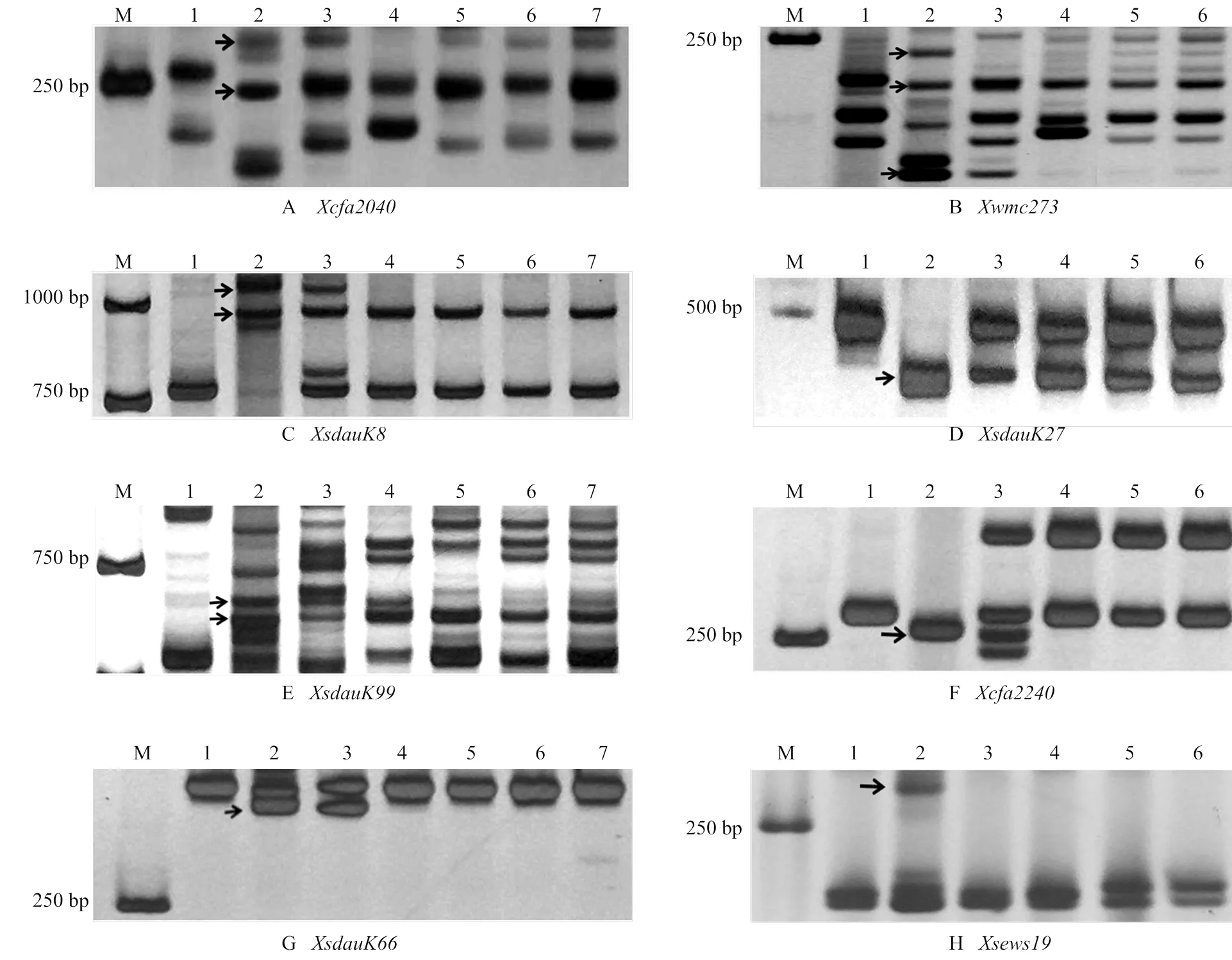
M:DEL2000;1:中国春Chinese Spring (CS);2:十倍体长穗偃麦草Th. ponticum;3:小偃693 Xiaoyan 693;4:小偃597 Xiaoyan 597;5:西农509 Xinong 509;6:西农511 Xinong 511;7:西农529 Xinong 529。左侧为分子量标记条帯大小(bp)。箭头所示为偃麦草染色体的特异条带The numbers at the top of the gels are lane numbers. The numbers on each photo is the marker bands size in bp. Arrows indicated the Thinopyrum specific bands linked toFhb7
3 讨论
在抗赤霉病育种方面,自20世纪70年代,中国研究者从普通小麦及其近缘物种3万多份材料中筛选出苏麦3号和望水白两个抗性强而稳定的普通小麦品种,但其农艺性状较差[50],应用过程中赤霉病抗性很难和丰产性结合起来,生产上亟需高产抗赤霉病的小麦品种。然而,自扬麦158后,大面积推广的品种均未解决好赤霉病抗性与丰产性负相关的问题[14]。因而开拓新的抗源以及如何高效利用小麦近缘种的抗病基因已成为小麦抗赤霉病育种亟待解决的重要问题[51]。
长穗偃麦草是小麦远缘杂交育种的重要亲本材料之一,研究表明其1E和7EL染色体上携带有抗赤霉病基因[13,18,21-23,26]。Jauhar等[31]将抗赤霉病的长穗偃麦草与硬粒小麦杂交,获得了赤霉病抗性提高显著的衍生系。张璐璐等[52]利用偃麦草7E染色体特异分子标记筛选结合GISH验证培育了小麦-长穗偃麦草7EL异位系和7ES附加系,赤霉病抗性鉴定表明7EL易位系的赤霉病抗性明显较好。西北农林科技大学培育的普通小麦-十倍体长穗偃麦草衍生新品种西农509、西农511和西农529目前已成为黄淮冬麦区南片和陕西省关中地区示范推广的高产优质的小麦新品种。据报道,上述3个小麦新品种在河南、安徽、江苏等赤霉病重发地田间表现赤霉病很轻[6,31-33]。本研究中,通过人工抗性鉴定,这3个品种的赤霉病抗性与扬麦158相当,属于中抗水平,且抗性稳定。西农509、西农511和西农529赤霉病轻且综合性状和丰产性好,有望在黄淮冬麦区南片赤霉病常发区和重发区大面积推广种植。
本研究利用长穗偃麦草E基因组特异的引物初步确定3个小麦新品种的长穗偃麦草的遗传物质来自7E染色体跨着丝粒区域的中段,说明其抗赤霉病基因来源于7E染色体,研究结果与前人的研究一致[13,18,22-23,52]。来自十倍体长穗偃麦草的抗赤霉病基因已定位于7EL染色体上,(7EL 156.27)和(7EL 158.02)分别与其紧邻[13]。为了探究上述3个小麦新品种中赤霉病抗性基因与的关系,选用偃麦草7EL染色体(7EL 140.03—7EL 160.00)上与连锁的分子标记对这3个新品种及其主要亲本小偃693、小偃597和十倍体长穗偃麦草进一步进行分子标记分析。结果发现,7EL上6个与紧密连锁的分子标记在3个新品种中扩增产生长穗偃麦草的特异产物,其中,和(7EL 153.27)3个标记与的遗传距离仅3.0—4.0 cM。Guo等[13]利用与抗赤霉病基因紧密连锁的分子标记(和)进行分子辅助选择,从济麦22与小麦-长穗偃麦草抗赤霉病短片段易位系SDAUl881杂交、回交后代中成功获得2个新的改良系SDAU2003和SDAU2028。然在本研究中,与紧邻的4个标记(7EL 153.27)、(7EL 154.70)、(7EL 156.27)和(7EL 158.02),在小偃597和3个长穗偃麦草衍生小麦新品种中均没有扩增出长穗偃麦草的特异性条带,只在八倍体小偃麦小偃693中有长穗偃麦草的特异性条带。同时,7EL末端的标记(7EL 158.97)和(7EL 160.00)不仅在3个小麦新品种中没有偃麦草特异条带,而且在小偃693和小偃597中也没有。何方[25]以十倍体长穗偃麦草总基因组DNA为探针对小偃693根尖细胞染色体进行GISH分析,发现小偃693中不仅含有4对Ee组染色体和4对Eb组染色体,同时还含有4条小麦-长穗偃麦草异位染色体。本研究中,西农509、西农511和西农529 这3个普通小麦-十倍体长穗偃麦草衍生新品种中含有长穗偃麦草7E染色体跨着丝粒区域的中段的遗传物质,但缺失了相关联的7EL末端片段,究其原因,也许是在八倍体小偃麦小偃693与普通小麦杂交、回交选育过程中,7E染色体与普通小麦染色体发生了异位,导致7EL末端片段丢失,这个推测有待借助GISH和FISH等技术进一步验证。
4 结论
西农509、西农511和西农529等3个普通小麦-十倍体长穗偃麦草衍生新品种在黄淮南片麦区赤霉病重发地区表现赤霉病很轻,同时人工接种鉴定表现为中抗赤霉病。赤霉病抗性基因分子鉴定表明,长穗偃麦草7ES 64.63—7EL 153.27之间有20对引物在3个新品种和它们的主要亲本小偃693和小偃597中均扩增出稳定清晰的长穗偃麦草特异条带,然而在这3个普通小麦-长穗偃麦草衍生新品种及小偃597中没有检测到7EL末端抗赤霉病主效QTL的分子标记产物。推测这3个普通小麦-长穗偃麦草衍生新品种含有来自于十倍体长穗偃麦草的不同于的抗赤霉病基因。
[1] GOSWAMI R S, KISTLER H C. Heading for disaster:on cereal crops., 2004, 5(6): 515-525.
[2] MATTHIES A, BUCHENAUER H. Effect of tebuconazole (Folicur®) and prochloraz (Sportak®) treatments on Fusarium head scab development, yield and deoxynivalenol (DON) content in grains of wheat following artificial inoculation with., 2000, 107(1): 33-52.
[3] PLACINTA C, DMELLO J P F, MACDONALD A M C. A review of worldwide contamination of cereal grains and animal feed withmycotoxins., 1999, 78(2): 21-37.
[4] GOSMAN N, SRINIVASACHARY A, STEED A, CHANDLER E, THOMSETT M, NICHOLSON P. Evaluation of type I Fusarium head blight resistance of wheat using non-deoxynivalenol-producing fungi., 2010, 59(1): 147-157.
[5] 曾娟, 姜玉英. 2012年我国小麦赤霉病暴发原因分析及持续监控与治理对策. 中国植保导刊, 2013, 33(4): 38-41.
ZEND J, JIANG Y Y. Outbreak reasons and control strategies of fusarium head blight in China during 2012., 2013, 33(4): 38-41. (in Chinese)
[6] 赵虹, 王西成, 胡卫国, 曹廷杰, 刘钊, 陈渝. 黄淮南片麦区小麦品种利用现状及建议. 河南农业科学, 2016, 45(8): 18-24, 38.
ZHAO H, WANG X C, HU W G, CAO T J, LIU Z, CHEN Y. Status and suggestion of wheat variety utilization in Southern Huang-Huai wheat region., 2016, 45(8): 18-24, 38. (in Chinese)
[7] CUTHERT P A, SOMERS D J, THOMAS J, CLOUTIER S, BRULE-BABEL A. Fine mapping, a major gene controlling fusarium head blight resistance in bread wheat (L.)., 2006, 112(8): 1465-1472.
[8] CUTHER P A, SOMERS D J, BRULE-BABEL A. Mapping ofon chromosome 6BS: a gene controlling fusarium head blight field resistance in bread wheat (L.)., 2007, 114(3): 429-437.
[9] QI L L, PUMPHREY M O, FRIEBE B, CHEN P D, GILL B S. Molecular cytogenetic characterization of alien introgressions with genefor resistance to fusarium head blight disease of wheat., 2008, 117(7): 1155-1166.
[10] XUE S L, LI G Q, JIA H Y, XU F, LIN F, TANG M Z, WANG Y, AN X, XU H B, ZHANG L X, KONG Z X, MA Z Q. Fine mapping, a major QTL conditioning resistance to Fusarium infection in bread wheat (L.)., 2010, 121(1): 147-156.
[11] XUE S L, XU F, TANG M Z, ZHOU Y, LI G Q, AN X, LIN F, XU H B, JIA H Y, ZHANG L X, KONG Z X, MA Z Q. Precise mapping, a major QTL conditioning resistance toinfection in bread wheat (L.)., 2011, 123(6): 1055-1063.
[12] CAINONG J C, BOCKUS W W, FEND Y G, CHEN P D, QI L L, SEHGAL S K, DANILOVA T V, KOO D H, FRIEBE B, GILL B S. Chromosome engineering, mapping, and transferring of resistance to Fusarium head blight disease frominto wheat., 2015, 128(6): 1019-1027.
[13] GUO J, ZHANG X L,HOU Y L, CAI J J, SHEN X R, ZHOU T T, XU H H, OHM H W, WANG H W, LI A F, HAN F P, WANG H G, KONG L R. High-density mapping of the major FHB resistance genederived fromand its pyramiding withby marker-assisted selection., 2015, 128(11): 2301-2316.
[14] 程顺和, 张勇, 别同德, 高德荣, 张伯桥. 中国小麦赤霉病的危害及抗性遗传改良. 江苏农业学报, 2012, 28(5): 938-942.
CHENG S H, ZHANG Y, BIE T D, GAO D R, ZHANG B Q. Damage of wheat Fusarium head blight (FHB) epidemics and genetic improvement of wheat for scab resistance in China., 2012, 28(5): 938-942. (in Chinese)
[15] GILBERT J, HABER S. Overview of some recent research developments in fusarium head blight of wheat,, 2013, 35(2): 149-174.
[16] GILL B S, FRIEBE B R, WHITE F F. Alien introgressions represent a rich source of genes for crop improvement., 2011, 108(19): 7657-7658.
[17] LIU S B, WANG H G. Characterization of a wheat-intermedium substitution line with resistance to powdery mildew., 2005, 143: 229-233.
[18] ZHANG X L, SHEN X R, HAO Y F, CAI J J, OHM H W, KONG L R. A genetic map ofchromosome 7E, haroring resistance genes to Fusarium head blight and leaf rust., 2011, 122(2): 263-270.
[19] ZHANG Z Y, XU J S, XU Q J, LARKIN P, XIN Z Y. Development of novel PCR markers linked to the BYDV resistance geneuseful in wheat for marker-assisted selection., 2004, 109(2): 433-439.
[20] AYALA-NAVARRETE L, THOMPSON N, OHM H, ANDERSON J. Molecular markers show a complex mosaic pattern of wheat-intermedium translocations carrying resistance to YDV., 2010, 121(5): 961-970.
[21] 英加, 陈佩度, 刘大钧. 将和的种质导入普通小麦的研究. 西北植物学报, 2000, 20(3): 321-326.
YING J, CHEN P D, LIU D J. Studies on transfer germplasm fromandinto common wheat., 2000, 20(3): 321-326. (in Chinese)
[22] SHEN X R, KONG L R, OHM H. Fusarium head blight resistance in hexaploid wheat ()-genetic lines and tagging of the alien chromatin by PCR markers., 2004, 108(5): 808-813.
[23] SHEN X R, OHM H. Molecular mapping ofderived Fusarium head blight resistance in common wheat., 2007, 20(2): 131-140.
[24] 李振声, 陈漱阳, 张楷. 普通小麦与长穗偃麦草的杂交育种及其遗传分析. 遗传学报, 1977, 4(4): 283-293.
LI Z S, CHEN S Y, ZHANG K. The cross breeding and its genetic analysis betweenand., 1977, 4(4): 283-293. (in Chinese)
[25] 何方. 小麦—长穗偃麦草杂种后代的分子细胞遗传学分析及种质材料的筛选鉴定[D]. 泰安: 山东农业大学, 2014.
HE F. Molecular cytogenetic analysis of wheat-hybrids and identification ofgermplasms [D]. Taian: Shandong Agricultural University, 2014. (in Chinese)
[26] 贺润丽, 畅志坚, 刘建霞, 詹海仙, 张晓军, 董春林. 源于长穗偃麦草的小麦新品系CH7034抗白粉病基因的染色体定位. 分子植物育种, 2008, 6(2): 251-256.
HE R L, CHANG Z J, LIU J X, ZHAN H X, ZHANG X J, DONG C L. Chromosomal location of powdery mildew resistance gene in-derived wheat germplasm line CH7034., 2008, 6(2): 251-256. (in Chinese)
[27] 白云, 李欣, 张丛卓, 张晓军, 詹海仙, 畅志坚. 小麦新抗源CH7103抗条锈基因的遗传及其与已知基因的关系. 麦类作物学报, 2011, 31(2): 364-369.
BAI Y, LI X, ZHANG C Z, ZHANG X J, ZHAN H X, CHANG Z J. Inheritance of stripe rust resistance gene in wheat line CH7103 introgressed fromand its allelism with known genes., 2011, 31(2): 364-369. (in Chinese)
[28] ZHENG Q, LV Z L, NIU Z X, LI B, LI H W, XU S S, HAN F P, LI Z S. Molecular cytogenetic characterization and stem rust resistance of five wheat-ponticum partial amphiploids., 2014, 41(11): 591-599.
[29] 张荣琦, 陈春环, 赵晓农, 钟冠昌. 利用远缘杂交技术选育小麦新品种之研究. 中国农学通报, 2006, 22(6): 186-188.
ZHANG R Q, CHEN C H, ZHAO X N, ZHONG G C. The study on selection of wheat varieties with distant hybrid technique., 2006, 22(6): 186-188. (in Chinese)
[30] 刘登才, 郑有良, 王志容, 侯永翠, 兰秀锦, 魏育明. 影响小麦赤霉病抗性的染色体定位. 四川农业大学学报, 2001, 19(3): 200-205.
LIU D C, ZHENG Y L, WANG Z R, HOU Y C, LAN X J, WEI Y M. Distribution of chromosomes in diploid(Host) A. Löve that influences resistance to head scab of common wheat., 2001, 19(3): 200-205. (in Chinese)
[31] JAUHAR P P, PETERSON T S, XU S S. Cytogenetic and molecular characterization of a durum alien disomic addition line with enhanced tolerance to Fusarium head blight., 2009, 52(5): 467-483.
[32] 陈士强, 黄泽峰, 张勇, 葛江燕, 朱雪, 高勇, 陈建民. 中国春背景下长穗偃麦草抗赤霉病相关基因的染色体定位. 麦类作物学报, 2012, 32(5): 839-845.
CHEN S Q, HUANG Z F, ZHANG Y, GE J Y, ZHU X, GAO Y, CHEN J M. Chromosomal location of the genes associated with FHB resistance ofin Chinese Spring background., 2012, 32(5): 839-845. (in Chinese)
[33] 张荣琦, 陈春环, 吉万全. 高产抗病优质小麦新品种西农509的选育及稳定性分析. 种子, 2014, 33(10): 100-101.
ZHANG R Q, CHEN C H, JI W Q. The selective breeding and stability analysis of high-yield, disease-resistant and good-quality wheat cultivar Xinong 509., 2014, 33(10): 100-101. (in Chinese)
[34] 吉万全, 张荣琦, 陈春环, 王长有, 张宏, 朱建峰, 王亚娟, 刘新伦, 田增荣, 蔡东明. 优质高产小麦新品种—西农529. 麦类作物学报, 2014, 34(7): 1019.
JI W Q, ZHANG R Q, CHEN C H, WANG C Y, ZHANG H, ZHU J F, WANG Y J, LIU X L, TIAN Z R, CAI D M. Good quality and high yield wheat cultivar Xinong 529., 2014, 34(7): 1019. (in Chinese)
[35] 严虎, 唐怀坡, 王瑞永. 2015-2016年度西农大新马桥点小麦新品种示范及产量分析. 安徽农学通报, 2016, 22(24): 148-149.
YAN H, TANG H P, wang R Y. New wheat varieties demonstration and yield analysis in Xinmaqiaodian Northwest A&F University during 2015 and 2016., 2016, 22(24): 148-149. (in Chinese)
[36] 孙道杰, 张玲丽, 冯毅, 陈春环, 张荣琦, 奚亚军, 何心尧, 王辉, 宋哲明. 西农系列小麦骨干新品种赤霉病抗源浅析. 麦类作物学报, 2016, 36(6): 822-823.
SUN D J, ZHANG L L, FENG Y, CHEN C H, ZHANG R Q, XI Y J, HE X Y, WANG H, SONG Z M. Analysis of FHB resistance sources for newly released Xinong varieties., 2016, 36(6): 822-823. (in Chinese)
[37] 蔡诚, 何流, 徐超艳, 单府, 陶珂, 杨磊, 汪文彦, 刘萍. 2015-2016年度12个小麦新品系在淮北地区的生态适应性试验. 安徽农学通报, 2016, 22(19): 48-49, 84.
CAI C, HE L, XU C Y, SHAN F, TAO K, YANG L, WANG W Y, LIU P. The experiment in the ecological adaptability of 12 new wheat varieties in Huaibei area during 2015 and 2016., 2016, 22(19): 48-49, 84. (in Chinese)
[38] 中国人民共和国农业部. 小麦抗病虫性评价技术规范. 第4部分:小麦抗赤霉病评价技术规范: NY/T 1443.4-2007[S]. 2007-01-14 [2017-05-17].
Ministry of Agriculture of the People’s Republic of China. Rules for resistance evaluation of wheat to diseases and insect pests part 4: Rules for resistance evaluation of wheat to wheat scab {Schwabe [Teleomorph(Schwein) Petch]}: NY/T 1443.4-2007[S]. 2007-01-14[2017-05-17]. (in Chinese)
[39] MULLAN D J, PLATTETER A, TEAKLE N L, APPELS R, COLMER, ANDERSON J M, FRANCKI M G. EST-derived SSR markers from defined regions of the wheat genome to identifyspecific loci., 2005, 48(5): 811-822.
[40] 秦树文, 戴毅, 陈士强, 张璐璐, 刘慧萍, 曹文广, FEDAK George, 高勇, 陈建民. 基于TRAP的长穗偃麦草SCAR标记的开发及应用. 麦类作物学报, 2014, 34(12): 1595-1602.
QIN S W, DAI Y, CHEN S Q, ZHANG L L, LIU H P, CAO W G, FEDAK G, GAO Y, CHEN J M. Development and applications of SCAR markers specific toby TRAP technology., 2014, 34(12): 1595-1602. (in Chinese)
[41] 张丽, 颜泽洪, 郑有良, 刘登才, 代寿芬, 张连全, 魏育明. 小麦中国春背景下长穗偃麦草Ee染色体组特异AFLP及STS标记的建立. 农业生物技术学报, 2008, 16(3): 465-473.
ZHANG L, YAN Z H, ZHENG Y L, LIU D C, DAI S F, ZHANG L Q, WEI Y M. Development of Ee-chromosome specific AFLP and STS molecular marker forin Chinese Spring wheat background., 2008, 16(3): 465-473. (in Chinese)
[42] 陈士强, 秦树文, 黄泽峰, 戴毅, 张璐璐, 高营营, 高勇, 陈建民. 基于SLAF-seq技术开发长穗偃麦草染色体特异分子标记. 作物学报, 2013, 39(4): 727-734.
CHEN S Q, QIN S W, HUANG Z F, DAI Y, ZHANG L L, GAO Y Y, GAO Y, CHEN J M. Development of specific molecular markers forchromosome using SLAF-seq technique., 2013, 39(4): 727-734. (in Chinese)
[43] HU L J, LIU C, ZENG Z X, LI G R, SONG X J, YANG Z J. Genomic rearrangement between wheat andrevealed by mapped functional molecular markers., 2012, 34: 67-75.
[44] XUE S, ZHANG Z, LIN F, KONG Z, CAO Y, LI C, YI H, MEI M, ZHU H, WU J, XU H, ZHAO D, TIAN D, ZHANG C, MA Z. A high-density intervarietal map of the wheat genome enriched with markers derived from expressed sequence tags., 117: 181-189.
[45] Ishikawa G, Yonemaru J, Saito M, Nakamura T. PCR-based landmark unique gene (PLUG) markers effectively assign homoeologous wheat genes to A, B and D genomes., 2007, 8: 135.
[46] 黄泽峰. 长穗偃麦草分子标记开发及硬粒小麦-长穗偃麦草附加系创建[D]. 扬州: 扬州大学, 2013.
HUANG Z F. Development of molecular markers forand establishment of-alien disomic addition lines[D]. Yangzhou: Yangzhou University, 2013. (in Chinese)
[47] 朱雪. 小麦中国春背景中长穗偃麦草E染色体组SCAR标记发展[D]. 扬州: 扬州大学, 2011.
ZHU X. Development of E-chromosome specific SCAR markers forin Chinese spring wheat background[D]. Yangzhou: Yangzhou University, 2011. (in Chinese)
[48] 郭军. 长穗偃麦草抗赤霉病基因遗传图谱的加密及其标记辅助转移[D]. 泰安: 山东农业大学, 2015.
GUO J. Saturation mapping of FHB resistance genederived fromand its marker assisted selection[D]. Taian: Shandong Agricultural University, 2015. (in Chinese)
[49] ZHANG X L, SHEN X R, HAO Y F, CAI J J, OHM H W, KONG L R. A genetic map ofchromosome 7E, harboring resistance genes to Fusarium head blight and leaf rust., 2011, 122: 263-270.
[50] 陆维忠, 程顺和, 王裕中. 小麦赤霉病研究. 北京: 科学出版社, 2001: 78-170.
LU W Z, CHENG S H, WANG Y Z.. Beijing: Science Press, 2001: 78-170. (in Chinese)
[51] 姚金保, 陆维忠. 中国小麦抗赤霉病育种研究进展. 江苏农业学报, 2000, 16(4): 242-248.
Yao J B, Lu W Z. Research advances in wheat breeding for scab resistance in China., 2000, 16(4): 242-248. (in Chinese)
[52] 张璐璐, 陈士强, 李海凤, 刘慧萍, 戴毅, 高勇, 陈建民. 小麦-长穗偃麦草7E抗赤霉病易位系. 中国农业科学, 2016, 49(18): 3477-3488.
ZHANG L L, CHEN S Q, LI H F, LIU H P, DAI Y, GAO Y, CHEN J M. Development of wheat -translocation lines resistant to Fusarium head blight., 2016, 49(18): 3477-3488. (in Chinese)
(责任编辑 岳梅)
附表1 所利用的长穗偃麦特异分子标记
Table s1 Specific molecular markers ofand their sequences used in this study
标记名称Marker name引物序列Nucleotides sequence of markers所在染色体genome location扩增片段大小Fragment size (bp)参考文献Reference左引物Left primer (5′-3′)右引物Right primer (5′-3′) 1Ee STS241TAACTAGCACACCAAATCGTTTTTGCAAACAATTCATTGGGGTGGGTAAAGGAAGTGG1E250[41] 1Ee STS318GAATTCATCATAAAAACAGATCAAAGGTAACGTAAACTTCCCGTCTGCTAC1E280[41] SP1E_No.1 TATCAAATAATATGTGCATG CATGACAGAGATATGAAAAG 1E276[42] SP1E_No.21 CATCATATAAGAATGCAGGA TCAACAGCACGCCAAC 1E230[42] SP1E_No.23 TAGGAAAGAAATCTGAAATA TGAGACAGCAGCAAAA 1E220[42] SP1E_No.27 GGCTGATGCACTGACGT TCCGAAATCTGAAAAGGAG 1E273[42] SP1E_No.29 ACTGTTTTTTTTTTCGAG AGCGCACAAGATGAGAC 1E151[42] SP1E_No.30 TTAGAAAAATTCAACCCG AATCTTGAAACATGGGAG 1E80[42] SP1E_No.31 CCGGTTTGAAAGAAGAGA CAACAAGCAAATGAGGGA 1E158[42] SP1E_No.33 TCACCTATGAGATCGCT TATCCGAAGAACCTGTAC 1E285, 175[42] SP1E_No.34 TGTTCCTTGCATCATCT AACTCACCTATCGGTCTAT 1E158[42] SP1E_No.35 TTGACAACCCAATCTG TCACTGATAAATAACAAGAG 1E162[42] SP1E_No.36 TGACGGCAACGGAGTG TCTGCCGAACATGGAATC 1E150[42] SP1E_No.37 AGCACTTGGGTTTTCAT GGATTGGAGGGTCACTA 1E260[42] SP1E_No.38 TTCAAGAAGTATAGCTCGTT AACCATAAAGTCCCAACA 1E300[42] SP1E_No.41 GGAAAGGGCACTCCAACC GCGTCACATCCTGGTTCG 1E210[42] SP1E_No.42 CTCCATCCCTCCACTT AACATCAGCAGTTGGG 1E190[42] SP1E_No.44 TTGACTTCTCCCAAGTTG AATGGAAAGGAAAATCTG 1E203[42] SP1E_No.45 TCTATTTTGCTATTTGCC TTTTGTCTTCATCACCAT 1E568[42] SP1E_No.47 TACTCGCTACTTACAATCT GAACATTATTGTGAAGCA 1E255[42] SP1E_No.48 CGGAATGTCCTCACA ATTGGCTAATGCTATGA 1E280[42] SP1E_No.8 AATATTTATGGCGCACGCAG TCATCAGGGGAGTGAGGAGC 1E400, 200[42] Xedm17TAGCTGCATCTCTGGTTTGGCTAGTTGTTGTTGCTGCGAAA1E396[39] Xedm74GTAAGTCGACCGAGGAGACGCCTTCTTGCTTGGCATTCTC1EL360[39] Xedm92ATCCGCGACGACAATGTAAGCTGGTGTGGATCTTGACGTG1EL185[39] Xmag2137 GAGTTCGATGACATGGCTGACCGATAACAAAGTGCGTGAA1E580, 250[43-44] Xmag3489CGATGTAAAGACTGTAGCCTGTAGGCGTCCATTTGGTTCGGTGAG1E330[43-44] Xmag834 ACGCCCTCAAACAAGAAAGACTTCCTCTCCTTCCCCAGAG1E500[43-44] Z34-1E200GATGTCACCAAGCCCTACTCCTACGAAACACCCAAG1E225[47] 2Ee STS116TTAACCAGGGATACAAAAACACCATAATGAATTCAATTACCTTGTGGATGCAAACAC2E150[41] 2Ee STS182AATTCATTTCTAATAGATACTCGAATGAATTAACTGAGGAGAGGCTACAACATCC2E182[41] SM2E-1CAGGTCCAAAAAACCAGCC GAATTCGAGGTTCCGCAG 2E480, 210[40] SM2E-3GTGTGTGCGCATGAACTACTT GGAGTCGTTGGTTGGACATG 2E225[40] SM2E-5ATGCTTTTCCCTTCCAACAG CAAACGAAAAGCTGACCAAAC 2E173[40] Xmag2090 AAAGGTGTAAGCGTGGCATCACACGCATAATGCACTCCAG2E300, 255[43-44] Xedm28GCTCACTCACGCATCATAGCGTTGGCGGAATCCTTCTTC2ES & 3EL150[39] Xedm8GACCCCCACTTCCTGCTCGGGCAAAACTGTGGCTATTG2ES & 3EL145[39] Xedm96TCCAAGACTGTGCCTCAAGAGCGTTGAACTCCTGTTCCAT2ES & 3ESL175[39] Xmag3253 CTGCTGCTTGGGATCATTCTGCTGGTGAGAGTTGGAAACC2E600, 310[43-44] Xmag3616 GTCCGTGCCATTCAACTTCTTCTATCCACTCCATTCCAAGC2E&3E260, 200[43-44] Xmag4137 CTCGTGTGGCTCATCAATGTTGACATCTGGTTGGTGGTCA2E&3E490[43-44] iPBS8TGCAGGTCGACGAGCGGGTATGCTTTG3E428[46] SM3E-1CACACGCAATTCAACAACTCAAG ACGAATTGACCGTCGATCAGA 3E195[40] SM3E-2GCAGCAGAGAACGTACACTTGTATGGCAAAAGTATTCTGAACCGT3E154[40] TNAC1248ATGATGCAGCAGCAAATTACACTGAGGAGCCTCTCCAACTCT3E700[43,45] Xmag905 ATGTGAATGGAAGGTCGGAGAGCACTTGCAGGCTCTTCAT3E260[43-44] SM4E-1ATTCCTAATGGTCCTACAGC ATCACGCACGAACAAGTC 4E580[40] Xmag1682 CGAATGCCAAGCTGTTCCCTACATGCCCCTTGAGAGTGTGG4E855[43-44] Xmag2055 TTCCCAGCAAAGTAGCACAATGTGGGATCGCCTAGAAATC4E550[43-44] Xmag3733 CACACAACTCCACGGAACAGCATAAACCAGCAGCCCAACT4E&6E300[43-44] Z8-4E374TGTTGCCAGTGGAGGAAGTTGAACTACTGAACCGAAT4E368[47] TNAC1503TGAGGTTGGTTCTCATCTGGACGTTGGAAACAATCTGAATGG5E700.00[43,45] TNAC1559AAACAAGGCCCTGAAACACTTCATTGTCAGGCTATGGGACAT5E1180[43,45] Xmag1293 GATTGTTCTTGGGGAGATGCTTGGCCTTTTGGACCGTCAGAGAC5E260, 300[43-44] Xmag1426 GCGAGTTTTCGTAGCAAAGGTCACAGGAGTGGAGGCTCAC5E650, 280[43-44] Xmag1579 AAGGCATTTTTCTCGGTCTCCACACTTCTTGAGGATTTGAGC5E550[43-44] Xmag4013 TCGACTCCCATCCTCAAGACCACCACCATTCCTTTGGAG5E650, 400[43-44] Xmag532 CGACGATCCAACCAGCTAATTAGCCAGCCTATGGTAACGG5E250[43-44] Xmag620 TAGTTGCATGGTCGCTTCTGCGTAGCTTTTCGTTGATCCC5E295[43-44] Xedm54CGTCCTTGTTCTTGCTTCAATGAGCAGGCCATAAGGAAAC5ES150[39] Xedm205AGTCGCCGTTGTATAGTGCCCGACCCGGTTCACTTCAG5ES240[39,49] Xgwm205CGACCCGGTTCACTTCAGAGTCGCCGTTGTATAGTGCC5ES180[39] Xedm46CCGCCGTCCGTCCTGCAGGAGGCCTCGTTGTAG5EL & 7EL215[39] Xedm335CGGTCCAAGTGCTACCTTTCCGTACTCCACTCCACACGG5EL & 7EL150[39,44] Xgwm335CGTACTCCACTCCACACGGCGGTCCAAGTGCTACCTTTC5EL & 7EL150[39] TNAC1674CCACCACAGAAGCAGATGAATGCTAGATGGCACACCAAGTG6E700[43,45] Xedm140CCTCCGACGAGATCTGCTACGCTTTGGCTTCCGGTACG6E271[39] Xgwm179AAGTTGAGTTGATGCGGGAGCCATGACCAGCATCCACTC6E162[39] Xmag2276 CCGCCCCGATACAGTTTATTTCTACCGCTCCGTCGTCAACTACTG6E320[43-44] Xmag3017 GGTGAGGGAGTGCGAGAATAGGCTTCCTGTTGCTGTGG6E260, 170[43-44] Xmag3113 TCATGCACCCATCTGGACTAAGCTATTCGTGGCAAACACA6E210[43-44] Xedm149ATCCACGCCAAGCAGAAGCTGTGGGAAGAAGTGCCTTG6EL186[39] Xedm80ATGCTCAAGCCGAGGAAGTATTGATGGAGCGACTGAAATG6ES126, 150[39] Xgwm149CATTGTTTTCTGCCTCTAGCCCTAGCATCGAACCTGAACAAG6ES85[39] Xgwm292TCACCGTGGTCACCGACCCACCGAGCCGATAATGTAC6ES163[39] Xedm158CTACGTCGTCTACCGCTGCTCTTGGGCATCATCGATCTTC7E120[39] Xbarc76ATTCGTTGCTGCCACTTGCTGGCGCGACACGGAGTAAGGACACC7EL210[49] TNAC1825GAAGCGGTTCAGGGTGACACCCATCACGTTGCTGTAGTC7EL750[43,45] TNAC1826CACATATGATGATGACGGCAATGGCAGGGAGGAAACTCTACTG7EL152.00[43,45] TNAC1829GCCACTTCCTCCCTCCTCGTCGGTCCTCCAGTATCAGC7EL210[43,45] TNAC1834CCGAGTGTCATCGTTAGGAAACCAGCAATCCTACCTGTTGAA7EL1900[43,45] TNAC1845AATGAACAGCTTGCTTTCTGCCAGATGCTCTGGATTTCATGG7EL120, 90[43,45] TNAC1956ACGAAGGACAATTGCTGCTAAGTGCACTTCTTGCCCTACTTG7EL1074[43,45] TNAC1957TCAACATTTGCAGGATTGTCATTTCACAGGAACCTCTGCATC7EL170[43,45] XBE399084CCTACCTACGACGCAAGTCCAGCGAGCAGAAAGCATCAAG7EL132[13] Xcfa2040TCAAATGATTTCAGGTAACCACTATTCCTGATCCCACCAAACAT7EL280, 240[49] Xcfa2106GCTGCTAAGTGCTCATGGTGTGAAACAGGGGAATCAGAGG7EL240[49] Xcfa2240TGCAGCATGCATTTTAGCTTTGCCGCACTTATTTGTTCAC7EL250[49] Xedm105ACCGCCAGGGAGCTCTGCGATGTCCTTCTGGCCGTACT7EL240[39] Xedm154GGAGAGGCCACTGTTTTTGAGGCACATCACTCCCTTCTTC7EL230[39] Xgwm130AGCTCTGCTTCACGAGGAAGCTCCTCTTTATATCGCGTCCC7EL250[49] Xgwm295CGTACTCCACTCCACACGGCGGTCCAAGTGCTACCTTTC7EL288[49] Xgwm333GCCCGGTCATGTAAAACGTTTCAGTTTGCGTTAAGCTTTG7EL300[49] Xgwm344CAAGGAAATAGGCGGTAACTATTTGAGTCTGAAGTTTGCA7EL130[49] Xgwm573AAGAGATAACATGCAAGAAATTCAAATATGTGGGAACTAC7EL182[49] Xgwm635TTCCTCACTGTAAGGGCGTTCAGCCTTAGCCTTGGCG7EL130[49] Xksum052 GTCGCTGAGTGCGAACTCCG CAGAGGAAGCCGTCTACTGG 7EL240[49] Xmag1759GTACGAGTATGGCAACAGCCCAACTCTAGGGGCAGATG7EL620, 250[13,43-44] Xmag1932TGGCGTCCTGCTGAAAACCCGAGCACAAACCCTTGG7EL285,235[13,43-44] Xmag2931GGGGGCAGAACTTGTATGAATCTTGAGGGACGTGAGGAAC7EL300[13,43-44] Xmag3283TGCCTGACCTACTGCAACACGAATAGCCATCAACCGCAAG7EL1820[13,43-44] Xpsp3003GATCGACAAGGCTCTAATGCCAGGAGGAGAGCCTCTTGG7EL200[13] Xpsp3123AGATTAGATGGCATAAGCAAAGAGCGATTAATCACCGCCCTTGGTCC7EL120[49] Xpsr680 GCTCCACTACTTCATCATCC TGTTTCCTCTATCACTGACTTG 7EL246[49] XsdauK107AAGACGCAGTGTCTCGAGGTTGCATCCTCAACATTCTCCA7EL100[13,48] XsdauK116TCTGCGCCTTGTATCCTTCTGGAGTTCCTGGATGTCGTGT7EL230[13,48] XsdauK118GTTCAGTTCCACGCCCAATAATTATGCGCCGGGCTTCT7EL170[13,48] XsdauK124CAGTGCTGCAGGAGCTACAAAGAACCCTTCGGTGGAAACT7EL500[13,48] XsdauK13GCCCTGTTACAGTCCACGATGAGCTCCTTGCTCTGATTGG7EL300[13,48] XsdauK130TCACACAACCTGCATTGGATATGCCATACGCATTTCCAG7EL900, 800[13,48] XsdauK138GCAACCCAGTACGCCTAGTTCAAATTCGAAAGCCCACATT7EL400[13,48] XsdauK140AGGGTCTGTTTGGTTGGATGAGGCGTGAGAAGTCAATGCT7EL390[13,48] XsdauK142GTCTCGTTGCAATTGAGGTGGAGGCGAGCTGGAAATACAA7EL375[13,48] XsdauK144GGGCAGGAAAGACAACACTTTTCCCAGCTTTGCTCTTCAT7EL440[13,48] XsdauK159GTCTCGTTGCAATTGAGGTGGAGGCGAGCTGGAAATACAA7EL370[13,48] XsdauK165ACTGCGTCAGATCCGAAGTTTGCAGCAATGAAGTCAGGAA7EL1600[13,48] XsdauK176AGGAGGCCTTCATGCAGATATCGCCATGACTTCCTCTTCT7EL775[13,48] XsdauK27TAAGGTGGCTCCGTTGTCTCCCTCCAGAGCCTCTTTCTCC7EL465[13,48] XsdauK32AGGCCGGAGTCGATTCTATTATCACAGTGCGCTCATCAAA7EL250[13,48] XsdauK336TCATGTATTTGGACAATGTTAAACGAGACAGCTCCATCGGAAAAA7EL400[13,48] XsdauK339CAATTTGCTTCAAGGGGCTATTGGGCTCTTCTTCCCCTAT7EL425[13,48] XsdauK340ACACTCATGGGAGGACGAAGGCTACTAGCAATGGCGGAAC7EL357[13,48] XsdauK341TCAGTCCCGTCACTGTCAACGTCGCCTGCAGGTAGGAGTA7EL250[13,48] XsdauK342TTTACCAAGTGGGTGGAAGCTCCATAAAACGGAAGGCAAC7EL325[13,48] XsdauK343ACGAGGGTGGGAAGAAAGATAGCCAATTTCGCTGCTTAAA7EL324[13,48] XsdauK345GAAACTAGGGGGTCGGGTAGTCAATTTACCAGGGGCATTC7EL500, 410[13,48] XsdauK348GCCAGATAAGAACCCCGTTTGTGGTAATTTTTAGGGAGTGC7EL280, 145[13,48] XsdauK350GAAAGGCAACGAAAATGGAAGGTACATGATTTCCCCATGC7EL240[13,48] XsdauK351AGTCACCCACAAGGACCAACTGGTGCCCATCTTCTGGTAT7EL775[13,48] XsdauK352CTTTCCAACGCCGTATCATTATTGCGCTGACTGTTGTTTG7EL440[13,48] XsdauK353GGCAGGCACCATATTGAAGTTTGCCAACCACGTAGTGAAA7EL272[13,48] XsdauK355GCGAGAACAATAGCCTGATTTTTTTAATGCATGTTCAGCATTTTT7EL400[13,48] XsdauK356ATAGGTTTCTCCCGGCTTTGAAATCTTCACGAATTCAAAACA7EL140[13,48] XsdauK36CCCTGCATGAGTTGTCAATGCATCCTCCAAGGTCATCACA7EL286[13,48] XsdauK38GAAGATGGCGAAGGTGTAGCTTACACTGTCATCGCCGTCT7EL345[13,48] XsdauK4CATCAAGTACGGCGTCGAGATCGGTCACAAGGCTATTCG7EL745, 640[13,48] XsdauK45GGGTTCCTGCTCAAGTTCCTGGCCTCCTTATGACCACAAA7EL860[13,48] XsdauK6CGCGCTCTTCTACTGGTTCTGTAGAAATCGTGGCCCTTGA7EL555[13,48] XsdauK60TGGCGAAGATTTCTTCAAGTCCTGATCGCACGCTATCATGG7EL430[13,48] XsdauK66ATTTGGCTGCGATCGATAAGCCATCTTGTTGTTGCCCTCT7EL330[13,48] XsdauK71GGAGCCAAATCATCCTTGAATGTGCTTCAGTTTCGACAGG7EL440[13,48] XsdauK75TCTGCACCCTAGTGTTGTGGGATCAGGATCCAATGCAGGT7EL520[13,48] XsdauK8CGAAGCAGAAGCACCTGAAGCGCTGAATCGATTACGGAAC7EL985[13,48] XsdauK99GTGATGGGCAACATGTTCTGCCGGTAATGGAAACCAAGAA7EL700, 660[13,48] Xsews19 AAGGCAATGGAGATGTTCG CCCGTTGATGACCGTGTT 7EL275, 195[49] Xwmc273AGTTATGTATTCTCTCGAGCCTGGGTAACCACTAGAGTATGTCCTT7EL200, 150[49] Xwmc702GAATCACATCGAATGGATCTCAGAGGCCTTTTTCGATATTCTGC7EL194[49] Xwmc606CCGATGAACAGACTCGACAAGGGGCTTCGGCCAGTAGTACAGGA7EL/7ES175[49] Xbarc154GTAATTCCGGTTCCACTTGACATTGGATGGGCAGCTTCAAGGTATGTT7ES520[49] Xbarc70GCGAAAAACGATGCGACTCAAAGGCGCCATATAATTCAGACCCACAAAA7ES100[49] Xcfa2049TAATTTGATTGGGTCGGAGCCGTGTCGATGGTCTCCTTG7ES257[49] Xcfa2174ACGGCATCACAGGTTAAAGGGGTCTTTGCACTGCTAGCCT7ES240[49] Xcfd14CCACCGGCCAGAGTAGTATTTCCTGGTCTAACAACGAGAAGA7ES103[49] Xcfd21CCTCCATGTAGGCGGAAATATGTGTCCCATTCACTAACCG7ES260[49] Xcfd31GCACCAACCTTGATAGGGAAGTGCCTGATGATTTTACCCG7ES340[49] Xcfd66AGGTCTTGGTGGTTTTGGTGTTTTCACATGCCCACAGTTG7ES119[49] Xedm109ATGAGGCACAAGTGGATGGTCTCCTGAAGGGAAAAGCTCA7ES446[39] Xedm16TCACCTAACAGCACCACGAGGCCGAGTACCAGCAGTACCA7ES195[39] Xedm34TTGTTTCTTGGCTGGGTTTCGATCTGCCAAAGCCTTCATAA7ES150[39] Xgwm154TCACAGAGAGAGAGGGAGGGATGTGTACATGTTGCCTGCA7ES325[49] Xgwm156CCAACCGTGCTATTAGTCATTCCAATGCAGGCCCTCCTAAC7ES158[39] Xgwm350ACCTCATCCACATGTTCTACGGCATGGATAGGACGCCC7ES160[49] Xgwm44GTTGAGCTTTTCAGTTCGGCACTGGCATCCACTGAGCTG7ES140[49] Xgwm471CGGCCCTATCATGGCTGGCTTGCAAGTTCCATTTTGC7ES141[15] Xgwm473TCATACGGGTATGGTTGGACCACCCCCTTGTTGGTCAC7ES230, 200[49] Xmag2934TGTTCACAAGCGATTCAAGGAATGCAAGATGGTGGCTTCT7ES280[13,43-44] Xwmc83TGGAGGAAACACAATGGATGCCGAGTATCGCCGACGAAAGGGAA7ES153[49] Xwmc653AGTGTTTTAGGGGTGGAAGGGACGGAACCCTAAACCCTAGTCG7ES190[49] Xwmc809CAGGTCGTAGTTGGTACCCTGAATGAACACGGCTGGATGTGA7ES360[49]
Molecular identification of FHB resistance gene in varieties derivedfrom common wheat-partial amphiploid
LIU XinLun1, WANG Chao1, NIU LiHua1, LIU ZhiLi2, ZHANG LuDe3, CHEN ChunHuan1, ZHANG RongQi1, ZHANG Hong1, WANG ChangYou1, WANG YaJuan1, TIAN ZengRong1, JI WanQuan1
(1College of Agronomy, Northwest A&F University/State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi;2Seed Administration Station of Chenggu County, Hanzhong 723200, Shaanxi;3Shaanxi provincial Office of Poverty Alleviation and Development, Xi’an 710006)
Fusarium head blight (FHB) is one of the most devastating diseases of wheat.is one of the important source resistance cultivars of wheat disease resistance breeding, with high tolerance to FHB. Three derived varieties of common wheat-Xinong 509, Xinong 511 and Xinong 529 showed moderate resistance to FHB in field. The objective of this study is to clarify the genetic constitution of resistance gene in three varieties.Single drop injection withwas performed to detect the reaction of the three derived varieties of common wheat-to FHB. Specific molecular markers located on homologous group 1 to 7 ofE chromosome were employed to detect the sources of resistance to FHB, screening the three derived varieties of common wheat-, and their major donor parents Xiaoyan 693, Xiaoyan 597 andSpecific molecular markers closely linked with flankingwere used to analyze the relationship between the gene conferring resistance to FHB andThree derived varieties were moderately resistant to FHB in the field experiment, and had no much difference compared with the medium-resistant control variety. Seven of the 105 chromosome E specific molecular markers ofcould be amplified polymorphism bands in the three derived varieties and, which located on the chromosome 7E. A total of 97 specific markers located on the chromosome 7E,20 showed specific bands ofin the three derived varieties of common wheat-and their major donor parents Xiaoyan 693, Xiaoyan 597The results indicate that the 89 cM genetic fragment derived from7E chromosome. Of them, six molecular markers (,,,,and), which were located on 7EL 149.00-7EL 153.77, tightly linked to. However,(7EL 153.77),(7EL 154.70),(7EL 156.27),(7EL 158.02),(7EL 158.97) and(7EL 160.00),which are next to, showed no specific bands ofamong the three derived varieties of common wheat-and Xiaoyan 597.Xinong 509, Xinong 511 and Xinong 529, derived fromwith good FHB resistance, possess a novel FHB resistance gene, which derived fromand was different from.
common wheat;;Fusarium head blight;; molecular marker
2017-05-17;接受日期:2017-07-23
国家重点研发计划(2016YFD0102000)、陕西省科技统筹创新工程计划(2015KTZDNY01-01-02)
刘新伦,E-mail:liuxxlun@126.com。王超,E-mail:1240026878@qq.com。刘新伦和王超为同等贡献作者。通信作者吉万全,E-mail:jiwanquan2008@126.com

