基于CSSL的水稻芽期耐盐性QTL定位
赵春芳,张善磊,赵庆勇,周丽慧,赵 凌,姚 姝,张亚东,王才林
(江苏省农业科学院 粮食作物研究所,江苏省优质水稻工程技术研究中心,国家水稻改良中心南京分中心,江苏 南京 210014)
基于CSSL的水稻芽期耐盐性QTL定位
赵春芳,张善磊,赵庆勇,周丽慧,赵 凌,姚 姝,张亚东,王才林
(江苏省农业科学院 粮食作物研究所,江苏省优质水稻工程技术研究中心,国家水稻改良中心南京分中心,江苏 南京 210014)
水稻耐盐遗传位点的发掘可为其耐盐遗传机制的研究提供理论基础,为耐盐品种培育提供基因资源。利用一套以9311为背景亲本导入了日本晴染色体片段的染色体片段置换系(CSSL)为试验材料,对芽期耐盐性进行快速鉴定,并分析了耐盐QTLs。结果表明,9个CSSLs表现出显著耐盐性,经过遗传背景的高密度分子标记检测,利用代换作图方法定位到4个芽期耐盐相关QTLs,分别位于水稻第1,2,4和11号染色体上,命名为qSAT1、qSAT2、qSAT4和qSAT11,其中qSAT1在含4个重叠片段的CSSLs中被检测到,其余3个QTLs均在含2个重叠置换片段的CSSLs中被检测到,经比较发现4个QTLs与已克隆水稻耐盐基因均不在同一染色体区间,说明为新的耐盐基因候选位点。结果对进一步发掘和利用新的水稻耐盐QTL具有重要意义。
水稻;芽期;耐盐;QTL
盐胁迫是农作物生长发育及产量提高的重要限制因素之一。水稻为中度感盐作物,在盐碱地上生长受到抑制,当土壤中可溶性盐达到0.3%时即表现受害症状。因此,挖掘和利用耐盐种质资源,培育耐盐水稻品种,对于高效利用盐碱地、保障全国粮食安全具有重要意义。
水稻的耐盐性为典型的数量性状遗传,受多基因协同控制并受到外界环境和发育时期的影响[1],可以利用数量性状的定位群体及其分子标记连锁图谱,对耐盐性状进行基因定位。关于水稻耐盐性状数量遗传的研究已有很多报道,其中苗期耐盐QTL定位的报道较多,已检测到与幼苗存活天数、盐害级别及茎干Na+/K+浓度等多种性状的70多个QTLs[2],位于第1,2,6和7号染色体上的较多,位于第10和11号染色体上的较少,但目前为止仅有少数耐盐基因被克隆。SKC1是第一个被克隆的耐盐基因,位于第1号染色体上,编码细胞膜定位的Na+转运蛋白,在Na+循环中起重要作用。当水稻处于高盐环境时,茎秆会积累大量Na+,SKC1蛋白可将茎秆中过量Na+回运至根系,从而减轻Na+毒害,增强水稻对高盐环境的耐性[3]。另一个克隆的耐盐基因DST位于第3号染色体上,是利用图位克隆方法从耐盐突变体中克隆到一个新型锌指转录因子,对水稻耐盐性及耐旱性具有负调控作用。该基因突变后导致过氧化氢代谢基因表达下降,使过氧化氢在保卫细胞中的超积累,造成气孔关闭,水分蒸发减少,从而表现出耐盐或耐旱表型[4]。最新研究发现DST能够与自身物理互作,同时与DCA1形成异源四聚体,这个转录复合物能够调控编码H2O2清除因子的Prx24基因的表达,从而正向调控气孔孔径大小和气孔中H2O2含量,最终影响植株的胁迫耐受性,认为抑制DCA1-DST功能互作能提高多种作物的耐盐性和耐旱性[5]。HST1 (Os06g0183100)是最近报道的耐盐基因,编码一个B型反应调节蛋白OsRR22,参与调节渗透或离子转运相关基因的表达,其耐盐程度强于SKC1基因,在日本品种的耐盐性改良上具有很高的利用价值[6]。
尽管已经定位了众多耐盐QTLs,克隆了几个耐盐基因,但是真正用于耐盐品种培育上的基因资源尚少,一方面是由于携带耐盐QTL的载体材料受遗传背景的影响很大,定位到的QTL很难直接应用于育种材料耐盐性的遗传改良;另一方面已克隆的耐盐基因具有多效性,导入耐盐基因的同时产生了不利的表型性状。为了筛选更多的耐盐基因资源,消除遗传背景对水稻耐盐QTL定位的影响,本研究利用一套以籼稻品种9311为受体、粳稻品种日本晴为供体的染色体片段置换系(Chromosome segment substitution line,CSSL)为试验材料,将处于萌发期的种子进行芽期耐盐处理,以幼芽成活率作为耐盐检测指标,进行了耐盐株系简单快速地筛选,并初步定位和分析了耐盐QTL,以期获得新的耐盐遗传位点。
1 材料和方法
1.1试验材料
本研究所用染色体片段置换系群体是以籼稻品种9311为受体亲本,粳稻品种日本晴为供体亲本,经杂交、多代回交至BC4F1,而后利用水稻基因组上均匀分布的SSR标记对BC4F1单株进行基因型检测,选择含少量杂合片段的单株进行种植,得到BC4F2群体,经自交纯合后获得染色体片段置换系群体,该套置换系共包含119个株系[7]。根据物理图谱[8],日本晴置换片段覆盖了水稻全基因组的83.98%,目前利用该群体已定位了重金属及氮磷养分胁迫等多个重要性状QTLs[9-11],获得了一批新的具有育种利用价值的遗传位点。
1.2水稻芽期耐盐性鉴定
将119个置换系及亲本种子浸没于自来水中,在30 ℃恒温培养箱中萌发2 d至种子露白,而后催芽20 h至露出芽尖。亲本及各置换系各挑选100粒露芽种子,放置于垫有滤纸的培养皿中,加入0.8%(m/V)的NaCl溶液至淹没种子,盖好培养皿盖子,置于30 ℃光照培养箱内进行芽期盐胁迫处理。盐溶液处理10 d后,用蒸馏水冲洗种子,并于蒸馏水中再培养5 d,统计每皿中幼芽成活粒数,计算幼芽成活百分率。盐胁迫处理设置3次生物学重复,每次生物学重复中每份材料设置3个重复,以3次生物学重复得到的幼芽成活率的平均值作为统计值。
1.3置换片段分析
根据该置换系的物理图谱(含250个分子标记),对筛选到的耐盐系中的置换片段进行全基因组检测,获得其置换片段的分子生物学信息。置换片段的长度按Young和Tanksley[12]的方法进行计算。在不考虑2个相邻标记间发生双交换事件的情况下,当相邻标记基因型均为供体亲本基因型时,认为这2个标记之间的区段为供体置换片段。而后在每个置换片段两端增加分子标记进行检测,直至亲本间多态性标记表现为受体亲本的纯合基因型为止。最后,以置换片段末端标记基因型与相邻的受体纯合基因型标记之间的中点为该末端边界点,两末端边界点之间的距离即置换片段长度。
1.4QTL定位
利用代换作图方法进行QTL定位[13],如果在含有重叠置换片段的不同CSSL上均检测到QTL,且遗传效应方向一致,则认为QTL存在于各置换片段的重叠片段上;如果在一个CSSL中检测到QTL,而在具有重叠置换片段的另一CSSL中未检测到,则认为QTL位于2个置换片段的非重叠区段上。QTL命名遵循McCouch等[14]制定的原则。
2 结果与分析
2.1亲本及置换系芽期耐盐的表型分析
通过对盐胁迫处理后CSSL及亲本种子的幼芽成活率统计分析,两亲本间存在极显著差异(P<0.01),亲本9311的幼芽成活率为15.8%,甚至出现零幼芽成活的现象,而供体亲本日本晴的幼芽成活率为76.2%,表现出较强的芽期耐盐能力(图1-A)。CSSL群体幼芽成活率集中在20%~60%,平均值为38.9%,变异范围为5.1%~93.2%,表现为单峰连续分布和超亲分离现象(图1-B)。其中CSSL群体中有9个CSSLs的幼芽成活率在70%以上,表现出明显的耐盐性(图1-A),此部分置换系可作为携带耐盐基因的材料进行QTL分析。

A.亲本及2个耐盐置换系的表型;B.幼芽成活率在置换系群体中的频率分布。A.Phenotype of parents and two CSSLs associated with salt tolerance;B. Frequency distribution of seedling survival rate in the CSSL population.
2.2耐盐置换系的遗传背景检测
利用250个水稻基因组上均匀分布的多态SSR/STS标记,以9311和日本晴为对照,对9个耐盐CSSLs的遗传背景进行全基因组检测。结果表明,9个CSSLs共含有13个置换片段,4个CSSLs(CSSL4、CSSL27、CSSL81和CSSL105)含有置换片段为2个,其余5个CSSLs(CSSL12、CSSL26、CSSL30、CSSL47和CSSL88)均为单片段置换系。图2显示了日本晴置换片段在水稻12条染色体上的分布。

黑框表示日本晴置换片段;双线表示受体亲本9311基因组。Black boxes indicate substituted segments of Nipponbare;Double lines indicate the genome of the recipient 9311.
2.3水稻芽期耐盐QTL的代换作图
通过对9个耐盐CSSLs携带的置换片段分析,采用重叠片段的代换作图法共检测出4个QTLs:qSAT1、qSAT2、qSAT4和qSAT11,分别位于水稻第1,

A.qSAT1代换作图;B.qSAT2代换作图;C.qSAT4代换作图;D.qSAT11代换作图。A.Substitution mapping of qSAT1;B.Substitution mapping of qSAT2;C.Substitution mapping of qSAT4;D.Substitution mapping of qSAT11.
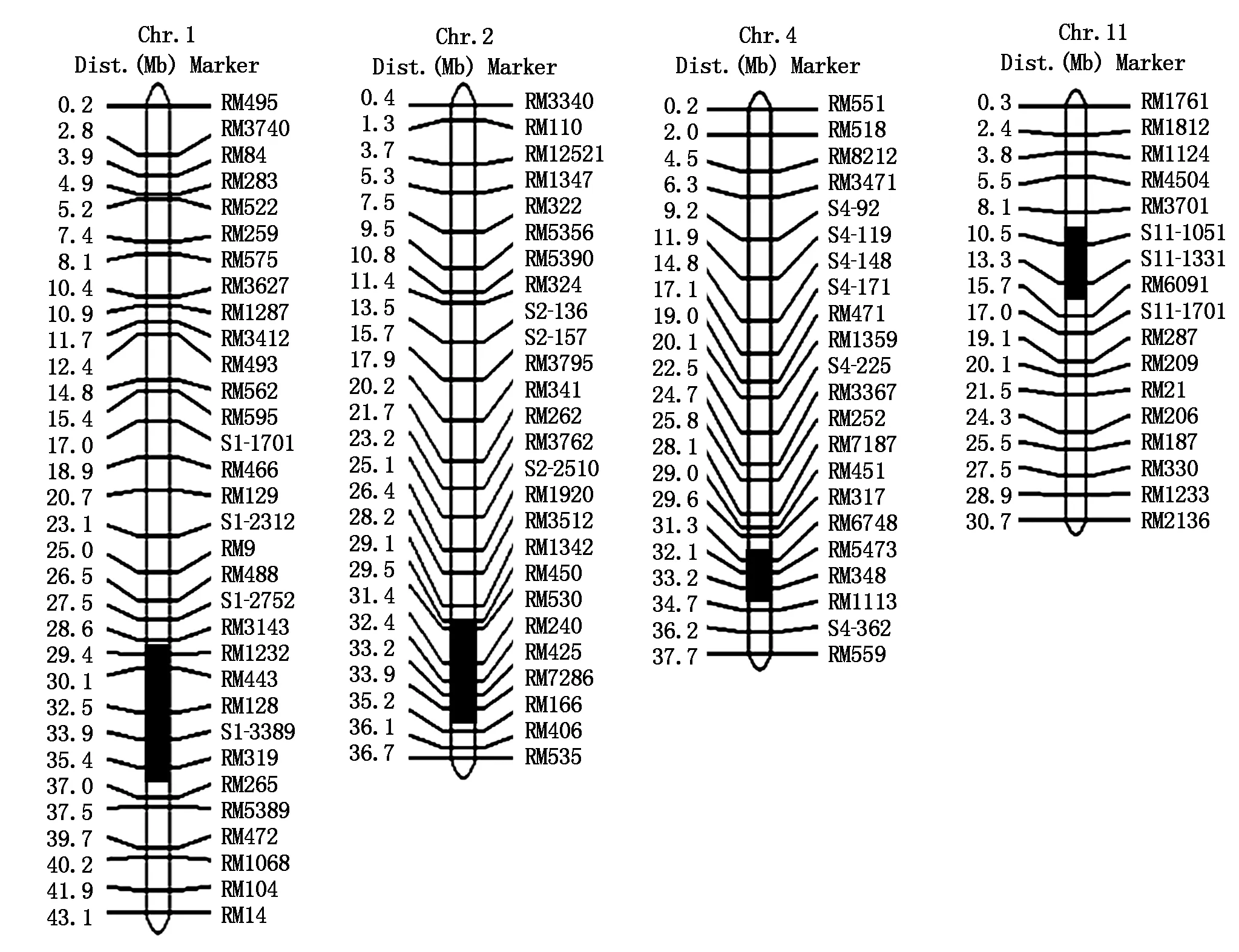
染色体右侧为分子标记名称;左侧数字表示物理距离(单位为Mb)。The molecular markers are in the right side of chromosomes and their corresponding physical distance are in the left side.
2,4,11染色体上。qSAT1在CSSL4、CSSL12、CSSL27和CSSL88中均被检测到,表明其位于重叠置换片段上,即qSAT1被定位于第1染色体的RM1232~RM319,物理距离为7.2 Mb的区段内(图3-A)。qSAT2在CSSL26和CSSL30中被检测到,而在与其有重叠关系的CSSL23中未被检测到,表明其位于与CSSL26和CSSL30重叠而与CSSL23非重叠的片段上,即qSAT2被定位在第2染色体RM530~RM7286,物理距离为4.1 Mb的区段内(图3-B)。qSAT4在CSSL47和CSSL81中被检测到,表明其位于两者的重叠置换片段上,即qSAT4被定位在第4染色体RM6748~RM348,物理距离为3.5 Mb的区段内(图3-C)。qSAT11在CSSL27和CSSL105中被检测到,而在与其有重叠关系的CSSL35中为被检测到,表明其位于与CSSL27和CSSL105重叠而与CSSL35非重叠的片段上,即qSAT11被定位在第11染色体S11-10.51~S11-13.31,物理距离为5.4 Mb的区段内(图3-D),值得注意的是,CSSL27同时携带了qSAT1和qSAT11,其耐盐强度高于携带单个QTL的系。图4综合了4个芽期耐盐QTLs在该置换系群体物理图谱上的分布及物理位置。
3 讨论
关于水稻对盐胁迫生理生化反应的研究,已有较多报道,一般来说,当水稻受到高盐胁迫时,摄取外界水分的能力受到抑制,细胞处于脱水状态,从而导致生长发育延缓甚至死亡[15]。目前,水稻耐盐性研究主要集中在苗期阶段,而芽期、生殖期及成熟期相对较少[16-17],其中苗期考察的性状较多,主要有盐胁迫下存活天数、盐害级别和Na+、K+浓度等[18-20]。但是,芽期耐盐是保障水稻全生育期耐盐的先决条件,特别是在水稻栽培逐渐转向直播种植的趋势下,对水稻芽期耐盐性的鉴定具有必要性。对于芽期耐盐的衡量指标,郭望模等[21]认为盐胁迫对水稻芽期的直接伤害是降低发芽率,可以用盐处理后的发芽率作为芽期耐盐检测指标。本研究统一选取已露芽的种子进行连续10 d的盐胁迫处理,一方面去除了发芽率和发芽势的影响,另一方面长时间的盐胁迫造成了幼芽死亡,因此选用幼芽成活率作为耐盐指标,相比以往的根长、苗高等测量指标更简单快捷。统计结果表明,9311和日本晴两亲本的幼芽成活率分别为15.8%和76.2%,表现为盐敏感和高度耐盐。CSSL群体的耐盐性呈现典型的正态分布,筛选到9个CSSLs的幼芽成活率在70%以上,说明以幼芽成活率作为芽期耐盐指标,适合对该置换系群体进行芽期耐盐性鉴定。
染色体片段置换系群体是在初级作图群体基础上通过杂交筛选到的次级分离群体,其遗传背景接近于受体基因组,仅含有少量的供体亲本的染色体片段,因而可以对控制目标性状的遗传位点进行简单、准确地定位,并便于对QTL进一步确证及在标记辅助选择的育种利用。本研究通过对筛选到的9个芽期耐盐CSSLs进行遗传背景检测和代换作图法检测到4个QTLs,分别位于水稻第1,2,4,11染色体上,将其与以往定位结果进行比较,发现位于第1染色体RM1232~RM319区间上的qSAT1与汪斌等[22]定位的地上部Na+含量QTL具有相同的染色体区间;位于第2染色体的RM530~RM7286区间上的qSAT2与以往定位的幼苗存活天数、茎干中Na+、K+浓度等多个耐盐性状QTLs具有相同的位置[18,23]。其余2个耐盐QTLs与以往的定位结果均不相同,为本研究鉴定的新QTLs。目前为止,水稻的耐盐性QTL定位已经进行了大量的研究,但是不同实验室利用不同群体的定位结果重现率较低[19],这可能是由于遗传材料的亲本、群体类型以及定位群体遗传背景不同而影响了定位结果的准确性;也可能是水稻芽期或苗期耐盐性易受到盐处理浓度及时间的影响,造成了性状调查时偏差较大;当然,耐盐性与所处发育时期亦有直接关系。另外,以往检测到的芽期或幼苗期耐盐性状QTLs的表型贡献率都不大[1,19,24],进一步说明了这些QTL的准确性和精确度较低,很难进行育种利用。而本研究方法筛选到的材料具有极其明显的芽期耐盐性,特别是CSSL27因携带了2个耐盐QTL而具有更高的耐盐性,因此真实性较高,后续可以考虑配制分离群体进行进一步验证并对QTL进行精细定位。本研究未鉴定到已克隆的水稻耐盐基因OsSKC1、DST、HST1,可能主要是由于这些基因在两亲本9311和日本晴间没有序列或基因表达差异的原因。
目前,科学家们对不同发育时期水稻耐盐性的认识存在较大争议,一些研究者认为水稻芽期、苗期及成熟期的耐盐性具有一定的相关性[25-26],而有些研究者认为同一水稻品种的耐盐能力在芽期和苗期表现不一致,两者相关性很低[27-28]。本研究对置换系群体芽期阶段的耐盐性遗传鉴定仅提供了初步数据,包括苗期、生殖期及成熟期在内的全生育期耐盐性鉴定更能增加该套置换系群体耐盐性的真实性,特别是对本研究筛选到的高耐盐品系的全生育期耐盐性还需要进行进一步的探讨。
[1] Lin H X,Zhu M Z,Yano M,et al. QTLs for Na+and K+uptake of the shoots and roots controlling rice salt tolerance[J]. Theoretical and Applied Genetics,2004,108(2):253-260.
[2] 胡时开,陶红剑,钱 前,等. 水稻耐盐性的遗传和分子育种的研究进展[J]. 分子植物育种,2010,8(4):629-640.
[3] Ren Z H,Gao J P,Li L G,et al. A rice quantitative trait locus for salt tolerance encodes a sodium transporter[J]. Nature Genetics,2005,37(10):1141-1146.
[4] Huang X Y,Chao D Y,Gao J P,et al. A previously unknown zinc finger protein,DST,regulates drought and salt tolerance in rice via stomatal aperture control[J]. Genes & Development,2009,23(15):1805-1817.
[5] Cui L G,Shan J X,Shi M,et al. DCA1 Acts as a transcriptional co-activator of DST and contributes to drought and salt tolerance in rice[J]. PLOS Genetics,2015,11(10):e1005617.
[6] Takagi H,Tamiru M,Abe A,et al. MutMap accelerates breeding of a salt-tolerant rice cultivar[J]. Nature Biotechnology,2015,33(5):445-449.
[7] Zhu W Y,Lin J,Yang D W,et al. Development of chromosome segment substitution lines derived from backcross between two sequenced rice cultivars,indica recipient 93-11 and japonica donor nipponbare[J]. Plant Molecular Biology Reporter,2009,27(2):126-131.
[8] 赵春芳,周丽慧,于 新,等. 基于CSSL的高密度物理图谱定位水稻分蘖角度QTL[J]. 植物学报,2012,47(6):594-601.
[9] 赵春芳,张亚东,陈 涛,等. 低磷胁迫下水稻苗期根长性状的QTL定位[J]. 华北农学报,2013,28(6):6-10.
[10] 赵春芳,周丽慧,丁 丹,等. 基于CSSL的水稻苗期耐亚铁离子胁迫QTL定位[J]. 江苏农业学报,2013,29(3):461-467.
[11] 赵春芳,赵 凌,张亚东,等. 水稻苗期耐低氮相关性状的QTL定位[J]. 华北农学报,2015,30(6):1-7.
[12] Young N D,Tanksley S D. Restriction fragment length polymorphism maps and the concept of graphical genotypes[J]. Theoretical and Applied Genetics,1989,77(1):95-101.
[13] Paterson A H,Deverna J W,Lanini B,et al. Fine mapping of quantitative trait loci using selected overlapping recombinant chromosomes,in an interspecies cross of tomato[J]. Genetics,1990,124(3):735-742.
[14] McCouch S R,Cho Y G,Yano M,et al. Report on QTL nomenclature [J]. Rice Genet Newsl,1997,14:11-13.
[15] 杨晓华,彭晓珏,杨国华,等. 水稻OsRab7耐盐功能的初步鉴定及其表达载体的构建[J]. 武汉植物学研究,2008,26(1):1-6.
[16] 郑洪亮,刘博文,赵宏伟,等. 利用连锁和关联分析定位粳稻芽期及幼苗前期耐盐性QTL[J]. 中国水稻科学,2014,28(4):358-366.
[17] Hossain H,Rahman M A,Alam M S,et al. Mapping of quantitative trait loci associated with reproductive-stage salt tolerance in rice [J]. J Agro Crop Sci,2015,201(1):17-31.
[18] 孙 勇,藏金萍,王 韵,等. 利用回交导入系群体发掘水稻种质资源中的有利耐盐QTL[J]. 作物学报,2007,33(10):1611-1617.
[19] 邢 军,常汇琳,王敬国,等. 盐碱胁迫条件下粳稻Na+、K+浓度的QTL分析[J]. 中国农业科学,2015,03:604-612.
[20] Liang J L,Qu Y P,Yang C G,et al. Identification of QTLs associated with salt or alkaline tolerance at the seedling stage in rice under salt or alkaline stress[J]. Euphytica,2015,201(3):441-452.
[21] 郭望模,傅亚萍,孙宗修. 水稻芽期和苗期耐盐指标的选择研究[J]. 浙江农业科学,2004,1(1):30-33.
[22] 汪 斌,兰 涛,吴为人. 盐胁迫下水稻苗期Na+含量的QTL定位[J]. 中国水稻科学,2007,21(6):585-590.
[23] Takehisa H,Shimodate T,Fukuta Y,et al. Identification of quantitative trait loci for plant growth of rice in paddy field flooded with salt water[J]. Field Crops Research,2004,89(1):85-95.
[24] Qiu X J,Yuan Z H,Liu H,et al. Identification of salt tolerance-improving quantitative trait loci alleles from a salt-susceptible rice breeding line by introgression breeding[J]. Plant Breeding,2015,134(6):653-660.
[25] Zang J,Sun Y,Wang Y,et al. Dissection of genetic overlap of salt tolerance QTLs at the seedling and tillering stages using backcross introgression lines in rice[J]. Science in China Series C-life Sciences,2008,51(7):583-591.
[26] 陈亚萍. 粳稻种质资源不同生育时期的耐盐性评价[D]. 银川:宁夏大学,2016.
[27] 谢留杰,段 敏,潘晓飚,等. 不同类型水稻品系苗期和全生育期耐盐性鉴定与分析[J]. 江西农业大学学报,2015,37(3):404-410.
[28] 郭望模,傅亚萍,孙宗修,等. 盐胁迫下不同水稻种质形态指标与耐盐性的相关分析[J]. 植物遗传资源学报,2003,4(3):245-251.
MappingofQTLsforBud-stageSalinityToleranceBasedonChromosomeSegmentSubstitutionLineinRice
ZHAO Chunfang,ZHANG Shanlei,ZHAO Qingyong,ZHOU Lihui,ZHAO Ling,YAO Shu,ZHANG Yadong,WANG Cailin
(Institute of Food Crops,Jiangsu Academy of Agricultural Sciences,Jiangsu High Quality Rice Research and Development Center,Nanjing Branch of Chinese National Center for Rice Improvement,Nanjing 210014,China)
The identification of salt tolerant genetic loci in rice can provide study basis for the molecular mechanism of salt tolerance,and gene resources for improving salt tolerant cultivars.A set of chromosome segment substitution lines (CSSLs) developed from two sequenced rice cultivars:theindicavariety 9311 as the recipient and thejaponicavariety Nipponbare as a donor were used to detect for salt tolerance in bud stage quickly,and to locate QTLs related to salt tolerance.9 CSSLs showed significant salt tolerance and 4 QTLs were mapped by genetic background detection based on high density molecular markers and the substitution mapping.These 4 QTLs located on rice chromosomes 1,2,4 and 11 chromosomes,designated asqSAT1,qSAT2,qSAT4 andqSAT11.qSAT1 was detected in 4 CSSLs containing over lapping fragments,while the other 3 QTLs were detected in 2 CSSLs.These QTLs were not included in the same chromosome interval as the cloned rice salt tolerance genes,and thus were described as new candidate gene loci associated with bud-stage salt tolerance.The results supplies important information for further exploration and utilization of new salt tolerant QTL in rice.
OryzasativaL.;Bud stage;Salt tolerance;QTL
2017-07-06
国家科技支撑计划项目(2015BAD01B02);江苏省重点研发项目(BE2016370);现代农业产业技术体系建设专项基金项目(CARS-01-62)
赵春芳(1981-),女,山东聊城人,副研究员,博士,主要从事水稻遗传育种研究。
王才林(1959-),男,江苏无锡人,研究员,博士,博士生导师,主要从事水稻遗传育种研究。
S511.03
A
1000-7091(2017)05-0106-06
10.7668/hbnxb.2017.05.016

