新铁炮百合朱丽叶器官差异基因表达分析
杜 方,王 婷,樊俊苗,张浩宇
(山西农业大学 园艺学院,山西 太谷 030801)
新铁炮百合朱丽叶器官差异基因表达分析
杜 方,王 婷,樊俊苗,张浩宇
(山西农业大学 园艺学院,山西 太谷 030801)
为了探明17个候选基因在新铁炮百合朱丽叶花、叶、鳞片3个器官中的表达情况,利用实时荧光定量PCR技术,对其相对表达量进行了研究。结果表明,根据表达情况,17个候选基因可以分为5类,CL10636.Contig1_All、CL6300.Contig2_All、CL7951. Contig5_All、 Unigene8314_All、Unigene19279_All和CL1306.Contig1_All 6个在花中显著高表达,分别与糖转运蛋白-类SWEET4基因、赤霉素调控蛋白基因、线粒体伴侣基因、光依赖性短下胚轴基因、1-脱氧葡萄糖-5-磷酸合成酶基因、牻牛儿基牻牛儿基焦磷酸合成酶基因高度同源;CL9292.Contig2_All、CL212.Contig5_All、Unigene1800_All、CL3468. Contig1_All、CL4520.Contig5_All、Unigene4212_All、CL4079.Contig1_All和Unigene10210_All 8个在叶中显著高表达,分别与GDSL酯酶/脂酶基因、类黄酮O-甲基转移酶、脂氢过氧化物裂解酶基因、4-羟基-3-甲基-2-丁烯基二磷酸合成酶基因、多酚氧化酶基因、类葡苷露聚糖4-β-甘露糖转移酶基因、左旋海松烷合成酶基因和反式罗勒烯合成酶基因高度同源;CL2405.Contig1_All在鳞片中显著高表达,功能未知;CL1114.Contig2_All在花和叶中同时显著高表达,与3-酮酯酰CoA-硫解酶高度同源;CL1772.Contig2_All为非器官差异表达基因,与Δ24-甾醇还原酶基因高度同源。此结果可为今后研究这些候选基因的功能奠定基础,对百合分子育种具有重要意义。
百合;差异基因;实时荧光定量PCR;器官
百合是百合科(Liliaceae)百合属(Lilium)所有植物的统称,其花朵硕大,色彩丰富,花姿优美,具有很高的观赏价值和药用价值[1]。百合的完整植株包括须根、鳞茎、花茎、叶片和花朵等器官,所有器官都来自于变态膨大的地下茎—鳞茎的鳞茎盘,这使得百合器官的起源变得十分有趣。
早期,对器官发生相关基因的研究主要是基于突变体库的建立,利用拟南芥模式系统,科学家对植物根、茎、叶、花等各器官的形态建成进行了研究,花发育的ABC模型是其中最重要的发现之一。后来,对矮牵牛胚珠发育和拟南芥花发育的深入研究,导致了花发育ABCDE模型的确认[2-3]。同时,对水稻[4]、人参[5]和大豆[6]等器官发育基因的研究,正推动着器官发育生物学的全面发展。
然而,对突变体进行筛选费时费力,该方法不适用于基因组特别大的物种(如百合,36 Gb)。近几年,随着测序技术的快速发展,可以在较短的时间内找到不同品种、不同器官或不同试验处理间的差异基因,为百合的分子生物学研究提供了发展机遇。近3年来,利用Illumina测序平台发展起来的数字基因表达谱技术,有学者深入挖掘了百合春化[7]、胁迫[8]、碳代谢[9]、矮化[10]、花粉萌发[11]、花发育[12-13]和花色生物合成[14]相关的基因。
为了开发百合的分子标记,山西农业大学园艺学院百合课题组利用Illumina测序平台构建了东方百合索邦(Sorbonne)根、鳞茎、茎皮、叶片、花瓣和柱头6种器官混合样的转录组数据库[15];在此基础上,又构建了东方百合索邦花、叶、鳞片3个器官的数字基因表达谱,找出了在索邦器官中差异表达的基因[16]。然而,基因的表达水平可能随品种和植株的发育时期而变化。朱丽叶是新铁炮百合系列的一个品种,花白色、长筒形,朝天开放,易于种子繁殖;与粉色、碟形花、花朵斜向开放的东方百合索邦完全不同。
为了探明在索邦花、叶、鳞片中差异表达的基因在朱丽叶中的表达情况,本研究利用实时荧光定量PCR(qPCR)对17个在索邦花、叶、鳞茎中差异表达的基因在朱丽叶中的表达进行了研究,以期为今后研究这些候选基因的功能奠定基础。
1 材料和方法
1.1试验材料
新铁炮百合朱丽叶(LiliumformolongiJulius),购于浙江虹越花卉有限公司,2015年4月栽植于山西农业大学园艺站,2015年7月开花,挖取刚开放的3株百合完整植株于实验室,分取5 cm长花蕾、顶端叶片(L)和内层鳞片(S),撕成小块后快速储藏于-80 ℃冰箱。
1.2试验方法
1.2.1 总RNA提取与反转录 RNA的提取根据GenStar公司的TRIGene 总RNA提取试剂盒说明书进行,用1.5%的琼脂糖凝胶电泳检测其质量,用MONODROP 2000c生物光度计检测RNA浓度和纯度,质量合格的RNA保存于-80 ℃超低温冰箱。
按照GenStar公司的StarScript Ⅱ cDNA 第1链合成试剂盒说明书反转录cDNA,将得到的cDNA溶液稀释至100 ng/μL,保存于-20 ℃冰箱备用。
1.2.2 引物设计 基因序列来源于山西农业大学园艺学院百合课题组前期所构建的百合转录组数据库L.-Unigene-All[15],采用Primer 3(http://primer 3.ut.ee/)在线设计引物(表1),交由生工生物工程有限公司合成。
1.2.3 实时荧光定量PCR 采用荧光染料法,以18S rRNA为内参基因(正向引物5′-CGCAAGGCTGAAACTTA
AAGG-3′;反向引物5′-CAGACAAATCGCTCCACCAAC-3′),利用TaKaRa公司 q-PCR试剂盒配置20 μL反应体系,包括 SYBR Ⅱ 10 μL,双蒸水 7 μL,cDNA 1 μL,Primer 1.6 μL,ROX 0.4 μL。在7500 Real time PCR System上运行qRT-PCR试验,每个样本重复3次。反应程序如下:95 ℃ 3 min;之后94 ℃ 30 s ,55 ℃ 30 s,72 ℃ 1 min(荧光),共40个循环。溶解曲线条件为95 ℃ 15 s,54 ℃ 1 min,从54 ℃每30 s上升0.4 ℃直到95 ℃,95 ℃ 30 s(荧光),54 ℃ 15 s。
1.3数据统计
采用2-ΔΔCt法计算相对表达量,采用SPSS和LSD法进行统计分析及多重比较,利用Excel 2003绘图。
2 结果与分析
2.1总RNA提取
使用生物光度计检测各样品RNA质量,各组织总RNA的OD260/280值均在1.8~2.0。取4 μL 总RNA进行琼脂糖凝胶电泳,凝胶成像系统成像如图1所示。相对而言,朱丽叶百合鳞片的RNA较难提取,提取浓度较低,但可以满足试验需求。
2.2花中的高表达基因
实时荧光定量PCR的结果表明,CL10636.Contig1_All、CL6300.Contig2_All、CL7951.Contig5_All、 Unigene8314_All 等4个转录本在朱丽叶百合花器官中的表达量显著高于在叶和鳞片中的表达量(P<0.05),且在叶片和鳞片中的表达量差异不显著。Unigene19279_All(7)和CL1306.Contig1_All(17)除在朱丽叶花中有显著高的表达量外,在叶中也有一定的表达量,显著低于花中的表达量,但显著高于鳞片中的表达量(P<0.05)(图2)。
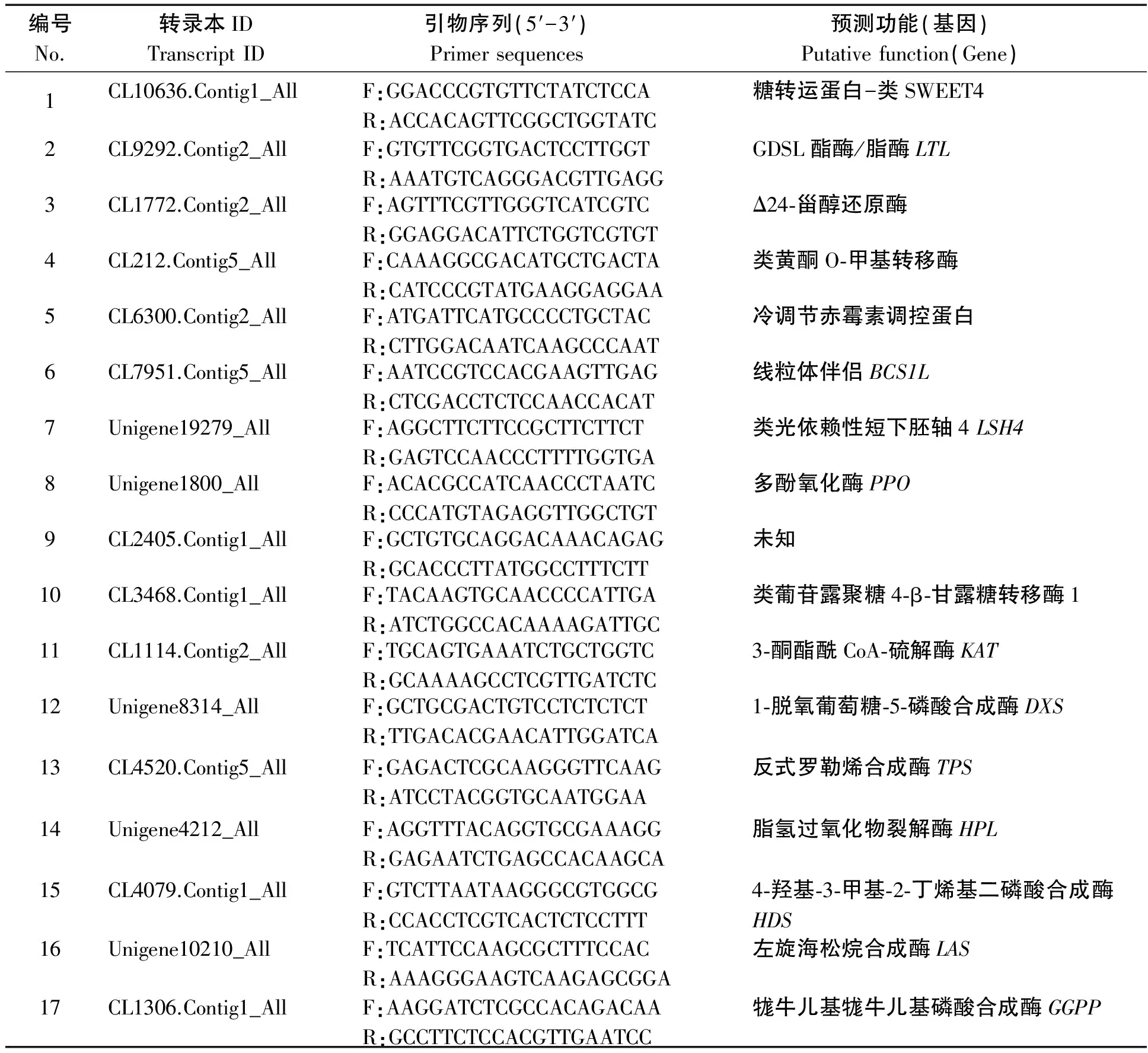
表1 引物信息Tab.1 Information of primers
注:F.正向引物;R.反向引物。
Note:F. Forward primer;R. Reverse primer.
F. 花;L.叶;S.鳞片;M.DL2000 Marker。图2-6同。F.Flower;L.Leaf;S.Scale;M.DL2000 Marker. The same as Fig.2-6.
2.3叶中的高表达基因
图3结果显示,CL9292.Contig2_All、CL212.Contig5_All、Unigene1800_All、CL3468. Contig1_All、CL4520.Contig5_All、Unigene4212_All 6个基因在朱丽叶叶器官中的表达量显著高于在花和鳞片中的表达量(P<0.05),且在叶和鳞片中的表达量差异不显著。CL4079.Contig1_All和Unigene10210_All在朱丽叶叶器官中的表达量显著高于在花和鳞片中的表达量,但花和鳞片中的表达量存在显著差异(P<0.05)。其中,CL4079.Contig1_All(15)在朱丽叶花中的表达量显著高于鳞片中的表达量,而Unigene10210_All(16)在朱丽叶鳞片中的表达量显著高于在花中的表达量(P<0.05)。
2.4鳞片中的高表达基因
仅CL2405.Contig1_All在朱丽叶鳞片中的表达量显著高于在花和叶中的表达量(P<0.05),且在花和叶中的表达量差异不显著(图4)。

1.CL10636.Contig1_All ;5.CL6300.Contig2_All ;6.CL7951.Contig5_All ;7.Unigene19279_All ;12.Unigene8314_All ;17.CL1306.Contig1_All。不同小写字母表示差异显著(P<0.05)。图3-6同。1.CL10636.Contig1_All;5.CL6300.Contig2_All;6.CL7951.Contig5_All;7.Unigene19279_All;12.Unigene8314_All;17.CL1306.Contig1_All;Different lowercase mean significant difference at 0.05 level.The same as Tab.3-6..
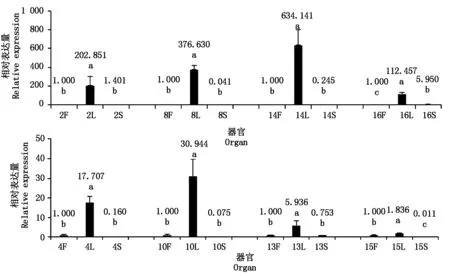
2.CL9292.Contig2_All;4.CL212.Contig5_All;8. Unigene1800_All;10.CL3468.Contig1_All;13.CL4520.Contig5_All;14.Unigene4212_All;15.CL4079.Contig1_All;16.Unigene10210_All。

图4 朱丽叶百合鳞片中高表达的基因Fig. 4 Highly expressed genes in scales
2.5花、叶中均高表达的基因
转录本CL1114.Contig2_All在朱丽叶花和叶器官中的表达量差异不显著,但均显著高于在鳞茎中的表达量(P<0.05)(图5)。
2.6非器官差异表达基因
转录本CL1772.Contig2_All在朱丽叶花、叶、鳞片中均表达,但差异不显著(图6)。

图5 朱丽叶百合花和叶中高表达的基因Fig.5 Highly expressed genes in flowers and leaves

图6 朱丽叶百合的非器官差异表达基因Fig.6 Insignificant expressed genes in flowers,leaves and scales
3 讨论
实时荧光定量PCR(qPCR)是一种快速、特异地检测单个基因表达变化的方法,现已在基础科学研究、食品安全检测、药物研发、海关检验检疫等科研和实践领域得到广泛应用[17]。本研究应用此项技术,将17个基因在朱丽叶3个器官中的表达情况分为5类,即在花中显著高表达的基因、在叶中显著高表达的基因、在鳞片中显著高表达的基因、在花和叶中同时显著高表达的基因和非器官差异表达基因。
供试转录本中有6个转录本在花朵中显著高表达,它们分别与糖转运蛋白-类SWEET4基因、赤霉素调控蛋白基因、线粒体伴侣基因、光依赖性短下胚轴基因、1-脱氧葡萄糖-5-磷酸合成酶基因、牻牛儿基牻牛儿基焦磷酸合成酶基因高度同源。SWEET蛋白起蔗糖转运体的作用,在植物体中常与育性有关[18]。线粒体伴侣蛋白对于线粒体电子传递复合物Ⅲ的组装必不可少[19],光依赖性短下胚轴基因在拟南芥的花和叶的边界细胞(Boundary cells)中被检测到[20],它们可能与花朵开放过程中能量的供应和形态发生有关。1-脱氧葡萄糖-5-磷酸合成酶基因和牻牛儿基牻牛儿基焦磷酸合成酶基因均是萜类代谢途径中2-C-甲基-D-赤藓糖醇-4-磷酸(MEP)途径的重要基因,可能与花香的形成有关[21]。
有8个转录本在叶片中高表达,它们分别与GDSL酯酶/脂酶基因、类黄酮O-甲基转移酶、脂氢过氧化物裂解酶基因、4-羟基-3-甲基-2-丁烯基二磷酸合成酶基因、多酚氧化酶基因、类葡苷露聚糖4-β-甘露糖转移酶基因、左旋海松烷合成酶基因和反式罗勒烯合成酶基因高度同源。其中,GDSL酯酶/脂酶已在多种植物中证明参与植物的生长发育和形态建成[22]。左旋海松烷合成酶和类黄酮O-甲基转移酶被发现在多种植物中具有防御病虫害的功能[23-26],4-羟基-3-甲基-2-丁烯基二磷酸合成酶基因和反式罗勒烯合成酶基因也是MEP途径中的重要基因,在多种植物的叶片和根中被克隆[27-28]。本试验发现,反式罗勒烯合成酶基因在朱丽叶花中也有少量表达,说明萜烯类是多种植物多种器官重要的次生代谢产物。多酚氧化酶是植物体内普遍存在的一类铜结合酶,主要存在于正常的光合组织内[29]。脂氢过氧化物裂解酶基因是脂氧合酶下游的一个关键性酶,与植物抗病虫、老化等生理进程有关[30]。也有试验证明,脂氢过氧化物裂解酶基因催化脂氢过氧化物裂解产生具有特异芳香气味的物质,在浓香型东方百合桑坦德中的表达量是香味稍差的东方百合和喇叭百合杂交种巴拉多纳的3倍[31],表明不同品种中该基因的表达水平不一样。本试验中的朱丽叶为清香型百合,该基因在花中的表达量显著低于在叶片中的表达量。
转录本CL1772.Contig2_All参与类固醇代谢,是与Δ24-甾醇还原酶基因高度同源的基因。甾醇存在于高等植物的细胞膜、细胞质和细胞器内,是调节细胞膜流动性与通透性的关键脂质成分,对植物生长发育有重要作用[32]。该基因属于管家基因,不具有器官特异性,这与该基因在朱丽叶花、叶、鳞片3个器官的表达特征无显著差异相一致。转录本CL1114.Contig2_All是与3-酮酯酰CoA-硫解酶高度同源的基因,参与脂质的代谢活动,催化β氧化循环中的硫解反应[33],而β氧化循环是个放能的过程,该基因在朱丽叶花和叶中高表达,暗示着刚刚开花的百合植株的地上部正进行着旺盛的生命活动。
[1] 陈敏敏,张建军,周 音,等. 百合根外植体体细胞胚胎再生影响因素研究[J]. 核农学报,2015,29(8):1494-1501.
[2] 钟军珺. 榆叶梅花发育相关MADs-box基因克隆和表达模式研究[D]. 北京:北京林业大学,2016:2-7.
[3] 张雪梅,赵 婧,赵银河. 莲瓣兰大雪素SEP1基因克隆和表达的研究[J]. 种子,2016,35(4):9-12.
[4] 徐 静,王 莉,钱 前,等. 水稻叶片形态建成分子调控机制研究进展[J]. 作物学报,2013,39(5):767-774.
[5] Chen S,Luo H,Li Y,et al. 454 EST analysis detects genes putatively involved in ginsenoside biosynthesis inPanaxginseng[J]. Plant Cell Reports,2011,30(9):1593-1601.
[6] Fan X D,Wang J Q,Yang N,et al. Gene expression profiling of soybean leaves and roots under salt,saline-alkali and drought stress by high-throughput Illumina sequencing[J]. Gene,2013,512(2):392-402.
[7] Villacorta-Martin C,Gonzalez F F,Huijben K,et al. Whole transcriptome profiling of the vernalization process inLiliumlongiflorum(cultivar White Heaven) bulbs[J]. BMC Genomics,2015,16:1-16.
[8] Wang J,Wang Q,Yang Y,et al.Denovoassembly and characterization of stress transcriptome and regulatory networks under temperature,salt and hormone stresses inLiliumlancifolium[J]. Molecular Biology Report ,2014,41(12):8231-8245.
[9] Li X E,Wang C X,Cheng J Y,et al. Transcriptome analysis of carbohydrate metabolism during bulblet formation and development inLiliumdavidiivar.unicolor[J]. BMC Plant Biology,2014,14:358.
[10] Zhu X P,Chai M,Li Y,et al. Global transcriptome profiling analysis of inhibitory effects of paclobutrazol on leaf growth in lily (Liliumlongiflorum-Asiatic Hybrid)[J]. Frontiers in Plant Science,2016,7:491.
[11] Lang V,Usadel B,Obermeyer G.Denovosequencing and analysis of the lily pollen transcriptome:an open access data source for an orphan plant species[J]. Plant Molecular Biology,2015,87(1/2):69-80.
[12] Liu X H,Huang J,Wang J M,et al. RNA-Seq analysis reveals genetic bases of the flowering process in oriental hybrid lily cv. Sorbonne[J]. Russian Journal of Plant Physiology,2014,61(6):880-892.
[13] Rhee S J,Seo M,Jang Y J,et al. Transcriptome profiling of differentially expressed genes in floral buds and flowers of male sterile and fertile lines in watermelon[J]. BMC Genomics,2015,16:914.
[14] Zhang M F,Jiang L M,Zhang D M,et al.Denovotranscriptome characterization ofLiliumSorbonne and key enzymes related to the flavonoid biosynthesis[J]. Molecular Genetics and Genomics,2015,290(1):399-412.
[15] Du F,Wu Y,Zhang L,et al.Denovoassembled transcriptome analysis and SSR marker development of a mixture of six tissues fromLiliumOriental Hybrid Sorbonne[J]. Plant Molecular Biology Report,2014,33(2):281-293.
[16] 杜 方. 百合不同器官转录组分析及SSR标记开发应用[D]. 杭州:浙江大学,2014:24-25.
[17] 王玉倩,薛秀花. 实时荧光定量PCR技术研究进展及其应用[J]. 生物学通报,2006,51(2):1-5.
[18] 刘 畅,姜 晶,韩晓雪,等. 植物中SWEET基因家族研究进展[J]. 植物生理学报,2014,50(9):1367-1373.
[19] Tamai S,Iida H,Yokota S,et al. Characterization of the mitochondrial protein LETM1,which maintains the mitochondrial tubular shapes and interacts with the AAA-ATPase BCS1L[J]. Journal of Cell Science,2008,121(15):2588-2600.
[20] Takeda S,Hanano K,Kariya A,et al. CUP-SHAPED COTYLEDON1 transcription factor activates the expression ofLSH4 andLSH3 ,two members of the ALOG gene family,in shoot organ boundary cells[J]. Plant Journal,2011,66(6):1066-1077.
[21] Muhlemann J K,Klempien A,Dudareva N. Floral volatiles:from biosynthesis to function[J]. Plant Cell and Environment,2014,37(SI):1936-1949.
[22] Chepyshko H,Lai C P,Huang L M,et al. Multifunctionality and diversity of GDSL esterase/lipase gene family in rice(OryzasativaL.japonica)genome:new insights from bioinformatics analysis[J]. BMC Genomics,2012,13(1):1-19.
[23] Keeling C I,Madilao L L,Zerbe P A,et al. The primary diterpene synthase products ofpiceaabieslevopimaradiene/abietadiene synthase (PaLAS) are epimers of a thermally unstable diterpenol[J]. Journal of Biological Chemistry,2011,286(24):21145-21153.
[24] Kim J H,Lee K I,Chang Y J,et al. Developmental pattern ofGinkgobilobalevopimaradiene synthase (GbLPS) as probed by promoter analysis inArabidopsisthaliana[J]. Plant Cell Reports,2012,31(6):1119-1127.
[25] Ro D K,Bohlmann J. Diterpene resin acid biosynthesis in loblolly pine(Pinustaeda):functional characterization of abietadiene/levopimaradiene synthase(PtTPS-LAS)cDNA and subcellular targeting of PtTPS-LAS and abietadienol/abietadienal oxidase(PtAO,CYP720B1)[J]. Phytochemistry,2006,67(15):1572-1578.
[26] 张传丽,仲月明,沈丹红,等. 植物类黄酮O-甲基转移酶研究进展[J]. 西北植物学报,2012,32(6):1274-1281.
[27] 刘攀峰,杜红岩,杜兰英,等. 1-羟基-2-甲基-2-(E)-丁烯基-4-二磷酸合酶基因cDNA全长克隆与序列分析[J]. 植物研究,2012(4):444-451.
[28] 程琪庆,何云飞,李 耿,等. 丹参4-羟基-3-甲基-2-丁烯基焦磷酸还原酶基因的全长克隆与诱导表达分析[J]. 药学学报,2013(2):236-242.
[29] 王曼玲,胡中立,周明全,等. 植物多酚氧化酶的研究进展[J]. 植物学通报,2005,22(2):215-222.
[30] 谢鑫鑫,吴卫东,林碧英,等. 脂氢过氧化物裂解酶基因对生菜遗传转化的研究[J]. 热带作物学报,2016,37(5):856-861.
[31] Johnson T S,Schwieterman M L,Kim J Y,et al.Liliumfloral fragrance:A biochemical and genetic resource for aroma and flavor[J]. Phytochemistry,2016,122:103-112.
[32] Silvestro D,Andersen T G,Schaller H A. Plant sterol metabolism. delta(7)-Sterol-C-5-Desaturase (STE1/DWARF7),delta(5,7)-Sterol-Delta(7)-Reductase (DWARF5) and delta(24)-Sterol-Delta(24)-Reductase (DIMINUTO/DWARF1) show multiple subcellular localizations inArabidopsisthaliana(heynh) L.[J].PLoS One,2013,8(2):1-11.
[33] 周延清,张永华,张 喻,等. 怀地黄3-酮酯酰CoA-硫解酶基因的克隆、序列特征和时空表达分析[J]. 中草药,2013,44(1):76-84.
AnalysisofDifferentiallyExpressedGenesinOrgansofLiliumformolongiJulius
DU Fang,WANG Ting,FAN Junmiao,ZHANG Haoyu
(College of Horticulture,Shanxi Agricultural University,Taigu 030801,China)
To understand the expression levels of 17 candidate genes in flower,leaf and scale ofLiliumformolongiJilius,Real time quantitative PCR was conducted. The results showed that the 17 candidate genes could be divided into five groups.CL10636.Contig1_All,CL6300.Contig2_All,CL7951. Contig5_All,Unigene8314_All,Unigene19279_All and CL1306.Contig1_All significantly highly expressed in flowers,which were highly homologous with Bidirectional sugar transporter SWEET4-like gene,cold-regulated gibberellin-regulated protein gene bidirectional,mitochondrial chaperone gene,Lightlight-dependent short hypocotyls 4-like gene,1-deoxyxylulose-5-phosphate synthase gene,geranylgeranyl pyrophosphate synthase gene. CL9292.Contig2_All,CL212.Contig5_All,Unigene1800_All,CL3468. Contig1_All,CL4520.Contig5_All,Unigene4212_All,CL4079.Contig1_All and Unigene10210_All eight significantly highly expressed in leaves,which were highly homologous with GDSL esterase/lipase gene,flavonoid O-methyltransferase gene,hydroperoxide lyase gene,4-hydroxy-3-methylbut-2-en-1-yl diphosphate gene,polyphenol oxidase gene,glucomannan 4-beta-mannosyltransferase 1-like gene,Levopimaradiene synthase gene and trans-ocimene synthase gene. CL2405.Contig1_All significantly highly expressed in scales,function unknown. CL1114.Contig2_All significantly highly expressed in both flowers and leaves,highly homologous with 3-ketoacyl-CoA thiolase gene. CL1772.Contig2_All,homologous with delta-(24)-sterol reductase,was non-organ specific gene. The results are valuable for the functional study of those genes and will be benefit for the molecular breeding of lily.
Lily;Differentially expressed genes;Real-time q-PCR;Organs
2017-05-02
山西省应用基础研究项目(201601D011077);山西农业大学引进人才科研启动项目(2014ZZ02)
杜 方(1973-),女,山西原平人,副教授,博士,硕士生导师,主要从事花卉种质资源创新及生物技术应用研究。
S682.29;Q78
A
1000-7091(2017)05-0025-06
10.7668/hbnxb.2017.05.005

