基于电子病历的临床医疗大数据挖掘流程与方法
阮彤,高炬,冯东雷,钱夕元,王婷,孙程琳
1. 华东理工大学,上海 200237;2. 上海曙光医院,上海 200025;3. 万达信息股份有限公司,上海 200233
基于电子病历的临床医疗大数据挖掘流程与方法
阮彤1,高炬2,冯东雷3,钱夕元1,王婷1,孙程琳1
1. 华东理工大学,上海 200237;2. 上海曙光医院,上海 200025;3. 万达信息股份有限公司,上海 200233
以医院电子病历为核心的临床数据记录了病人的疾病、诊断和治疗信息。挖掘此类数据,可以辅助医生进行临床科研与临床诊疗。首先提出了临床大数据挖掘过程中碰到的各项难题,总结了临床医疗大数据挖掘的核心流程,流程包括以临床数据集成、基于知识图谱的临床专病库的构建过程、电子病历数据质量的评估方法以及以临床疗效分析与疾病预测为核心的临床医疗大数据应用等任务,进而对流程中的每个任务提出了解决方案,给出了实验结果。最后,展望了未来临床电子病历挖掘应用和技术的发展。
医疗知识图谱;临床专病库;数据质量评估;电子病历;疾病预测;疗效对比
1 引言
医疗健康大数据研究对辅助医生给病人选择更好的治疗方案,进而提升医疗服务质量,降低医疗成本有积极的作用,得到了各国政府的大力支持。从2013年起,美国、英国在医疗大数据应用方面投入了大量资金[1,2]。2015年3月,我国在国家卫生计划生育委员会网络安全和信息化工作组全体会议上提出“推进健康医疗大数据应用,制定促进健康医疗大数据应用的相关方案,推动健康医疗大数据有序发展”的意见。2016年6月,国务院办公厅颁发了《关于促进和规范健康医疗大数据应用发展的指导意见》,明确指出健康医疗大数据是国家重要的基础性战略资源,要通过其应用,激发深化医药卫生体制改革的动力和活力,提升健康医疗服务效率和质量。
医疗健康大数据包含来自于移动终端的个人健康数据、医院临床数据、基因数据以及疾病预防控制的流调数据。从长远来说,上述多个来源的数据的融合,能为个人的健康规划、疾病防治以及国家卫生策略提供更好的数据基础。但高质量的数据采集和融合不是一蹴而就的,鲜有机构能够采集到大规模的关联的包含个人健康、基因以及临床信息的病人数据。
相比而言,过去十余年中,随着医疗信息化的不断推进,医疗机构经过长期的历史积累已拥有大量的电子病历(electronic medical record,EMR)数据。对于临床科研而言,与临床实验获得的数据或是人工构造的专病队列数据相比,EMR数据具有采集成本低和数据实时等优势。当前已有越来越多的研究[3]将EMR数据用于疗效分析与转归分析等临床科研中。因此,以医院电子病历为基础的临床大数据挖掘工作具有较好的数据基础。
笔者项目团队3年前依托于国家“863”计划项目,建立了包括医院临床医生、医院临床信息化、计算机工程师、数据分析师以及卫生管理的跨学科团队,以心衰和大肠癌两个慢性疾病为核心,展开了临床大数据研究。在研究过程中,碰到了下列问题。
整体挖掘流程问题。挖掘过程是由应用驱动、方法驱动,还是由数据驱动?换而言之,是先整理数据,根据数据找问题,还是基于问题采集数据,寻找合适的挖掘方法。是否存在一个理想的数据挖掘方法,在数据有噪音的情况下,无需数据清洗,也会有比较好的数据结果。
病历文本问题。在临床中,大量的医疗文书以文本形式存在。电子病历的文本包含了病人病史、家族史、症状以及医生根据症状、理化指标等基础数据做出的诊断等描述。更重要的是,临床文本中记录了医生的判断依据以及对各种诊疗行为的效果跟踪。如果说各种明细记录是结果跟踪,那么文本数据就是过程跟踪的基础。而这些重要的信息保存在非结构化信息中,不能被计算机理解和处理。
数据质量(可用性)问题。由于EMR数据来源于多个不同的信息系统,经历了多次版本变化,数据的统一表示、关联和集成存在各种问题。同时,医生录入缺乏语义规范,同一诊断与治疗方案,不同医生的录入结果会不同。另外,EMR数据产生于病人真实的诊疗情况记录,目的并不直接面向科学研究。一个诊疗质量良好的病人记录,未必可以产生满足科研需求的数据记录。
分析与挖掘方法问题。传统医学使用随机临床实验证明疗效,是传统医学研究的基础方法。在大数据场景下,不存在临床对照组,如何证明医学事件之间的因果关系,是目前医学界真实事件研究的话题之一[4]①。与此同时,以深度学习为核心的机器学习方法,在疾病的预测、诊疗方法方面会有比较好的效果,然而,这些学习方法可解释性比较差,难以被医学领域的科研工作者认同。
本文针对上述问题进行了研究,介绍了医疗大数据挖掘的整体流程、基于知识图谱的临床文本结构化过程、电子病历数据质量的评估方法及部分挖掘应用的成果。
2 基于电子病历的临床大数据挖掘整体流程
图1展示了基于电子病历的临床医疗大数据的整体流程。第一步,对来自不同医院信息系统的病人数据进行数据集成,形成临床数据中心(clinical data repository,CDR)。数据来源包括医院信息系统(hospital information system,HIS)、临床信息系统(clinical information system,CIS)、实验室信息系统(laboratory information system,LIS)、放射信息管理系统(radioiogy information system,RIS)、影像归档和通信系统(picture archiving and communication system,PACS)和病案系统等信息系统。第二步,基于CDR构造面向特殊疾病的专病库,如大肠癌病例库、心衰病例库等。在构建临床专病库时,要确定符合疾病特征的病例;确定需要的病例字段,对于结构化的字段,需要从原始的电子病历库中抽取,例如年龄与性别,对于半结构化或非结构化字段,需要使用文本抽取等技术,结合知识库对其进行结构化。在这个过程中,需要建立知识图谱,以方便自动化的病例数据抽取。第三步,需要对病例库进行数据质量评估,评估其是否适用于挖掘。评估指标包括数据完整性、一致性、医疗实体及其编码的一致性、数据精确性等。若病历库达到评估要求,即可进行第四步的数据挖掘,如果不能,则需要回到前面步骤,重新抽取和整理数据。第四步,确定挖掘目标,选择合适的模型,设计并实施实验。如果实验发生问题,可能需要改进算法,也有可能是数据质量缘故,需要回到前面步骤,重新抽取和整理数据。
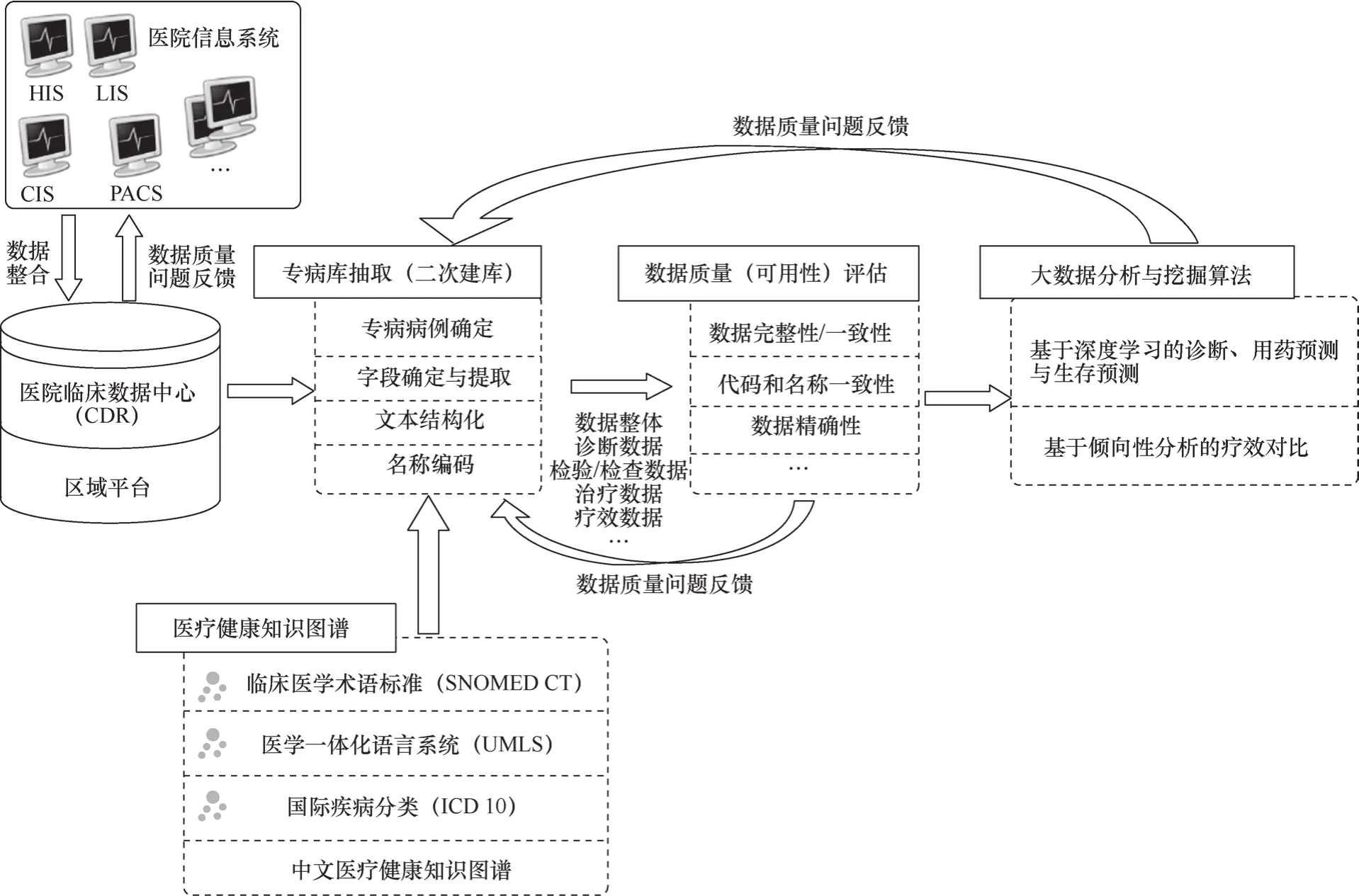
图1 基于电子病历的临床医疗大数据挖掘整体流程
3 基于中文医疗健康知识图谱构建临床专病库
挖掘与预测算法通常处理的是结构化数据。然而,在临床中,大量的医疗文书是以文本形式存在的。电子病历的文本包含了病人病史、家族史、症状以及医生根据症状、理化指标等基础数据做出的诊断等描述,更重要的是,临床文本中记录了医生的判断依据以及对各种诊疗行为的效果跟踪。因此,需要将文本结构化。
然而,仅仅结构化也是不够的,因为医疗术语存在大量的同义词或上下位词,比如,同一症状具有多种多样的文本表达形式,如“期前收缩”“过早搏动”与“早搏”是同义词。再比如,一个症状常常被不同的词语修饰,以表达略有不同的语义含义,如“急性背痛”“慢性背痛”都可以是“背痛”的下位词。
再以疾病为例,目前医学诊断大量采用了国际疾病分类(international classification of diseases,ICD)编码,但ICD编码结构并不包含完整的上下位关系。以中文ICD编码[5]中的“特指急性风湿性心脏病”为例,它的上位词有“特指风湿性心脏病”和“急性风湿性心脏病”,这两种疾病拥有共同的上位词“风湿性心脏病”,“风湿性心脏病”又有上位词“心脏病”。而这几种疾病之间的关系和层次结构并没有在ICD 10中通过编码结构表示出来,只是通过编码的首字母“I”将它们划分到了循环系统类疾病中。如果希望找到某一类患者,无法通过一个ICD编码获得,而是需要人工地选择多个ICD编码。同时,医生在编写一个疾病的ICD编码时,可粗可细,也会给病历的自动处理带来困难。
为此,需要建立一个标准化的、包含疾病、症状等在内的医疗健康知识图谱,然后通过文本挖掘与实体链接手段,将结构化的文本与知识库相关联,如图2所示。一段医疗文本中,可能包含具体的家族史、时间事件、症状、检查、诊断与用药等信息,这些信息依赖于知识图谱抽取出来后,变成结构化的信息,如症状部位、症状的有无、诊断编码、检查结果与病理分期等。这样结构化的病例,可以更方便后续数据的挖掘。
3.1 中文医疗健康知识图谱构建
近年来,生物医疗领域的海量数据迅速形成。然而,目前医疗行业数据存在封闭、分散且表示方式不一致的问题。生物医疗领域缺乏公开的中文基础数据与公共的数据服务,不同来源的数据缺乏关联与融合,制约了整个行业的发展。
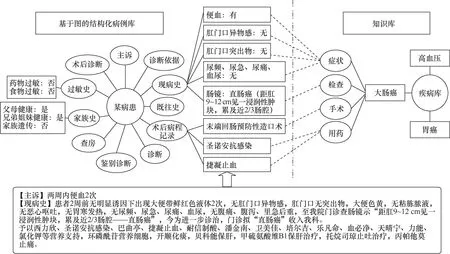
图2 基于知识图谱/知识库的结构化病例库的构建
与此形成鲜明对比的是,国外的生物医疗数据涉及领域内的方方面面。一方面,国外构建了丰富的生物医疗分类体系和本体,如一体化医学语言系统(unified medical language system,UMLS)[6]、医学主题词表(medical subject headings,MeSH)②、临床医疗术语集(systematized nomenclature of medicine -clinical terms,SNOMEDC T)③等通用的分类系统,还有面向药物的命名系统RxNorm④、针对观测指标的编码系统LOINC⑤、基因本体(gene ontology)⑥和被广泛应用的疾病分类系统ICD9⑦、ICD10⑧等细分的本体和系统。此外,国外还发布了临床病人数据集,例如,由美国国家癌症研究所领导的项目癌症和肿瘤基因图谱(the cancer genome atlas,TCGA)[7]收集并发布了癌症病人的临床数据以及美国国立卫生研究院发布的面向全球人类受试者的临床研究数据库ClinicalTrail.gov⑨。
基于这些分类体系和标准,国外的研究工作者构建了多个生物医药数据集平台,发布了大量的链接数据集,较为知名的数据集平台有Linked Open Drug Data[8]、Liked Life Data⑩和Bio2RDF[9]。其中,Linked Open Drug Data整合了14个数据集,包含超过800万的RDF三元组和超过37万的RDF链接。Liked Life Data提供了25个公共生物医疗数据集的统一访问点,覆盖了基因、蛋白质、分子反应、信号通路、靶点、药物、疾病和临床试验相关的信息。Bio2RDF利用语义网络技术建立并提供生命科学领域最大的链接数据网络,其最新版本包含了35个数据集,共110亿条三元组。这些开放链接数据集的发布大大促进了国外生物医药领域研究工作的发展。
目前为止,中文缺乏比较好的知识图谱,而英文知识图谱的汉化也存在版权问题。因此,为方便后续的电子病历结构化以及大数据挖掘工作,笔者项目组利用互联网数据与百科数据,构造了自己的知识图谱。从医学角度来说,可能存在不精准之处,但用于数据的预处理过程确是有效的。笔者团队的知识图谱的构建过程如下。
(1)模式图定义
在领域专家的帮助下,根据医疗知识手工创建医疗知识图谱的模式图,包含概念、概念的属性以及概念之间的层次关系。图3展示了笔者定义的医疗知识图谱的模式。笔者定义了5个顶层概念:症状、疾病、药品、科室和检查。“症状”概念又细分为“中医症状”和“西医症状”两个子概念,“药品”细分为“中药”和“西药”两个子概念。概念之间通过“症状相关疾病”“疾病相关科室”等属性进行关联。每个概念都给出了实例,这些实例形成了临床实践中一个场景:一位“头部”患有“头痛”的患者同时患有“打喷嚏”“恶寒”等症状,则该患者需要去“内科”就诊,并进行“血检”和“测温”等相关检查。该患者最终被诊断为“夏季感冒”,并伴有“扁桃体发炎”,建议服用西药“阿司匹林”和中药“小柴胡”。
(2)医疗知识抽取
基于上文定义的模式图,抽取实体(症状、疾病与检查等)、属性和属性值,用来构建医疗知识图谱。知识抽取分为医疗健康网站的知识抽取和中文百科站点的知识抽取两部分。
笔者收集了多个医疗健康网站作为知识抽取的数据源,医疗健康网站包含症状、疾病、药品、检查和科室5种类型的实体,每一类实体都有两种类型的页面:实体列表页面和实体详情页面。其中,实体列表页面列举了该网站上所有属于该类型的实体,实体详情页面则展示了某个实体的详细信息。
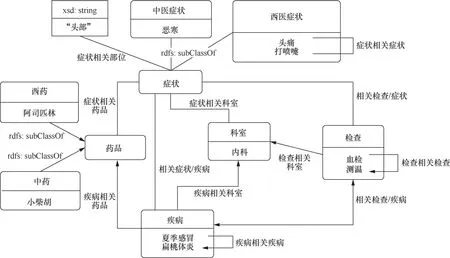
图3 医疗知识图谱的模式
医疗健康网站的知识抽取过程为:从实体列表页面出发,爬取所有实体的详情页面,这一过程抽取了实体的类型。对于相同类型的实体,它们的详情页面具有相同的页面结构,因此基于统一的超文本标记语言(hypertext markup language,HTML)封装器抽取页面中的“信息框”。“信息框”是一种半结构化的数据,包含了实体的属性信息。最后,基于人工总结的Hearst模板[10]从详情页面的摘要中抽取实体的同义词。
接着,选取了3个最大的中文百科站点(百度百科、互动百科和中文维基百科)进行知识抽取,包括抽取和分类两个阶段。首先将医疗健康网站抽取得到的实体作为种子集,获取它们在百科页面中的分类。然后抽取分类中包含的所有实体,形成一个实体集合。这些集合中包含了和目标无关的噪声实体,因此训练一个分类器对抽取阶段得到的结果进行分类。训练数据的正例来自医疗健康网站不同类型的实体,负例则由医疗健康网站中“美容”“养生”和“心理”列表页面下的实体组成。分类器的特征来自百科实体页面的“实体名”“摘要”“目录”“正文”和“分类”5个字段。笔者基于启发式规则将百科实体页面的5个字段转化成一系列二值型特征。
(3)医疗知识融合
知识融合阶段对抽取结果进行实体对齐、实体类型对齐和实体属性对齐。实体对齐主要是建立实体之间的同义关系。为保证数据的可靠性,将医疗健康网站和中文百科站点抽取的同义关系加入医疗知识图谱中,并不通过算法计算实体间新的同义关系。
实体类型对齐解决了一个实体对应多个互斥类型的数据冲突问题。笔者采用基于投票和数据源优先级的方法确定实体类型。整体思路是:票数最高的结果作为实体的最终类型;当出现多个类型获得最高票数时,根据最高票数中权重最大的数据源确定最终结果。
实体属性对齐主要建立抽取的实体属性三元组的谓词到模式图中属性的映射关系。对于医疗健康网站,由于同一网站下相同类型实体的“信息框”包含了相同的实体属性,笔者手工制定“信息框”到模式图的映射规则。例如,从“信息框”中抽取的“关节疼痛”的3个属性为症状部位、相关科室和相关疾病,分别映射到模式图中的症状相关部位、症状相关科室和症状相关疾病。
3.2 临床专病库的构建
为了对特定疾病进行挖掘分析,常用的方法是构建专病病例库。专病病例库的构建有3个步骤:专病病例确定、专病病例库所需字段确定与提取以及专病病历文本结构化。
(1)专病病例确定
专病病例主要根据疾病的ICD编码和疾病名称从医院信息系统中抽取。考虑到医院信息系统在时间上经历了多次版本变化,在抽取专病病历时,使用ICD 9以及ICD 10编码中涉及该疾病的所有编码集合抽取相关病历。ICD中疾病编码和名称有完整的规范,考虑到很多医护人员不了解ICD体系,难以分辨ICD中疾病名称之间的细微差别,因此系统中常出现ICD编码与疾病名称不对应的情况,单使用ICD编码难以抽全该疾病的所有病历,还需使用该疾病名称及其同义词从疾病名称字段进行抽取。这个过程目前是手动完成的,未来会对现有的ICD编码库补充部分层次结构,并自动对疾病名称进行编码,进而寻找某一类疾病的所有病例。
(2)专病病例库所需字段确定与提取
本文中,专病库的字段使用Delphi过程[11]向专家收集。根据临床医生定义、疾病的诊疗指南、挖掘需求、相关文献等多个来源的需求,明确用户使用数据的目的和重点关注的数据。Delphi过程通过多轮咨询问卷向领域专家开展问卷调查,可以比较好地找到共性需求,已被用在医疗电子病历实施的关键因素分析、诊疗方案的调查等多个场合。
在使用Delphi过程向专家收集专病库字段时,选择了3类专家:第一类是从事临床科研的临床医生;第二类是从事医疗大数据挖掘的科研人员;第三类是医院信息科的数据管理人员以及负责系统构建与数据集成的IT工程师。由临床专家和数据挖掘专家填写需求字段,医院信息科工作人员根据需求字段填写字段来源。然后进行多轮调查,确定对临床症—治—效分析及医疗大数据挖掘所需的字段。采用电子邮件形式发放和回收调查表,调查一共进行3轮。每一轮的调查结果会以匿名的方式将报告提供给下一轮的参与者。调查过程中参与者在任何时间都可以退出。
(3)专病病历文本结构化
医疗病历中很大一部分都是由医生用自然语言书写而成,内容繁复,形式多样,无法直接对其进行处理,因而需要将其转化为结构化数据,抽取出其中的症状、疾病、检查等信息,或与知识库中的实体进行链接,或对检查指标进行统一转换(包括书写格式的统一与计量单位的统一等),从而实现病历文本的结构化与病历信息的标准化。
下面以病历文本中症状的结构化为例进行说明。首先需要识别出文本中的症状,其识别方法参见上文医疗实体抽取方法的相关介绍。然后需要对识别出的症状进行构成成分分析。中文症状可以拆分为以下16种组成成分:原子症状、连词、否定词、存在词、程度词、发展词、能够词、不能词、动作词、情景限定词、方位词、部位词、中心词、感觉词、特征词、修饰词,见表1。
其中,原子症状是最基本的症状描述;连词可以连接多个构成元素;否定词、存在词、程度词是一类构成元素,用于对原子症状或中心词的多寡有无进行度量;发展词用于描述症状的发展状况,好转或恶化;能够词与不能词是一类构成元素,用于描述是否具有某种能力;动作词用来表示特定的动作;情景限定词对症状发生的情景进行限定;方位词用来表示方位,一般是对部位词的进一步描述;部位词用来表示身体部位;中心词是症状所要描述的除身体部位外的客观实体;感觉词则是症状所要描述的主观感受;特征词用于描述事物的特征,是对症状描述主体的进一步刻画;剩下的均为修饰词。

表1 症状构成元素
对中文症状进行构成分析,类似于中文分词与词性标注,可以把它看成序列标注任务,运用条件随机场(conditional random field,CRF)或双向长短期记忆(long short-term memory,LSTM)网络+CRF等方法进行实现。在得到每个症状的构成成分之后,便可以对其进行归一化处理,如对于原子症状“疼”“痛”“疼痛”,统一为“疼痛”;对于程度词及否定词,“无”可以量化成0,“轻微”可以量化成0.2,“有点”可以量化成0.4,“明显”可以量化成0.6,“广泛”可以量化成0.8,“极度”可以量化成1。此外,还可以根据切分出的症状构成成分,将抽取出的症状与知识库中的症状实体进行软链接,从而实现症状的标准化。
4 电子病历数据质量评估
电子病历数据来源于医院实际业务系统,医疗系统主要由医疗工作人员人工录入,难免存在一些数据质量问题,而质量问题是影响医疗挖掘结果准确性的重要因素。因此,评估电子病历数据能否或多大程度上能用于以症—治—效分析为核心的临床科研,对于目前的医疗挖掘以及未来电子病历数据质量的提升,都具有重要的意义。
数据质量评估过程分为6个步骤。
步骤1 使用Delphi过程收集评估需求。根据临床医生定义、疾病的诊疗指南、相关文献等多个来源的需求,明确用户使用数据的目的和重点关注的数据。
步骤2 确定和采集评估数据。根据评估需求,明确评估的数据范围,抽取出待评估数据集。电子病历主要有两类,即门诊病历和住院病历。门诊病历通常较短,包含信息较少,也缺乏对患者治疗情况的跟踪,因而,电子病历信息抽取和文本挖掘研究大多关注于住院病历。
步骤3 建立评估需求与评估数据之间的映射关系。根据临床科研人员、大数据挖掘人员的需求,补充需求字段来源与字段类型,其中需求字段来源用于说明字段来源于哪几个系统的哪几个字段,字段类型用于说明是文本、结构化还是影像类型。
步骤4 提出质量评估指标。根据用户使用数据的目的选择评估度量或自定义评估度量。针对研究人员的心血管疗效分析需求,提出心血管疗效分析评估度量指标,具体对数据整体质量、患者基础数据质量、诊断数据质量、治疗数据质量以及疗效数据质量建立评估度量指标,得到的指标体系见表2。
步骤5 执行数据质量评估,针对每个评估度量进行数据质量评估,根据评分标准得到评估,该过程可以自动执行或者人工评估。
步骤6 分析评估结果。根据评估结果分析数据集的质量问题,判定是否适合于研究目的。
通过对项目中电子病历数据的分析可知,电子病历数据用于疗效分析研究具有一定的可用性,但现有数据质量在很多方面还存在一些问题。考虑以下几方面的改进措施。

表2 心血管疾病质量评估指标体系
首先,需要集成更多的医院系统。例如,心电图和心脏彩超的数据影响着心血管疾病的诊疗,也是疗效评估的依据。而HIS和LIS中缺乏此类检查数据,系统需要集成医院的RIS和PACS,确保用于疗效分析研究数据的可用性。其次,改进与规范数据录入规程,加强各环节的管理,例如,一些家族史或是症状信息可由患者自助录入。最后,引入更多的元数据规范,现有症状与检查名称缺乏规范,需要大量的数据后处理工作,可以引入更为完整的元数据规范,如SNOMED以及LOINC。
5 临床医疗大数据挖掘应用
5.1 基于深度学习的疾病预测
目前,大多数医疗领域相关工作都集中于疾病风险预测和疗效预测[12-18],诊疗模式预测的相关工作较少,而且诊疗模式预测的工作目前使用的方法大多数还是基于规则和传统机器学习算法[19,20]。深度学习在医疗领域涉及还不深,典型的工作见参考文献[21],该文献通过对病人的电子病历进行时间维度上的建模,然后使用卷积神经网络(convolutional neural network,CNN)模型进行疾病风险的预测。循环神经网络(recurrent neural network,RNN)模型目前还主要集中于疾病风险预测和疗效预测的范围[22,23]。
一个病人可能有多次住院的电子病历信息,在对其进行疾病预测的时候,需要考虑多次住院的电子病历序列,而不是某次住院的电子病历,使用传统的特征抽取方法难以捕捉到历次住院之间的变化信息。RNN模型可以用来处理序列数据,但是如果RNN的循环序列过长,它的性能就会有所下降。LSTM模型是对RNN的一种改进,它能够选择性地记忆前面节点的信息,因而可以获得更长的最大稳定序列长度。这也更加符合病人的时间关系特点,即一个病人的前一次住院情况总是部分地影响下一次住院时的情况。因此,使用LSTM模型对病人历次住院病历进行建模较为合理,具体建模使用参考文献[24]中提出的序列到序列(sequence to sequence,Seq2Seq)思想构造住院病人向量特征。
(1)住院病人的向量表示
对于如何生成病人的向量表示,采用了Seq2Seq模型的思想。如图4所示,将模型编码出的中间编码C向量作为病人的特征。与原来模型不同的是,Seq2Seq的模型通常被用在机器翻译中,所以输出层选择的是softmax +交叉熵。而这里由于是自动编码器的思路,所以输出层和输入层的数据是一致的。
使用深度学习的自动编码器,将病人的每一次住院记录编码成一个低维稠密的向量,用于病人的特征表示。然而,病人的住院记录通常不止一次,那么对于编码出来的向量就需要用来表示其历次住院时的一个信息的总和。即对于一个病人,其就诊记录为x1,x2,…,xn,那么就需要生成对应的一组向量V={v1,v2,…,vn},对于一个向量vn,需要能够表示从x1一直到xn中所有记录的信息。
通过对出院次数分布进行统计,发现超过10次住院的病人仅占很少的比例,因此考虑到训练性能以及信息损失的问题,将Seq2Seq模型中的最大步长设置为10。对于超过10次和不满10次住院的病人采取如下的方法进行预处理。
● 首先,将一个病人多次住院的记录进行拆分,即将x1,x2,…,xn拆分成n条训练数据:{x1},{x1,x2},…,{x1,x2,…,xn}。
● 对于超过10次住院的病人,由于最大步长为10,故需要进行裁剪,笔者选择保留最后10次的数据,将剩余的数据进行裁剪。即当n>10时,仅保留{xn-9,xn-8,…,xn}这10次记录。
(2)疾病预测

图4 Seq2Seq模型
对比Seq2Seq模型构造的特征与其他方法在预测病人疾病上的优劣,实验结果以及部分设置见表3。本实验预测的对象是10种常见的心衰患者的伴随疾病,具体见表3第一列。对比使用Seq2Seq产生的特征向量与使用主成分分析(principal component analysis,PCA)、K均值(K-means)、高斯混合模型(gaussian mixture model,GMM)等方法生成的特征向量预测疾病的效果。通过实验对比发现,使用支持向量机(support vector machine,SVM)进行疾病预测的效果比使用K最近邻(K-nearest neighbor,KNN)、朴素贝叶斯(naive Bayes)、随机森林(random forest)、梯度提升决策树(gradient boosting decision tree,GBDT)、逻辑回归(logistic regression)好,以下直接使用SVM进行实验。对于一些疾病,由于其出现在实验数据中的样本较少,笔者采用了NearMiss[25]进行采样(当百分率低于30%时进行采样),进行采样的疾病由sample列(sample为表3最后一列列名)标识。其中NearMiss是通过与TomekLinks[26]、簇中心、oneside selection(OSS)[27]、edited nearest neighbour rule[28]、neighbourhood clearning rule(NCR)[29]、synthetic minority over-sampling technique(SMOTE)[30]、随机欠采样(random under-sampling,RUS)对比得到效果最好的采样模型。
表3对比实验结果为各个方法在各个疾病预测上的曲线下面积(area under curve,AUC)值。第一列是需要预测的疾病名称,第二列是本文的方法,第三列到第五列是PCA、K-means、GMM对比方法,第六列hand表示未对原始特征做预处理,第七列count是患者中有并发疾病的数目,第八列percent是患者中有并发疾病的百分比,第九列sample表示是否用了NearMIss进行采样。从实验结果可知,使用Seq2Seq模型构造的特征在6项疾病预测中排第一,一项疾病排第二,明显优于其他特征生成方法。然而该方法并不是在所有疾病预测中占优。该方法优势在于不需要手工进行特征选择,而且在实践中发现,传统特征构造方法经常受限于窗口大小的选择(即在窗口范围内是否有再住院),不同的窗口大小会影响分类效果,不如基于Seq2Seq模型构造特征的方法简单方便。因此,本文方法是疾病预测任务的最佳选择。
5.2 基于倾向值匹配的疗效对比
倾向值(propensity score)这一概念在1983年由Rosenbaum P R[31]提出,倾向值指被研究的个体在控制可观测到的混淆变量的情况下,受到某种自变量影响的条件概率。
倾向值匹配后的结果不仅仅指出了变量之间有关系,还进一步确定了二者之间的因果性,可以从科技哲学[32]和统计学[33]两个方面阐述。
考虑到医院信息系统中关于死亡的数据不完整,笔者使用180天内是否再入院替代疗效。因此,因变量是再入院,笔者关注的自变量是心衰患者的十大伴随疾病,即高血压、糖尿病、冠心病、房颤、慢性肾功能不全、心脏瓣膜疾病、扩张性心肌病、肥厚性心肌病、慢性阻塞性肺疾病和脑梗塞或一过性脑缺血。需要控制的混淆变量包括患者的年龄、性别、用药、脉搏、检查等信息。
表4是倾向值匹配后,进行逻辑回归后的结果,其中高血压、糖尿病、冠心病、房颤、慢性肾功能不全、扩张性心肌病、脑梗塞或一过性脑缺血对心衰患者180天再入院有显著影响(其中,P值<0.05时,变量具有显著性影响)。

表3 疾病预测对比实验
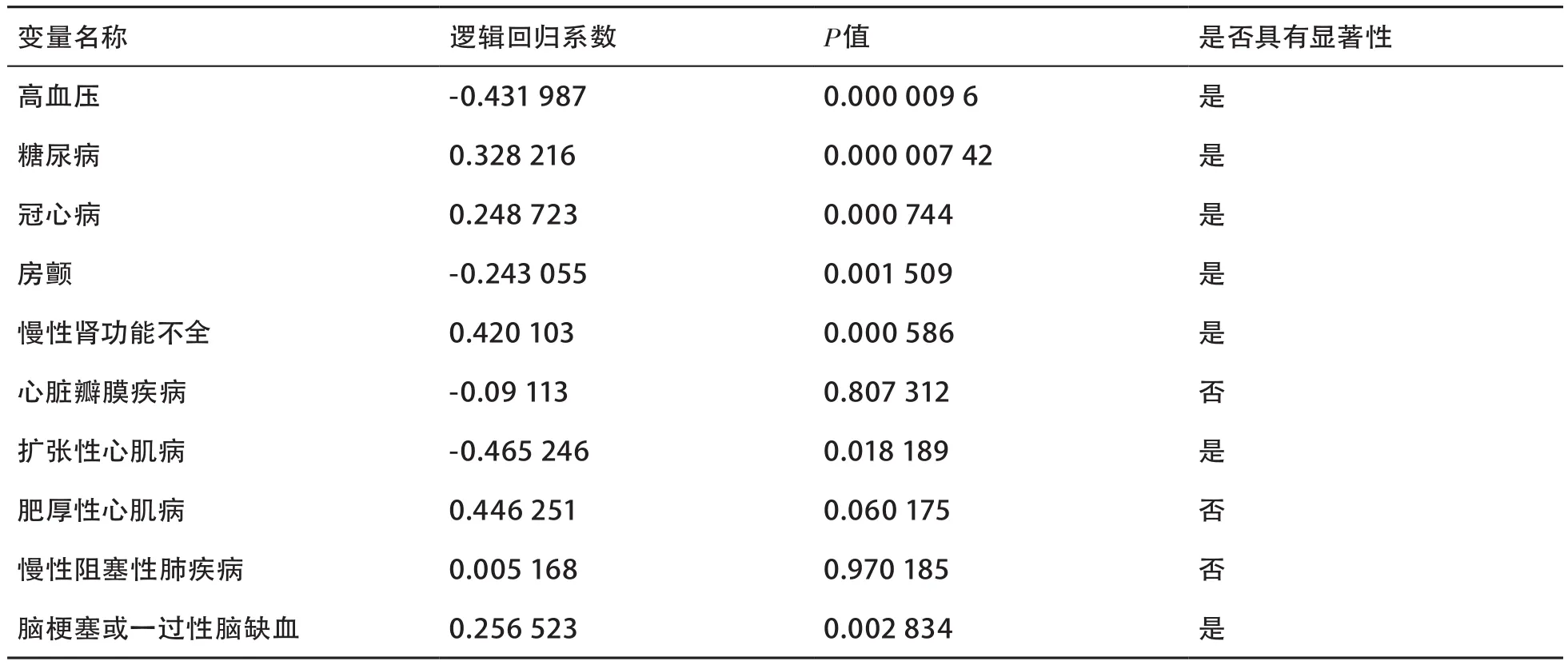
表4 伴随疾病显著性影响
6 结束语
医院信息系统数据优点在于获取代价低,缺点在于数据质量低,为此,本文给出了如何基于医院电子病历数据进行大数据挖掘的流程与应用示例。对于未来的工作,从数据角度,需要融合更多数据字段的病人数据;从方法角度,需要找到能够支撑真实世界研究更细致、更有说服力的统计学的方法,并且需要让现有的方法更有可解释性;从信息技术角度,可以进一步地将工作流程工具化,以便为医疗工作者提供更好的科研支撑。
[1] 王茜. 英国大数据战略分析[J]. 全球科技经济瞭望, 2013(8): 24-27.WANG X. British state strategy of developing big data[J]. Global Science,Technology and Economy Outlook,2013(8): 24-27.
[2] 王忠. 美国推动大数据技术发展的战略价值及启示[J]. 中国发展观察, 2012(6): 44-45.WANG Z. The strategic value and enlightenment of promoting big data technology development in America[J].China Development Observation, 2012(6):44-45.
[3] BROWN J S, HOLMES J H, SHAH K, et al.Distributed health data networks: a practical and preferred approach to multiinstitutional evaluations of comparative effectiveness, safety, and quality of care[J]. Med Care, 2010, 48(6): 45-51.
[4] S H E R M A N R E, A N D E R S O N S A,DALPAN G J, et al. Real-world evidencewhat is it and what can it tell us[J]. New England Journal of Medicine, 2016,375(23): 2293.
[5] 董景五. 疾病和有关健康问题的国际统计分类第十次修订本(ICD-10)[M]. 北京: 人民卫生出版社, 1996.DONG J W. The international statistical classification of diseases and related health problems 10th revision[M]. Beijing:People’s Medical Publishing House, 1996.
[6] BODENREIDER O. The unified medical language system (UMLS): integrating biomedical terminology[J]. Nucleic Acids Research, 2004, 32(suppl 1): D267-D270.
[7] W E I N S T E I N J N, CO L L I S S O N E A,MILLS G B, et al. The cancer genome atlas pan-cancer analysis project[J].Nature Genetics, 2013, 45(10): 1113-1120.
[8] S A M W A L D M, J E N T Z S C H A,BOUTON C, et al. Linked open drug data for pharmaceutical research a n d d e v e l o p m e n t[J]. J o u r n a l o f Cheminformatics, 2011, 3(1): 19.
[9] BELLEAU F, NOLIN M A, TOURIGNY N,et al. Bio2RDF: towards a mashup to build bioinformatics knowledge systems[J].Journal of Biomedical Informatics, 2008,41(5): 706-716.
[10] HEARST M A. Automatic acquisition of hyponyms from large text corpora[C]//The 14th Conference on Computational Linguistics, August 23-28, 1992, Nantes,France. New York: ACM Press, 1992:539-545.
[11] D A L K E Y N C, R O U R K E D L.Experimental assessment of Delphi procedures with group value judgements:advanced research projects agency[J].Cluster Analysis, 1971: 58.
[12] CHENG Y, WANG F, ZHANG P, et al.Risk prediction with electronic health records: a deep learning approach[C]//The 2016 SIAM International Conference on Data Mining, May 5-7, 2016, Miami,USA. [S.l.:s.n.], 2016: 432-440.
[13] S U T H E R L A N D S M, C H AW L A L S,K A N E-G I L L S L, e t a l. U t i l i z i n g electronic health records to predict acute kidney injury risk and outcomes:workgroup statements from the 15th,ADQI consensus conference[J]. Canadian Journal of Kidney Health & Disease, 2016,3(1): 1-14.
[14] WO LFS O N J, BANDYO PAD H YAY S,ELIDRISI M, et al. A naive Bayes machine learning approach to risk prediction using censored, time-to-event data[J]. Statistics in Medicine, 2014, 34(21): 2941-2957.
[15] 马宗帅. 基于深度学习的心脑血管疾病预测方法研究[D]. 西安: 西安建筑科技大学, 2015.MA Z S. Research on cardiovascular disease prediction based on deep learning technical[D]. Xi’an: Xi’an University of Architecture and Technology, 2015.
[16] AULI M, GALLEY M, QUIRK C, et al.Joint language and translation modeling with recurrent neural networks[J].American Journal of Psychoanalysis,2013, 74(2): 212-213.
[17] RUFFINI G, IBAÑEZ D, CASTELLANO M,et al. EEG-driven RNN classification for prognosis of neurodegeneration in at-risk patients[C]//International Conference on Artificial Neural Networks, September 6-9,2016, Barcelona, Spain. Berlin: Springer,2016: 306-313.
[18] MIOT TO R, LI L, DUDLEY J T. Deep learning to predict patient future diseases from the electronic health records[M].Berlin: Springer International Publishing,2016.
[19] LIU L, TANG J, CHENG Y, et al. Mining diabetes complication and treatment patterns for clinical decision support[C]//The 22nd ACM international conference on Information & Knowledge Management,October 27 - November 1, 2013, San Francisco, USA. New York: ACM Press,2013: 279-288.
[20] HUANG Z, DONG W, BATH P, et al. On mining latent treatment patterns from electronic medical records[J]. Data Mining& Knowledge Discovery, 2015, 29(4):1-36.
[21] CHENG Y, WANG F, ZHANG P, et al.Risk prediction with electronic health records: a deep learning approach[C]//The 2016 SIAM International Conference on Data Mining, May 5-7, 2016, Miami,USA. [S.l.:s.n.], 2016: 432-440.
[22] S U T H E R L A N D S M, C H AW L A L S,KANEGILL S L, et al. Utilizing electronic health records to predict acute kidney injury risk and outcomes: workgroup statements from the 15th ADQI consensus conference[J]. Canadian Journal of Kidney Health and Disease, 2016, 3(1):1-14.
[23] WO LFS O N J, BANDYO PAD H YAY S,ELIDRISI M, et al. A naive Bayes machine learning approach to risk prediction using censored, time-to-event data[J]. Statistics in Medicine, 2011, 34(21): 2941-2957.
[24] SUTSKE VER I, VINYAL S O, LE Q V.Sequence to sequence learning with neural networks[C]//The 27th International Conference on Neural Information Processing Systems, December 8-13,2014, Montreal, Canada. New York: ACM Press, 2014: 3104-3112.
[25] ZHANG J, MANI I. kNN approach to unbalanced data distributions: a case study involving information extraction[C]//The ICML 2003 Workshop on Learning from Imbalanced Datasets, December 3-8,2003, Piscataway, USA. [S.l.:s.n.], 2003.
[26] TOMEK I. Two modifications of CNN[J].IEEE Transactions on Systems Man and Communications, 1976, SMC-6(11): 769-772.
[27] KUBAT M,MATWIN S. Addressing the course of imbalanced training sets: onesided selection[C]// The 14th International Conference on Machine Learning (ICML 1997), July 8-12, 1997, Nashville, USA.[S.l.:s.n.], 1997: 179-186.
[28] WILSOND L. Asymptotic properties of nearest neighbor rules using edited data[J].IEEE Transactions on Systems, Man,and Communications, 2007, SMC-2(3):408-421.
[29] LAURIKKALA J. Improving identification of difficult small classes by balancing class distribution[C]// Conference on Artificial Intelligence in Medicine in Europe, July 1-4,2001, Cascais, Portugal. Berlin: Springer Berlin Heidelberg, 2001: 63-66.
[30] CHAWLAN V, BOWYER K W, HALL L O,et al. SMOTE: synthetic minority oversampling technique[J]. Journal of Artificial Intelligence Research, 2002(16): 321-357.
[31] ROSENBAUM P R, RUBIN D B. The central role of the propensity score in observational studies for causal effects[J].Biometrika, 1983, 70(1): 41-55.
[32] SOBEL M E. Causal inference in the social and behavioral sciences[M]//Handbook of Statistical Modeling for the Social and Behavioral Sciences. New York: Springer US, 1995: 1-38.
[33] HOLLAND P W. Statistics and causal inference[J]. Journal of the American Statistical Association, 1986, 81(396):945-960.
Process and methods of clinical big data mining based on electronic medical records
RUAN Tong1, GAO Ju2, FENG Donglei3, QIAN Xiyuan1, WANG Ting1, SUN Chenglin1
1. East China University of Science and Technology, Shanghai 200237, China 2. Shanghai Shuguang Hospital, Shanghai 200025, China 3. Wonders Information System Co. Ltd., Shanghai 200040, China
Electronic medical records from hospitals record the patient's disease, diagnosis and treatment information. It forms the basis of clinical data. Mining such data can assist doctors in clinical research and clinical diagnosis and treatment. Firstly,challenges encountered in the process of big data mining on EMR were raised, then the core process was summarized.The process includes tasks such as clinical data integration, the construction of clinical specialist disease database based on knowledge graph, the quality assessment methods on EMR, and comparative effectiveness and risk prediction of diseases as the core of clinical big data applications. A solution for each task was proposed, and the experimental results were given.Finally, the future directions of technologies and applications of big data mining on healthcare were presented.
medical knowledge graph, clinical specialist disease database, evaluation of data quality, electronic medical record, risk prediction of diseases, comparative effectireness
s: The National High Technology Research and Development Program of China (863 Program)(No.2015AA020107), National Key Technology Research and Development Program of the Ministry of Science and Technology of China (No.2015BAH12F01-05)






TP311.13
A
10.11959/j.issn.2096-0271.2017054
阮彤(1973-),女,博士,华东理工大学计算机技术研究所教授、所长,自然语言处理与大数据挖掘实验室主任,主要研究方向为文本抽取、知识图谱、数据质量评估等。
作者简介
高炬(1966-),男,上海曙光医院副院长、主任医师,主要研究方向为医院行政管理及中西医结合肝胆病研究。
冯东雷(1972-),男,博士,万达信息股份有限公司教授级高级工程师,主要研究方向为健康医疗大数据+人工智能、健康医疗+互联网、区域人口健康信息化、卫生信息标准化等。
钱夕元(1968-),男,博士,华东理工大学教授,主要研究方向为统计计算、数值软件等。
王婷(1993-),女,华东理工大学硕士生,主要研究方向为知识图谱、信息抽取。
孙程琳(1993-)女,华东理工大学硕士生,主要研究方向为知识图谱、问答系统。
2017-06-07
国家高技术研究发展计划(“863”计划)基金资助项目(No.2015AA020107);国家科技支撑基金资助项目(No.2015BAH12 F01-05)

