小桐子转化酶基因家族的生物信息学分析及表达特性
王海波 辛胡 刘潮 唐利洲
(曲靖师范学院 云南高原生物资源保护与利用研究中心 云南省高校云贵高原动植物遗传多样性及生态适应性重点实验室,曲靖 655011)
小桐子转化酶基因家族的生物信息学分析及表达特性
王海波 辛胡 刘潮 唐利洲
(曲靖师范学院 云南高原生物资源保护与利用研究中心 云南省高校云贵高原动植物遗传多样性及生态适应性重点实验室,曲靖 655011)
转化酶是植物蔗糖代谢与抗逆性形成的关键酶。基于小桐子基因组数据,鉴定到16个小桐子转化酶基因,并对其系统进化、基因结构、理化性质、保守基序及表达特性进行了分析。结果表明,小桐子转化酶基因家族聚类为3个亚家族,中性/碱性转化酶α亚组与酸性转化酶基因包含6或7个外显子,β亚组包含4或5个外显子。同时,酸性转化酶N端都包含信号肽序列,且都鉴定到GH32家族保守的-NDPNG/A-与-MWECV/P-基序。qRT-PCR表达分析显示,JcC-INVD基因在小桐子子叶中表达量最高,而在根中表达量较低,且在叶中属于低温诱导基因。
小桐子;转化酶;基因家族;表达分析
蔗糖是植物体内光合产物由源器官如叶片向库器官如果实、块茎、种子等运输的主要形式,在源库关系调节、糖分组成调控、胁迫响应及己糖信号转导等方面发挥重要作用。在植物体内,蔗糖通过蔗糖酶/β-呋喃果糖苷酶(Sucrase/βfructofuranosidase,即转化酶Invertase,INV,EC3.2. 1.26)与蔗糖合成酶(Sucrose synthase,SS,EC2.4. 1.13)催化分解生成葡萄糖与果糖,被库器官直接利用,另外还可以通过浓度效应[1]或己糖激酶(Hexokinase)磷酸化产生信号[2,3]进而调控一系列基因的表达。按照亚细胞定位,转化酶可分为细胞质转化酶(Cytoplasm invertase,C-INV/CINV/SNAI)、细胞壁转化酶(Cell wall invertase,CW-INV/cwINV/ CWI)及液泡转化酶(Vacuole invertase,V-INV/VI-NV/SAI)[4]。又根据低离子强度缓冲液中的溶解性,分为酸性转化酶(Acidic invertase,EC3.2.1.26)与中性/碱性转化酶(Neutral/alkaline invertase,EC3. 2.1.27),前者包括细胞壁转化酶(不溶或可溶性的,最适pH值3.5-5.0之间)与液泡转化酶(可溶性的,最适pH值5.0-5.5之间),且都属于糖基化蛋白,不仅可以催化蔗糖的水解,还可以催化寡聚糖如棉子糖、水苏糖等的水解[5],同时活性受到重金属离子如Cu2+、Ag+、Hg2+、Co2+等的抑制作用;后者主要指细胞质转化酶(可溶性的,最适pH值6.8-8.0之间),属于单纯蛋白,具有蔗糖水解特异性[6,7],其活性不受重金属离子的抑制。另外研究发现中性/碱性转化酶还存在于叶绿体、线粒体、淀粉体及细胞核中[8-11]。
第一个被克隆的植物转化酶基因是胡萝卜(Daucus carota)细胞壁转化酶基因[4]。随着多种植物基因组测序的完成,更有利于从全基因组范围内对转化酶基因家族进行分子结构、进化特性及表达调控等的深入研究。酸性转化酶(GH32家族,Glycoside hydrolase family 32)与碱性/中性转化酶(GH100家族)由不同的基因家族编码[12],且不同的植物其基因数量也不同,如拟南芥细胞质转化酶基因、细胞壁转化酶基因、液泡转化酶基因分别有9、6、2个,而水稻对应分别有8、9、2个。小桐子属大戟科(Euphorbiaceae)能源植物,可生产清洁生物质柴油,兼具良好的经济与生态效益。对于小桐子转化酶基因家族的鉴定及分析还未见报道。本研究基于小桐子基因组数据库,利用生物信息学方法鉴定到16个小桐子转化酶基因,并对其理化性质、基因结构、系统进化及差异表达进行了分析,以期为研究小桐子转化酶基因的生物学特性、克隆转化、及功能验证提供理论依据。
1 材料与方法
1.1 材料
1.1.1 实验材料及处理 选取饱满的小桐子种子(云南省楚雄州元谋县),按照李忠光的方法[13]进行发芽处理,将发芽的种子播于消毒的培养土中并于恒温培养箱(温度26℃、相对湿度75%、光周期16/8 h)中生长15 d至第二片真叶展开,每天用无菌水润湿培养土。将生长15 d的小桐子幼苗置于12℃、相对湿度75%、16/8 h光周期的低温培养箱中进行低温胁迫处理,分别取低温胁迫0.5、3、12、24 h与对照(正常培养)的第二片真叶、子叶、茎及根,用铝箔纸包好,液氮速冻后保存于-80℃冰箱中用于RNA的提取。
1.1.2 实验试剂与仪器 RNA提取试剂盒TransZol Up、反转录试剂盒TransScript Two-Step RT-PCR SuperMix、实时荧光定量PCR试剂盒TransStart Top Green qPCR SuperMix购自北京全式金生物技术有限公司;DNase I购自天根生化科技有限公司;其余试剂为进口或国产分析纯。引物合成由深圳华大基因有限公司完成。实时荧光定量PCR仪型号为Roche Lightcycler 96。
1.2 方法
1.2.1 小桐子转化酶基因家族的鉴定 从GenBank下载小桐子最新注释蛋白质数据库(http://www. ncbi.nlm.nih.gov/genome/915/),根据模式植物拟南芥数据库(http://www.arabidopsis.org/)中已经鉴定出的17个转化酶基因的蛋白质序列(细胞质转化酶At1g56560、At4g34860、At3g06500、At1g22650、At5g22510、At1g7200、At1g35580、At3g05820、At4g09510;细胞壁转化酶At3g13790、At3g52600、At1g55120、At2g36190、At3g13784、At5g11920;液泡转化酶At1g62660、At1g12240),利用NCBI Blast程序对小桐子蛋白质数据库进行本地BlastP相似性比对(阈值E<1e-10,序列相似性>50%),得到候选的小桐子转化酶蛋白质序列,并手工去除重复序列。将非冗余的候选序列利用Pfam与CCD在线工具分析转化酶结构域,得到小桐子转化酶家族蛋白序列。
1.2.2 小桐子转化酶基因家族序列分析 利用ExPaSy提供的在线工具ProtParam(http://web.expasy. org/protparam/)对得到的小桐子转化酶蛋白质序列进行基本性质的分析。利用获得的小桐子转化酶家族蛋白序列对GenBank小桐子基因组数据库(Annotation release 100,JatCur_1.0)进行tblastn相似性检索得到其基因与CDS(Coding sequence)序列,并利用GSDS(Gene Structure Display Server,http://gsds.cbi.pku.edu.cn/)绘制基因结构图。根据CDS序列得到小桐子各转化酶基因起始密码子ATG上游1 500 bp的调控序列,并通过PlantCARE工具对其顺式作用元件进行鉴定。将鉴定的小桐子转化酶蛋白序列利用ClustalX(Version2.0)进行序列相似性比对,然后用MEGA6.0软件通过邻接法(NJ)构建系统进化树,并采用自展法(Bootstrap)进行检验。
1.2.3 小桐子JcC-INVD转化酶基因的表达分析 利用TransZol Up提取各材料的总RNA,并利用DNase I消化RNA中的残余基因组DNA,得到纯化的总RNA。分别取3 μg总RNA,利用TransScript Two-Step RT-PCR SuperMix合成第一链cDNA,以18S rRNA为内参(GenBank登录号:AY823528),利用TransStart Top Green qPCR SuperMix进行小桐子转化酶C-INVD基因的qRT-PCR扩增,20 μL反应体系,每个样品重复3次。JcC-INVD基因扩增引物序列JcC-INVD_F(5'-CGGATTGGGTATTTG-3'),JcC-INVD_R(5'-TCTGGTATTCTTGGGAT-3');内参基因18S rRNA扩增引物序列18S rRNA_F(5'-AGAAACGGCTACCACATC-3'),18S rRNA_R(5'-CCAAGGTCCAACTACGAG-3')。扩增条件为:94℃预变性30 s;94℃变性10 s,55℃退火15 s,72℃延伸15 s,45个循环,之后增加溶解曲线程序:95℃ 10 s,65℃ 60 s,97℃ 1 s,连续检测信号。采用2-ΔΔCt法进行相对表达量分析。
2 结果
2.1 小桐子转化酶基因家族的鉴定及系统进化分析
以拟南芥17个转化酶蛋白质序列为种子序列对小桐子蛋白质数据库进行BlastP相似性检索,结合结构域分析共鉴定到16个小桐子转化酶,其中包括细胞质转化酶8个、细胞壁转化酶6个及液泡转化酶2个(表1)。进化树分析(图1)显示,16个小桐子转化酶聚类为两大家族,即酸性转化酶家族(GH32家族,8个)与中性/碱性转化酶家族(GH100家族,8个),酸性转化酶家族又聚类为细胞壁转化酶(Jccw-INV1-Jccw-INV6)与液泡转化酶(JcV-INV1与JcV-INV2)两个分支,而中性/碱性转化酶(细胞质型转化酶)家族也分支为2个亚组(α亚组与β亚组),而且与细胞质型转化酶的亚细胞定位一致,其中,α1亚组包括JcC-INVA与JcC-INVC,定位线粒体;α2亚组包括JcC-INVE与JcC-INVH,定位叶绿体;β亚组包括JcC-INVB、JcC-INVD、JcC-INVF及JcC-INVG,定位细胞质。
同时,利用基因组数据库得到小桐子转化酶蛋白序列对应的CDS序列与基因序列,并利用GSDS绘制其基因结构(图1)。与进化树对比分析表明,6个小桐子细胞壁转化酶基因包含6(Jccw-INV2、Jccw-INV3、Jccw-INV4、Jccw-INV5,除Jccw-INV5都不包含5'-UTR与3'-UTR区)或7(Jccw-INV1与Jccw-INV6,都包含5'-UTR与3'-UTR区)个外显子,与进化树中细胞壁转化酶聚类为2个分支一致,2个液泡转化酶都包含7个外显子,且都包含5'-UTR与3'-UTR区。另外,8个酸性转化酶基因都包含该家族中典型的9 bp外显子(第2个外显子),编码3个氨基酸残基-DPN-,参与构成植物酸性转化酶保守基序Motif1(-NDPNG/A-)。在细胞质型转化酶家族中,α1亚组都包含6个外显子,而α2亚组都包含7个外显子,且都包含5'-UTR与3'-UTR区;β亚组中,JcC-INVB与JcC-INVD都包含5个外显子且都包含5'-UTR与3'-UTR区,而JcC-INVF与JcCINVG都包含4个外显子,但都不包含5'-UTR与3'-UTR区(表1)。
2.2 小桐子转化酶的理化性质及结构特征分析
小桐子16个转化酶蛋白单体序列长度分布在560-735 aa之间,其中酸性转化酶氨基酸长度变化较小,而中性/碱性转化酶氨基酸长度差异较大。等电点分布在4.85-9.40之间,其中细胞壁转化酶除Jccw-INV6外都偏碱性,液泡转化酶都偏酸性,而中性/碱性转化酶变化差异较大(表1)。通过SignalP分析显示,小桐子中性/碱性转化酶都不含信号肽,而细胞壁转化酶在N端都含有一段约21-25 aa的信号肽,液泡转化酶则在N端含有一段101 aa(JcVINV1)与129 aa(JcV-INV2)的长信号肽,具有辅助转化酶前体蛋白运输及靶标识别的作用。
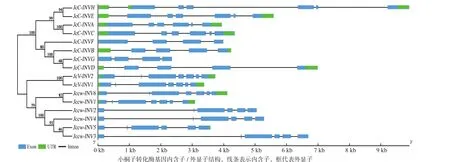
图1 小桐子转化酶基因家族的进化与基因结构分析
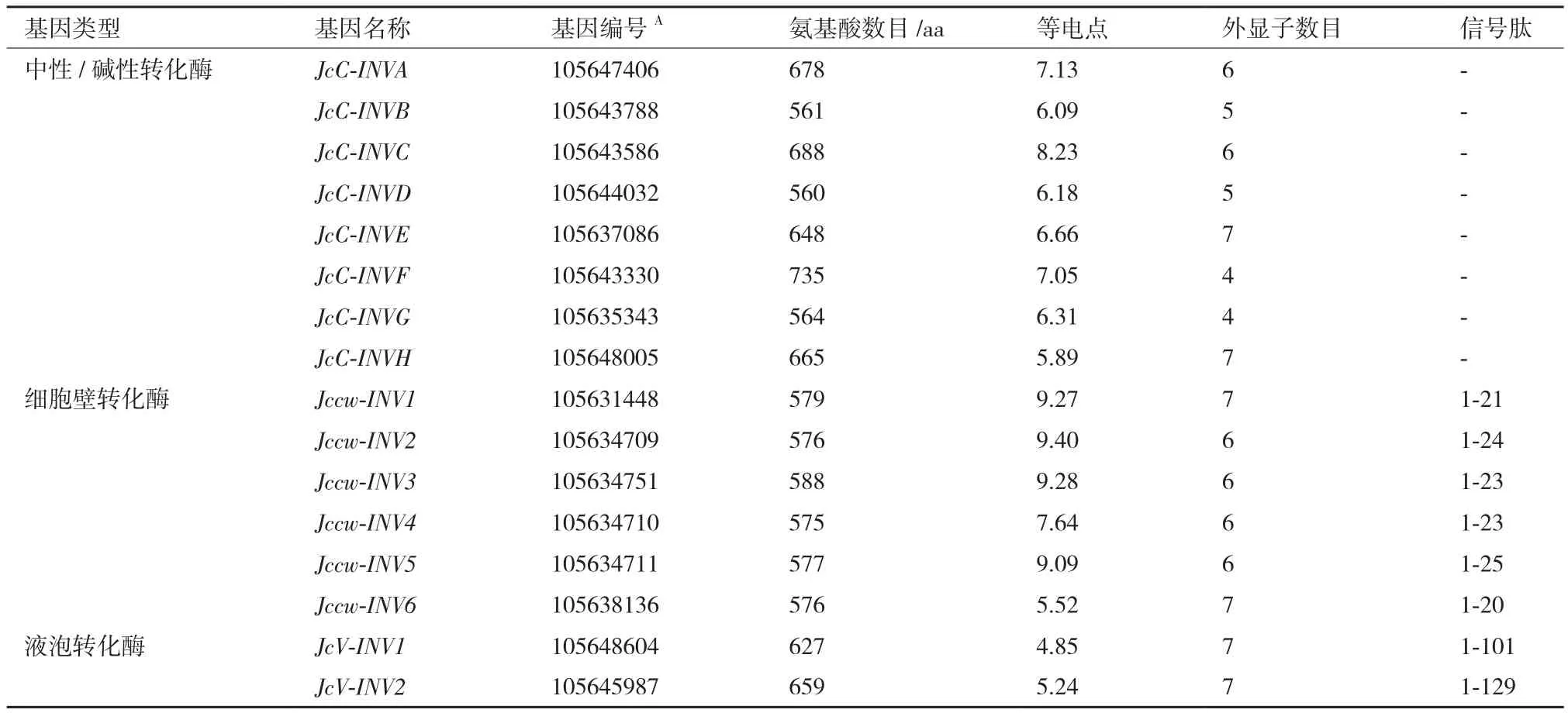
表1 小桐子转化酶蛋白的理化性质分析
通过GenBank结构域分析显示,在小桐子8个酸性转化酶中都鉴定到GH32家族结构域(以Jccw-INV4为例见图2)。同时,对小桐子酸性转化酶进行多序列比对发现,细胞壁转化酶与液泡转化酶在N端都包含信号肽,但序列相似性较低。另外,小桐子酸性转化酶活性中心主要由保守基序Motif1(-NDPNG/A-)与Motif2(-MWECV/P-)及催化必需氨基酸残基(Asp)组成,催化中心则由核心氨基酸残基-DPN/D-组成,而在Motif2中的V/P残基则决定了小桐子酸性转化酶的最适pH值及催化底物的特异性(图2)。2.3 小桐子转化酶JcC-INVD基因的表达分析

图2 小桐子酸性转化酶多重序列比对及保守结构域
文献报道,转化酶是与植物抗逆性密切相关的基因家族,多种非生物胁迫可以诱导不同类型转化酶基因的表达。利用PlantCARE工具对小桐子定位细胞质的中性/碱性转化酶JcC-INVD基因上游调控序列进行了顺式作用元件的鉴定,结果表明,其启动子中存在脱落酸、茉莉酸甲酯、水杨酸、生长素、高温及干旱等调控元件,说明转化酶在调节小桐子蔗糖运输与代谢、抵抗非生物环境胁迫等方面起重要的作用。以18S rRNA为内参基因,对小桐子JcCINVD基因在不同组织与12℃低温胁迫下的表达情况进行了qRT-PCR分析,结果(图3)表明,JcCINVD基因在小桐子各组织中都有表达,其中在子叶中表达量最高,其次是叶片,而在根中表达量较低。另外,随着低温处理时间的延长,JcC-INVD在叶片中的表达量显著增加,并在3 h时达到最大表达量,较对照提高19.03倍,之后逐渐降低。说明小桐子JcC-INVD基因也是低温诱导基因,与小桐子的抗冷性形成直接相关。
3 讨论

图3 小桐子JcC-INVD转化酶基因的差异表达分析
根据高等植物酸性转化酶基因的结构分析表明,大部分都属于6/7外显子基因构型[5],其中第2个外显子仅9 bp,编码3个氨基酸(DPN),属呋喃果糖苷酶及酸性转化酶的保守区-NDPNG/A-[14,15],与第3个外显子编码的催化位点-MWECV/P-(液泡转化酶为V、细胞壁转化酶为P)共同稳定并维持酸性转化酶的催化活性[5,7,16,17],而小桐子液泡转化酶JcV-INV1的第2个外显子却编码DPD,根据GenBank结构域分析显示该氨基酸残基并不属于催化必需氨基酸,因而推测JcV-INV1中N(天冬酰胺)由D(天冬氨酸)取代并不影响其催化活性。
植物转化酶基因家族的进化历程可以通过内共生学说解释[18],Sturm等[4]推测植物酸性转化酶来自于呼吸真菌与好氧细菌,细胞壁转化酶基因首先通过内吞好氧细菌进入植物细胞,之后发生突变在N端增加了一段信号肽序列使其定位于液泡形成液泡转化酶[16,19],而中性/碱性转化酶则来源于20-35亿年前蓝藻中形成的一个原始基因,且在内共生事件发生之前,蓝藻中性/碱性转化酶基因就已经进化形成α与β组[20,21]。Bocock等[14]、Ji等[18]、Fridman等[22]根据转化酶的基因结构、保守区位置、染色体的定位及转化酶基因上下游基因的类型等特征对毛果杨、水稻、番茄、马铃薯及拟南芥的转化酶基因家族进化历程进行分析也证明转化酶基因是由一个共同的原始基因通过多次的基因丢失与复制进化而来的。同样规律,小桐子细胞壁转化酶中Jccw-INV2-Jccw-INV4-Jccw-INV5-Jccw-INV3按顺序串联在一起,而两个液泡转化酶的上游均含有Trehalose-phosphate phosphatase的ORF,下游均含有Monocopper oxidase的ORF,与Bocock等[14]对毛果杨转化酶基因结构分析相似。
Liao等[23]对竹子转化酶基因启动子序列分析发现包含大量生物与非生物响应元件,显示转化酶广泛参与植物多种抗逆性过程,如赤霉素处理降低甜菜液泡转化酶BVInv-V3基因的表达[24];番茄受到真菌浸染细胞壁转化酶IncW1表达量上调[25];低温胁迫降低水稻OsINV4的表达从而导致花粉败育[26]。本研究中小桐子中性/碱性转化酶JcC-INVD基因在其调控序列中并没有鉴定到低温调控元件,但通过qRT-PCR分析显示该基因受低温诱导显著,说明其参与小桐子抗冷性的形成过程或信号转导过程。
4 结论
本研究基于小桐子基因组数据库鉴定到16个小桐子转化酶基因家族成员,聚类为3个亚家族,其中,细胞质转化酶、细胞壁转化酶及液泡转化酶分别为8、6、2个。实时荧光定量PCR表达分析显示,JcC-INVD基因在小桐子各组织中存在表达特异性,在叶中属于低温诱导基因,可以作为抗冷小桐子新品种选育的候选基因。
[1]Moore BD, Sheen J. Plant sugar sensing and signaling a complex reality[J]. Trends Plant Sci, 1999, 4(7):250-251.
[2]Sheen J, Zhou L, Jiang JC. Sugar as signaling moleculaes[J]. Curr Opin Plant Biol, 1999, 2(5):410-418.
[3]Rook F, Bevan MW. Genetic approaches to understanding sugarresponse pathways[J]. J Exp Bot, 2003, 54(382):495-501.
[4]Sturm A, Chrispeeels M. cDNA cloning of carrot extracellular β-fructosidase and its expression in response to wounding and bacterial infection[J]. Plant Cell, 1990, 2(11):1107-1119.
[5]Roitsch T, Gonzalez MC. Function and regulation of plant invertase:sweet sensations[J]. Trends Plant Sci, 2004, 9(12):606-613.
[6]Sturm A. Invertase:primary structures, functions and roles in plant development and sucrose partitioning[J]. Plant Physiol, 1999, 121(1):1-7.
[7]Hsiech CW, Liu LK, Yeh SH, et al. Molecular cloning and functional identification of invertase isozymes from green bamboo Bambusa oldhamii[J]. J Agr Food Chem, 2006, 54(8):3101-3107.
[8]Gerrits N, Turk SC, van Dun KPM, et al. Sucrose metabolism in plastides[J]. Plant Physiol, 2001, 125(2):926-934.
[9]Lou Y, Gou JY, Xue HW. PIP5K9, an Arabidopsis phosphatidylinositol monophosphate kinase, interacts with a cytosolic invertase to negatively regulate sugar-mediated root growth[J]. The Plant Cell, 2007, 19(1):163-181.
[10]Murayama S, Handa H. Genes for alkaline/neutral invertase in rice:alkaline/neutral invertases are located in plant mitochondria and also in plastids[J]. Planta, 2007, 225(5):1193-1203.
[11]Maruta T, Otori K, Tabuchi T, et al. New insights into the regulation of greening and carbon-nitrogen balance by sugar metabolismthrough a plastidic invertase[J]. Plant Signal Behav, 2010, 5(9):1131-1133.
[12]Hothorn M, Van den Ende W, Lammens W, et al. Structural insights into the pH-controlled targeting of plant cell-wall invertase by a specific inhibitor protein[J]. Proc Natl Acad Sci USA, 2010, 107(40):17427-17432.
[13]李忠光, 龚明. 不同化学消毒剂对小桐子种子萌发和幼苗生长的影响[J]. 种子, 2010, 30(2):4-7, 12.
[14]Bocock P, Morse A, Dervinis C, et al. Evolution and diversity of invertase genes in Populus trichocarpa[J]. Plant, 2008, 227(3):565-576.
[15]Nonis A, Ruperti B, Pierasco A, et al. Neutral invertase in grapevine and comparative analysis with Arabidopsis, poplar and rice[J]. Planta, 2008, 229(1):129-142.
[16]Goetz M, Roitsch T. The different pH optima and substrate specificities of extracellular and vacuolar invertases from plants are determined by a single amino acid substitution[J]. Plant Journal, 1999, 20(6):707-711.
[17]Ruan YL, Jin Y, Yang YJ, et al. Sugar input, metabolism, and signaling mediated by invertase:roles in development, yield potential, and response to drought and heat[J]. Mol Plant, 2010, 3(6):942-955.
[18]Ji X, Van den Ende W, Van Laere A, et al. Structure, evolution, and expression of the two invertase gene families of rice[J]. J Mol Evol, 2005, 60(5):615-634.
[19]Nielsen H, Engelbrecht J, Brunak S, et al. Identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage sites[J]. Protein Eng, 1997, 10(1):1-6.
[20]Vargas WA, Cumino A, Salerno GL. Cyanobacterial alkaline/neutral invertase. Origin of sucrose hydrolysis in the plant cytosol?[J]. Planta, 2003, 216(6):951-960.
[21]Vargas WA, Salerno GL. The cinderella story of scurose hydrolysis:alkaline/netural invertases, from cyanobacteria to unforeseen roles in plant cytosol and organelles[J]. Plant Sci, 2010, 178(1):1-8.
[22]Fridman E, Zamir D. Functional divergence of a syntenic invertase gene family in tomato, potato and Arabidopsis[J]. Plant Physiol, 2003, 131(2):603-609.
[23]Liao SC, Lin CS, Wang AY, et al. Differential expression of genes encoding acid invertases in multiple shoots of bamboo in response to various phytohormones and environmental factors[J]. J Agric Food Chem, 2013, 61(18):4396-4405.
[24]Gonzalez C, Cejudoand FJ. Gibberellin-regulated expression of neutral and vacuolar invertase genes in petioles of sugar beet plants[J]. Plant Science, 2007, 172(4):839-846.
[25]Schaarshmidt S, Roitsch T, Hanuse B. Arbuscular mycorrhiza induces gene expression of the apoplastic invertase LIN6 in tomato(Lycopersicon esculentum)roots[J]. J Exp Bot, 2006, 57(15):4015-4023.
[26]Oliver SN, Van Dongen JT, Alfred SC, et al. Cold-induced repression of the rice anther-specific cell wall invertase gene OsINV4 is correlated with sucrose accumulation and pollen sterility[J]. Plant, 2005, 28(12):1534-1551.
(责任编辑 朱琳峰)
Genome-wide Identification and Expression Characteristics of Invertase Gene Family in Jatropha curcas
WANG Hai-bo XIN Hu LIU Chao TANG Li-zhou
(Center for Yunnan Plateau Biological Resources Protection and Utilization,Key Laboratory of Yunnan Province Universities of the Diversity and Ecological Adaptive Evolution for Animals and Plants on Yungui Plateau,Qujing Normal University,Qujing 655011)
Plant invertase is one of critical enzymes in sucrose metabolism and stress resistance formation. The 16 invertase genes were identified based on complete genome data of Jatropha curcas,then the evolutionary relationship,gene structure,physical and chemical characteristics,conserved motif,and expression feature were analyzed. The results showed that invertase gene family in J. curcas was clustered to three subfamilies according to phylogenetic analysis. The α subgroup of neutral/alkaline invertases and acidic invertase gene owned 6-7 exons,and β subgroup owned 4-5 exons in gene structure. Signal peptides in N-terminal and conserved motifs of -NDPNG/A- and -MWECV/P-belonging to GH32 family were identified in acidic invertase protein sequences. qRT-PCR analysis revealed that JcC-INVD expressed differently in different tissues,with abundantly in cotyledon,but scarcely in root,and remarkably cold-induced expression in leaf.
Jatropha curcas;invertase;gene family;expression analysis
10.13560/j.cnki.biotech.bull.1985.2017-0303
2017-04-14
国家自然科学基金项目(31460179)
王海波,男,博士,副教授,硕士生导师,研究方向:植物逆境分子生物学;E-mail:bocai0406@163.com

