基于随机森林算法的小鼠micro- CT影像中骨骼关节特征点定位
屠睿博 陈中华 王洪凯
(大连理工大学生物医学工程系,辽宁 大连 116024)
基于随机森林算法的小鼠micro- CT影像中骨骼关节特征点定位
屠睿博 陈中华 王洪凯#*
(大连理工大学生物医学工程系,辽宁 大连 116024)
随着小动物成像技术的发展,技术人员每天需要处理的小动物影像数量急剧增长,这使得自动化的小动物图像分析方法成为迫切的需求。在小鼠图像分析方面,小鼠灵活多变的身体姿态给自动化的图像分析带来困难。基于随机森林算法实现小鼠micro- CT图像中骨骼关节点的自动定位,为解决小鼠影像中身体姿态的自动识别打下基础。该算法主要分3步:先通过分类随机森林算法得到小鼠骨骼关节点的粗定位,再通过回归随机森林算法进一步减小定位误差,最后通过图匹配的方法在备选点中挑选正确位置上的关节点。对49例不同身体姿态的小鼠全身三维micro- CT图像进行测试,全身关节点定位的成功率为98.27%,定位误差的中值为0.68 mm。同时验证联合使用分类与回归随机森林的必要性,并探究训练数据的数量对不同骨关节的识别效果的影响。研究为小鼠micro- CT影像中身体姿态的识别提供一种新方法,为后续的自动化图像配准、图像分割以及自动化图像测量提供重要的定位信息。
小动物影像分析; 骨关节点定位; 随机森林; 模式识别; 显微CT
引言
在癌症研究和新药试制的临床阶段,小动物成像起着重要的作用。近些年来,随着小动物成像研究的增多和小动物成像设备的发展[1- 4],小动物影像的数量剧烈增长。与此同时,研究人员处理分析小动物影像的压力也随之增大,不得不每天从事繁冗、单一的基本操作。因此,对小动物影像自动分析系统的需求愈加迫切。在小鼠全身影像的自动分析算法中,数字解剖图谱的全身配准是最基础的一步。通过配准,图谱中包含的丰富的器官解剖结构信息可被映射到个体图像中,从而为后续的精确器官分割以及各种影像参数的测量打下基础。
目前,阻碍小鼠图谱与个体影像精确配准的最大障碍之一,就是图谱与个体之间的姿态差异。由于小鼠的骨骼关节灵活,不同个体之间姿态差异可以很大。为解决这一问题,研究人员尝试了多种方案,主要可分为3类:非线性变形配准、活动关节配准、统计形状配准。Kesner等通过人工定义PET影像中的特征点实现图谱与PET影像的配准是一种非线性变形配准的方法[5],但是非线性变形在姿态差异较大情况下容易导致器官和骨骼的扭曲变形。Baiker等通过定义小鼠活动骨骼图谱的方式,在一定程度上克服了小鼠姿态变化对配准的影响[6- 8],但是仍然无法自动配准较大的姿态差异。笔者前期研究通过统计训练数据形状进行建模,实现对身体姿势和器官形态同时有差异图像的配准[9- 10],但是这种统计形状模型只适合配准训练样本集所包含的身体姿态。
为了解决大幅度自由姿态变形的小鼠图谱配准问题,骨关节点的定位至关重要。近年来,随机森林算法在医学影像和人体表面扫描影像中实现快速准确的关节定位。Shotton使用分类随机森林算法和Mean- Shift聚类方法在人类体表扫描深度图中预测关节点位置,由随机森林算法得到全身关节点的类别判断[11],Mean- shift算法实现最后关节点的筛选;由于体表扫描影像的获取较为便利,而且他们还通过人体数字模型模拟方式产生巨大的训练数据,所以可以提供充足的训练数据。这样庞大的数据量,对于临床CT图像的收集是不可能的。不同于体表扫描的深度图,CT图像数据的干扰因素也很多,比如设备的差异带来像素分布范围和图像分辨率的不同。因此在CT图像数据上使用随机森林算法,后期对分类结果的进一步处理是必要的。Donner等针对CT图像,在随机森林算法得到的定位基础上,进一步根据相邻关节点之间的相对位置关系来最终确定每个关节点的最优位置[12],在人体CT影像的骨骼关节定位方面取得了良好的效果。
在小动物成像领域,微型断层成像(micro- computed tomography, micro- CT)技术具有高分辨率、方便监测、易于建立多模态影像、提供精确的解剖信息等优点[13], 因此获得了广泛的应用。在小鼠的micro- CT影像中,骨组织的对比度好,包含较为清晰的全身关节图像特征。因此本文着重研究小鼠micro- CT影像中骨关节的自动识别问题。虽然在人体三维CT影像领域,骨关节的定位已经取得较好的效果,但是在小鼠的骨关节定位领域还未见相关的研究。本研究针对小鼠micro- CT影像,使用随机森林的分类算法首先得到对医学图像关节点的初步预判,随后使用回归随机森林对已有的分类结果进行优化,得到高精准度的关节点备选点,最后通过基于训练数据的结构图进行关节备选点的全局优化,在已有的关节识别结果中挑选最优的备选点。以下章节将先简要介绍随机森林算法的原理,然后重点介绍本研究方法,并给出实验结果和总结。
1 方法
1.1 随机森林算法简介
随机森林是由多颗决策树并联而成的集成分类系统,每一棵决策树都是一个能力较弱的模式识别分类器(简称“弱分类器”),而多棵树的集成则构成了一个强分类器[14]。
在20世纪80年代,早期决策树的研究工作主要在“分类回归树”(CART)[15]中体现,研究者则主要集中于使用已有数据构造最优的决策树。在20世纪90年代,学者们发现使用集成学习器(比如一般的弱习器)可以得到更高的准确率[16]。集成方法结合决策树便产生了决策森林,首先应用在手写数字的识别上[17]。20世纪90年代末,Breiman提出“随机森林”的概念[18],并且实现更广泛的使用。
为了解随机森林的工作原理,首先需要了解森林中每一棵决策树的原理。如图1所示,待识别数据的n维特征向量v从树状结构最顶层的根节点输入,并逐层向下走,每到达一个节点就根据该节点所保存的判别式进行一次判别,决定继续走向左下还是右下节点,直到到达最底层某个叶子节点(见图中方块节点),由最终所达到的叶子节点给出最终输出结果。图中虚线表示了特征向量从根节点到叶子节点所走过的一个可能的路径。
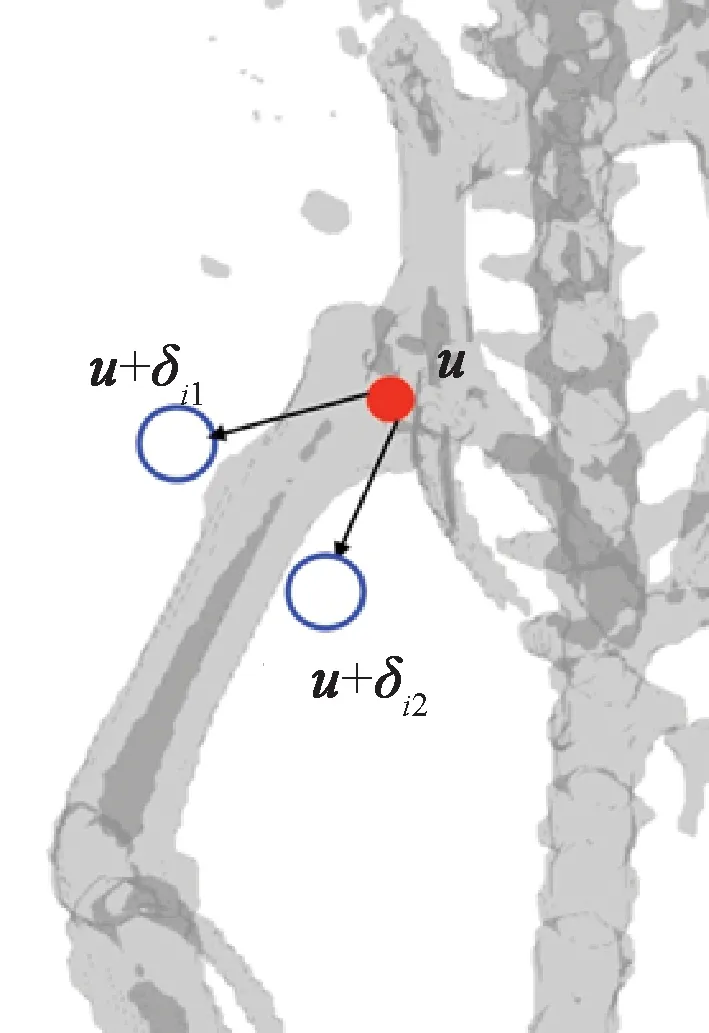
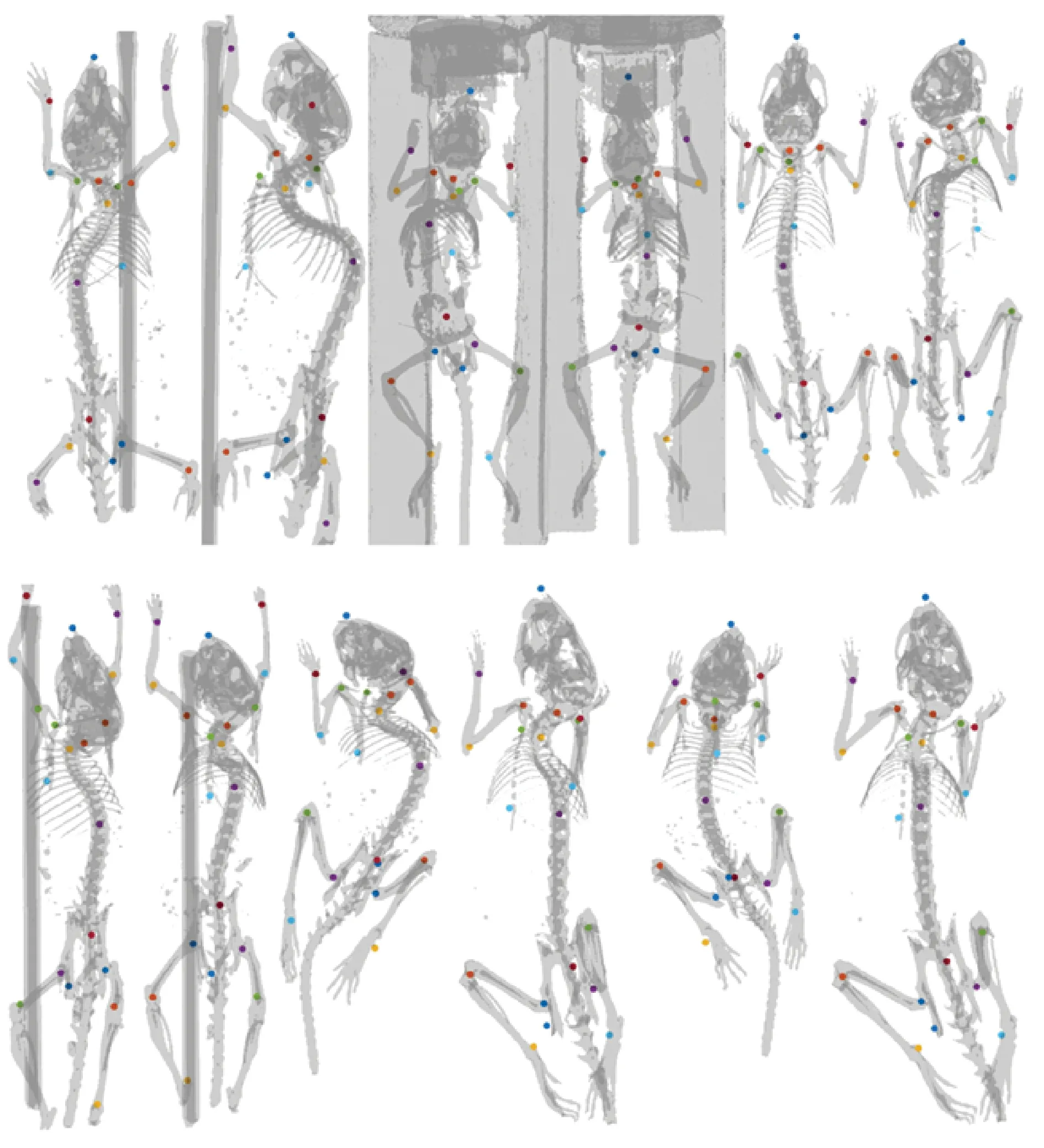
在树中每一个节点,判别过程如下:随机抽取v中的d个特征构成一个d维(d 图1 决策树原理Fig.1 The decision tree 根据应用目的的不同,随机森林可以分为分类森林(classification forest)和回归森林(regression forest)[19]。分类森林的作用是解决传统的模式分类问题,当输入特征到达决策树最底层的叶子节点后,以叶子节点中所保存的训练样本中最大概率类别作为分类的结果。回归森林则是为了解决函数拟合的问题,根据输入的特征向量v来预测一个连续的输出值y=f(v),整个森林就相当于是对函数f(v)的拟合;在回归森里中,当输入特征到达决策树最底层的叶子节点后,会以叶子节点中所保存的训练样本的条件概率函数来预测该叶子的y值。举例说明,在本研究的应用中,分类森林用来识别图像中的像素属于哪个关节点,而回归森林用来预测像素到关节点中心的偏移量。 分类森林与回归森林在决策树每个节点的判别原理上是基本相似的,只是叶子节点的输出原理不同。关于分类森林和回归森林的详细介绍推荐参考文献[20]。对于这两类随机森林的训练和测试方法,本研究均采用文献[20]所发布的sherwood C++函数库,因此在后面章节中将不做累述,本研究重点介绍针对小鼠micro- CT图像的算法设计。 1.2 算法总体流程 所处理的小鼠micro- CT影像都是将二维断层影像在三维空间中堆叠重构成的三维图像。处理的过程是逐个扫描三维图像的体素,对每个体素提取100维的特征向量作为分类森林和回归森林的输入。分类森林被用来识别每个像素位于哪个关节点附近,从而在每个关节点附近得到一群备选点组成的点云,并得到每个点属于每个关节的概率;进一步,回归森林会计算备选点云中每个点到关节中心的偏移量,更加精确地定位关节的中心位置;针对分类森林和回归森林可能产生错误识别的问题,用图匹配的方法、利用相邻关节之间的相对位置关系做约束,筛选掉可能产生的错误识别点,得到最终的定位结果。 本方法中的分类森林、回归森林和匹配图模型均通过有监督的训练得到。在训练阶段,每幅训练图像中的关节点位置由人类专家手工标定。如图2所示,算法分为测试阶段和训练阶段。训练数据是标记关节的小鼠三维CT图像,用于训练分类、回归随机森林和匹配图。测试阶段以小鼠的膝关节识别为例说明。先通过分类随机森林得到初步测试结果,再通过回归随机森林缩小识别结果范围,最后通过匹配图筛选正确的关节识别结果。 1.3 分类森林 分类随机森林算法是关节点定位的第一步,对小鼠全身micro- CT图像进行处理,预测每一个体素属于不同类别的概率,得到初步的类别概率预测结果。 1.3.1 分类森林的数据预处理 分类森林的训练数据是n幅不同身体姿态的小鼠全身CT图像,其中骨关节点的位置坐标已经被人为标记。为了满足训练正样本的数量,需要扩充每幅图像中关节标记点的个数。由于关节点的标记是人工标记的,并且关节是一定范围内存在的,所以扩充关节点标记的个数是有必要的。另外,为了准确识别关节并且增加算法的鲁棒性,也需要扩充每个关节的标记点。扩充正样本的方式是用已标记关节点为中心、边长为5个体素的正方体范围内的体素作为关节点标记点,这样每幅图像有20个关节,每个关节有125个标记点,由原来的每幅图像20个正样本扩充到2 500个正样本。 图2 算法的流程Fig.2 Algorithm Workflow 除了训练需要的正样本,算法需要提供负样本,即背景类。背景样本的采样方式有很多,这里只介绍其中的两种。第一种方式是背景采样,在去除标定的关节点后,对图像剩余的空间进行均匀采样,或者随机采样一定数量的负样本。第二种方式是在标记的关节点周围采样负样本点。注意,对于负样本采样的数量,应该遵循负样本类别的数量与正样本数量近似相等的原则。因为当负样本数量远大于正样本数量时,随机森林的训练将是一种不平衡训练,影响对各类别的预测。同时,若采用第二种负样本采样方式,负样本的采样空间应该距离正样本的标记空间一定的距离,以免出现对有歧义的空间进行标记,导致随机森林学习失败、分类能力减弱。 为了有效控制负样本的存在范围,采用简单的阈值分割得到骨骼,并对分割结果用半径为5 mm的球体算子做形态学膨胀,以膨胀后的结果为骨骼遮罩,从中提取负样本进行训练。在后期算法测试的过程中,也对测试图像生成同样的遮罩并从中寻找骨骼关节点,实验证明这种方法比全局采样得到负样本的方法识别结果敏感性要好。同时,骨骼遮罩的方式还有利于缩小关节点搜索范围,提升算法速度,并防止小鼠其他部位影响识别效果。 1.3.2 分类森林的训练 特征设计是随机森林的基础,特征的好坏直接决定后续识别能力的强弱。体素灰度值作为特征,这种简单的特征不仅可以提升算法的速度,而且由随机森林在计算机视觉中应用的经验得知,以体素或者像素值作为特征符合随机森林的学习习惯,可以得到很好的学习效率和效果。本研究采用随机森林图像分类算法中常用的双体素差值特征,如图3所示,对于一个体素的位置u,定义其特征为d维特征向量,有 (1) (2) 式中,δi1和δi2为随机产生的三维偏移向量,函数代表像素灰度值。 图3 双体素差值特征Fig.3 Double voxel difference feature 这种定义实际上是将体素u的第i维特征定义为其周围随机抽取的两个体素的灰度差值。这种差值可以摒除因为图像整体明暗偏差造成的影响。随机抽取的过程遵循高斯分布,其均值为0,方差为7个体素(实验尝试确定),共抽取100次,从而形成100维的特征向量v(u)。这里引入随机性,因为随机森林要求每一棵树要尽可能不同,这样可以避免学习得到的森林只能识别某一特定模式的图像,从而具备较强的鲁棒性,可以识别多种姿态小鼠CT图像的骨关节点。 最后,关于分类森林训练的最优化方法,采用训练数据划分后的信息增量作为判决条件,随机地计算若干次划分结果,并选取可以最大信息增益的参数作为本次训练的参数[20]。 1.3.3 分类森林的测试 由于分类森林的训练是基于骨骼遮罩,并提取了100维的特征向量,因此在测试过程中要使用相同的阈值提取骨骼遮罩,并遵照训练过程为每个节点确定的偏移量来提取100维像素特征。 1.4 回归森林 1.4.1 数据预处理 回归森林使用的原始训练数据同分类随机森林相同。由于回归随机森林的输出是连续的向量,所以在训练阶段需要给算法提供每个训练样本点指向关节中心点的偏移向量。所有训练数据是采用以关节点为中心,21为边长的正方体范围内的点,并以每个点指向关节点的三维向量为训练的输出。因此每幅图像20个关节,每个关节有9 261个训练数据,这要比之前的分类随机森林的训练数据要多,因此回归随机森林的训练耗时更长。与分类随机森林不同,回归随机森林不需要提供负样本的数量,因为不需要提供指向背景类的向量。 1.4.2 回归随机森林的训练 回归随机森林训练目的是根据图像的局部特征学习出某一位置指向关节点的向量。所采用的输入特征与分类随机森林相同,定义同式(1)、(2),同样是通过随机抽取形成100维的特征向量v(u)。在训练方法上,回归随机森林也是采用信息增量作为判决条件,它与分类随机森林的不同点主要体现在两个方面:其一,由于分类森林的输出是不连续的类别变量,而回归森林的输出是连续的实数变量,因此在训练每个节点的时候,分类森林采用离散的信息熵,而回归森林采用连续的信息熵;其二,在叶子节点的训练方式上,分类森林选取叶子节点中出现次数最多的训练样本类别作为输出类别,而回归森林则首先获取叶子节点中每个训练样本指向关节中心的位移向量,然后对这些向量拟合一个三维的条件高斯分布,以像素特征v(u)为条件,计算该条件下的高斯分布均值作为输出的位移向量。受篇幅所限,推荐读者参考文献[18]进一步了解算法细节。 本研究针对20个不同的关节点训练20个不同的森林,预测某一点到某一个关节的距离只需输入到相应的森林中进行预判。 1.4.3 回归随机森林的测试 在回归随机森林的测试阶段,需要分别预测每一个体素测试距离20种不同类别关节点的偏移向量。测试时,需要先统计分类森林结果中不同类别识别概率中的最大概率,只针对那些识别概率大于最大概率一半的点进行测试,从而摒除不必要的干扰点,减小计算量。 得到初步的偏移向量后,移动测试点以递归的方式继续进行回归测试。当递归的次数过少时由于偏移向量指示范围有限而导致误差较大,而当递归次数过多时,将会导致测试点的过度移动反而加大定位的误差。因此,本研究选用递归3次进行测试。得到的测试结果是每一个类别的备选点经过3次移动后的新位置。 1.5 使用图匹配方法筛选关节备选点 回归随机森林得到的结果是某一个关节点的多个备选点,需要进一步从这些备选点中挑选出一个最优点作为最终的定位结果,本研究使用基于随机场的图匹配方法来实现选取最优点。 1.5.1 匹配图的训练 若想在多个位置中挑选出一个关节点,除了考虑该点的识别概率,还应该考虑与它相邻的关节点的相对位置关系,因此需要根据训练数据学习出对某一关节点识别最有影响力的几个相邻关节点。 假设一共有N个关节点,每一个关节点有N-1个关节点与其相连,首先需要根据训练数据找到对每个关节点影响较大的几个关节点。倘若两个关节点在多幅训练数据中分布的相对位置关系是稳定的,则认为二者之间有较强的相互影响,其相对位置关系对于关节备选点的筛选十分有利。 关节点之间相对位置关系的稳定性可以通过熵的概念来描述,相对位置关系稳定的两个关节点之间的熵最小。在N幅训练数据中,第i幅图像的两个关节点s、t(s是待训练关节点,t是与s相邻关节点),二者的三维坐标分别是Isi和Iti,t相对于s的偏移向量是ΔIs,ti=Iti-Isi。由于训练数据的数目是有限的,为了以有限训练样本模拟无穷多样本中关节点之间的相对位置分布,假设每个训练样本都可扩充为无穷多个相似的样本,以三维向量ΔIs,ti为均值构造一个三维高斯分布,gs,ti=N(ΔIs,ti,σg)来代表s与t之间的相对位置分布,其中分布半径σg是一个经验参数(其取值对最终结果影响很小,本研究中设为2个像素)。对于N幅训练图像,会得到N个gs,ti高斯分布,对这N个分布的求均值并计算均值熵,即可作为对关节s与t之间相对位置稳定性的衡量,有 (3) 计算s同其他所有关节点之间的熵并排序,选出最小的前T个作为对s影响最大的关节点(本研究选取T=2),用于在后面的图匹配过程中帮助挑选关节s的最优备选点。 1.5.2 图匹配方法的测试 图匹配的作用是为每个关节点在多个备选点中挑选出最优的点作为最终结果,其挑选过程不仅利用了相邻关节点之间的相对位置关系作为先验知识,还利用了之前分类随机森林的每个备选点属于该关节的概率。图匹配算法的代价函数定义为 (4) 这个典型的随机场问题可以通过经典的图割方法[21]来求解,如果优化问题的规模足够小,也可以通过遍历所有组合的方式来求解。这里解释一下一元项和二元项的定义。一元项的计算是关节识别概率的信息量的计算,有 (5) (6) (7) (8) 在使用图割方法求解该随机场时,需要采用多标签的图割方法[22],这种方法采用迭代机制,通过多次双标签的图割运算来近似求得最优的多标签分割结果。在本研究中,骨骼关节l对应随机场中的节点,关节对e代表随机场中的边,式(4)中的一元项对应图割中的t- link,二元项对应图割中的n- link。由于每个关节具有多个备选点,因此所有关节的备选点可以组成一个备选点集∏;假设∏中共有K个点,则编号1~K对应了图割问题中的K个标签,从而将备选点选取问题转化成了多标签的图割问题。需要注意的是,如果某个备选点k是属于关节i的备选点,则须将k与其他关节之间的t- link值设为无穷大,以避免图割算法将k赋值给其他关节。 1.6 算法评价方法 本研究所采用的训练数据是49幅不同身体姿态的小鼠的全身micro- CT图像,采集自美国加州大学洛杉矶分校Crump分子影像中心。图像由256×256×496个体素构成,每个体素的尺寸为0.204 mm×0.203 mm×0.202 mm。笔者训练的分类森林包含50棵树,每棵树深度为15层;回归森林包含20棵树,深度为10层;以上参数通过实验尝试得到。被识别的骨骼关节点包括20种,分别是鼻尖、脊柱与头骨连接处、胸椎上端,胸椎下端、胸骨柄上端、胸骨柄下端、骨盆后延、骨盆前端结合处、左肩、左肘、左手、右肩、右肘、右手、左胯、左膝、左踝、右胯、右膝、右踝。 测试采用leave- one- out的交叉验证方式进行,即每测试一幅图像,就以另外48幅作为训练数据。对于被测试图像,人类专家的手工标记相当于衡量算法精度的金标准;对于训练图像,手工标记则是训练数据的一部分。 图4展示了多幅不同姿态的下小鼠骨骼关节定位结果。结果图以骨骼阈值分割结果的半透明三维面绘制的方式展现,突出小鼠的骨骼结构,算法给出的关节点以圆点的形式标出。部分图像中的CT床由于像素值较高,同骨骼一起被绘制出,但是我们的算法并没受到CT床的干扰。可以看出算法对不同姿态的小鼠均实现了较好的关节定位,图中第一行中间两个小鼠图像中的CT床没有被去除,第二行左侧两个图像没有小鼠的足部,但是这些因素都不影响小鼠关节的识别。 图4 多种姿态小鼠的关节识别结果。小鼠的骨骼以阈值分割的半透明三维面绘制方式展现,识别出的骨骼关节圆点形式标记图像上Fig.4 Bone joints localization results of various body postures. The skeletons are displayed as surface rendering of the threshold bones, the detected joints are marked as dots. 2.1 算法准确性分析 这里采用均值误差和中位数误差评估定位关节点的准确率。 图5展示算法对每个关节识别的均值误差和中位数误差,由于踝关节的训练数据和测试数据不足没有将踝关节加入考虑。定位误差定义为算法定位的位置与人类专家标记位置之间的欧氏距离。误差计算是基于49幅图像的测试结果统计得到,可以看到大部分关节的中值误差在0.8 mm以下,均值误差在1.0 mm以下,所有关节点的中值误差为0.63 mm,均值误差为0.97 mm。(中值误差为3.16个体素,均值误差为4.80个体素),说明本方法对于小鼠micro- CT图像骨关节有较好识别和定位能力。 图5 关节定位误差Fig.5 Bone joints localization errors 图5中误差较大的两个关节是左膝和右膝,主要原因并不是算法对这两个关节本身的识别能力弱,而是由于部分测试图像的踝关节缺失,会造成算法把一部分膝关节备选点识别为踝关节,使膝关节的识别精度受到影响。 2.2 算法识别成功率分析 这里首先定义识别失败,当某一关节点识别到其他关节的位置时记作识别失败,当识别的关节点距离正确位置与其他错误位置的距离相等或者大于到错误关节点的距离也记作识别失败。当识别的关节点在正确位置,以及相对于错误位置的距离远大于正确关节点的距离,定义为识别成功。 算法对绝大多数关节均能正确识别,唯一的错误识别来自少数缺少踝关节图像:当小鼠的脚没有被扫描到图像中,膝盖的识别会受到影响,因为膝盖和踝关节相关性比较高,在通过匹配图筛选关节点时,有踝关节可以帮助筛选膝关节的备选点。 如图6所示,横坐标为定义的关节各类别,识别成功率为成功识别的关节个数与同类关节全部参与识别的关节个数的比值。可以发现,由于部分数据的踝关节缺失的干扰,使膝关节的识别率也受到影响,但是单个关节的最低识别率仍达92%。综合考虑所有关节,一共49×20=980个关节,有17个错误识别的关节(包含识别错误的踝关节),占所有关节的1.73%,即全身关节点定位的成功率为98.27%。 图6 关节识别成功率Fig.6 Bone joints recognition rate 2.3 回归随机森林的必要性探究 本研究方法中,回归森林用于进一步改善关节识别的精度。但是,回归森林的使用会延长算法执行时间,因此有必要验证回归森林是否真的十分必要。这里将单独使用分类森林结合匹配图进行关节的定位,并同分类森林+回归森林+匹配图进行关节定位进行比较,探究回归森林发挥的作用。 图7 关于是否使用回归森林的误差对比。(a)“分类森林+回归森林+匹配图”的误差;(b)“分类森林+匹配图”的误差Fig.7 Identification error comparison between with and without the regression forest. (a) The errors of “classification forest +regression forest+graph matching”; (b) The errors of “classification forest + graph matching”. 图7(a)、(b)所示分别是“分类森林+回归森林+匹配图”和“分类森林+匹配图”的结果箱线图,纵坐标表示定位误差,横坐标为骨关节类别。可以看到,图7(a)中各关节识别误差的四分位间距超过5个体素的只有一个关节点(上胸骨柄),其他识别误差结果稳定在上下四分位相差为2~3个体素,证明本研究所用方法识别小鼠骨关节稳定性高。而图7(b)中多数关节的中值误差是图7(a)的2倍左右,且上下四分位的间距也更大,说明如果没有回归森林,大部分关节的定位精度和稳定性都会下降。 2.4 训练数据量对算法精度的影响 这里研究训练数据数量和训练数据小鼠姿态对小鼠骨关节定位的影响。如图8所示,49幅测试图像中的身体姿态主要呈现为3类:最左侧的姿态双腿张开,中间的腿部收缩,右侧的躯干扭曲,这三种姿势定义为1、2、3类,其他各类非主流姿势定义为4类。 图8 不同姿态的小鼠。(a)正常放置姿态;(b)脊柱与后肢蜷缩姿态;(c)脊柱侧向弯曲姿态。Fig.8 Mice with different postures. (a)Normal posture; (b)Spine and lower limbs curled up; (c)Spine lateral-bending. 作为测试,首先对以上4类姿态每类选出6幅图像,共24幅图像。然后,从第1类姿态的6幅中选出一幅作为测试图像,其他23幅作为训练图像。一共进行了5次实验,第1次只用了6幅属于第2类姿态的训练图像,第2次增加了6幅第3类姿态的图像,第3次再增加6幅第4类姿态的图像。第4次实验,从已有的18幅训练图像中去掉6幅(每类姿态2幅),增加5幅1类姿态的训练图像。第5次实验,使用全部23幅测试图像。 图9展示了这5次实验的结果,图的横轴为实验的序号,纵轴为识别的误差。由于小鼠大多数姿态中身体躯干部分变化很少,而四肢的姿态变化很大,所以将躯干与四肢分开进行误差分析。通过观察箱线图的四分位间距,可以发现,位于四肢关节的识别稳定性收敛比躯干关节稳定性收敛快,这是由于躯干脊柱上各个脊椎的相似性大,其脊柱周围的灰度值分布复杂,算法更难精确定位脊柱上的关节点,而四肢关节特征明显辨识度强,识别的稳定性收敛更快。 图9 训练数据量与识别误差箱线图。(a)全身关节误差分析;(b)四肢关节误差分析;(c)躯干关节误差分析Fig.9 Training data number and localization error.(a)Whole- body joints error analysis; (b)Limbs joints error analysis; (c)Trunk joints error analysis 对比5次实验,前3次实验的训练数据数目每次增加6幅图像,但是都不包含与测试图像相似的小鼠姿态。得出结论是:训练数据越多,识别的误差越小。第4次与第3次的训练样本数量相当,但是第4次包含了与测试小鼠姿态相同的图像,对比实验3、4的结果可以得出结论:当训练数据包含测试小鼠的姿态,识别的误差将减小并且更加稳定。所以训练数据应该尽可能包含多种小鼠姿态。第5次实验包含全部23幅训练图像,而误差却没有比第4次明显改善,对于躯干关节的稳定性甚至比第4次略差,说明训练样本的数目已经基本达到了饱和。 从实验结果可以看出,本研究方法对各个关节的定位误差中位数可以控制在0.63 mm,相当于3.16个体素,关节识别的成功率达到92%以上。定位精度主要受回归森林影响,而成功率主要由分类森林决定。影响定位精度和成功率的主要因素都是部分图像中踝关节的缺失。因此,今后的研究有必要重点解决踝关节缺失的问题。 通过对比“分类森里+回归森林+图匹配”和“分类森林+图匹配”,可以明显看出后者在精度和稳定性方面的优势。但是,两种方法中位数误差之间的差距在两个体素以内,并且脊柱和头骨连接处、骨盆前和右手联合使用回归森林的效果略差于单独使用分类森林。所以在严格要求识别精度的情况下,有必要使用回归森林,但是考虑到定位时间和相对低精度的要求,可以考虑只使用分类森林结合匹配图的方式进行定位。 通过5次不断增加训练样本的实验,可以看到训练样本数目对于精度和稳定性的重要作用,训练样本越多,精度和稳定性越好。为了使算法的精度和稳定性达到饱和区,本方法所需的训练样本数最小应该在18以上。还可看到,在训练集中包含于测试样本相同姿态的训练数据对于提高精度和稳定性也很重要。因此,为了达到对不同姿态的良好定位精度,应尽量在训练集中包含各种身体姿态。在未来的研究工作中,需要收集更多不同姿态的影像,将其加入训练集,从而进一步提高算法对不同姿态的普适性。 本研究仅涉及到了micro- CT这一种小动物成像模式,为了进一步拓宽算法的适用范围,今后还将考虑对核磁、PET、SPECT等影像模式开发类似的关节点定位算法。由于其他影像模式对于骨组织的对比度分辨率不如CT好,后续研究的难度可能会更大。 虽然本研究的小鼠关节点定位问题对于小动物影像分析具有较重要意义,但是国内外尚无同类研究。正如本文引言部分所介绍,现有的各种小鼠micro- CT图像分析方法多侧重于全身图谱的配准,但是其配准精度又都受到小鼠姿态变化的限制,足以说明本研究方法对于现有研究有促进意义。对比人类临床CT图像中的关节定位精度,Donner等[4]采用随机森林算法得到的中值误差为2.23个体素,与本研究相当。 本研究提出了一种较为准确稳定的算法用于定位小鼠micro- CT图像中的骨骼关节点。本方法使用分类和回归随机森林实现了对CT图像关节点备选点的选择,然后应用图匹配算法根据关节相对位置关系来筛选备选关节点。基于49幅包含不同身体姿态的测试图像,本研究方法的中值误差为0.63 mm,均值误差为0.97 mm(中值误差为3.16个体素,均值误差为4.80个体素)。回归森林的使用,对于提高定位精度有重要作用,在训练集中包含各种不同的身体姿态对于提高精度十分关键。本研究方法对于脚踝缺失的CT数据的识别效果不够理想,但是对完整的CT数据包括小鼠全身的图像识别成功率很好。本研究为小鼠micro- CT影像的姿态识别和自动分析提供了新的方法途径。 (致谢:衷心感谢美国加州大学洛杉矶分校Crump分子影像中心的Arion Chatziiannou教授为本研究提供小动物CT影像数据。) [1] Zhang Guanglei, Liu Fei, Pu Huangsheng, et al. A direct method with structural priors for imaging pharmacokinetic parameters in dynamic fluorescence molecular tomography [J]. IEEE Transactions on Biomedical Engineering, 2014, 61(3): 986- 990. [2] Wang Luyao, Zhu Jun, Liang Xiao, et al.Performance evaluation of the Trans- PET©BioCaliburn©LH system: A large FOV small- animal PET system [J]. Physics in Medicine and Biology, 2015, 60(1): 137- 50. [3] Wang Huina, Liu Chengbo, Gong Xiaojing, et al. In vivo photoacoustic molecular imaging of breast carcinoma with folate receptor- targeted indocyanine green nanoprobes [J]. Nanoscale, 2014, 6(23): 14270- 14279. [4] 杨昆, 李真, 杨永鑫. 小动物正电子发射断层成像仪探测器发展 [J]. 中国生物医学工程学报, 2014, 33(2): 218- 226. [5] Kesner AL, Dahlbom M, Huang SC, et al. Semiautomated analysis of small- animal PET data [J]. Journal of Nuclear Medicine, 2006, 47(7): 1181- 1186. [6] Baiker M, Vastenhouw B, Branderhorst W, et al. Atlas- driven scan planning for high- resolution micro- SPECT data acquisition based on multi- view photographs: a pilot study [C]// Michael IM, Kenneth HW, eds. SPIE Medical Imaging 2009: Visualization, Image- Guided Procedures, and Modeling. Lake Buena Vista: SPIE, 2009: 72611L1- 72611L8. [7] Baiker M, Staring M, Löwik CWGM, et al. Automated Registration of whole- body follow- up micro CT data of mice [C]//Medical Image Computing and Computer- Assisted Intervention- MICCAI 2011. Berlin: Springer Berlin Heidelberg, 2011: 516- 523. [8] Baiker M, Milles J, Dijkstra J, et al. Atlas- based whole- body segmentation of mice from low- contrast Micro- CT data [J]. Med Image Anal, 2010, 14(6): 723- 737. [9] Wang H, Stout DB, Chatziioannou AF. A method of 2D/3D registration of a statistical mouse atlas with a planar X- ray projection and an optical photo [J]. Med Image Anal, 2013, 17(4): 401- 416. [10] Wang H, Stout DB, Chatziioannou AF. Estimation of mouse organ locations through registration of a statistical mouse atlas with micro- CT images [J]. IEEE Trans Med Imaging, 2012, 31(1): 88- 102. [11] Shotton J, Sharp T, Kipman AA, et al. Real- time human pose recognition in parts from single depth images [J]. Communications of the ACM, 2013, 56(1): 116- 124. [12] Donner R, Menze BH, Bischof H, et al. Global localization of 3D anatomical structures by pre- filtered Hough forests and discrete optimization [J]. Med Image Anal, 2013, 17(8): 1304- 1314. [13] 林修煅. Micro- CT成像系统及其应用研究 [D].西安:西安电子科技大学,2012. [14] 方匡南,吴见彬,朱建平,等. 随机森林方法研究综述 [J].统计与信息论坛, 2011, 26(3):32- 38. [15] Van Ryzin J, Breiman L, Friedman JH, et al. Classification and Regression Trees [J]. Journal of the American Statistical Association, 1986,33(1):128- 128. [16] Schapire RE. The strength of weak learnability[J].Machine Learning, 1990, 5(2): 197-227. [17] Amit Y, Geman D. Shape quantization and recognition with randomized trees [J]. Neural Computation, 1997, 9(7): 1545- 1588. [18] Breiman L. Random forests [J]. Machine Learning, 2001, 45(1): 5- 32. [19] 李欣海. 随机森林模型在分类与回归分析中的应用 [J]. 应用昆虫学报, 2013, 50(4): 1190- 1197. [20] Criminisi A, Jamie Shotton. Decision Forests for Computer Vision and Medical Image Analysis[M]. Berlin: Springer Science & Business Media, 2013:340- 366. [21] Pushmeet K, Philip HST. Efficiently solving dynamic markov random fields using graph cuts [C]// IEEE International Conference on Computer Vision (ICCV 05). Beijing: IEEE, 2005: 922- 929.. [22] Boykov Y, Veksler O, Zabih R. Fast approximate energy minimization via graph cuts[J]. IEEE Transactions on Pattern Analysis & Machine Intelligence, 2001, 23(11):1222- 1239. Bone Joints Localization in Mouse Micro- CT Images Using Random Forests Algorithm Tu Ruibo Chen Zhonghua Wang Hongkai#* (Department of Biomedical Engineering, Dalian University of Technology, Dalian 116024, Liaoning, China) Along with the rapid development of imaging techniques for small animals, more and more images obtained from small animals need to be analyzed per day, therefore automated image analysis method has become an urgent demand. For mice images, the significant inter- subject posture variations become a major difficulty for automated image analysis. In this paper, an automatic bone joint localization method was developed for mouse micro- CT images, so as to help with posture identification of mouse body. The proposed method was composed of three steps: (1) classification random forests for rough joint localization, (2) aggregating the results of classification through regression forest, and (3) picking up landmarks in the right position by the mapping graph. The method achieved automatic bone joint localization for 49 test images of different body postures. The median localization error of the whole body CT images was 0.68 mm. The success rate of localization was 98.27%. We also demonstrated the necessity of combining classification and regression random forest and discussed the influence on localization with different number of training data. With this new method for mouse micro- CT posture identification was expected to provide helpful information for the subsequent image registration, segmentation and measurements. small animal image analysis;bone joint localization;random forest;pattern recognition;micro- CT 10.3969/j.issn.0258- 8021. 2017. 03.001 2016-08-19, 录用日期:2016-10-21 国家自然科学基金(61571076);国家自然科学基金青年基金(81401475);辽宁省自然科学基金(2015020040) R318 A 0258- 8021(2017) 03- 0257- 10 *通信作者(Corresponding author),E- mail: wang.hongkai@dlut.edu.cn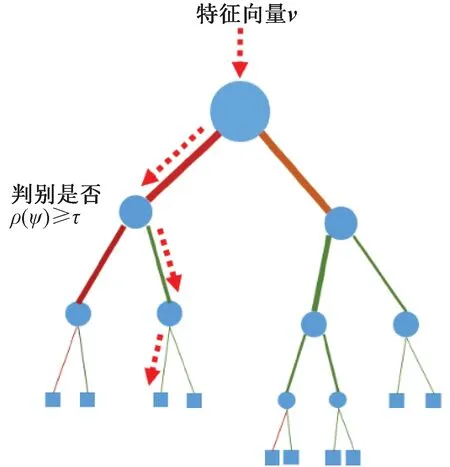


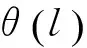
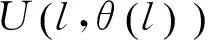

2 结果


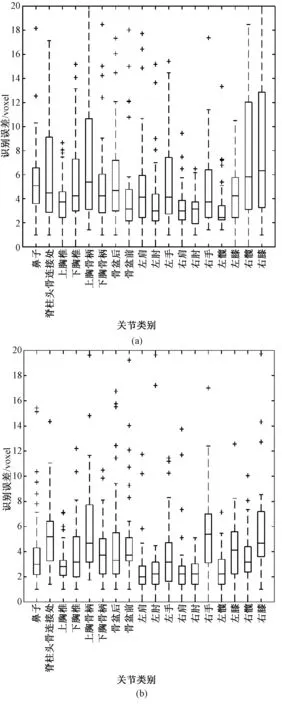


3 讨论
4 结论

