内蒙古地区绿豆品种遗传多样性SSR分析及DNA指纹图谱构建
赵雅楠,王 颖,2,张东杰,*,王丽侠*,佐兆杭
内蒙古地区绿豆品种遗传多样性SSR分析及DNA指纹图谱构建
赵雅楠1,王 颖1,2,张东杰1,*,王丽侠3,*,佐兆杭1
(1.黑龙江八一农垦大学食品学院,黑龙江 大庆 163319;2.国家杂粮工程技术研究中心,黑龙江 大庆 163319;3.中国农业科学院作物科学研究所,北京 100081)
目的:分析内蒙古地区绿豆品种的遗传多样性并构建其DNA指纹图谱,为探明绿豆种质资源遗传背景及真伪鉴定提供科学依据。方法:以内蒙古地区92 份绿豆品种为模板,利用筛选得到的10 对多态性丰富的简单重复序列(simple sequence repeat,SSR)引物对其进行遗传多样性分析及DNA指纹图谱构建。结果:10 对SSR引物共检测到58 个等位基因,每对引物检测到3~10 个不等;不同引物揭示的多态信息含量变幅介于0.371 4~0.773 7,平均为0.52。92 份绿豆种质的遗传相似系数变幅介于0.033 4~1.000 0之间,平均为0.46。非加权组平均法聚类分析结果显示,在遗传相似系数为0.294 4时,可将92 份绿豆种质分为两大类群。构建了92 份绿豆品种的SSR指纹图谱及分子身份证,除个别品种外,大部分具有唯一性,可用于品种鉴定。结论:内蒙古地区绿豆种质资源的遗传多样性相对较丰富,但存在一定程度的近交现象,有待进一步加强引进基因资源,创新绿豆育种材料;构建绿豆SSR指纹图谱和分子身份证,对绿豆品种真伪鉴定、身份识别、溯源管理及原产地保护具有重要意义。
SSR荧光标记;绿豆;遗传多样性分析;指纹图谱构建
绿豆(Vigna radiate)是豇豆属、亚洲豇豆亚属的栽培作物,是起源于中国的重要粮食作物,在我国已有2000多年的栽培历史[1-3]。绿豆生育期短,具有较强的耐瘠性和固氮能力,是禾谷、棉花、薯类等作物间套种的适宜作物,也是良好的减灾救荒的填闲作物。同时绿豆富含蛋白质、淀粉等,是调解营养平衡的重要补充粮[4-5]。随着人们生活水平的提高,绿豆以其特殊的药食同源性深受青睐。然而对我国近些年来育成的绿豆品种进行深入分析不难发现,少数优良亲本被重复集中使用,种质资源应用同质化的趋势日趋显著,导致我国绿豆种质资源遗传基础狭窄,优异基因流失,且绿豆种子侵权、机械混杂和人为掺假现象时有发生,传统鉴定方法越来越难以区分鉴别[6-7]。因此,探究绿豆种质资源遗传多样性、构建DNA指纹图谱对明确绿豆种质遗传分化水平,挖掘、利用优质绿豆种质资源及名优品种保护具有重要意义。
随着分子生物学的快速发展,分子标记技术逐渐成熟,尤其是简单重复序列(simple sequence repeat,SSR)标记技术因其操作简便、多态性丰富、重复性好等特点在作物遗传多样性分析及品种鉴定等研究中广泛应用。Rana等[8]对48 个梨品种进行遗传多样性分析,将参试品种划分为5 个类群。罗兵等[9]对太湖地区42 份粳稻品种进行遗传多样性分析,认为SSR标记可将常规粳稻和杂交粳稻进行区分。宫慧慧等[10]利用18 对SSR引物对80 份小豆种质进行分析,证明SSR技术是探究种群聚类与种质地理起源相关性的重要途径。薛建峰等[11]利用10 对SSR引物构建了国内外31 份具有品种特异性的蓖麻DNA指纹图谱;姚全胜等[12]构建了11 个具有代表性的芒果品种的指纹图谱,利用4 对引物即可将其完全区分开;陆徐忠等[13]利用48 对SSR引物构建了133 份杂交水稻的分子身份证,并将其与品种形态学信息结合,构建了具有查询功能的SSR指纹网络数据库,为水稻品种的选育及鉴别提供参考。
我国绿豆的主产区主要分布在黄淮流域及东北地区,其中内蒙古在绿豆播种量及产量方面均位居全国前列,并且在漫长的自然进化和人工选择过程中形成了丰富的绿豆种质资源。然而随着绿豆品种的大面积推广,遗传基础愈发狭窄,品种间相似度高,生产存在潜在危险性[14-16]。同时,关于内蒙古地区绿豆品种遗传多样性分析及指纹图谱构建的研究报道十分有限[17-18],研究中涉及的品种数量较少,目前鲜见针对内蒙古地区大部分绿豆资源遗传多样性研究的报道,同时,前人研究中均利用聚丙烯酰胺凝胶电泳进行检测,耗时费力,在不同批次分析中易产生误差,检测精确度不高。基于此,为进一步充分了解内蒙古地区的绿豆种质资源,本研究对中国农业科学院作物科学研究所提供的100 对绿豆SSR引物筛选得到条带清晰稳定、多态性丰富的10 对SSR引物,利用SSR荧光标记技术对内蒙古地区92 份绿豆种质资源进行遗传多样性分析并构建其SSR指纹图谱,为探明内蒙古地区绿豆种质资源遗传背景提供理论依据,同时也为我国绿豆品种真伪鉴定、种质资源评价及资源保护利用提供科学依据。
1 材料与方法
1.1 材料与试剂
本研究选取来自内蒙古自治区包头市、呼和浩特市等23 个地区的92 份绿豆地方品种,绿豆种质材料均由中国农业科学院作物科学研究所提供,供试绿豆信息见表1。
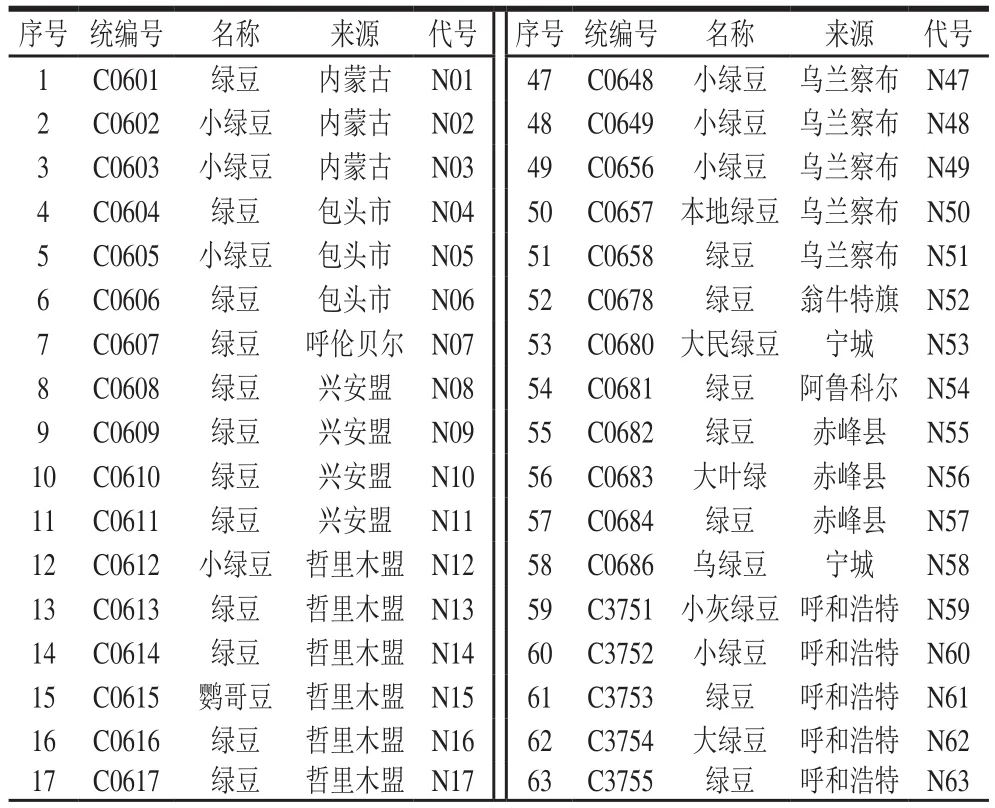
表1 内蒙古地区92 份参试绿豆品种信息Table 1 Information about 92 mungbean varieties in different regions of Inner Mongolia
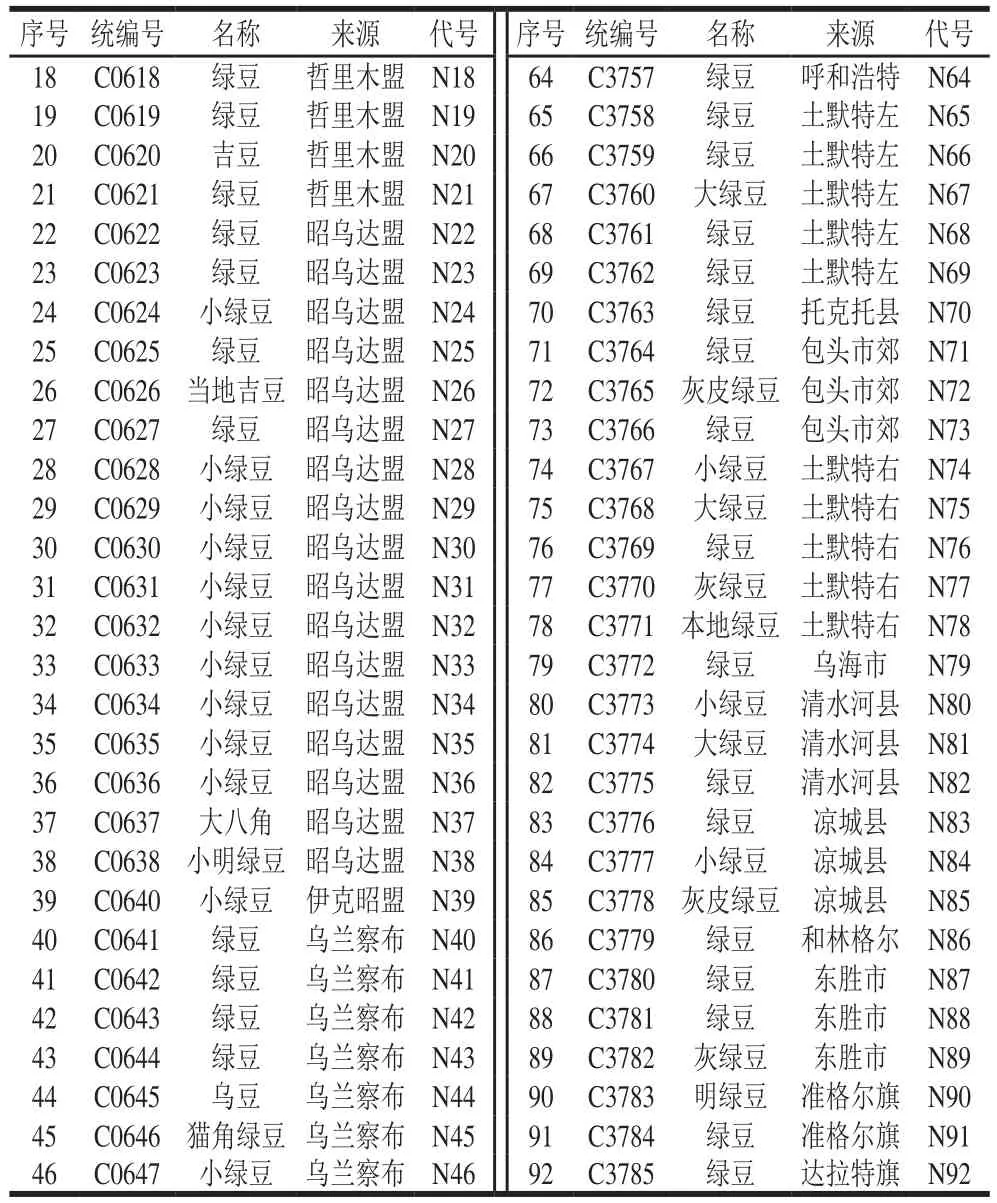
续表1
植物基因组DNA提取试剂盒、dNTPs、Taq酶、Mg2+、Buffer、琼脂糖、标准分子质量等均购于北京天根生物技术有限公司。
1.2 SSR引物
10 对SSR引物均由中国农业科学院作物科学研究所提供,普通引物由上海生工生物工程公司合成;荧光标记引物由美国ABI公司合成,在正向引物上加注的荧光染料分别是5’HEX(绿色)、5’FAM(蓝色)和5’TMRA(黄色),见表2。
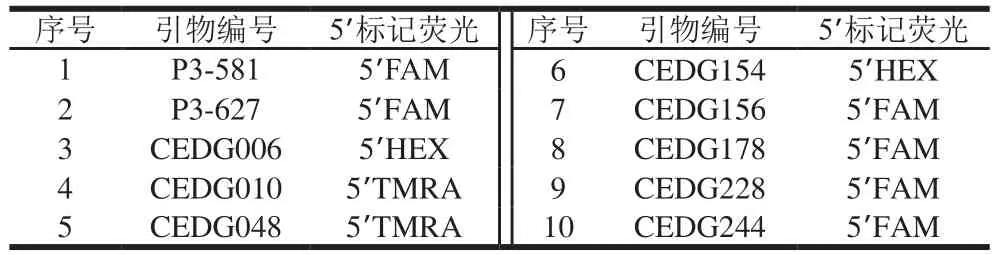
表2 10 对SSR引物信息Table 2 Information about 10 pairs of SSR primers
1.3 仪器与设备
ABI Prism 3730XL型基因分析仪 上海艾研生物科技有限公司;WD-9402C基因扩增仪 北京六一生物科技有限公司;QT-88A智能核酸蛋白检测仪 上海琪特分析仪器有限公司;2-16PK高速冷冻离心机 德国Sigma公司;Alpha凝胶成像系统 美国ProteinSimple公司;SW-CJ-1D超净工作台 上海皓庄仪器有限公司;SANyO SIM-F14全自动制冰机 上海迭戈生物科技有限公司;LDZX-40B立式自动电热压力蒸汽灭菌锅河北蓝梦生物医药科技有限公司。
1.4 方法
1.4.1 DNA提取
取室温条件下种植12 d左右的新鲜嫩叶,液氮环境中用球磨机打碎成粉状,采用改良后的CTAB法[19]提取DNA。用1%琼脂糖凝胶电泳检测DNA提取质量,用QT-88A智能核酸蛋白检测仪检测DNA质量浓度,并稀释成30 ng/μL于4 ℃保存备用。
1.4.2 聚合酶链式反应(polymerase chain reaction,PCR)扩增
PCR体系为20 μL,包括30 ng/μL DNA模板2 μL,2 μmol/L引物0.6 μL、2.5 U/μL Taq酶0.2 μL、10 mmol/L dNTP 0.4 μL、20 mmol/L MgCl21 μL、Buffer 2 μL,其余用ddH2O补齐。PCR程序为94 ℃预变性5 min;94 ℃变性30 s,退火温度35 s,72 ℃延伸40 s,共35 个循环;最终72 ℃延伸3 min;4 ℃保存。
1.4.3 毛细管电泳
将甲酰胺与分子质量内标按100∶1的体积比混匀后,取9 μL加入上样板中,再加入1 μL稀释10 倍的PCR产物。然后使用3730XL测序仪进行毛细管电泳,利用Genemarker中的Fragment(Plant)片段分析软件对测序仪得到的原始数据进行分析,将各泳道内分子质量内标的位置与各样品峰值的位置作比较分析,得到片段大小。
1.5 数据处理
用POPGENE version 1.32[20]软件计算平均观测等位基因数、有效等位基因数、多态位点百分率、期望杂合度、观察杂合度、扩增位点的多态性信息含量、遗传相似度。基于遗传相似度,采用非加权组平均法(unweighted pair-group method with arithmetic means,UPGMA)法对各种群进行聚类分析[21]。
2 结果与分析
2.1 SSR引物多态性分析
如表3所示,10 对SSR引物在92 份绿豆品种中共扩增出58 个等位基因,每对引物检测到的等位基因数变幅为3~10 个不等,平均为5.8 个。其中,引物CEDG156检测到的等位变异最多,为10 个,而引物P3-581、P3-627和CEDG178检测到的等位基因数最少,仅为3 个。10 对引物共检测到28.38 个有效等位基因,占48.9%。多态位点的有效等位基因数变幅介于1.64~4.98中间,平均为2.58。不同引物揭示的多态信息含量变幅介于0.371 4~0.773 7,平均为0.52,其中引物CEDG048、CEDG154、CEDG156、CEDG228、CEDG244多态性较高,多态信息含量均大于0.5,为高度多态性位点,而其余5 对引物均为中度多态性位点(0.25<多态信息含量<0.5)。Nei’s基因多样性变幅介于0.388 8~0.799 2之间,平均为0.575 2。Shannon信息指数变幅范围介于0.700 4~1.779 7之间,平均为1.140 0。观察杂合度变幅介于0.804 3~1.000 0之间,平均为0.896 0。期望杂合度变幅介于0.390 9~0.803 7之间,平均为0.578 4。引物CEDG244和CEDG048无论观测从等位基因数目(仅次于CEDG156,为8和7)还是多态信息含量(0.773 7和0.649 6)来看,均较高,因此它们将是内蒙古地区绿豆遗传多样性分析研究中的骨干引物。

表3 10 对SSR引物多态性信息Table 3 Information about genetic diversity of 92 mungbean germplasms based on 10 SSR
2.2 遗传相似性分析

图1 遗传相似系数的次数分布Fig. 1 Genetic similarity distribution
依据10 对SSR引物在92 份内蒙古地区绿豆种质资源中检测到的58 个等位基因,利用NTSySpc2.0统计分析软件按Nei的方法计算实验材料间的遗传相似系数,共获得遗传相似系数4 278 个,92 份绿豆种质资源的遗传相似系数变幅介于0.033 4~1.000 0之间,平均为0.46。其中昭乌达盟绿豆(C0623)与准格尔旗绿豆(C3784)的遗传相似系数最小,为0.033 4,说明二者遗传相似性较低,亲缘关系较远;昭乌达盟小绿豆(C0628)与乌兰察布绿豆(C0641)和乌兰察布绿豆(C0643)与乌兰察布绿豆(C0641)的遗传相似系数较大,为0.983 3,说明彼此间遗传距离较小,亲缘关系较近。以0.1为组距,对4 278 个遗传相似系数进行次数分布分析,如图1所示。参试材料的遗传相似系数近似呈正态分布,有2 928 个遗传相似系数在0.21~0.60之间,占全部数据的68.4%;有435 个在0.2以下,占全部数据的10.2%;有489 个在0.7以上,占所有数据的21.4%。结果表明内蒙古地区绿豆种质资源的遗传多样性相对较高,但也存在一定程度的近交现象。
2.3 聚类分析
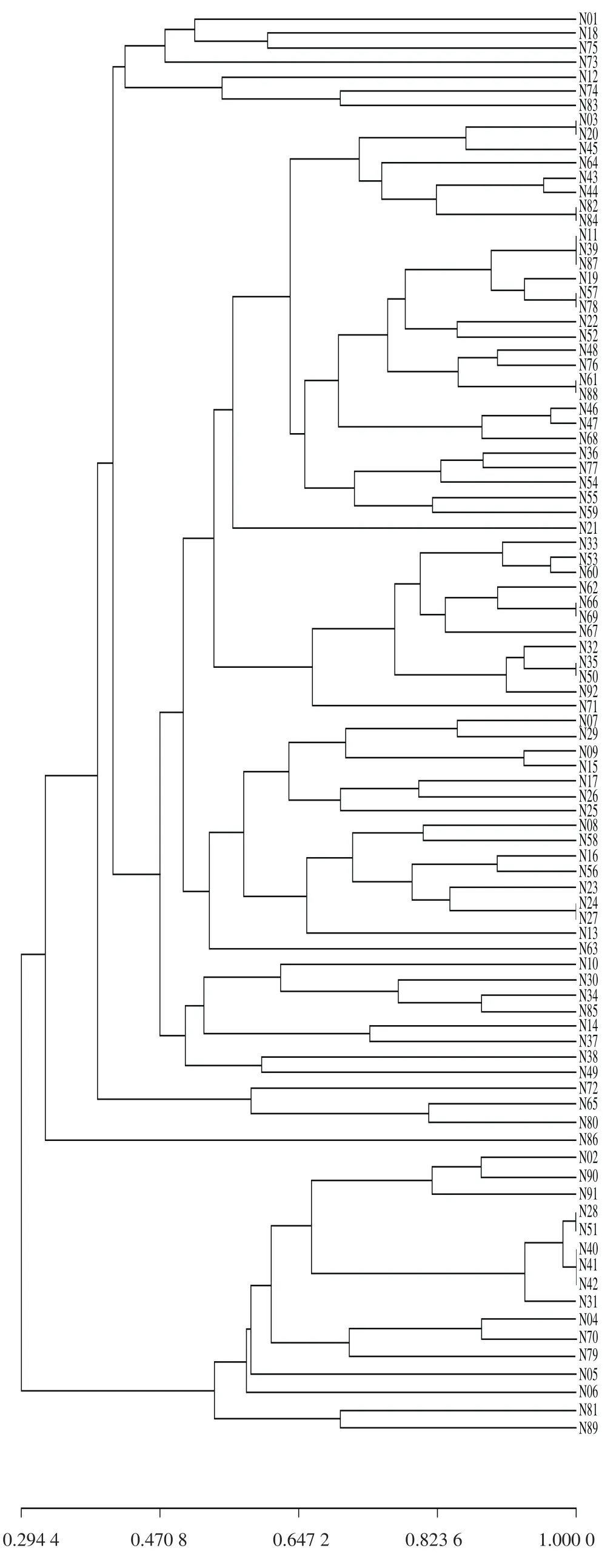
图2 基于SSR标记的92 份绿豆聚类分析Fig. 2 Dendrogram of 92 mungbean varieties generated by SSR markers
通过UPGMA法利用遗传相似系数对92 份内蒙古地区绿豆种质资源材料进行聚类分析,如图2所示。在遗传相似系数为0.294 4时,可将92 份绿豆种质分为2 个类群。第1类群由76 份种质组成,其余16 份种质聚集在第2类群。在遗传相似系数为0.412 0时,可将第1类群分为4 个亚群。第1个亚群包括绿豆(C0601)、绿豆(C0618)、大绿豆(C3768)、绿豆(C3766)、小绿豆(C0612)、小绿豆(C3767)和绿豆(C3776)共7 份绿豆种质;第2个亚群包括65 个绿豆种质。在遗传相似系数为0.576 6时,可将第2个亚群分成7 个小组。第1个小组包括29 份种质,其中来自内蒙古中部地区(乌兰察布、东胜、呼和浩特)的绿豆种质17 份,来自内蒙古东部地区(哲里木盟、兴安盟、昭乌达盟)的绿豆种质10 份,仅小绿豆(C0640)来自内蒙古西南部地区的伊克昭盟,未知来源的小绿豆(C0603)与来源于哲里木盟的吉豆(C0620)聚在一起,推测其可能也来源于哲里木盟或者内蒙古东部其他地区;第2小组包括12 份绿豆种质,除了C0633等4 份来自昭乌达盟的种质外,其余均来自内蒙古中部呼和浩特市附近;第3小组包括16 份绿豆种质,其中除绿豆(C0607)和绿豆(C3755)分别来自内蒙古北部地区呼伦贝尔和中部呼和浩特外,其余所有品种均来自内蒙古东部地区;第4、5、6、7小组分别由绿豆(C0610)、小绿豆(C0630)、小绿豆(C0634)、灰皮绿豆(C3778)和绿豆(C0614)、大八角(C0637)和小明绿豆(C0638)、小绿豆(C0656)以及灰皮绿豆(C3765)、绿豆(C3758)、小绿豆(C3773)组成。第2亚群包括16 份绿豆种质,其中除小绿豆(C0628)和小绿豆(C0631)来自昭乌达盟外,其余所有品种均来自内蒙古中部地区,未知来源的小绿豆(C0602)与来自准格尔旗的明绿豆(C3783)聚在一起,推测其可能也来源于内蒙古中部地区。第3个亚群包括3 份绿豆种质,即灰皮绿豆(C3765)、绿豆(C3758)和小绿豆(C3773)单独聚为一类;第4亚群仅包含1 份种质,即绿豆(C3779),说明该品种与其他材料间的基因交流较少,亲缘关系相对较远。
2.4 SSR指纹图谱及分子身份证构建

表4 92 个绿豆品种在10 个SSR位点的指纹数据Table 4 Fingerprint data at 10 SSR loci of 92 mungbean varieties

续表4

续表4

图3 绿豆(C0627)(A)和小绿豆(C0628)(B)在位点P3-581座位上的毛细管电泳峰图Fig. 3 SSR fingerprint patterns of mungbean varieties C0627 (A) and C0628 (B) using primer P3-581
利用10 对核心引物对内蒙古地区92 份绿豆品种进行扩增,准确获得不同材料在不同位点的等位基因片段大小及相应的毛细管电泳图,如表4、图3所示。例如小绿豆(C0648)在SSR位点P3-581的扩增片段为165 bp和168 bp。按表2的引物顺序,即P3-581、P3-627、CEDG006、CEDG010、CEDG048、CEDG154、CEDG156、CEDG178、CEDG228、CEDG244将每对引物在92 份参试绿豆品种中扩增出的等位基因按片段大小升序排列,依次编号。将每个样品在10 对引物上扩增条带的数据串联起来,即得到由至少10 位数字或字母(有杂合带时则多于10 位)组成的分子身份证。如绿豆(C0601)的分子身份证为2152723326,第1个数字是“2”,即绿豆(C0601)在1号引物P3-581上扩增出在片段排列中排位第2的片段,其余9位依此类推。92份参试绿豆品种的分子身份证如表5所示。

表5 92 份绿豆品种的分子身份证Table 5 List of digital molecular identifications of 92 mungbean accessions
3 讨 论
SSR标记因其多态性丰富、操作简便等特点在农作物遗传多样性分析及指纹图谱构建中广泛应用[22-24]。王丽侠等[25]利用28 对小豆SSR引物分析60 份绿豆种质,检测到的等位变异数变幅范围为2~7 个,平均为2.9,多态信息含量变幅范围为0.02~0.69,平均为0.36;Wang Xiaoqiang等[26]利用15 对引物分析65 份绿豆种质的遗传多样性,共扩增出47 个变异位点,每对引物扩增出2~6 个变异位点不等,平均多态信息含量为0.28。而本研究利用10 对SSR引物对内蒙古地区的92 份绿豆种质进行分析,得到的平均等为基因数(5.8)和平均多态信息含量(0.52)明显优于前人的研究,说明选取的核心引物多态性较丰富,在绿豆种质研究中表现出较大潜力。
遗传相似性分析结果显示,内蒙古地区绿豆种质资源间遗传相似系数平均值为0.46,小于0.5的遗传相似系数占整个数据的62.8%,说明内蒙古地区绿豆品种的遗传多样性水平相对较丰富,与任红晓等[17]分析中国北方名优绿豆品种时得到北方参试地区中内蒙古是绿豆资源最丰富地区的结果相符。但大于0.7的遗传相似系数占整个数据的21.4%,其中10 组(22 个)品种间的遗传相似系数为1.00,考虑这几组品种的遗传背景相同,但不排除“同种异名”现象,同时也说明内蒙古地区绿豆种质资源存在一定程度的近交现象,个别品种在DNA水平上差异较小,亲缘关系较近。原因可能是内蒙古是春绿豆的主栽区,一些优良品种由于长时间的自然驯化、地区间资源共享,导致遗传变异率降低,遗传基础狭窄,多样性水平降低。因此,在今后的育种工作中应加大引进省外或国外的绿豆材料以拓宽内蒙古地区的遗传背景。
聚类分析发现,各组内聚类均呈现出一定的地域相关性,即地域相近或纬度相近地区材料间遗传关系较近,来自同一地理区域的品种聚在一起的机率比较大。如第2亚群第3小组的16 份种质中有14 份来自内蒙古东部地区,其中来自哲里木盟和昭乌达盟的种质各自聚在一起;来自昭乌达盟的绿豆种质除小绿豆(C0628)和小绿豆(C0631)外,其余均聚为一类,其他地区种质也呈现相类似的情形,如呼和浩特的6 份种质、包头的3 份种质均被聚在相同类群中。此外,不同地区来源品种间也存在相互聚集的情况,如第1亚群中的7 份种质分别来自哲里木盟、土默特旗等7 个不同地区,体现出内蒙古绿豆种质可能来源于共同的祖先以及品种之间较近的亲缘关系,或在基因进化的过程中,不同地理来源的品种为适应环境发生了一定的基因交流。可见SSR分子标记技术能够在一定程度上反映品种的地理来源情况,来自相同地理条件和生态环境的种质在分子水平上的遗传相似度较高。但从遗传距离分析,地理来源不同的品种遗传距离却比较相近,如兴安盟绿豆(C0611)和伊克昭盟小绿豆(C0640)2 个品种间的遗传距离为0,土默特旗灰绿豆(C3770)和东胜市绿豆(C3781)遗传距离为0.223 1。来源不同的绿豆品种地理距离远而遗传距离却比较相近,这可能与品种具有相似的环境条件或常年引种及相近的选择标准有关。
DNA指纹图谱能够在分子水平上反映出生物个体间差异,多态性丰富、环境稳定性强,在品种真伪鉴定、种权保护、产地溯源研究中发挥重要作用[27-30]。本研究将10 个SSR标记扩增的电泳结果按照一定规律制成类似于人类指纹的图谱即SSR指纹图谱,对其进行数字化转化即形成了分子身份证,具有高度的遗传稳定性及个体特异性,对绿豆品种真伪鉴定及身份识别具有重要意义。然而本研究中选用的10 对核心引物并未将参试的92 个品种完全区分开,少数品种的指纹图谱及分子身份证不具备唯一性。究其原因,一方面可能是由于绿豆种质的基因组结构特性决定的;另一方面可能是因为绿豆种质研究起步较晚,多态性引物的开发还不够全面,目前公开的绿豆SSR引物多态性水平较低,鉴别能力较弱,同时也可能是因为参试品种全部来自内蒙古地区,品种间遗传背景相似,甚至在长期的发展种植过程中出现“同种异名”情况。
4 结 论
本研究利用10 对SSR引物对内蒙古地区92 份绿豆种质进行遗传多样性分析,从分子水平上揭示了内蒙古地区绿豆品种的遗传多样性,初步明确了现阶段内蒙古地区绿豆品种的遗传分化程度及遗传多样性水平,为内蒙古地区绿豆品种选育及种质创新提供参考;构建了92 份绿豆品种的SSR指纹图谱及分子身份证,为绿豆品种真伪鉴定提供理论基础,对绿豆品种溯源管理及原产地保护具有重要意义。研究结果表明,虽然内蒙古地区绿豆种质资源多样性相较丰富,但仍存在一定程度的近交现象,因此今后的研究一方面要加大对内蒙古地区优质绿豆资源的收集及保存,保护资源的遗传多样性;另一方面需加强种质创新,引进和利用其他地区丰富的绿豆种质资源,拓宽绿豆育种的遗传基础。
[1] LESTARI P, KIM S K, REFLINUR, et al. Genetic diversity of mungbean (Vigna radiata L.) germplasm in Indonesia[J]. Plant Genetic Resources, 2014, 12(S1): S91-S94. DOI:10.1017/ S1479262114000343.
[2] AMIN M R, KARIM M A, ISLAM M R, et al. Effect of flooding on growth and yield of mungbean genotypes[J]. Bangladesh Journal of Agricultural Research, 2016, 41(1): 151-162. DOI:10.3329/bjar. v41i1.27680.
[3] 王兰芬, 武晶, 景蕊莲, 等. 绿豆种质资源芽期抗旱性鉴定[J]. 植物遗传资源学报, 2014, 15(3): 498-503. DOI:10.13430/j.cnki. jpgr.2014.03.007.
[4] ROy S, BARMAN M, PUSTE A M, et al. Growth, yield, water use efficiency and competitive functions of intercropping system of maize (Zea mays L.) and mungbean (Vigna radiata L.) as influenced by irrigation[J]. Saarc Journal of Agriculture, 2016, 13(2): 94-107.
[5] RAM H, SINGH G, AGGARWAL N. Effect of irrigation, straw mulching and weed control on growth, water useefficiency and productivity of summer mungbean[J]. Legume Research, 2016, 39(2): 1-5. DOI:10.18805/lr.v39i2.9533.
[6] CHOUDHARy S, CHOUDHARy A K, SHARMA O P. Screening of mungbean (Vigna radiata) genotypes to identify source of resistance to dry root rot[J]. Journal of Food Legumes, 2011, 24: 117-119.
[7] LIU C y, CHENG X Z, WANG S H, et al. The screening of SSR and STS markers for genetic diversity analysis of mungbean germplasm[J]. Journal of Plant Genetic Resources, 2007, 8(3): 298-302. DOI:10.13430/j.cnki.jpgr.2007.03.009.
[8] RANA J C, CHAHOTA R K, SHARMA V, et al. Genetic diversity and structure of Pyrus accessions of Indian Himalayan region based on morphological and SSR markers[J]. Tree Genetics and Genomes, 2015, 11(1): 821. DOI:10.1007/s11295-014-0821-2.
[9] 罗兵, 徐港明, 孙海燕, 等. 利用简单重复序列(SSR)标记分析太湖稻区现代粳稻品种的遗传多样性[J]. 农业生物技术学报, 2014, 22(12): 1502-1513. DOI:10.3969/j.issn.1674-7968.2014.12.005.
[10] 宫慧慧, 谢华, 马荣才, 等. 利用SSR分析小豆种质遗传多样性[J]. 农业生物技术学报, 2008, 16(5): 872-880. DOI:10.3864/ j.issn.0578-1752.2009.08.004.
[11] 薛建峰, 谭美莲, 严明芳, 等. 我国部分蓖麻品种遗传资源SSR分析及DNA指纹图谱[J]. 中国油料作物学报, 2015, 37(1): 48-54. DOI:10.7505/j.issn.1007-9084.2015.01.008.
[12] 姚全胜, 詹儒林, 黄丽芳, 等. 11 个芒果品种SSR指纹图谱的构建与品种鉴别[J]. 热带作物学报, 2009, 30(11): 1572-1576.
[13] 陆徐忠, 倪金龙, 李莉, 等. 利用SSR分子指纹和商品信息构建水稻品种身份证[J]. 作物学报, 2014, 40(5): 823-829. DOI:10.3724/ SP.J.1006.2014.00823.
[14] 刘慧. 我国绿豆生产现状和发展前景[J]. 农业展望, 2012, 8(6): 36-39. DOI:10.3969/j.issn.1673-3908.2012.06.010.
[15] 赵雪英, 王宏民, 李赫, 等. 绿豆种质资源的ISSR遗传多样性分析[J].植物遗传资源学报, 2015, 16(6): 1277-1282. DOI:10.13430/j.cnki. jpgr.2015.06.020.
[16] 郭鹏燕, 王彩萍, 左联忠, 等. 绿豆种质资源创新研究及应用[J].农业科技通讯, 2015(4): 213-214.
[17] 任红晓, 程须珍, 徐东旭, 等. 应用SSR标记分析中国北方名优绿豆的遗传多样性[J]. 植物遗传资源学报, 2015, 16(2): 395-399. DOI:10.13430/j.cnki.jpgr.2015.02.029.
[18] 刘岩, 程须珍, 王丽侠, 等. 基于SSR标记的中国绿豆种质资源遗传多样性研究[J]. 中国农业科学, 2013, 46(20): 4197-4209. DOI:10.3864/j.issn.0578-1752.2013.20.003.
[19] 陆敏佳, 莫秀芳, 王勤, 等. 藜麦基因组DNA提取方法的比较[J]. 江苏农业科学, 2014(4): 42-45. DOI:10.3969/j.issn.1002-1302.2014.04.016.
[20] YEH F C, YANG R C, BOYLE T. POPGENE, Microsoft Windowsbased freeware for population genetic analysis release 1.31[M]. Edmonton: University of Alberta, 1999.
[21] SUN M, SUN M. Genetic diversity in three colonizing orchids with contrasting mating systems[J]. American Journal of Botany, 1997, 84(2): 224-232. DOI:10.2307/2446084.
[22] EL-ESAWI M A, GERMAINE K, BOURKE P, et al. Genetic diversity and population structure of Brassica oleracea germplasm in Ireland using SSR markers[J]. Comptes Rendus Biologies, 2016, 339(3/4): 133-140. DOI:10.1016/j.crvi.2016.02.002.
[23] SONKAR S, SINGH S K, VENNELA P R, et al. Molecular marker based genetic diversity analysis in rice (Oryza sativa L.) using SSR markers[J]. Euphytica, 2016, 9(1): 45-51. DOI:10.5958/2230-732X.2016.00007.3
[24] KRISHNAMURTHY S L, SHARMA S K, KUMAR V. Analysis of genomic region spanning Saltol using SSR markers in rice genotypes showing differential seedlings stage salt tolerance[J]. Journal of Plant Biochemistry and Biotechnology, 2016, 25(3): 331-336. DOI:10.1007/ s13562-015-0335-5.
[25] 王丽侠, 程须珍, 王素华, 等. 小豆SSR引物在绿豆基因组中的通用性分析[J]. 作物学报, 2009, 35(5): 816-820. DOI:10.3724/ SP.J.1006.2009.00816.
[26] WANG Xiaoqiang, KWON S W, PARK Y J. Comparison of population genetic structures between Asian and American mungbean accessions using SSR markers[J]. Journal of Agricultural Science, 2012, 4(9): 150-158. DOI:10.5539/jas.v4n9p150.
[27] BROWN S, HIGHAM T, SLON V, et al. Identification of a new hominin bone from Denisova Cave, Siberia using collagen fingerprinting and mitochondrial DNA analysis[J]. Scientific Reports, 2016, 6: 23559. DOI:10.1038/srep23559.
[28] KOSMOWSKI F, ARAGAW A, KILIAN A, et al. Varietal Identification in household surveys: results from an experiment using DNA fingerprinting of sweet potato leaves in Southern Ethiopia[J]. Policy Research Working Paper, 2016, 7812: 1-25.
[29] SETIMELA P S, WARBURTON M L, ERASMUS T. DNA fingerprinting of open-pollinated maize seed lots to establish genetic purity using simple sequence repeat markers[J]. South African Journal of Plant and Soil, 2016, 33(2): 1-8. DOI:10.1080/02571862.2015.1104737.
[30] 麻丽颖, 孔德仓, 刘华波, 等. 36 份枣品种SSR指纹图谱的构建[J]. 园艺学报, 2012(4): 647-654. DOI:10.16420/j.issn.0513-353 x.2012.04.007.
Genetic Diversity Analysis and DNA Fingerprint Construction Based on Fluorescent Labeled SSR Markers for Mungbean Varieties (Vigna radiate L.) from Inner Mongolia
ZHAO yanan1, WANG ying1,2, ZHANG Dongjie1,*, WANG Lixia3,*, ZUO Zhaohang1(1. College of Food Science, Heilongjiang Bayi Agricultural Universitiy, Daqing 163319, China; 2. National Coarse Cereals Engineering Research Center, Daqing 163319, China; 3. Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China)
Objective: The aim of this study was to analyze the genetic diversity of mungbean varieties from Inner Mongolia region and to establish a simple sequence repeat (SSR) fingerprinting database for providing scientific evidence to explore the genetic background of mungbean germplasms and authenticate mungbean varieties. Methods: A total of 92 mungbean varieties from Inner Mongolia area were taken to establish their genetic diversity and DNA fingerprint maps by using 10 pairs of SSR primers. Results: A total of 58 alleles were detected, and each pair of primers detected 3–10 alleles with an average of 5.8. The genetic diversity coefficients among the 92 mungbean varieties varied from 0.033 4–1.000 0 with an average of 0.46. Unweighted pair-group method with arithmetic means (UPGMA) clustering analysis showed that these mungbean varieties were clustered into 2 groups at theof genetic similarity coefficient of 0.294 4. SSR fingerprinting database and molecular identification of the 92 mungbean cultivars were established, and it was found that most of the tested varieties, with different SSR fingerprints, which could be molecularly discriminated from each other, could serve as cultivars-specific patterns and as an important basis for cultivar identification. Conclusions: There was a high level of genetic diversity among mungbean germplasms from Inner Mongolia, but a certain degree of inbreeding also existed. Therefore, further efforts are needed to develop new mungbean breeding materials. SSR fingerprinting and molecular identification using fluorescent labeled SSR marker technique is of great significance for genetic authentication, identity validation, traceability management and origin protection of mungbean varieties.
10.7506/spkx1002-6630-201716007
S522
A
1002-6630(2017)16-0043-08
2017-01-08
“十二五”农村领域国家科技计划项目(2012BAD34B02);黑龙江省应用技术与开发重大项目(GA14B104);
大庆科技局创新项目(sjh-2013-65);黑龙江省研究生创新科研项目(YJSCX2017-Y50)
赵雅楠(1993—),女,硕士研究生,研究方向为农产品加工及贮藏工程。E-mail:zyn658@163.com
*通信作者:张东杰(1966—),男,教授,博士,研究方向为农产品加工及贮藏工程。E-mail:byndzdj@126.com
王丽侠(1972—),女,副研究员,博士,研究方向为食用豆种质资源。E-mail:wanglixia03@caas.cn
赵雅楠, 王颖, 张东杰, 等. 内蒙古地区绿豆品种遗传多样性SSR分析及DNA指纹图谱构建[J]. 食品科学, 2017, 38(16): 43-50. DOI:10.7506/spkx1002-6630-201716007. http://www.spkx.net.cn
ZHAO Yanan, WANG Ying, ZHANG Dongjie, et al. Genetic diversity analysis and DNA fingerprint construction based on fluorescent labeled SSR markers for mungbean varieties (Vigna radiate L.) from Inner Mongolia[J]. Food Science, 2017, 38(16): 43-50. (in Chinese with English abstract)
10.7506/spkx1002-6630-201716007. http://www.spkx.net.cn
Key words: fluorescent labeled SSR markers; mungbean varieties; genetic diversity analysis; fingerprint construction

