猪7种腹泻相关病毒的临床检测及猪嵴病毒的遗传进化分析
孟丽,陶洁,李本强,马玉飞,程靖华,张春玲,刘惠莉
1 上海市农业科学院畜牧兽医研究所,上海种猪工程技术研究中心,上海 201106 2 上海海洋大学 水产与生命学院,上海 201306
猪7种腹泻相关病毒的临床检测及猪嵴病毒的遗传进化分析
孟丽1,2,陶洁1,李本强1,马玉飞1,2,程靖华1,张春玲1,刘惠莉1
1 上海市农业科学院畜牧兽医研究所,上海种猪工程技术研究中心,上海 201106 2 上海海洋大学 水产与生命学院,上海 201306
孟丽, 陶洁, 李本强, 等. 猪 7种腹泻相关病毒的临床检测及猪嵴病毒的遗传进化分析. 生物工程学报, 2017, 33(8):1292-1303.
Meng L, Tao J, Li BQ, et al. Clinical detection of seven porcine diarrhea-associated viruses and evolution analysis of porcine kobuvirus. Chin J Biotech, 2017, 33(8): 1292-1303.
为了解引起猪腹泻疫病的病毒性病原种类及感染现状,本研究首先针对 7种报道较少但可引起猪腹泻的病毒性病原建立了多重RT-PCR检测方法,包括猪捷申病毒 (PTV)、猪萨佩罗病毒 (PSV)、猪丁型冠状病毒 (PDCoV)、猪嵴病毒 (PKV)、猪札幌病毒 (PSaV)、猪星状病毒 (PAstV)和猪环曲病毒 (PToV)。利用该方法对419份临床腹泻粪便样品进行筛检,结果显示PKV检出率最高,阳性率达26.98%–45.79%;PKV和其他病原混合感染率达9.52%–18.54%。基于PKV的高检出率及其可能在猪腹泻疾病中发挥的作用,本试验进一步选取3个PKV阳性样品进行全基因组序列测定及遗传进化分析。结果表明,这3个阳性样品PKV均归属于猪嵴病毒属,且与其他动物嵴病毒遗传距离较远,分别标记为CM-PKV、JS-PKV和PD-PKV;它们的全基因组同源性为88.1%–89.1%;CM-PKV与2013年报道的中国株JS-02a-CHN/2013同源性最高,而JS-PKV和PD-PKV则与2008年报道的匈牙利株K-30-HUN/2008/HUN同源性最高。表明上海不同地区猪群中存在的PKV有明显的遗传差异,但毒株遗传特性的差异与其致病力的相关性需进一步研究。本研究为深入了解PKV的流行现状及探讨其在猪腹泻疫情中的作用提供了参考。
多重RT-PCR,猪嵴病毒,全基因组序列,遗传进化分析
近年来,猪腹泻病的流行越来越广,且疾病的复杂程度日益增加,给我国畜牧业造成了严重的经济损失。猪流行性腹泻病毒 (Porcine epidemic diarrhea virus, PEDV)、猪传染性胃肠炎病毒 (Transmissible gastroenteritis virus, TGEV)和猪轮状病毒 (Porcine rotavirus, PoRV) 是引起猪腹泻的三种重要的病毒性病原[1]。但近年来不断报道有新的腹泻病原出现,导致猪腹泻病原复杂化,疾病防控难度增加。兰道亮等[2]报道华东地区猪群中猪萨佩罗病毒 (Porcine sapelovirus,PSV) 平均阳性率达 17.2%。蔡雨涵等[3]报道湖北、湖南、河南省各大猪场中猪博卡病毒(Porcine bocavirus, PBoV) 的阳性率达73.95%。刘玲玲等[4]利用RT-PCR方法对江西省地区腹泻粪便检测,结果显示腹泻样本中猪丁型冠状病毒 (Porcine deltacoronavirus, PDCoV) 的检出率为31.33%。Xiao等[5]检测湖南猪场中猪星状病毒 (Porcine astrovirus, PAstV) 感染率达11.5%。猪嵴病毒 (Porcine kobuvirus, PKV) 也广泛存在于猪群中,其主要临床症状表现为胃肠炎,因此容易与TGEV相混淆,从而导致所谓的“疫苗使用效果不佳”[6]。所以,开展猪病毒性腹泻病原调查十分必要,有助于对猪腹泻病的针对性治疗。
猪嵴病毒是小RNA病毒科嵴病毒属成员,该病毒于 2007年首次在匈牙利健康猪腹泻粪便样品中发现。PKV主要危害3周龄以下的仔猪,可引起仔猪胃肠炎,出现严重腹泻,甚至死亡[7]。PKV呈地方性流行,中国、泰国、韩国、日本、新西兰及巴西等[8-13]均有PKV感染的相关报道。PKV与其他病毒混合感染现象也较普遍[14],不仅会促进仔猪腹泻疾病的发生,同时还会加重病情,其感染也不仅仅局限于肠道,还可以侵入血液并造成病毒血症。国内从2009年开始PKV阳性检出率呈逐年递增的趋势。很多学者推断PKV和目前的猪腹泻有着很大的关系,但也有报道指出PKV在无腹泻症状猪群中的阳性率比腹泻症状猪群还要高[15]。因此,对猪群中PKV监测并进一步了解其致病特性和遗传规律具有重要意义。
本研究首先建立了针对7种报道较少的猪病毒性腹泻相关病原的多重RT-PCR检测方法,通过对临床样品的检测,明确了上海地区PKV的感染率及其混合感染情况;进一步测定了不同猪场来源的PKV毒株的全基因组序列,并分析其遗传进化规律,为后续深入研究PKV的致病性奠定基础。
1 材料与方法
1.1 材料
1.1.1 病毒及病料样品采集
PEDV、TGEV、猪瘟病毒 (Classical swine fever virus, CSFV)、猪源牛病毒性腹泻病毒(Bovine viral diarrhea virus, BVDV)、猪流感病毒(Swine influenza virus, SIV) 均由上海市农业科学院畜牧兽医研究所分离保存。
419份粪便样品采自上海及周边猪场中有腹泻症状的猪。
1.1.2 主要试剂和仪器
100 bp DNA ladder marker、pMD-18 T 载体、rTaq DNA聚合酶、TRIzol、M-MLV (RNase H-)、dNTPs均购自 TaKaRa公司;普通琼脂糖凝胶 DNA回收试剂盒和质粒提取试剂盒购自Tiangen公司;DH5α菌株由本实验室保存,其他常规试剂均为国产分析纯级产品。
主要仪器包括梯度PCR仪 (ABI)、低温台式高速离心机 (Beckman)、涡旋振荡仪 (Vision)、凝胶成像分析系统 (Bio-Rad)、Q5000蛋白核酸测定仪等。
1.2 方法
1.2.1 引物设计
参照文献[2-6],根据GenBank中已有PKV(EU787450、KT892971、JX401523、KM977675)、PAstV (JX556692、KU764486、JX556693、JX556691)、PSaV (KT922089、FJ387164、KT922087、KF204570)、PDCoV (KJ769231、KR265865、KT021234、KX022605)、PToV(GU196786、JQ740195、FJ555569)、PTV(AF231767、AF296096、GU446660、JQ664746、AJ516011)、PSV (HQ875059、KJ821020、KJ821021) 全基因组序列,通过比较其保守区域,利用Oligo7.37软件分别针对PTV 3D基因、PKV 3D基因、PDCoV M基因、PSV 5′UTR基因、PSaV ORF1基因、PAstV ORF2基因、PToV S基因设计7对特异性检测引物 (表1)。引物由生工生物工程 (上海) 股份有限公司合成。
1.2.2 样品处理与RNA提取
采集的粪便溶于适量 PBS中,反复冻融2次,12 000 r/min离心2 min,吸取250 μL上层样品进行总RNA的提取,并反转录成cDNA。

表1 猪7种病毒性腹泻病原的多重PCR引物序列Table 1 Multiplex PCR primers of the seven porcine diarrhea-associated viruses
1.2.3 多重RT-PCR的建立与优化
以 cDNA为模板,分别利用相对应的病原检测引物进行单一RT-PCR扩增,并克隆各目的片段,测序鉴定,制备阳性质粒模板。
进一步将 7种分别含有特定病原基因片段的质粒的混合物作为模板,进行多重RT-PCR反应条件的优化,包括最佳退火温度、引物浓度、多重RT-PCR特异性和灵敏性等。
1.2.4 临床样品检测
用建立的多重RT-PCR方法对临床419份腹泻样品进行检测。
1.2.5 全基因组序列的测定
参照文献[16],合成8对PKV全基因组扩增引物,选取3例阳性样本分别进行基因组序列的测定,得到3株PKV全基因组。其中5′末端序列利用5′ RACE kit测定。每个片段送样3份,由生工生物工程 (上海) 股份有限公司测序。
1.2.6 序列分析
应用Lasergene分析软件对各测定片段进行拼接,获得PKV全基因组序列并进行分析,利用MEGA6.0软件制作进化树分析其亲缘性。
2 结果与分析
2.1 阳性质粒的克隆和鉴定
分别利用 7种腹泻病原的检测引物,对临床采集样品进行检测,获得扩增出阳性目的片段的样品 (图 1)。进一步回收各目的片段,利用 pEASY-T1 Simple Cloning Kit进行“TA”连接,然后转化DH5α。通过菌液PCR方法鉴定,获得疑似阳性质粒 pEASY-PTV/3D、pEASY-PKV/3D、pEASY-PDCoV/M、pEASYPSV/5′UTR、pEASY-PSaV/ORF1、pEASY-PAstV/ORF2、pEASY-PToV/S。测序证实,各片段均与相应的腹泻病原序列吻合,获得正确的阳性质粒。
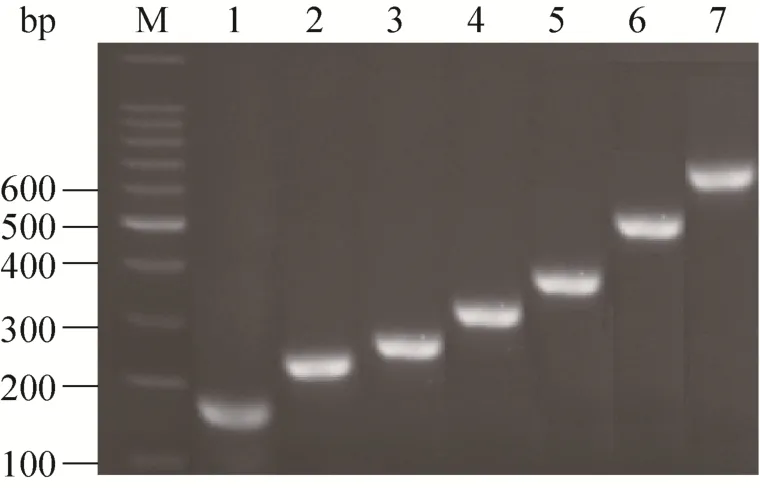
图1 猪7种病毒性腹泻病原的目的片段扩增Fig. 1 Specific fragments amplification of the seven porcine diarrhea-associated viruses. M: 100 bp DNA marker; 1: PTV 3D fragment; 2: PAstV ORF2 fragment;3: PKV 3D fragment; 4: PSaV ORF1 fragment; 5:PToV S fragment; 6: PDCoV M fragment; 7: PSV 5′UTR fragment.
2.2 多重PCR的建立
将 7种阳性质粒模板逐渐增加,依次进行多重RT-PCR扩增,最终建立了7重RT-PCR方法 (图 2),7个目的片段大小依次为 624 bp、504 bp、421 bp、340 bp、280 bp、249 bp 和176 bp,大小正确,条带特异。进一步优化退火温度,如图3所示,退火温度为52 ℃、55 ℃、58 ℃和61 ℃时,7重PCR均能扩增成功;其中,当退火温度为58 ℃时,PCR扩增条带最清晰。
2.3 多重RT-PCR特异性和灵敏性的鉴定
2.3.1 特异性试验
用建立的多重 RT-PCR方法检测 PEDV、TGEV、CSFV、BVDV、SIV,结果如图4所示,没有扩增出片段;而以含有PTV、PKV、PDCoV、PSV、PSaV、PAstV、PToV特异性目的片段的阳性质粒为模板进行多重 RT-PCR,扩增出了7条特异的目的片段;证实该方法特异性较好(图 4)。
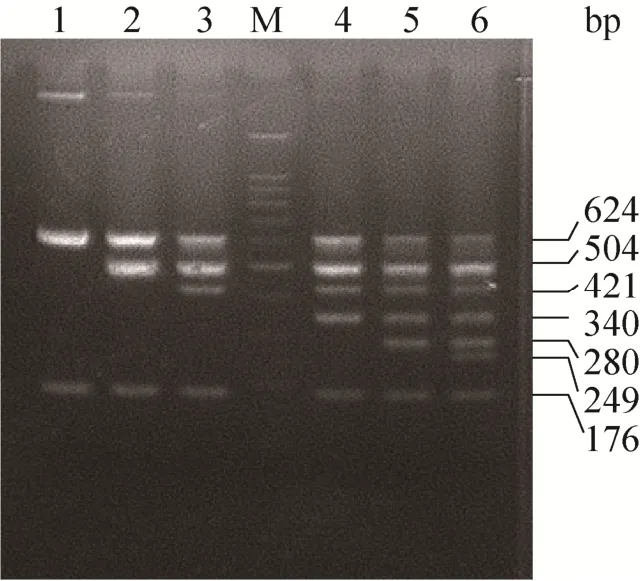
图2 多重RT-PCR的建立Fig. 2 Development of the multiplex RT-PCR. M:100 bp DNA marker; 1: multiplex RT-PCR for PTV and PSV; 2: multiplex PCR for PTV, PDCoV and PSV; 3:multiplex RT-PCR for PTV, PToV, PDCoV and PSV; 4:multiplex RT-PCR for PTV, PSaV, PToV, PDCoV and PSV; 5: multiplex RT-PCR for PTV, PKV, PSaV, PToV,PDCoV and PSV; 6: multiplex RT-PCR for PTV, PAstV,PKV, PSaV, PToV, PDCoV and PSV; M: marker.
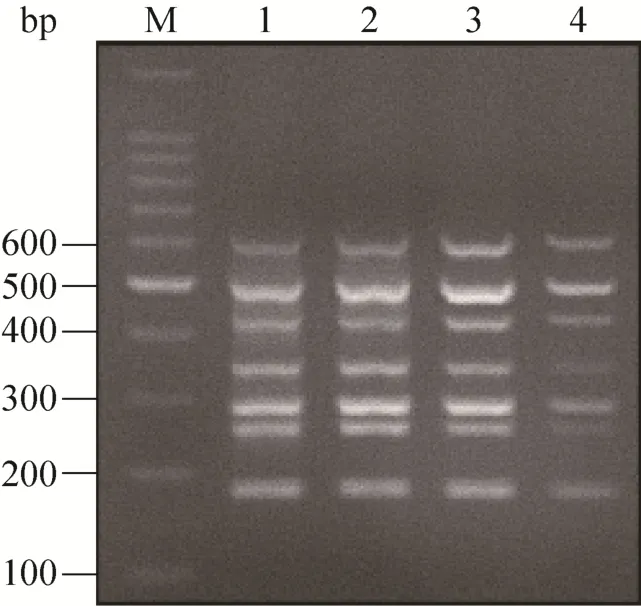
图3 多重RT-PCR的退火温度筛选Fig. 3 Temperature screening of the multiplex RT-PCR.1–4: Tm52 ℃, 55 ℃, 58 ℃ and 61 ℃; M: marker.
2.3.2 灵敏性试验
分别以7个阳性质粒模板10倍倍比稀释后,进行多重PCR,检测其灵敏性。结果发现,当质粒稀释至10–5倍时,多重PCR条带很浅。因此,我们判定该多重 PCR方法可检测 PTV、PKV、PDCoV、PSV、PSaV、PAstV和PToV的质粒最低检测限量分别为 5.0×104、5.5×104、6.0×104、6.0×104、3.8×104、6.1×104和 6.9×104copies/μL,对应的最低核酸检测浓度分别为 0.32、0.36、0.41、0.41、0.18、0.39 和 0.43 μg/L (图 4)。
2.3.3 重复性试验
应用多重 RT-PCR对同一阳性模板进行检测,5次试验检测结果均一致,表明本方法的重复性较好。
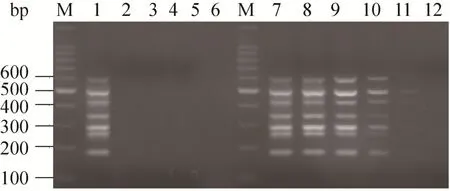
图4 多重RT-PCR特异性和灵敏性鉴定Fig. 4 Specificity and sensitivity of the multiplex RT-PCR. M: 100 bp DNA ladder; 1: multiplex RT-PCR used PTV, PKV, PDCoV, PSV, PSaV, PAstV and PToV as the templates; 2: multiplex RT-PCR used PEDV as the template; 3: multiplex RT-PCR used TGEV as the template; 4: multiplex RT-PCR used BVDV as the template; 5: multiplex RT-PCR used CSFV as the template; 6: multiplex RT-PCR used SIV as the template; 7-12: sensitivity detection of the multiplex RT-PCR with the plasmids of 10-1-10-6.
2.4 多重RT-PCR与单项RT-PCR临床检测结果的比较
利用建立的多重 RT-PCR方法对上海地区采集的419份临床腹泻样品进行检测,结果见表2,其中PKV的感染率最高,2015和2016年分别为26.98%和45.79%。2种以上病原混合感染率较高,其中PKV和其他病原的混合感染率达9.52% (2015年)、18.54% (2016年)。与用单一RT-PCR检测7种腹泻相关病原结果的符合率均为 100%,表明该方法能够快速准确地筛检出这7种腹泻相关病原的混合感染病例。
2.5 PKV全基因组序列的测定
选择来源于不同猪场的PKV阳性样品3例,进行PKV全基因组序列的测定,如图5所示,共有 11个目的片段,大小正确且特异。利用Lasergene和Cluster X软件拼接,获得3株PKV全基因组序列,分别编号为 PD-PKV、JS-PKV和CM-PKV。PD-PKV、JS-PKV和CM-PKV全长分别为8 054 bp、8 053 bp和8 057 bp,均包含 5′UTR、3′UTR 和一个大的 ORF。PD-PKV和 JS-PKV编码一个 2 459 aa的多聚蛋白,CM-PKV编码2 460 aa的多聚蛋白。
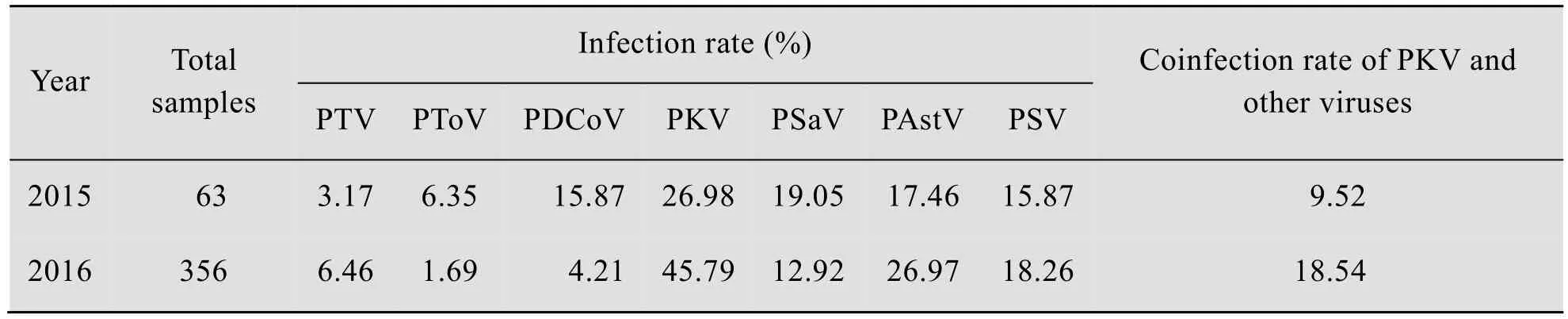
表2 2015-2016年猪病毒性腹泻病原的阳性检出率Table 2 Infection rate of the porcine diarrhea-associated viruses during 2015 to 2016
图5 PKV全基因组PCR扩增Fig. 5 PCR amplification of the complete genomic sequence of PKV.
2.6 全基因组遗传进化分析
从GenBank中选取不同地区、年代、种属的嵴病毒全基因组序列,利用MEGA6.0软件构建全基因组进化树,具体参考毒株见表3。结果发现 (图6),JS-PKV、PD-PKV和JS-PKV均归属于猪嵴病毒,分别与中国北京株 JS-2010a PDPKV、中国甘肃株 K-4/2012/CH、中国江苏株 JS-01-CHN/2013/China的亲缘性最近,与其他动物嵴病毒遗传距离较远。
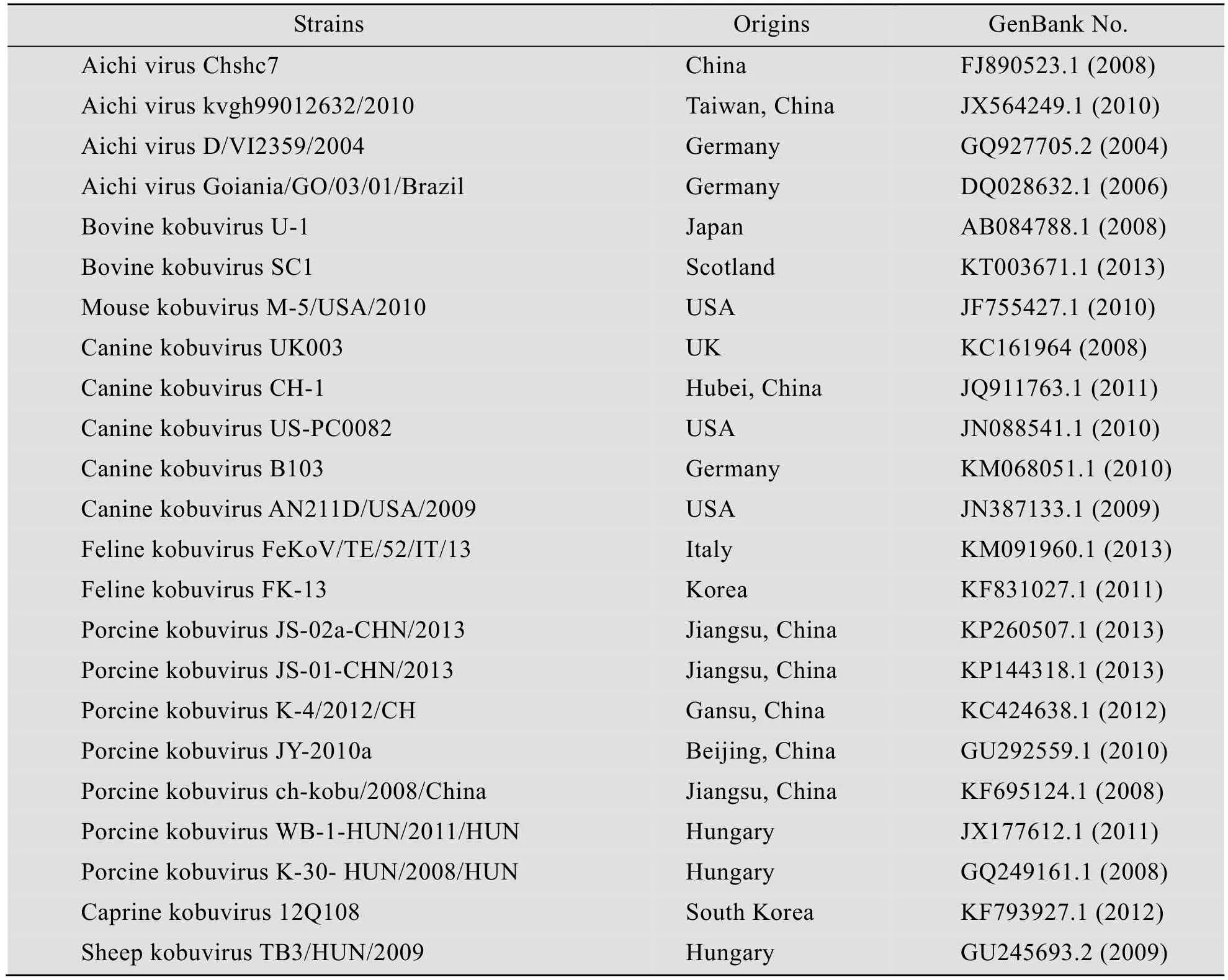
表3 全基因组进化树参考毒株Table 3 Reference strains of the phylogenetic tree based on the complete genomic sequences
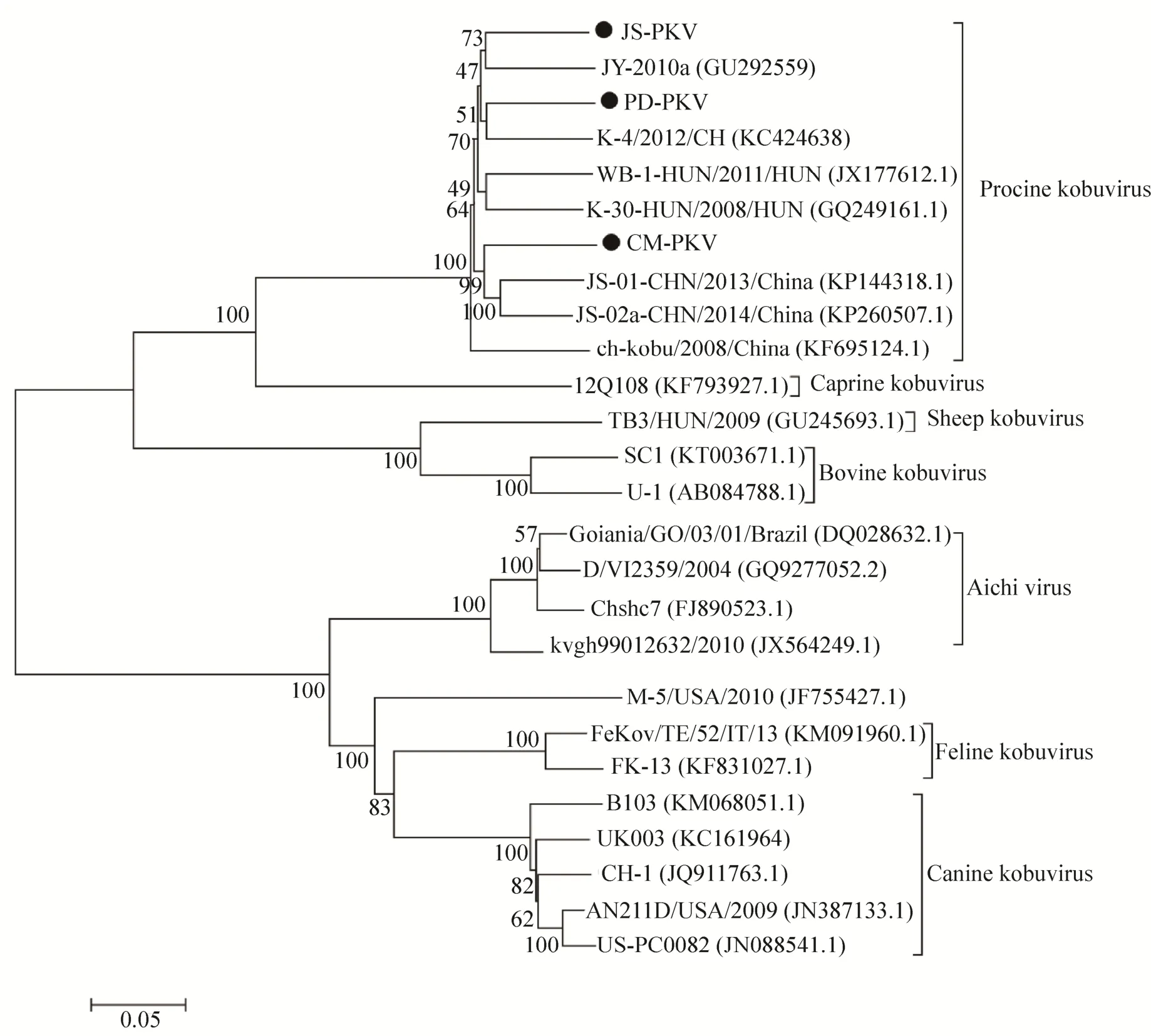
图6 PKV全基因组进化树分析Fig. 6 Phylogenetic tree based on the complete genomic sequences of PKV.
这3株PKV的全基因组序列同源性为88.1%-89.1% (图 7),CM-PKV 和中国株 JS-02a-CHN/2013/China同源性最高 (89.5%),而JS-PKV和 PD-PKV则与匈牙利株 K-30-KUN/2008/HUN的同源性最高 (89.6%),说明近年来不同国家流行的猪嵴病毒之间存在着一定的相似性。
2.7 VP1基因的遗传进化分析
VP1是PKV的主要结构蛋白,具有较大变异性。通过构建VP1基因的进化树 (图8),我们发现虽然JS-PKV、PD-PKV和CM-PKV处于不同分支,但它们均与日本株的亲缘性最近;JS-PKV、CM-PKV分别与 2007年日本报道的CMP03/07株、CMP17/07-THA株处于同一分支,而PD-PKV与2009年日本报道的T247/2009/JP株处于同一分支。说明本试验中的3株PKV很有可能是由日本株变异而来。

图7 PKV全基因组序列的同源性比对Fig. 7 Identity analyzation of the complete genomic sequences of PKV.
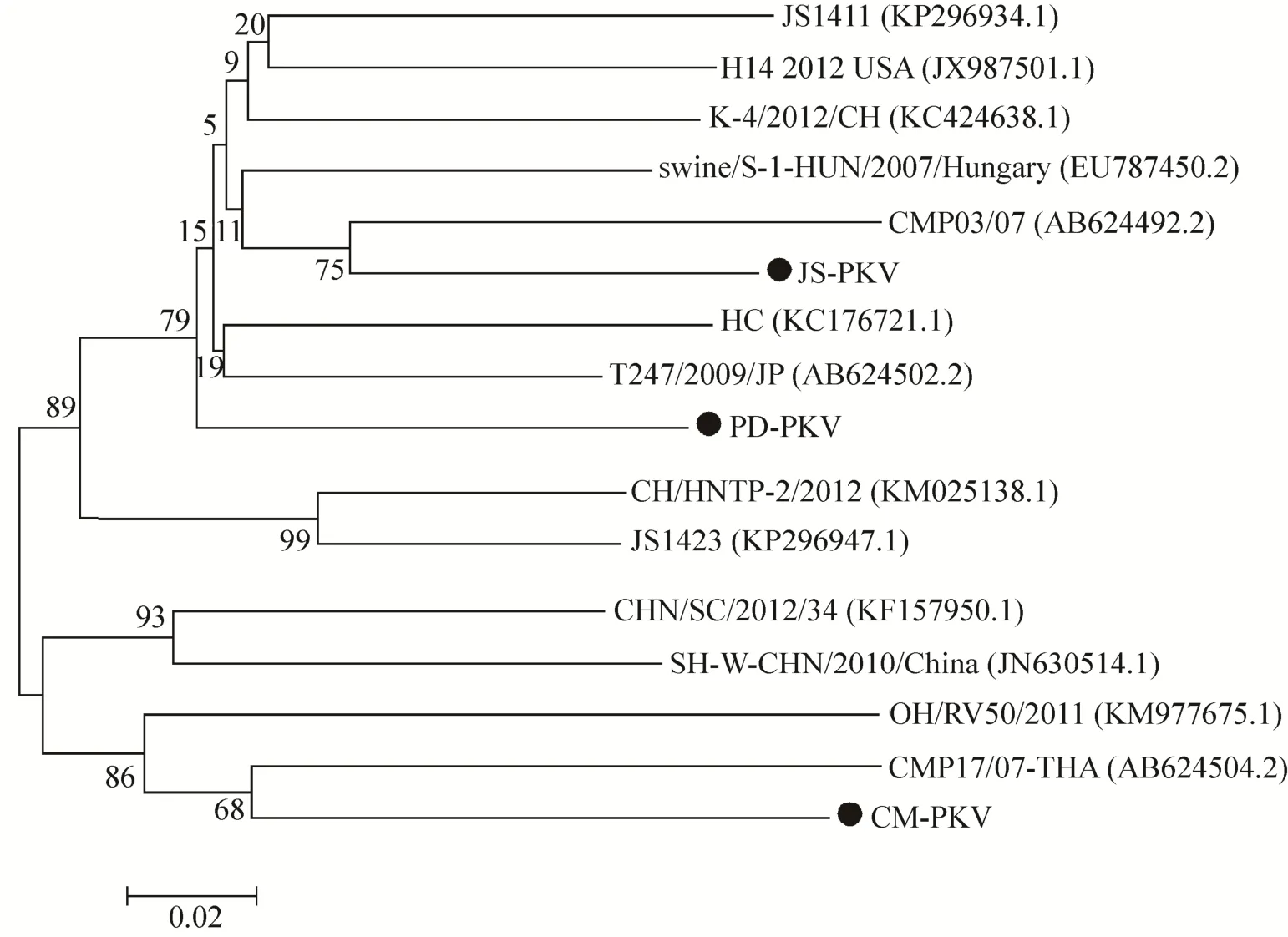
图8 VP1基因的进化树分析Fig. 8 Phylogenetic tree based on the VP1 genes.
3 讨论
近年来,猪病毒性腹泻流行甚广,对引起腹泻病原的诊断、检测技术也有很多报道。特别是多病原检测方法研究较多。目前,关于 PEDV、TGEV和PoRV等腹泻病原的多重RT-PCR方法较多[1,17-21],且多是3重RT-PCR或4重RT-PCR。本研究首次建立了可同时检测 7种猪病毒性腹泻病原的多重 RT-PCR方法。该方法特异性较好,只可特异性扩增出 PTV、PKV、PDCoV、PSV、PSaV、PAstV和PToV的条带,不会出现其他病毒性腹泻病原的交叉扩增现象;最低核酸检测浓度分别为0.32、0.36、0.41、0.18、0.39和0.43 μg/L,与已有多重 RT-PCR方法[22-26]相比,灵敏性较高。利用该方法对临床腹泻样品进行筛检,共检出PKV阳性样品180例、PTV和PDCoV阳性各25例、PToV阳性10例、PAstV阳性108例、PSV阳性75例;PKV和其他病原混合感染 72例。测序证实PCR检测正确。说明本研究建立的7重RT-PCR方法可适用于临床大批量样品的筛查。
鉴于PKV的感染率较高,研究进一步选取了 3个地区的 PKV毒株进行全基因组序列测定。结果表明,JS-PKV、PD-PKV和CM-PKV的基因组构成符合PKV的基因组结构,依次分为 5′ UTR、一个大的 ORF和3′ UTR。除 polyA尾巴外,它们的全基因组长度依次为8 053 bp、8 054 bp和 8 057 bp;G+C百分含量依次为52.07%、52.04%和52.09%;A+T百分含量依次为 47.93%、47.96%和 47.91%。它们的全基因组同源性不高,仅达 88.1%–89.1%,说明上海不同地区流行的PKV毒株之间存在着一定的变异。与参考毒株相比,JS-PKV和 PD-PKV与匈牙利株K-30-KUN/2008/HUN的同源性最高,达89.6%;CM-PKV和中国株JS-02a-CHN/2013/China同源性最高,达 89.5%。全基因组进化树表明,这3株PKV均与中国株的亲缘性最近;但VP1基因进化树表明,它们与日本株PKV的亲缘性最近。由此推测,近年来我国流行的PKV和日本株PKV之间有着一定的相似性,很有可能是从日本株变异而来。后续我们将开展这3株PKV的致病性及其在腹泻疾病中的作用研究。
REFERENCES:
[1] Ogawa H, Taira O, Hirai T, et al. Multiplex PCR and multiplex RT-PCR inclusive detection of major swine DNA and RNA viruses in pigs with multiplex infections. J Virol Methods, 2009,160(1/2): 210–214.
[2] Lan DL, Ji WH, Wang CS, et al. Molecular epidemiology investigation of porcine sapelovirus in pigs in East China. Prog Vet Med, 2012, 33(12):116–121 (in Chinese).兰道亮, 吉文汇, 王长松, 等. 华东部分地区猪群中猪萨佩罗病毒分子流行病学调查. 动物医学进展, 2012, 33(12): 116–121.
[3] Cai YH, Zhu L, Zhou YC, et al. Development and application of the duplex PCR for detection of porcine bocavirus. Chin Vet Sci, 2013, 43(8):833–838 (in Chinese).蔡雨涵, 朱玲, 周远成, 等. 猪博卡病毒双重PCR检测方法的建立及其应用. 中国兽医科学,2013, 43(8): 833–838.
[4] Liu LL, Cao BB, Zhang LW, et al. Establishment and preliminary application of duplex RT-PCR for detection of emerging porcine deltacoronavirus and porcine transmissible gastroenteritis virus. J Agricul Biotechnol, 2016, 24(12): 1955–1963 (in Chinese).刘玲玲, 曹贝贝, 张利卫, 等. 新发 PDCoV 和TGEV双重RT-PCR检测方法的建立及初步应用.农业生物技术学报, 2016, 24(12): 1955–1963.
[5] Xiao CT, Luo Z, Lv SL, et al. Identification and characterization of multiple porcine astrovirus genotypes in Hunan province, China. Arch Virol,2017, 162(4): 943–952.
[6] Jackova A, Sliz I, Mandelik R, et al. Porcine kobuvirus 1 in healthy and diarrheic pigs: Genetic detection and characterization of virus and co-infection with rotavirus A. Infect Genet Evol,2017, 49: 73–77.
[7] Jin WJ, Yang Z, Zhao ZP, et al. Genetic characterization of porcine kobuvirus variants identified from heathy pigs in China. Infect Genet Evol, 2015, 35: 89–95.
[8] Yu JM, Jin M, Zhang Q, et al. Candidate porcine kobuvirus, China. Emerg Infect Dis, 2009, 15(5):823–825.
[9] Wang C, Lan D, Hua X. Porcine kobuvirus from pig stool specimens in Shanghai, China. Virus Genes, 2011, 43(3): 350–352.
[10] Khamrin P, Maneekarn N, Kongkaew A, et al.Porcine kobuvirus in piglets, Thailand. Emerg Infect Dis, 2009, 15(12): 2075–2076.
[11] Park SJ, Kim HK, Moon HJ, et al. Molecular detection of porcine kobuvirus in pigs in Korea and their association with diarrhea. Arch Virol,2010, 155(11): 1803–1811.
[12] Khamrin P, Maneekarn N, Hidaka S, et al.Molecular detection of kobuvirus sequences in stool samples collected from heathy pigs in Japan.Infect Genet Evol, 2010, 10(7): 950–954.
[13] Barry AF, Ribeiro J, Alfieri AF, et al. First detection of kobuvirus in farm animals in Brazil and the Netherlands. Infect Genet Evol, 2011,11(7): 1811–1814.
[14] Zhao ZP, Yang Z, Lin WD, et al. The rate of co-infection for piglet diarrhea viruses in China and the genetic characterization of porcine epidemic diarrhea virus and porcine kobuvirus.Acta Virol, 2016, 60(1): 55–61
[15] Phan NT, Trinh QD, Khamrin P, et al. Sequence analysis of the capsid gene of Aichi viruses detected from Japan, Bangladesh, Thailand, and Vietnam. J Medical Virol, 2008, 80(7): 1222–1227.
[16] Peng Q, Song D, Huang D, et al. Complete genome sequence of a porcine kobuvirus variant strain from Jiangxi, China. Genome Announc,2017, 5(5): pii: e01580-16.
[17] Ben Salem AN, Chupin Sergei A, Bjadovskaya Olga P, et al. Multiplex nested RT-PCR for the detection of porcine enteric viruses. J Virol Methods, 2010, 165(2): 283–293.
[18] Zhao J, Shi BJ, Huang XG, et al. A multiplex RT-PCR assay for rapid and differential diagnosis of four porcine diarrhea associated viruses in field samples from pig farms in East China from 2010 to 2012. J Virol Methods, 2013, 194(1/2): 107–112.
[19] Song DS, Kang BK, Oh JS, et al. Multiplex reverse transcription-PCR for rapid differential detection of porcine epidemic diarrhea virus,transmissible gastroenteritis virus, and porcine group A rotavirus. J Vet Diagn Invest, 2006, 18(3):278–281.
[20] Ren YP, Zhang B, Tang C, et al. Development and application of a multiplex RT-PCR for detecting PEDV, TGEV and PDCoV. Chin Vet Sci, 2016,46(6): 756–762 (in Chinese).任玉鹏, 张斌, 汤承, 等. 同时检测 PEDV 和TGEV及PDCoV的多重RT-PCR方法的建立及初步应用. 中国兽医科学, 2016, 46(6): 756–762.
[21] Xiong NN, Tao J, Zhang QZ, et al. Establishment and application of the multiplex PCR for detection of four porcine viral diarrhea viruses.Chin J Vet Sci, 2016, 46(8): 972–978 (in Chinese).熊年年, 陶洁, 张清真, 等. 猪四种病毒性腹泻病原多重PCR检测方法的建立及应用. 中国医科学, 2016, 46(8): 972–978.
[22] Li LM, Zhu WX, Wang XB, et al. Establishment and application of a multiplex reverse transcription-PCR for detecting porcine epidemic diarrhea virus, porcine transmissible gastroenteritis virus and porcine group A rotavirus. Chin J Vet Sci,2016, 36(2): 216–220 (in Chinese).李丽敏, 朱卫霞, 王晓波, 等. 猪流行性腹泻病毒、猪传染性胃肠炎病毒、猪A型轮状病毒多重R-PCR方法的建立及其应用. 中国兽医学报,2016, 36(2): 216–220.
[23] Long FX, Shi KC, Zhang Z, et al. Establishment and application of a multiplex RT-PCR assay for detection of PEDV, TGEV and PRoV. Chin Anim Husb & Vet Med, 2016, 43(10): 2518–2526 (in Chinese).龙飞翔, 施开创, 张珍, 等. PEDV、TGEV 和PRoV多重RT-PCR检测方法的建立及应用. 中国畜牧兽医, 2016, 43(10): 2518–2526.
[24] Yang WY, Zhou YC, Han Y, et al. Establishment and clinical application of a multiplex reverse transcription-PCR for simultaneous detection of porcine group A rotavirus, porcine group C rotavirus and porcine astrovirus. Chin Vet Sci,2014, 44(4): 394–400 (in Chinese).杨文宇, 周远成, 韩燕, 等. 猪A群轮状病毒和C群轮状病毒以及猪星状病毒多重 RT-PCR同时检测方法的建立及应用. 中国兽医科学, 2014,44(4): 394–400.
[25] Gu F, Zhou YC, Huang JB, et al. Establishment and clinical application of a multiplex RT-PCR for detection of porcine kobuvirus, porcine astrovirus and porcine torovirus. Chin Vet Sci, 2015, 45(7):661–667 (in Chinese).顾凡, 周远成, 黄剑波, 等. 猪嵴病毒和猪星状病毒及猪环曲病毒多重 RT-PCR检测方法的建立及临床应用. 中国兽医科学, 2015, 45(7):661–667.
[26] Xu YD, Zhang QX, Li JL, et al. Establishment and application of multiple RT-PCR method for diagnosis of PEDV, TGEV and RV infection. Chin J Vet Drug, 2014, 48(11):10–13 (in Chinese).徐引弟, 张青娴, 李建林, 等. PEDV、TGEV、RV多重RT-PCR检测方法的建立与应用. 中国兽药杂志, 2014, 48(11): 10–13.
(本文责编 陈宏宇)
Clinical detection of seven porcine diarrhea-associated viruses and evolution analysis of porcine kobuvirus
Li Meng1,2, Jie Tao1, Benqiang Li1, Yufei Ma1,2, Jinghua Cheng1, Chunling Zhang1,and Huili Liu1
1 Shanghai Engineering Research Center for Breeding Pigs, Institute of Animal Science and Veterinary Medicine, Shanghai Academy of Agricultural Sciences, Shanghai 201106, China 2 College of Fisheries and Life Sciences, Shanghai Ocean University, Shanghai 201306, China
In order to analyze the epidemic situation of porcine viral diarrhea-associated diseases, a multiplex RT-PCR method was developed for detection of Seven diarrhea-associated viruses, including porcine teschovirus (PTV), porcine sapelovirus(PSV), porcine deltacoronavirus (PDCOV), porcine kobuvirus (PKV), porcine sapovirus (POSAV), porcine astrovirus (PASTV)and porcine torovirus (PTOV). A total of 419 samples were screened using the method, the results showed that PKV has the highest positive rate of 26.98%–45.79% and the mixed infection rate of PKV with others viruses were 9.52%–18.54%. On account of high positive rate of PKV and its potential important role in diarrhea disease, complete genome sequences of three PKV positive samples were further sequenced and genetic evolution were established. Sequences analysis indicated that three PKV-positive samples, designated as PD-PKV, JS-PKV and CM-PKV, respectively, were classified into the porcine kobuvirus genus. The genomic homologies of three viruses were 88.1%–89.1%. CM-PKV had the highest homology with a kobuvirus JS-02a-CHN/2013 reported in 2013, while JS-PKV and PD-PKV were most closed to a kobuvirusv K-30-HUN/2008/HUN reported in Hungary in 2008. This illustrated the significant genetic differences were existed among the PKV isolates in Shanghai.The relationship between the difference among the PKV isolates and the viral pathogenicity still needs to be explored. This research provides references for understanding the prevalence of PKV and its role in swine diarrhea disease.
multiplex RT-PCR, porcine kobuvirus, complete genomic sequences, evolution analysis
March 24, 2017; Accepted: May 10, 2017
Huili Liu. Tel: +86-21-62202473; E-mail: huilil@163.com
Supported by: Shanghai Agricultural Committee Key Project (Nos. 2013-5-5, 2013-3-6), Pig Industry Project of Shanghai Municipal(No. 2016-6).
上海市科技兴农重点攻关项目 (Nos. 2013-5-5,2013-3-6),上海市生猪产业体系项目 (No. 2016-6) 资助。
时间:2017-07-03
:http://kns.cnki.net/kcms/detail/11.1998.Q.20170705.1051.004.html

