抑癌DNA甲基化在乳腺癌早期筛查和辅助诊断中的价值比较:间接比较Meta分析
刘 迪 赵中垚 孙 琦 王友信
(首都医科大学公共卫生学院流行病与卫生统计学系 临床流行病学北京市重点实验室,北京 100069)
抑癌DNA甲基化在乳腺癌早期筛查和辅助诊断中的价值比较:间接比较Meta分析
刘 迪 赵中垚 孙 琦 王友信*
(首都医科大学公共卫生学院流行病与卫生统计学系 临床流行病学北京市重点实验室,北京 100069)
目的 本研究利用间接比较Meta 分析的方法评价在乳腺癌中众多甲基化的基因中价值较大的基因。方法 计算机检索 Cochrane Library、PubMed、EMbase、Chinese BioMedical Literature Database(CBM)、Web of Science、中国知网(China National Knowledge Infrastructure,CNKI)和万方数据库,收集涉及抑癌基因启动子甲基化与乳腺癌关系的Meta研究。采用间接比较Meta分析的方法评价在乳腺癌病人中甲基化的抑癌基因的诊断价值。结果 最终共纳入10个甲基化基因(10个Meta研究),共计130个原始研究。在汇总的Meta分析中,甲基化的同源性磷酸酶-张力蛋白基因(phosphatase and tensin homolog,PTEN)比值比[odds ratio(OR)=66.16(24.48~178.82)]、人类相关转录因子基因(Runt-related transcription factor 3,RUNX3)[OR=28.88(15.48~54.25)]对乳腺癌的风险最大,虽然乳腺癌1号基因(breast cancer 1,BRCA1)[OR=2.11(1.88~2.35)]最小,但是在10个Meta研究中BRCA1的权重是57.1%。根据灵敏度相比之下,RUNX3基因[0.62(0.56~0.67)]最大,其次是脆性组氨酸三联体基因(fragile histidine triad protein,FHIT)[0.59(0.54~0.63)],但是RUNX3基因的特异度[0.95(0.91~0.97)]大于FHIT基因[0.65(0.61~0.69)]。综合甲基化基因的灵敏度和特异度,利用间接比较的方法,RUNX3基因对乳腺癌的诊断价值最大。结论 在乳腺癌的早期筛查和辅助检查中,提倡对RUNX3和BRCA1甲基化的基因加以利用。
乳腺癌;甲基化;诊断试验;间接比较;Meta分析
乳腺癌是女性最常见的恶性肿瘤,我国乳腺癌病死率位居女性恶性肿瘤第5位,而且有逐年上升的趋势,严重威胁女性的身心健康和生活质量[1]。研究[2]表明乳腺癌病人的五年生存率低,早期诊断及早期治疗对于病人的预后有重要意义,而通过筛查从表面健康的无症状人群中发现癌前病变者或早期癌病人更具有重要意义。DNA甲基化是重要的表观遗传修饰,虽不改变DNA序列,但DNA甲基化状态的改变导致基因结构和功能的异常[3]。DNA甲基化被认为是乳腺癌形成的重要分子机制,并且甲基化异常的现象早于明显的恶性表型出现,很多甲基化的基因譬如结肠腺瘤样息肉(adenomatous polyposis coli,APC)、乳腺癌1号(breast cancer 1,BRCA1)、人类相关转录因子(Runt-related transcription factor 3,RUNX3)、Ras相关区域家族亚型1 A(Ras association domain family member 1A,RASSF1A)等[4-8]出现在乳腺癌病人的早期,这为DNA甲基化可以作为乳腺癌早期诊断的生物标志物提供了重要依据。越来越多的抑癌基因启动子区高甲基化在乳腺癌的辅助诊断、疗效观察、预后评估方面的临床意义被众多学者广泛研究,其对于乳腺癌的筛查具有较高的灵敏度和特异度[8]。
但在众多被发现甲基化的基因中作为用于乳腺癌筛查价值较大的甲基化基因尚不清楚,而且目前尚无2种或者多种甲基化基因进行直接比较的结果,笔者利用间接比较的思想评价在乳腺癌中众多甲基化的基因的价值。近年来间接比较的Meta分析或多种干预措施比较的Meta分析(网状Meta分析)被广泛应用[9-10],为本研究提供研究思路和研究方法。甲基化的基因在乳腺癌筛查价值的评价指标为,甲基化的基因对疾病的风险评估[比值比(odds ratio,OR)],诊断的灵敏度、特异度及一致性。本研究旨在采用系统分析方法对甲基化的基因用于乳腺癌筛查的价值大小进行评价,以期为其临床转化应用提供最佳证据。
1 对象与方法
1.1 研究对象
纳入标准:①乳腺癌病人的甲基化基因的Meta分析,所有标本术后均经病理科医师确诊,乳腺浸润性导管癌分级与分期参照乳腺癌世界卫生组织(World Health Organization, WHO)分类和分级标准;基因的甲基化检测采用甲基化特异PCR方法;②基因甲基化诊断乳腺癌的诊断性试验,可提取四格表数据;③Meta分析有异质性检验[11]、敏感性分析、发表偏倚评估、研究质量评价[12]。
评价指标:合并灵敏度、合并特异度、综合灵敏度和特异度的正确指数。
1.2 文献检索
计算机检索 Cochrane Library、PubMed、EMbase、Chinese BioMedical Literature Database(CBM)、Web of Science、中国知网(China National Knowledge Infrastructure,CNKI)和万方数据库,检索时限为1990年1月1日至2016年10月10日。并补充检索纳入文献的参考文献,搜索相关文献如需相关信息,与本领域相关专家或作者联系,获取以上检索时未发现的相关信息。英文检索词包括:breast cancer、methylation、Meta analysis;中文检索词包括乳腺癌、甲基化、Meta分析。并检索涉及的甲基化的基因从Meta截止时间后的研究,并统一分析。
1.3 资料提取与质量评价
由2名评价者独立按照纳入和排除标准筛选文献、提取资料并交叉核对,如遇分歧讨论解决或由第三者研究者协助解决。提取资料包括:①一般信息:文献作者、出版时间、甲基化基因标志物名称、纳入Meta研究个数、纳入样本数、Meta检索截止时间;②诊断参考信息:从原始文献中提取或通过计算获得真阳性(true positive,TP)、真阴性(true negative,TN)、假阳性(false positive,FP)、假阴性(false negative,FN);③检查Meta分析中是否包括异质性检验、敏感性分析、发表偏倚评估、研究质量评价。
1.4 统计学方法
计数资料采用OR值,并计算效应量的95%可信区间(95%confidence interval,95%CI)。由于本研究基于Meta分析筛选的甲基化基因进行间接比较,纳入Meta分析均有异质性检验、敏感性分析、发表偏倚评估、研究质量评价;而且本研究是基于病理诊断的金标准比较各个甲基化的基因的间接比较,符合网状Meta分析的相似性条件;本研究属于网状Meta类型中星形网状Meta分析,不存在直接比较的数据,只存在间接比较的数据,因此不需要进行一致性检验[10]。采用Review manager 5.3软件绘制森林图及呈现间接比较的结果,进行Meta分析中甲基化基因对癌症的风险评估的汇总,对纳入甲基化基因作为乳腺癌诊断标志物的灵敏度和特异度进行分析比较,对纳入甲基化基因诊断的一致性进行分析比较,OR(95%CI)为评价甲基化异常的基因对乳腺癌诊断一致性的比较,若AvsB,OR>1且置信区间不包括1,相对于B,A的诊断价值大;若置信区间包括1,则A和B诊断价值一样;若OR<1且置信区间不包括1,相对于B,A的诊断价值小。检验水准为α=0.05,双侧P<0.05表示差异具有统计学意义。
2 结果
2.1 检索结果
检索相关数据库,最初检索到相关文献33篇,严格依据纳入和排除标准,进一步仔细阅读摘要及全文后剔除22篇,最终有11篇文献符合要求并纳入本研究(图1)。10篇Meta中的原始研究均提供了较为完整的数据资料,如表1为10篇Meta研究的信息。

图1 文献筛选流程及结果

表1 纳入研究的基本特征
2.2 甲基化基因对乳腺癌风险评估及诊断的灵敏度和特异度
如图2,在汇总的Meta分析中,甲基化的同源性磷酸酶-张力蛋白基因(phosphatase and tensin homolog, PTEN)[OR=66.16(24.48~178.82)]、RUNX3 [OR=28.88(15.48~54.25)]对乳腺癌的风险最大,虽然BRCA1[OR=2.11(1.88~2.35)]最小,但是在10个Meta研究中BRCA1的权重是57.1%。根据灵敏度相比之下,RUNX3基因[0.62(0.56~0.67)]最大,其次是FHIT [0.59(0.54~0.63)],但是RUNX3基因的特异度[0.95(0.91~0、97)]大于脆性组氨酸三联体(fragile histidine triad protein,FHIT)基因[0.65(0.61~0.69)]。
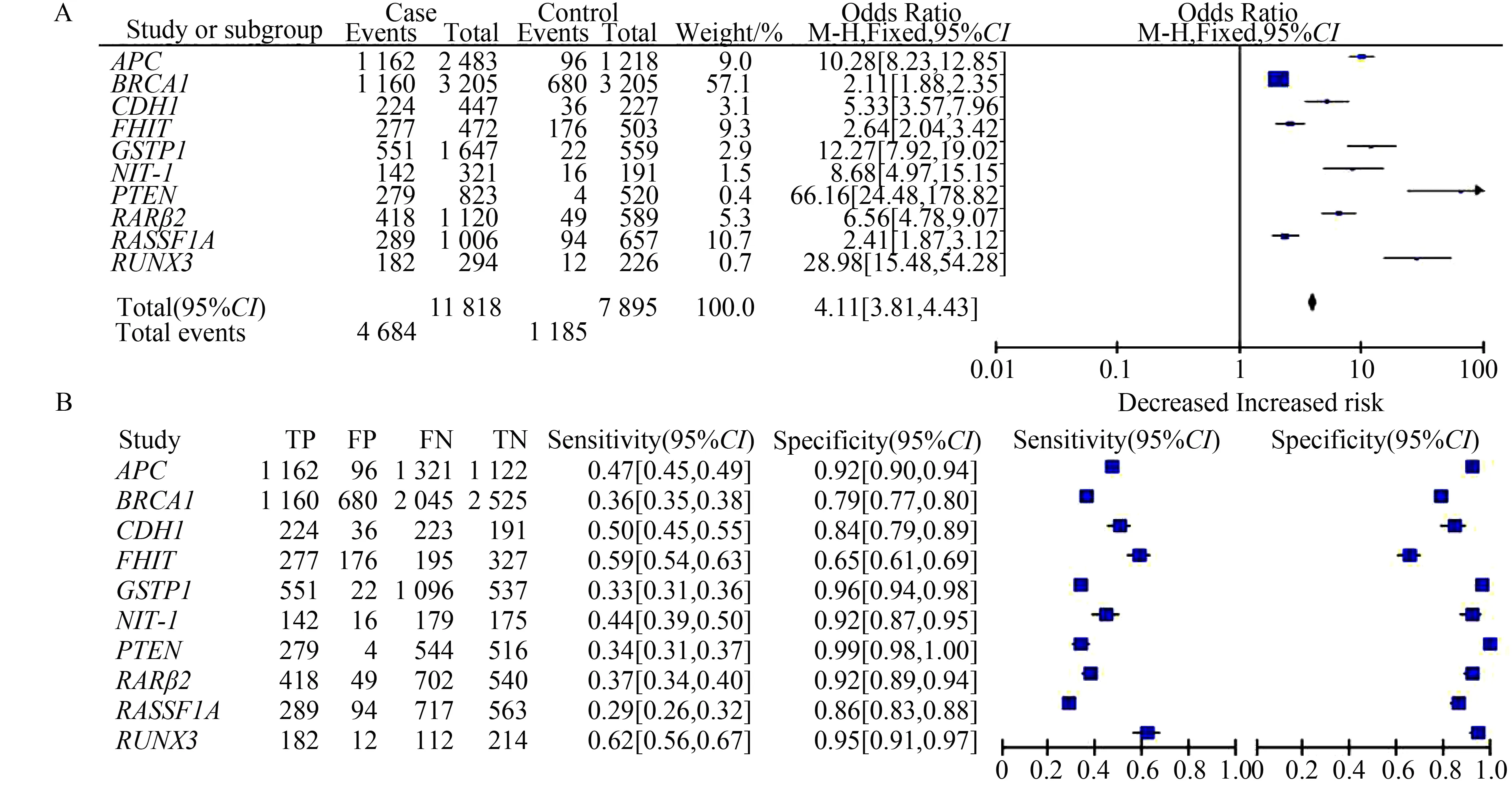
图2 Meta分析的汇总森林图
A: forest plots of Meta analysis of the higher risk of DNA methylation in breast cancer;B: forest plots of Meta analysis of sensitivity and specificity of DNA methylation in breast cancer;APC:adenomatous polyposis coli; BRCA1:breast cancer 1; CDH1:cadherin 1; FHIT:fragile histidine triad protein; GSTP1:glutathione S-transferase pi 1; HIN-1:high in normal-1; PTEN:phosphatase and tensin homolog;RARβ2:retinoic acid receptor beta2;RASSF1A: ras association domain family member 1;RUNX3:Runt-related transcription factor 3; TP:true positive; FP:false positive; FN:false negative;TN:true negative.
2.3 甲基化基因对乳腺癌诊断价值的间接比较
综合甲基化基因的灵敏度和特异度,利用间接比较的方法,RUNX3基因相比于结合珠蛋白基因(high in normal-1,HIN-1)[1.96(1.50~2.57)]、FHIT[1.96(1.54~2.49)]、APC[1.98(1.60~2.45)]、上皮-钙粘连素基因(cadherin 1,CDH1) [1.99(1.55~2.57)]、PTEN[2.20(1.75~2.77)]、BRCA1[2.36(1.92~2.91)]、维甲酸受体β2基因(retinoic acid receptor beta2,RARβ2)[2.50(2.00~3.13)]、RASSF1A3.04[(2.43~3.80)]、谷胱甘肽S-转移酶P1(glutathione S-transferase pi 1,GSTP1) [3.28(2.64~4.08)],对乳腺癌的诊断价值较大,详见表2。
3 讨论
乳腺癌的发生、发展是一个涉及多基因改变和多阶段致癌的复杂过程[13],其中癌基因的激活和抑癌基因的失活在乳腺癌发生、发展过程中发挥着关键作用,其中DNA高甲基化被认为是使抑癌基因沉默的重要分子机制,在乳腺癌形成及其侵袭性不断增强的过程中,参与DNA损伤修复、细胞周期调控、细胞黏附以及细胞信号转导等功能的基因,都可能因为其启动子区CpG岛的甲基化而发生功能失活相关[8]。在笔者汇总的甲基化的基因对乳腺癌的风险评估的Meta中发现了APC、BRCA1、RUNX3、RASSF1A等[4-7, 14-19]10个甲基化的基因与乳腺癌密切相关,这些基因的甲基化不仅发生在乳腺癌癌变前期,而且甲基化的水平与乳腺癌的分期相关。基因异常甲基化不仅在组织能检测到,在病人的血液中亦能检测到,而在正常人的组织或者血液中基因的甲基化率很低[8]。对其甲基化的基因进行早期筛查可以早期发现乳腺癌病人或癌前病变,提高筛查的灵敏度,尽可能较多地发现可疑病人,因此寻找对乳腺癌诊断灵敏度较高的甲基化基因。利用间接比较的方法评估对乳腺癌诊断价值较大的甲基化的基因,笔者发现RUNX3基因对乳腺癌诊断的灵敏度[(0.62(0.56~0.67)]最大,特异度[(0.95(0.91~0.97)],综合灵敏度和特异度,相比于其他9个甲基化的基因,RUNX3基因的诊断价值较大。

表2 甲基化的基因在乳腺癌诊断价值中间接比较结果
The group of interest versus the referent are shown in the cells above,OR(95%) is used to evaluste the differences between alterations in DNA methylation. For example, 10vs1,OR=3.28,OR>1: compared with 1, 10 had a greater value for the diagnose of the breast cancer. Blue represents the differences arestatistically significant, red represents the differences are not statistically significant. GSTP1:glutathione S-transferase pi 1; RASSF1A: ras association domain family member 1 A; RARβ2:retinoic acid receptor beta2; BRCA1:breast cancer 1; PTEN:phosphatase and tensin homolog;CDH1:cadherin 1; APC:adenomatous polyposis coli; FHIT:fragile histidine triad protein; HIN-1:high in normal-1; RUNX3:Runt-related transcription factor 3.
RUNX3是RUNT基因家族的成员之一,其编码蛋白是一组DNA结合转录因子,在细胞的生长、发育及凋亡过程中起着重要的作用[20]。RUNX3转录的蛋白是转化生长因子(transforming growth factor-β,TGF-β)信号通路下游的一个转录因子,TGF-β信号通路对维持细胞正常生长发育起重要作用,并可能与肿瘤发生发展密切相关。笔者汇总了RUNX3基因甲基化对癌症的风险评估的Meta分析中发现RUNX3基因在食管癌、肺癌、肝癌、胰腺癌、乳腺癌中[5, 21-23]呈现高甲基化状态,而此抑癌基因高甲基化使其表达缺失与癌症的发生密切相关,这提示RUNX3基因是一个很有前景的用于癌症的筛查的生物学标志物。在汇总的评估乳腺癌风险的Meta分析中,发现BRCA1基因的权重占所有研究的57.1%,BRCA1突变基因是乳腺癌易感基因中头号关键的基因,而甲基化可能使其突变的原因,并且在乳腺癌和卵巢癌中发生,BRCA1甲基化具有明显的组织特异性[24-25]。因此,在乳腺癌早期筛查和辅助诊断的方案即选择对乳腺癌诊断价值较大的RUNX3基因及对乳腺癌发生机制贡献较大且具有特异性的BRCA1基因。
本研究基于乳腺癌病理诊断的金标准比较各个甲基化的基因的诊断价值,在没有直接比较的证据下根据间接比较的数据来推断甲基化的基因的诊断价值优劣。本研究最大的优越性在于可以将用于乳腺癌诊断的甲基化的基因汇总后进行定量化的比较,满足网状Meta分析的基本假设条件-异质性、相似性、一致性,对分析结果的有效性和可靠性提供保障[10]。但是本研究亦存在以下局限:①考虑到甲基化的Meta研究非常多,本研究只纳入Meta研究的甲基化基因,虽然对Meta研究后期的研究进行了搜索汇总,但是漏掉了其他没有Meta研究但在乳腺癌中甲基化的基因。②所纳入Meta的研究人群存在差异,而对于不同人群甲基化率会有所差异,这样对于结果会带来一定的误差。③虽然研究文献[26]表明甲基化异常存在于病人组织及血液中,但是在组织和血液中会存在差异,这在以后的研究中要分亚组分析再讨论。④虽然本研究针对Meta分析的甲基化的基因进行了比较,但仍然存在小样本效应,运用本研究结论仍需谨慎。⑤本研究所纳入的研究基于病例对照设计,作为诊断标志物最大挑战就是寻找其截断值或临界点(cut off point),而不同的研究其临界点可能不一致,而对于病例对照研究的临界值的外推性不高,这对本研究的结果也会产生影响。
综上所述,甲基化异常的现象早于明显的恶性表型出现,是乳腺癌发生的早期事件及危险因素;甲基化的异常能从病人较易获取的血液或者体液标本中检测到,无创伤;随着甲基化技术的不断发展,甲基化的检测更加成熟、稳定、廉价。这些为甲基化作为乳腺癌的早期筛查及辅助诊断的生物标志物提供了优势及必要条件。本研究基于间接比较的思想,首次在标志物作为疾病的诊断上进行比较,筛选出诊断价值较大的标志物,这为在众多筛选的候选基因的选择上提供了参考和思路,但尚需高质量、大样本的研究进一步论证。
[1] Chen W, Zheng R, Baade P D, et al. Cancer Statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132.
[2] Li T, Mello-Thoms C, Brennan P C. Descriptive epidemiology of breast cancer in China: incidence, mortality, survival and prevalence[J]. Breast Cancer Res Treat, 2016, 159(3): 395-406.
[3] Mathe A, Wong-Brown M, Locke W J, et al. DNA methylation profile of triple negative breast cancer-specific genes comparing lymph node positive patients to lymph node negative patients[J]. Sci Rep, 2016, 6:33435.
[4] Zhou D, Tang W, Wang W, et al. Association between aberrant APC promoter methylation and breast cancer pathogenesis: a Meta-analysis of 35 observational studies[J]. Peer J, 2016, 4: e2203.
[5] Wang Y, Qin X, Wu J, et al. Association of promoter methylation of RUNX3 gene with the development of esophageal cancer: a Meta analysis[J]. PLoS One, 2014, 9(9): e107598.
[6] Wu L, Wang F, Xu R, et al. Promoter methylation of BRCA1 in the prognosis of breast cancer: a Meta-analysis[J]. Breast Cancer Res Treat, 2013, 142(3): 619-627.
[7] Jiang Y, Cui L, Chen W D, et al. The prognostic role of RASSF1A rromoter methylation in breast cancer: A Meta-analysis of published data[J]. PLoS One, 2012, 7(5):e367805.
[8] Faryna M, Konermann C, Aulmann S, et al. Genome-wide methylation screen in low-grade breast cancer identifies novel epigenetically altered genes as potential biomarkers for tumor diagnosis[J]. FASEB J, 2012, 26(12): 4937-4950.
[9] Guo T, Ren L, Wang Q, et al. A network Meta-analysis of updated haemostatic strategies for hysterectomy[J]. Int J Surg, 2016, 35: 187-195.
[10]刘国伟,于莉莉,贾洪霞,等. 系统评价中的间接比较与网状Meta分析方法研究进展[J]. 中国循证医学杂志,2014(10): 1276-1280.
[11]王丹,翟俊霞,牟振云,等. Meta分析中的异质性及其处理方法[J]. 中国循证医学杂志,2009(10): 1115-1118.
[12]Whiting P, Rutjes A W, Reitsma J B, et al. The development of QUADAS: a tool for the quality assessment of studies of diagnostic accuracy included in systematic reviews[J]. BMC Med Res Methodol, 2003, 3: 25.
[13]Rojas K, Stuckey A. Breast cancer epidemiology and risk factors[J]. Clin Obstet Gynecol, 2016, 59(4): 651-672.
[14]Dai D, Dong X H, Cheng S T, et al. Aberrant promoter methylation of HIN-1 gene may contribute to the pathogenesis of breast cancer: a Meta-analysis[J]. Tumour Biol, 2014, 35(8): 8209-8216.
[15]Huang R, Ding P, Yang F. Clinicopathological significance and potential drug target of CDH1 in breast cancer: a Meta-analysis and literature review[J]. Drug Des Devel Ther, 2015, 9: 5277-5285.
[16]Fang C, Jian Z Y, Shen X F, et al. Promoter methylation of the retinoic acid receptor beta2 (RAR beta2) is associated with increased risk of breast cancer: a PRISMA Compliant Meta-analysis[J]. PLoS One, 2015, 10(10): e140329.
[17]Luo S, Chen J, Mo X. The association of PTEN hypermethylation and breast cancer: a Meta-analysis[J]. Onco Targets Ther, 2016, 9: 5643-5650.
[18]Su Y, Wang X, Li J, et al. The clinicopathological significance and drug target potential of FHIT in breast cancer, a Meta-analysis and literature review[J]. Drug Des Devel Ther, 2015, 9: 5439-5445.
[19]Song X Y, Li B Y, Zhou E X, et al. The clinicopathological significance of RUNX3 hypermethylation and mRNA expression in human breast cancer, a Meta-analysis[J]. Onco Targets Ther, 2016, 9: 5339-5347.
[20]闫战涛,聂艳红,吴永平,等. 不同乳腺癌分子亚型中RUNX3蛋白表达和启动子的甲基化[J]. 临床与实验病理学杂志,2014,30(1): 10-14.
[21]Xia Y, Zhang M, Zhang X, et al. A systematic review and Meta-analysis of runt-related transcription factor 3 gene promoter hypermethylation and risk of gastric cancer[J]. J Cancer Res Ther, 2014, 10 Suppl: 310-313.
[22]Xu L, Lan H, Su Y, et al. Clinicopathological significance and potential drug target of RUNX3 in non-small cell lung cancer: a Meta-analysis[J]. Drug Des Devel Ther, 2015, 9: 2855-2865.
[23]吴丹,董莹,张矛.RUNX3基因启动子区甲基化与肝癌临床病理特征的Meta分析[J]. 临床肿瘤学杂志,2014,19(8): 705-710.
[24]Gross E, van Tinteren H, Li Z, et al. Identification of BRCA1-like triple-negative breast cancers by quantitative multiplex-ligation-dependent probe amplification (MLPA) analysis of BRCA1-associated chromosomal regions: a validation study[J]. BMC Cancer, 2016, 16(1): 811.
[25]Weren R D, Mensenkamp A R, Simons M, et al. Novel BRCA1 and BRCA2 tumour test as basis for treatment decisions and referral for genetic counselling of patients with ovarian carcinomas[J]. Hum Mutat, 2017,38(2):226-235.
[26]Huang Y T, Chu S, Loucks E B, et al. Epigenome-wide Profiling of DNA Methylation in Paired Samples of Adipose Tissue and Blood [J]. Epigenetics, 2016, 11(3): 227-236.
编辑 孙超渊
Comparison of DNA hypermethylation of tumor suppressor genes as diagnostic biomarkers in screening and auxiliary detection of breast cancer: an indirect comparison Meta-analysis
Liu Di, Zhao Zhongyao, Sun Qi, Wang Youxin*
(BeijingKeyLaboratoryofClinicalEpidemiology,DepartmentofEpidemiologyandBiostatistics,SchoolofPublicHealth,CapitalMedicalUniversity,Beijing100069,China)
Objective To assess the comparative efficacy of diagnostic accuracy of DNA methylation biomarkers for the screening and detection of breast cancer, we conducted an indirect comparison Meta-analysis of published reports. Methods Databases including the Cochrane Library, PubMed, EMbase, Chinese BioMedical Literature Database (CBM), Web of Science, China National Knowledge Infrastructure (CNKI) and WanFang Data were searched to collect the diagnostic trials on the Meta-analysis of aberrant DNA hypermethylation for breast cancer published until October 2016. The indirect comparison Meta-analysis was used to evaluate the diagnostic value of the included genes, and the Meta-analysis was conducted via Review manager 5.3 software. Results Ten Meta-analysis of 130 original studies were included. In summary of Meta-analysis, compared with others gene, methylation of phosphatase and tensin homolog gene (PTEN) [iodds ratio (OR)=66.16 (24.48-178.82)], methylation of Runt-related transcription factor 3 gene (RUNX3) [OR=28.88 (15.48-54.25)] had greater risk in breast cancer. Although breast cancer 1 gene (BRCA1) [OR=2.11 (1.88-2.35)] was the smallest risk for breast cancer, the weight ofBRCA1 gene was up to 57.1% in all the Meta. According to the sensitivity, compared with others gene,RUNX3 [0.62 (0.56-0.67)] was the largest, followed by fragile histidine triad protein gene (FHIT) [0.59 (0.54-0.63)], but the specificity ofRUNX3 [0.95 (0.91-0.97)] was greater thanFHIT[0.65 (0.61-0.69)]. Comprehensive sensitivity and specificity of ten genes, the largest value ofRUNX3 in the diagnosis of breast cancer was found. Conclusion We advocate thatRUNX3 andBRCA1 gene methylation can be used in the screening and auxiliary detection of breast cancer.
breast cancer; methylation; diagnostic test; indirect comparison; Meta-analysis
国家自然科学基金(81673247, 81370083, 81530087),北京市科技新星项目(Z141107001814058)。This study was supported by National Natural Science Foundation of China (81673247, 81370083, 81530087), Beijing Nova Program (Z141107001814058).
时间:2017-07-16 17∶34 网络出版地址:http://kns.cnki.net/kcms/detail/11.3662.r.20170716.1734.042.html
10.3969/j.issn.1006-7795.2017.04.014]
R195
2016-10-25)
*Corresponding author, E-mail:wangy@ccmu.edu.cn

