贵州小麦条锈菌群体遗传结构分析
袁文娅++孙振宇++梁丹++时晓伟++陈静++张梅茹++马占鸿++刘秀峰


摘要:为了解贵州省小麦条锈菌群体的遗传多样性,本研究采用扩增片段长度多态性标记(AFLP)对其进行研究。结果显示:贵州省6个县市分离到的128个小麦条锈菌单孢子堆分离物中共获得32个AFLP基因型,没有检测到频率占绝对优势的AFLP基因型,其中1个AFLP基因型(zy6)频率(0.195)较高,AFLP基因型频率介于0.050~0.100的有3个,其余28个AFLP基因型的频率均低于0.050。香农系数及Φpt方法均显示亚群体间遗传差异不明显;Mantel test显示小麦条锈菌贵州群体遗传差异程度与地理距离无关;最大简约树步长评估结果显示小麦条锈菌贵州群体自由重组的可能性较低。表明,贵州省小麦条锈菌群体具有丰富的遗传多样性。
关键词:小麦条锈菌;群体遗传结构;AFLP标记
中图分类号:S435.121.4+2文献标识号:A文章编号:1001-4942(2017)06-0095-06
AbstractThe population genetic diversity of wheat stripe rust (Puccinia striiformis f. sp. Tritici) in Guizhou Province was analyzed by amplified fragment length polymorphism (AFLP). The results showed that 32 AFLP genotypes were obtained from 128 isolates collected from 6 counties in Guizhou Province. No predominant genotype was identified. In which, 1 genotype,zy6,had the highest frequency of 0.195, 3 genotypes had the frequency between 0.050~0.100, and the frequency of the other 28 genotypes was less than 0.050. Shannon coefficient and Φpt statistics results showed that the genetic differentiation among 6 counties was not significant. Mantel test suggested that there was no significant correlation between genetic differentiation and geographical distances. The most parsimonious tree length results showed that the possibility of free recombination for population of Puccinia striiformis f. sp. tritici in Guizhou was very lower. The results indicated that the genetic diversity of P. striiformis was rich in Guizhou Province.
KeywordsPuccinia striiformis f. sp. tritici; Population genetic structure; AFLP marker
貴州省地处云贵高原,毗邻云南越夏区。海拔300~2 400 m地带均有小麦种植,不同海拔地区小麦生长时期重叠,加之播种下茬作物时常不翻耕保留着大量自生麦苗,致使田间周年都有麦苗[1]。夏季低温,具有独特的环境条件,从理论上推测具备小麦条锈菌(Puccinia striiformis f.sp tritici)越夏条件。小麦条锈菌在贵州境内能否安全越夏长期以来并不清楚,直到2006年证实小麦条锈菌在赫章等地海拔1 700 m以上地区能够安全越夏,引起秋苗发病[2]。2006年11月农业部印发的《小麦条锈病中长期治理指导意见》指出,贵州等地的低山、河谷、山坝是我国小麦条锈菌冬繁区之一,也是当地和流行区小麦条锈病春季流行的重要菌源地。小麦条锈菌在贵州地区既能越夏也能越冬,在一定海拔范围内在当地能够完成周年循环,非常适合小麦条锈菌产生遗传变异。目前贵州菌源与周边省份的菌源传播关系及其对我国小麦条锈病发生流行的影响等研究资料稀少。贵州地区小麦条锈菌群体作为西南流行区系的重要组成,了解其群体遗传结构显然具有重要意义。本研究从贵州省6个县市分离出128个小麦条锈菌单孢子堆分离物,通过AFLP标记和重组信号检测进行群体遗传结构分析,以期为了解小麦条锈病的传播途径提供理论依据。
1材料与方法
1.1试验材料
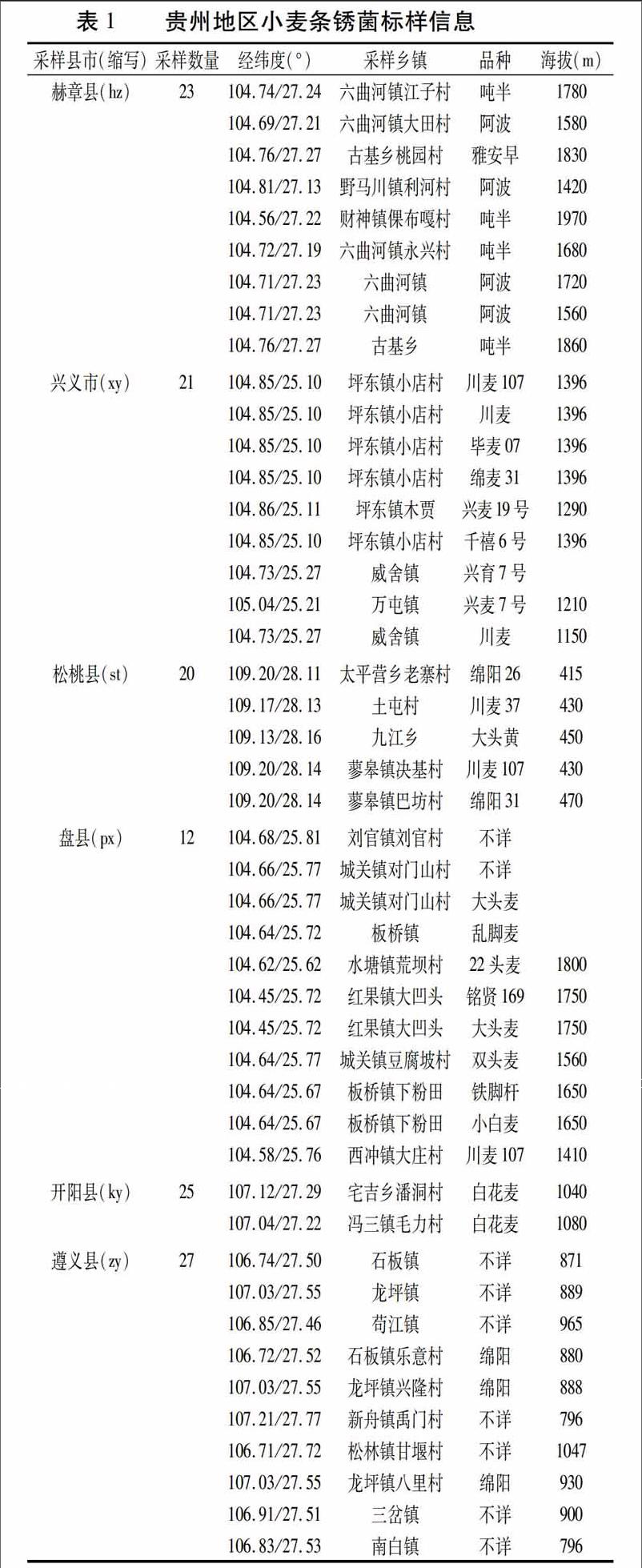
小麦条锈菌标样采集于贵州省6个县市,这些地区基本覆盖了贵州省东北、西北、西南以及贵州中部。样本具体信息见表1。
1.2试验方法
小麦条锈菌单孢子堆基因组DNA提取、AFLP体系和反应条件采用Enjalbert等[3]报道的方法,略有修改。
1.3数据分析
同一对AFLP引物的扩增产物中电泳迁移率一致的条带被认为具有同源性,属于同一位点的产物。AFLP条带在两个以上供试小麦条锈菌分离物共有的作为多态性条带,多态性条带的有和无分别记作1和0。排除无法准确标识的带,然后构建多态性条带二进制原始数据矩阵。
1.3.1贵州小麦条锈菌群体和亚群体的AFLP基因型调查每个县市采集到的小麦条锈菌分离物看作一个亚群体,贵州省标样共计分为6个亚群体。采用MULTILOCUS 1.3软件[4]计算各亚群体及贵州小麦条锈菌群体的不同AFLP基因型频率。
1.3.2贵州小麦条锈菌亚群体内的遗传多样性分析用Nei 氏多样性系数(Neis diversity index)、Nei 氏無偏多样性系数(Neis unbiased diversity index)、香农系数(Shannons information index)以及多态位点百分率(percentage of polymorphic loci)评估贵州小麦条锈菌6个亚群体内的多样性。
1.3.3贵州小麦条锈菌亚群体间的遗传分化比较φpt类似于F统计,可用于评估遗传结构,但其不需要假设可直接对显性标记数据进行群体遗传结构的分析[5]。Fst和φpt两个参数用于评估贵州小麦条锈菌亚群体间的遗传变异。同时采用GENALEX 6.0软件[6]中的分子方差分析(analysis of molecular variance,AMOVA)评估贵州各亚群体间的遗传差异。
1.3.4贵州小麦条锈菌遗传距离与地理距离相关性分析根据各采样地点的经纬度计算地理矩阵,根据Nei 氏遗传距离计算遗传矩阵,利用GENALEX 6.0中的Mantel test方法计算贵州地区小麦条锈菌各亚群体遗传距离与地理距离的相关性[3] ,同时对两个矩阵相关系数的显著性进行1 000次permutation自举检验,评估贵州小麦条锈菌各亚群体遗传结构是否具有地理隔离现象(isolation-by-distance)。该相关性检测中的无效假设是地理距离矩阵和遗传距离矩阵间没有相关性,即各亚群体间遗传距离不随地理距离的改变而改变。Mantel test 产生的相关系数范围在 -1和 +1之间,如果各亚群体间遗传距离和地理距离不相关,则真实进化值应在自举检测的相关系数范围之内。
1.3.5贵州小麦条锈菌群体重组信号检测采用连锁不平衡系数(rd)计算连锁不平衡时不依赖于所用位点个数,可提高不同研究者间结果比对的可行性[4]。利用rd对贵州地区小麦条锈菌各亚群体间是否有重组现象进行检验。应用MULTILOCUS 1.3软件计算贵州各亚群体间的rd,同时进行1 000次permutation自举检验。该检验中无效假设是群体间没有连锁不平衡,可以自由重组。如果贵州各亚群体间的连锁不平衡系数值偏离无效假设的数据分布范围,则表明各亚群体间不能自由重组。同时利用最大简约树步长评估法(parsimony tree length permutation test, PTLPT)检测贵州地区小麦条锈菌群体是否有重组信号。PTLPT的运算在PAUP 4.0 β 10 中进行[7]。
2结果与分析
2.1贵州小麦条锈菌群体和亚群体的AFLP基因型
贵州地区128个小麦条锈菌分离物中共获得32个AFLP基因型,群体的AFLP基因型多态性频率为0.250。各AFLP基因型个数及其分布见表2。如表2所示,贵州小麦条锈菌群体没有检测到频率占绝对优势的AFLP基因型,在遵义县南白镇获得的分离物的基因型(zy6)频率占有一定优势(频率为0.195),基因型频率介于0.050~0.100的有3个(xy2、xy1、px1),其余28个AFLP基因型的频率均低于0.050。
2.2贵州小麦条锈菌亚群体内的遗传多样性
香农系数、Nei 氏多样性系数、Nei 氏无偏系数和多态性位点百分率这几个参数都是通过计算群体内的各对个体间距离后,得出一个衡量该群体遗传差异的平均数。其中,香农系数计算方法更侧重于存在条带的权重,Nei 氏多样性系数更关注于所比较个体的相似性,Nei 氏无偏系数与Nei 氏多样性系数相似,但更适合于在群体研究数量少时采用。多态性位点百分率不能确定各条带在频率上的均匀程度,且受到样本大小和条带总数的影响,它只是衡量群体遗传多态性的一个粗略估计值,但它计算简单直观,能够反映一定的遗传多样性程度,在研究中得到了广泛应用。由表3可知, 尽管这几个参数的统计侧重点有少许差异,但结果均显示贵州小麦条锈菌各亚群体内都存在较丰富的遗传多样性。
2.3贵州小麦条锈菌亚群体间的遗传变异
由表4可知,将AFLP 基因型数据作为共显性(codominant)的双等位基因基因型数据,用φpt参数衡量亚群体两两之间的遗传差异性。利用φpt值推断,除开阳县-松桃县间的φpt值稍高(φpt=0.301)外,其它亚群体两两间φpt值都很小,显示贵州省小麦锈菌6个亚群体两两之间的遗传差异不明显。
本研究根据小麦条锈菌的特点选用欧氏距离平方计算和矫正基因型之间的进化距离,矫正后再进行分子方差分析,AMOVA结果(表5)表明,89%的变异来源于亚群体之内。
这些结果显示贵州小麦条锈菌群体和6个亚群体间都存在着一定遗传分化。
2.4贵州小麦条锈菌遗传距离与地理距离的相关性
图1结果显示,贵州群体的遗传距离和地理距离的相关系数为0.11,在1 000次假设的相关系数分布范围内,表明小麦条锈菌贵州群体遗传差异程度与地理距离无关。
2.5贵州小麦条锈菌群体重组信号检测
rd值计算结果显示,rd值明显偏离无效假设的自由交配群体的值(rd=0.032, P<0.001),因此拒绝无效假设,表明小麦条锈菌贵州群体自由重组的可能性较低。图2结果显示,PTLPT构建的贵州群体最大简约树步长(165步)明显短于无〖CM(21〗效假设的自由重组的简约树步长范围,所以拒绝无效假设。连锁不平衡系数rd和PTLPT结果均表明贵州小麦条锈菌群体自由重组的可能性较低。
3讨论
贵州全省贯穿我国南北走向和东北-西南走向两组山脉交汇的云贵高原,地理环境复杂,地势起伏较大,种植小麦品种繁杂。本研究采样地点涵盖了松桃、兴义、遵义等6个县市,代表黔东北、黔西南、黔中不同地理区域,西南部的盘县与云南省的富源县接壤,而东北部的松桃县与湖南省接壤,又结合了南亚热带等贵州小麦种植的主要气候带。鉴定的标样采自20余个小麦品种共128份,具较好的地域代表性、气候代表性及寄主品种代表性。
总体上比较,小麦条锈菌贵州群体的各基因型频率相对均衡,没有频率非常高的优势基因型。贵州小麥条锈菌群体的基因型中,只有zy6基因型的频率占有一定优势(频率为0.195),但明显低于小麦条锈菌云南群体中的qj3基因型频率[8]。zy6基因型在遵义县检测到,但其出现频率在遵义县比较低,而在开阳县检测到该基因型的分离物较多,暗示小麦条锈菌遵义群体和开阳群体间有较高的基因型交流。与湖南接壤的松桃县检测到的5个基因型频率基本相当,没有优势的AFLP基因型。西北部的赫章县各个基因型的频率也比较均衡,没有优势基因型或者频率非常低的基因型。Mantel test检验结果表明贵州小麦条锈菌群体遗传差异程度与地理距离无关,暗示贵州各亚群体间存在着基因型交流。与此一致,对贵州小麦条锈菌群体进行的分子方差分析结果显示,贵州群体中变异仍然主要来自于群体内部,种群间变异较小,只占遗传总变异的11%。小麦条锈菌贵州群体没有发现重组信号,可将其看作克隆繁殖方式。与北美[9]、西北欧[10]、法国[11]、地中海[12] 、澳大利亚1979—2006年的小麦条锈菌群体[13]的繁殖方式相同。克隆繁殖方式使得寄生适合度高的个体得以迅速有效增长,这样的群体能够保持适合度高的个体迅速侵染寄主[13]。
美国Pst-130[14]和中国CY32小种[15]的基因组序列为研究小麦条锈菌提供了新的工具。利用CY32小种的基因组序列已经筛选出20个具有多态性的SSR引物,比利用表达序列标签筛选的SSR引物具有更高的多态性[16]。若将本研究中的各AFLP基因型分离物进行重测序或某一时刻的转录组分析,对测序所得的数据进行全面分析,可能会对贵州地区小麦条锈菌越夏菌源与本地和周边地区小麦发病的关系、与我国西南云贵川小麦条锈菌菌源关系以及西南地区小麦条锈菌越夏、越冬后对我国小麦条锈病发生流行的影响等有更深入认识。
4小结
贵州小麦条锈菌群体的变异主要来源于亚群体之内,亚群体间的遗传差异不明显,群体遗传差异程度与地理距离不相关,贵州小麦条锈菌群体自由重组的可能性较低。结果表明贵州小麦条锈菌群体具有丰富的遗传多样性。
参考文献:
[1]王海光,谈孝凤,金星,等.2006年贵州省赫章县秋季小麦条锈病调查[J].中国农学通报, 2007,23(3):360-364.
[2]姜玉英,金星,谈孝凤,等. 黔西部小麦条锈病菌越夏考察初报[J]. 植物保护, 2007,33(1):133-134.
[3]Enjalbert J, Duan X Y, Vautrin D, et al. Isolation of twelve microsatellite loci, using an enrichment protocol, in the phytopathogenic fungus Puccinia striiformis f. sp. tritici[J]. Moecular Ecology Notes, 2002,2(4):563-565.
[4]Agapow P M, Burt A. Indices of multilocus linkage disequilibrium[J]. Molecular Ecology Notes, 2001, 1:101-102.
[5]Excoffier L, Smouse P E, Quattro J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data [J].Genetics, 1992, 131:479-491.
[6]Peakall R, Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research[J]. Molecular Ecology Notes, 2006, 6:288-295.
[7]Swofford D L. PAUP: phylogenetic analysis using parsimony (and other methods), version 4.0. Sinauer Associates[M]. Sunderland MA. , 1996.
[8]Liu X F, Huang C, Sun Z Y, et al. Analysis of population structure of Puccinia striiformis in Yunnan Province of China by using AFLP[J]. European Journal of Plant Pathology, 2011,129:43-55.
[9]Chen X M, Line R F, Leung H. Relationship between virulence variation and DNA polymorphism in Puccinia striiformis[J]. Phytopathology, 1993, 83:1489-1497.
[10]Hovmller M S, Justesen A F, Brown J K M. Clonality and long-distance migration of Puccinia striiformis f. sp. tritici in north-west Europe[J]. Plant Pathology, 2002,24:24-32.
[11]Enjalbert J, Duan X Y, Leconte M, et al. Genetic evidence of local adaptation of wheat yellow rust (Puccinia striiformis f. sp. tritici) within France[J]. Molecular Ecology, 2005, 14:2065-2073.
[12]Bahri B, Leconte M, Ouffroukh A, et al. Geographic limits of a clonal population of wheat yellow rust in the Mediterranean region[J]. Molecular Ecology, 2009, 18:4165-4179.
[13]Wellings C R. Puccinia striiformis in Australia: a review of the incursion, evolution, and adaptation of stripe rust in the period 1979-2006[J]. Australian Journal of Agricultural Research, 2007, 58:567-575.
[14]Cantu D, Govindarajulu M, Kozik A, et al. Next generation sequencing provides rapid access to the genome of Puccinia striiformis f. sp. tritici, the causal agent of wheat stripe rust[J]. PLoS ONE, 2011, 6:1-8.
[15]Zheng W M, Huang L L, Huang J Q, et al. High genome heterozygosity and endemic genetic recombination in the wheat stripe rust fungus[J]. Nature Communication, 2013,4:1-10.
[16]Zhan G M, Wang F P, Luo H Y, et al. Screening for simple sequence repeat markers in Puccinia striiformis sp.tritici based on genomic sequence[J]. Journal of Zhejiang University-Science B, 2015,16(8):727-732.

