汝州白三叶草根瘤菌的分离与分子鉴定
张俊杰, 杨 旭, 陆 勇, 张志炎, 王志伟, 魏喜旺
(郑州轻工业学院食品与生物工程学院,河南 郑州 450002)
汝州白三叶草根瘤菌的分离与分子鉴定
张俊杰, 杨 旭, 陆 勇, 张志炎, 王志伟, 魏喜旺
(郑州轻工业学院食品与生物工程学院,河南 郑州 450002)
本研究采用16S rDNA PCR-RFLP、IGS PCR-RFLP和16S rDNA基因测序和系统发育分析等分子鉴定方法,对分离的汝州白三叶草共生根瘤菌进行了多样性研究。结果表明,21株供试菌株仅产生了一种16S rDNA酶切图谱组合类型,通过IGS PCR-RFLP又把所有的供试菌株分为了6种酶切图谱类型。经过对代表菌株的16S rDNA基因测序及系统发育分析,6个代表菌株之间具有99.9%~100%的相似性,且供试菌株与根瘤菌属的已知种群Rhizobiumanhuiense和Rhizobiumleguminosarum等也具有99.9%~100%的相似性,因此,分离自汝州的白三叶草根瘤菌属于根瘤菌属(Rhizobium)。
白三叶草;根瘤菌; 16S rDNA;IGS;RFLP
长期以来,中国农业生产依赖化学肥料,造成生产成本的增加,引起土壤板结。由于作物只能在生长期吸收利用化肥中的部分营养元素,化肥中未被利用的氮磷钾将随降雨流失到地下水与江河等生态环境中,严重破坏了自然生态平衡和农业的可持续发展[1-2]。生物固氮是一条很重要的向土壤输氮的途径,是豆科植物有别于其他农作物最重要的优势。根瘤菌与豆科植物的共生体系是生物固氮中固氮能力最强的体系,占生物固氮量的65%以上,它为豆科植物提供氮素营养,培肥地力,从而增加了作物产量、改善作物品质。高效的固氮根瘤菌应用于生产,不仅可提高大豆的固氮能力,而且可以有效减少因长期使用化肥带来的环境污染[3-6]。有研究表明,在含速效氮中等偏低肥力的土壤中,接种大豆根瘤菌可以有效提高大豆地上部植株生长和地下部根瘤形成,并且可以提高植株高度和鲜质量[6]。白三叶草(TrifoliumrepensL.)是一类重要的优良豆科绿肥和牧草,是本属植物中在中国很有推广前途的种群[7]。在长期进化过程中,根瘤菌由于受到环境胁迫和寄主的选择,各自向着不同的方向进化,从而形成了丰富的生物多样性[8-9]。目前,对白三叶草根瘤菌的研究资料不多,国内外对白三叶草根瘤菌多样性及系统分类研究的报道也很少,尤其是河南省境内,尚未见白三叶草根瘤菌多样性研究的报道。因此,了解白三叶草根瘤菌的生物多样性,选育高效与抗逆性能优良的菌株用于农林生产具有非常重要的经济和生态学意义。
1 材料和方法
1.1 供试与参比菌株
本研究所用的材料为分离自河南省汝州市煤山公园野生白三叶草根瘤的21株共生根瘤菌,样品采集采用张俊杰[10]采样方法,根瘤菌的分离按照VINCENT[11]经典方法进行。在系统发育分析过程中用到的模式及参比菌株有来自根瘤菌属(Rhizobium)的R.anhuienseCCBAU 23252T、R.phaseoliATCC 14482T、R.acidisoliFH13T、R.indigoferaeCCBAU71042T、R.laguerreaeFB206T、R.leguminosarumUSDA2370T和R.sophoraeCCBAU03386T等;来自土壤农杆菌属(Agrobacterium)的A.tumefaciensB6T等;来自中华根瘤菌属的Sinorhizobiumsp. CCBAU65101;来自慢生根瘤菌属的BradyrhizobiumjaponicumUSDA6T;和近期发表的根瘤菌新属Pararhizobiumsp. F5.3及NeorhizobiumgalegaeG323T等。这些标准菌株的16S rDNA序列均从NCBI数据库下载获得。
1.2 培养基
YMA (Yeast Extract D-mannitol Agar)培养基:酵母粉 3 g, K2HPO40.25 g,甘露醇10 g,无水MgSO40.1 g, KH2PO40.25 g, NaCl 0.l g,琼脂粉(agar)18 g,去离子水(distilled water)1 000 mL,pH值6.8~7.2,121 ℃灭菌30 min;
TY(Tryptone-Yeast)培养基: CaCl2·2H2O 0.7 g,胰蛋白胨5 g,酵母粉 3 g,去离子水(distilled water)1 000 mL,pH值6.8~7.2,121 ℃灭菌30 min。
1.3 根瘤菌DNA的提取
先将供试菌株活化在YMA平板上,挑去单菌落并接种于5 mL TY液体培养基,放至于28 ℃摇床上以130 r·min-1的转速振荡培养至对数生长期。DNA的提取方法参照Terefework的GUTC方法[12],该方法基本过程是对收集的样品通过GUTC(60%乙醇,20mmol·L-1pH值7.5 Tris-HCl,1 mmol·L-1EDTA和400 mmol·L-1NaCl)作用裂解细胞,然后去除样品中的蛋白质等杂质,最终用硅藻土吸附沉淀核酸DNA,并用双蒸水溶解并获得样品DNA。提取的DNA用1.0 %琼脂糖凝胶电泳估测浓度,所有DNA样品在-20 ℃冰箱中保存备用。
1.4 16S rDNA和IGS PCR-RFLP
16S rDNA和IGS基因扩增程序方法等参见文献[10],PCR过程使用伯乐C1000 PCR扩增仪,所用的DNA聚合酶则来自生工生物工程(上海)股份有限公司。16S rRNA PCR产物用4种限制性内切酶:AluI、HaeIII、MspI和HinfI分别进行酶切,而IGS产物则选用3种限制性内切酶酶切:HaeIII 、MspI和HhaI。所用限制性内切酶均来生工生物工程(上海)股份有限公司。酶切体系参见文献[10],结果用2.5%的琼脂糖凝胶(含10% GoldView I型核酸染色剂,索莱宝)在60~80 V电压下电泳1~2 h,然后用DNI凝胶成像系统(DNI Bio-Imaging System,MiniBIS Pro)检测酶切电泳结果。
1.5 代表菌株的16S rDNA基因序列测定和系统发育分析
对16S rDNA和IGS PCR-RFLP结果进行整理和酶切图谱类型分析,从每个图谱组合类型中找出一个代表菌株,扩增其16S rDNA基因,并送公司进行测序和拼接,拼接好的序列经过NCBI BLASTn比对找到序列相似性较高的菌株,最后利用Mega 6.0软件对供试序列进行系统发育分析,具体方法同文献[10]中的方法,采用Kimura 2-parameter模型,bootstrap值为1000,获得Neighbour-Joining系统发育树。
2 结果
2.1 供试菌株16S rDNA 和IGS基因序列PCR扩增结果
16S rDNA 和IGS基因序列PCR产物大小分别为1.5 kb和1 kb。
2.2 三叶草根瘤菌16S rDNA RFLP分析
21株待测菌株的16S rDNA PCR产物分别经AluI、HaeIII和MspI共3种限制性内切酶分别酶切后,酶切产物在2.5%的琼脂糖凝胶电泳中分别呈现出相同的单一的酶切图谱类型,如图1所示,所有待测菌株的16S rDNA具有相同的酶切图谱组合类型。由于HinfI在所有供试菌株的16S rDNA PCR产物中均无酶切位点,所以未给出相关电泳图谱。结果表明,21株三叶草根瘤菌同属于1个属。
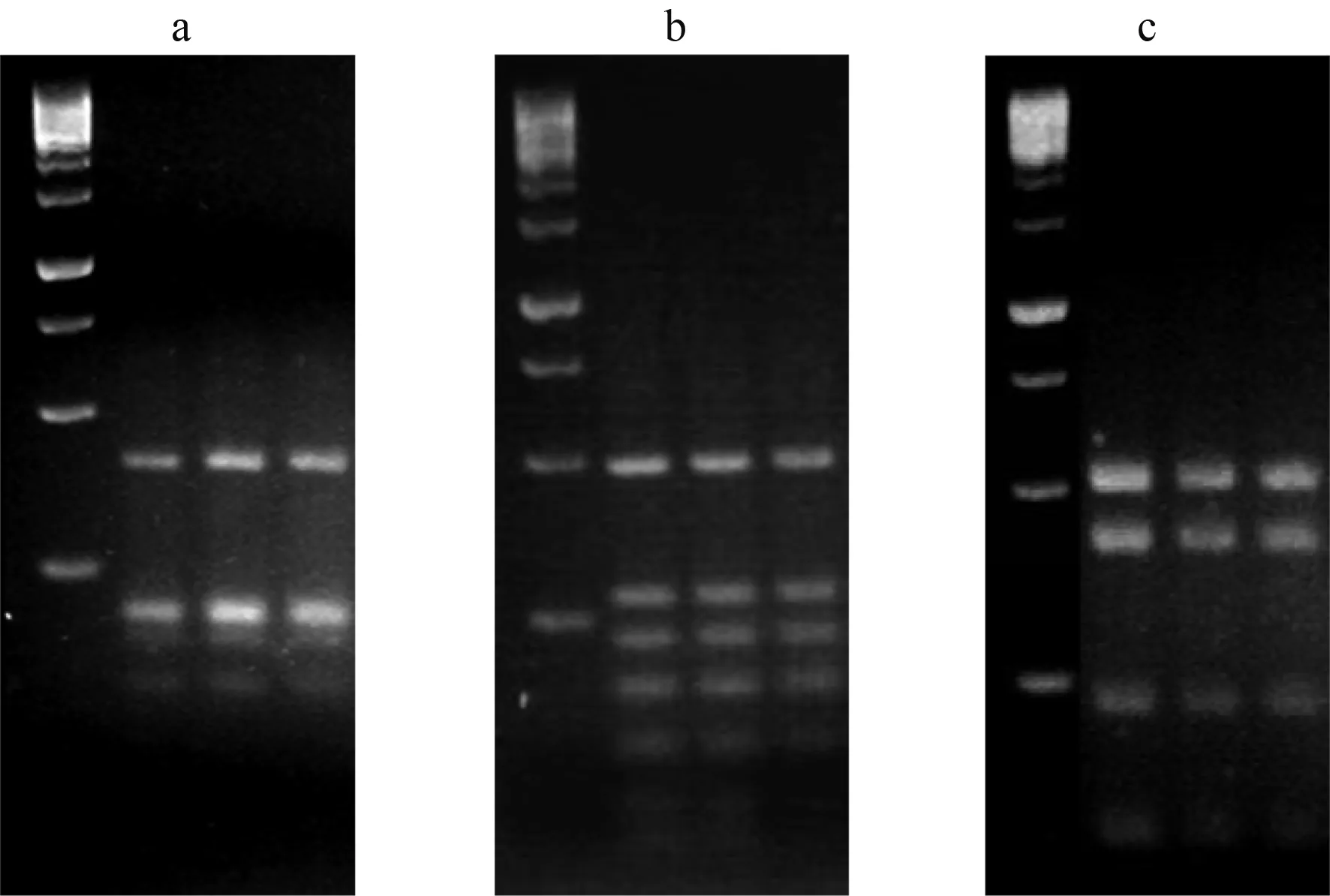
a:AluI酶切图谱;b:HaeIII酶切图谱;c:MspI酶切图谱(每种酶的酶切图谱都相同,仅显示部分代表酶切结果,下同)。
a.AluI finger-print; b.HaeIII finger-print; c.MspI finger-print (each enzyme with the same kind of finger-print, and only some representative bands showed here. The same as below).
图1 供试菌株16S rDNA PCR产物酶切电泳图谱
Fig.1 The electrophoresis results of the 16S rDNA PCR RFLP for the representatives
2.3 IGS PCR RFLP结果与分析
21株待测菌株的IGS PCR产物经过MspI、HaeIII
和HhaI共3种限制性内切酶的酶切,在2.5%的琼脂糖凝胶电泳检测得到RFLP酶切图谱(图2),图2中列出了每种限制性内切酶酶切后所有的酶切图谱代表类型,其中HaeIII酶切图谱共有5种图谱类型,HhaI酶切图谱共有2种图谱类型,而MspI酶切图谱则有4种图谱类型。
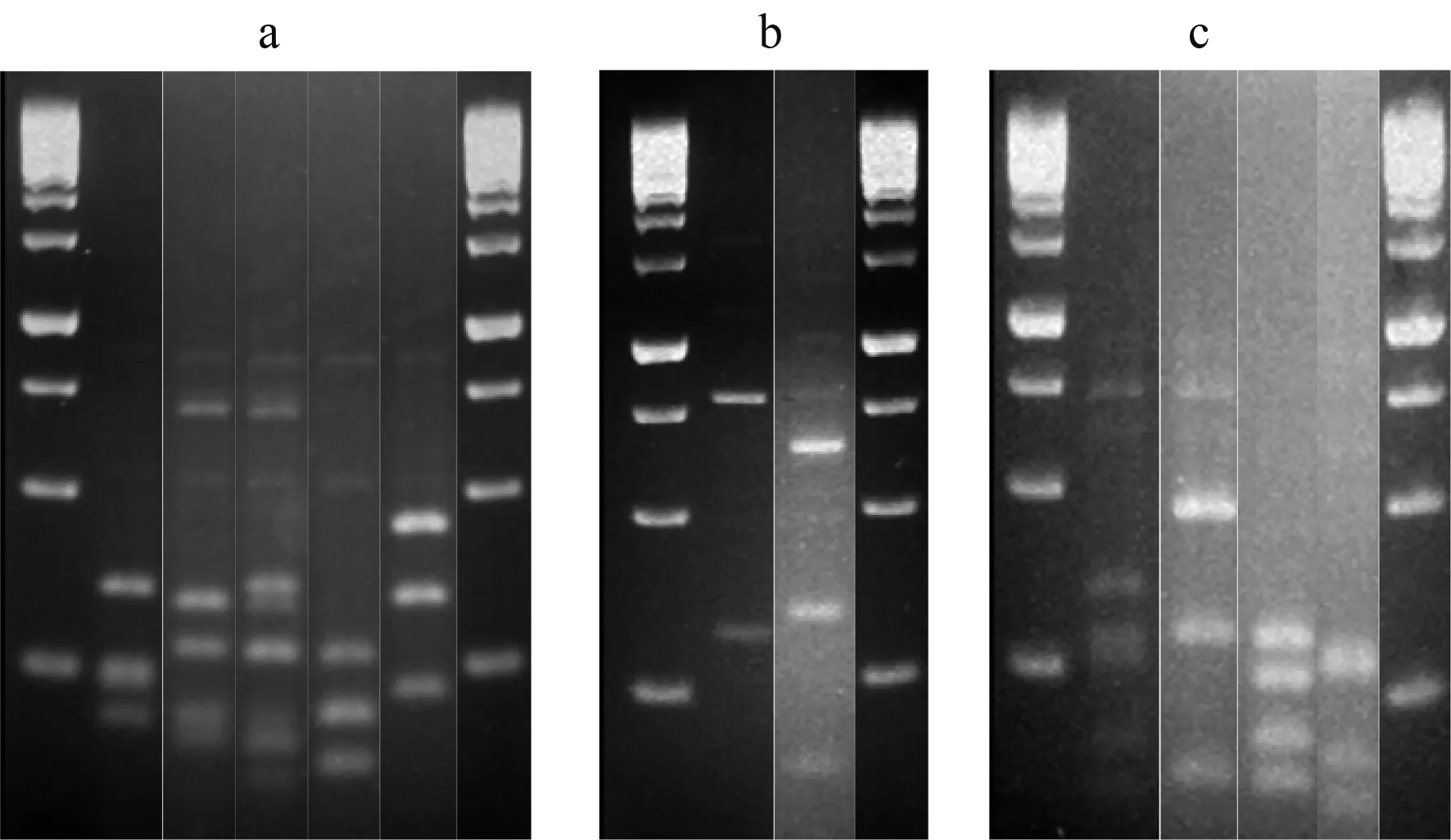
a:HaeIII酶切图谱;b:HhaI酶切图谱;c:MspI酶切图谱。a.HaeIII finger-print; b.HhaI finger-print; c.MspI finger-print.图2 供试菌株IGS PCR-RFLP电泳图谱Fig.2 The IGS PCR-RFLP for the representatives
经过对所有供试菌株酶切图谱的分析,21株供试菌株一共被分为6个酶切图谱组合类型(T1~T6),见表1。T1占所有菌株的47.6 %,T2占9.5 %,T3占23.8 %,T4占9.5 %,而T5和T6各占4.8 %。 其中,T1和T3为主要图谱类型,占到供试菌株的71.4 %,而T5和T6是其中占少数的酶切图谱类型。
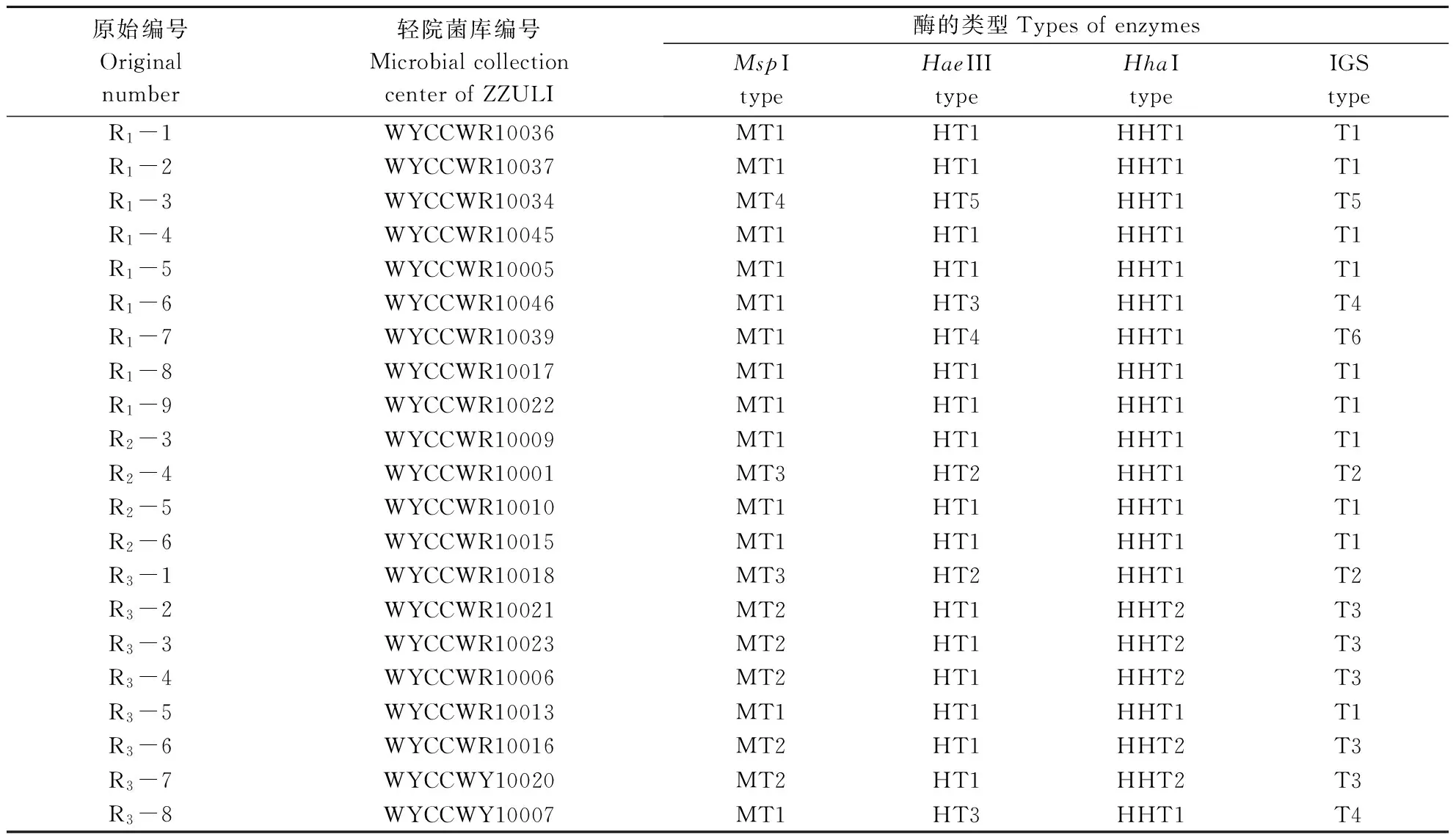
表1 供试菌株的IGS PCR RFLP分型结果Table 1 The IGS PCR RFLP typing results of all the strains slected
2.4 代表菌株16S rDNA基因序列测定及系统发育分析
菌株16S rDNA基因序列测定及系统发育分析,结果见图3。根据IGS基因酶切结果从6种IGS图谱类型中一共选择了6个代表菌株:T1代表菌株R2-6,T2代表菌株R2-4,T3代表菌株R3-7,T4代表菌株R1-6,T5代表菌株R1-3和T6代表菌株R1-7,用于16S rDNA基因的序列测定和分析。经PCR反应得到的16S rDNA基因产物送公司进行序列测定,然后分析和拼接测序结果,并应用NCBI BLASTn工具进行序列比对,选取序列相似性较高的参考序列及相关种属的已知种群部分序列,利用Mega 6.0软件进行系统发育分析,结果见图3。由系统发育树状图可以得知,6个代表菌株与根瘤菌属的代表菌株聚为一个大的分支,所以可以判定汝州三叶草根瘤菌都属于根瘤菌属,并且6个代表菌株序列相似性为99.9% ~100 %。具体来看,6个代表菌株都分布在树状图的小分支I(Clade-I),并且与该分支的所有已知代表菌株的序列相似性范围为99.9%~100 %,如:R.anhuienseCCBAU 23252T等;6个代表菌株与小分支II(Clade-II)中已知代表菌株的序列相似性为99.7%~99.3 %,如:R.phaseoliATCC 14482T等。而代表菌株测序后的拼接序列都已经提交Genbank并获得相应的序列登录号,见图3。
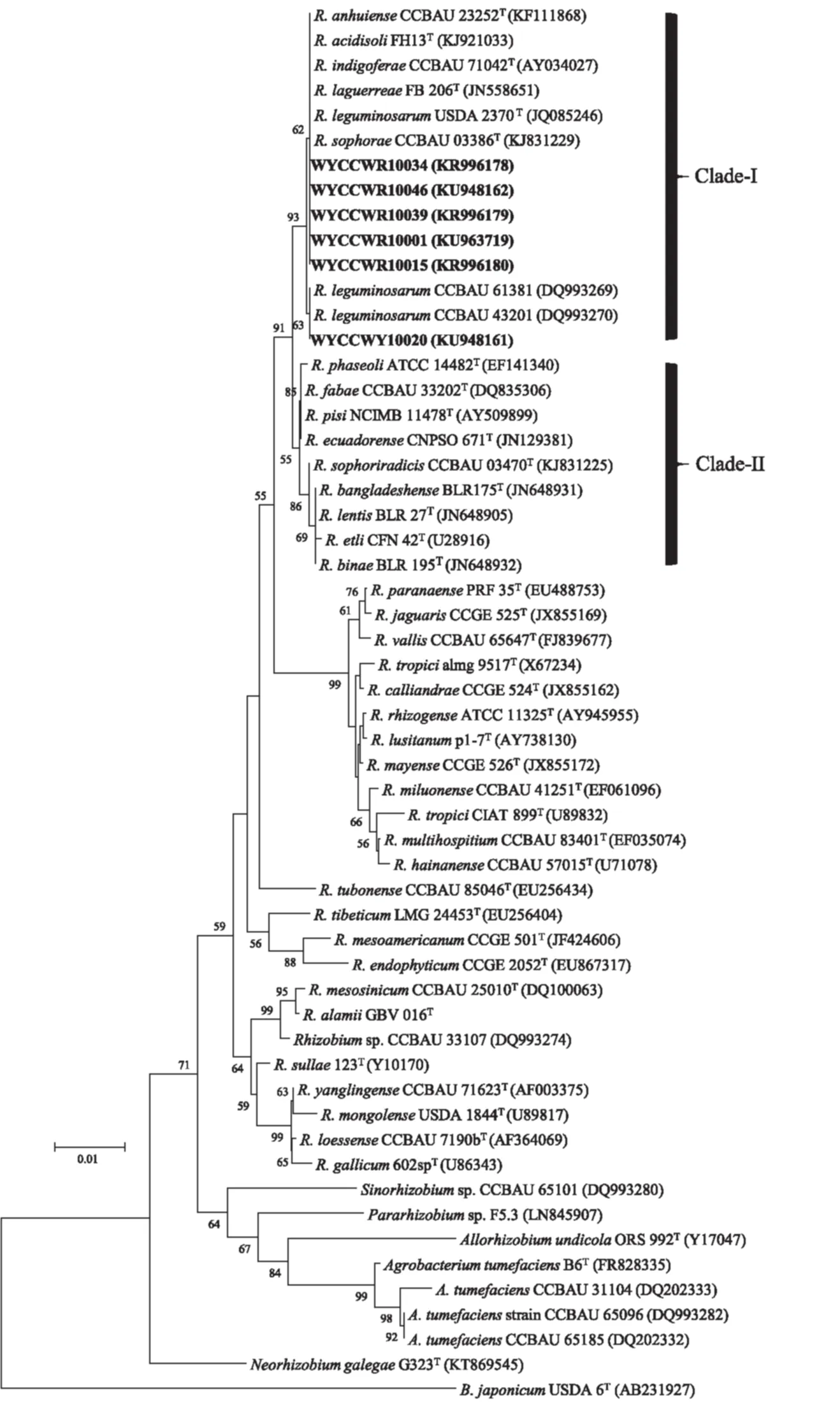
采用Neighbour-Joining方法的Kimura 2-parameter 模型和1000的bootstrap值构建系统发育树,其中菌株B.japonicumUSDA 6T被作为发育树的外群,仅大于50%的bootstrap值被保留在系统发育树上。字体加粗的菌株为本试验中三叶草根瘤菌代表菌株,Genbank登录号在相对应的括号中标出。
The Neighbour-Joining tree was derived from a distance matrix (Kimura 2-parameter model) and 1000 of the bootstrap value. Bootstrap confidence levels >50% are indicated at the internodes.B.japonicumUSDA 6Twas taken as the outgroup. The representatives were in boldface and the correspondence accession numbers were in the bracket following the strain numbers.
图3 代表菌株16S rDNA序列的系统发育树
Fig.3 The phylogenetic tree of 16S rDNA gene sequences of the representatives
3 讨论
本研究以分离自河南省汝州市煤山公园草坪白三叶草根瘤的21株根瘤菌菌株为研究对象,充分结合16S rDNA PCR-RFLP、IGS PCR-RFLP和16S rDNA基因测序及系统发育分析方法进行了遗传多样性的研究。经过16S rDNA PCR-RFLP分析,所有的供试菌株仅产生了一种酶切图谱组合类型,并初步判定所有供试菌株属于同一个属;又通过IGS PCR-RFLP把所有的供试菌株分为了6种酶切图谱类型。然后经过对代表菌株的16S rDNA基因测序及系统发育分析,6个代表菌株之间具有99.9%~100%的相似性,且供试菌株与根瘤菌属的已知种群R.anhuiense和R.leguminosarum等也具有99.9%~100 %的相似性,最终确认供试菌株均属于Rhizobium。
21株供试菌株都属于根瘤菌属,较为单一。这与刘晓云等[13]在江西、湖北、云南等地分离出的三叶草根瘤菌分别归属于快生根瘤菌属、中华根瘤菌属、中慢生根瘤菌属和慢生根瘤菌属的研究结果不同,显示出三叶草根瘤菌在地区分布的局限性,可能与当地的地理环境和气候条件有关。刘晓云等[13]分离的根瘤菌主要采自江西、湖北、云南等地的三叶草属植物,范围较广,具有良好的多样性,而本研究的三叶草根瘤菌均来源于河南汝州。而我们过去的研究中也发现来自中国新疆木垒鹰嘴豆主产区的鹰嘴豆共生根瘤菌种群也是很单一的一个种群——木垒中慢生根瘤菌种群[17],这说明长期的地理隔离可能导致某一地区某种豆科植物的共生根瘤菌种群比较单一。杨亚珍[14]在对西北干旱地区三叶草根瘤菌的研究中发现,不同地理来源、同一地理来源、甚至同一植株不同根瘤的菌株在各方面存在着差异的生理生化性状,本研究一定程度上也证明了三叶草地理分布的差异,导致其共生根瘤菌类群组成的差异,二者是一致的。通过IGS PCR-RFLP的结果分析来看,21株白三叶草的根瘤菌分为6种类型,这说明尽管在同一采样地点的根瘤菌在很大程度上受地域、生态环境影响而表现为相似的类群,但是进化过程中却表现出较好的群内多样性,这与潘明洪等[15]分离自四川地区的白三叶草根瘤菌的属较为单一,但却含有丰富的群内多样性的结果相一致。在今后的研究中,需要扩大地理范围和菌株数量,以便更全面探索白三叶草根瘤菌的多样性分布。
[1] 金晓梅,Синеговская В Т,赵念力. 根瘤菌、微肥和作物生长调节剂对大豆氮磷钾积累和产量的影响[J]. 大豆科学,2009,28(4):751-754.
[2] 陈文新. 豆科植物根瘤菌-固氮体系在西部大开发中的作用[J]. 草地学报,2004,12(1):1-2.
[3] 赵宇枢,段玉玺,王媛媛,等. 辽宁省大豆根瘤菌资源抗逆性及生防潜力研究[J]. 大豆科学,2009,28(1):113-117.
[4] 马中雨,李俊,张永芳,等. 大豆根瘤菌与大豆品种共生匹配性研究[J]. 大豆科学,2008,27(2):221-227.
[5] 戴廷波,孙传范,荆奇,等. 不同施氮水平和基追比对小麦籽粒品质形成的调控[J]. 作物学报,2005,31(2):248-253.
[6] 孟庆英,张立波,张春峰,等. 根瘤菌对大豆生理及农艺性状的影响[J].黑龙江农业科学,2015(1): 27-28.
[7] 徐广. 西安地区三叶草根瘤菌生物学特性及共生固氮效应的研究[D]. 西宁:青海师范大学, 2009.
[8] 姚竹云,陈文新. 根瘤菌的现代分类及其系统发育[J]. 微生物学杂志,1998,18(1):38-43.
[9] 张小平,李阜棣. 根瘤菌的遗传多样性与系统发育研究进展[J]. 应用与环境生物学报,2002,8(3):325-333.
[10]张俊杰. 新疆鹰嘴豆根瘤菌的生物学特征研究[D]. 北京:中国农业大学,2014.
[11]VINCENT J M.A manual for the practical study of the root-nodule bacteria[M].Oxford and Edinburgh: Blackwell Scientific Publications,1970: 1-13.
[12]TEREFEWORK Z, KAIJIALAINEN S, LINDSTROM K. AFLP fingerprinting as a tool to study the genetic diversity ofRhizobiumgalegaeisolated fromGalegaorientalisandGalegaofficinalis[J]. Journal of Biotechnology, 2001, 91:169-180.
[13]刘晓云,陈文新. 三叶草、猪屎豆和含羞草植物根瘤菌16S rDNA PCR-RFLP分析和数值分类研究[J]. 中国农业大学学报,2003,8(3):1-6.
[14]杨亚珍. 西北干旱地区甘草根瘤菌的表型多样性及系统发育[D]. 杨凌:西北农林科技大学, 2004.
[15]潘明洪,凌瑶,景文,等. 四川白三叶根瘤菌遗传多样性及系统发育研究[J]. 草业学报,2014,3(5): 143-152.
[16]吴云汉. 大豆根瘤菌菌株接种效果的研究[J].河南农业大学学报,1996,30(1):99-101.
[17]张俊杰,郭晨,杨旭,等. 鹰嘴豆根瘤菌多样性研究进展[J].轻工学报,2016,31(6):1-7.
(责任编辑:朱秀英)
The isolation and molecular identification ofRhizobiaassociated with white clover from Ruzhou
ZHANG Junjie, YANG Xu, LU Yong, ZHANG Zhiyan, WANG Zhiwei, WEI Xiwang
(College of Food and Bioengineering, Zhengzhou University of Light Industry, Zhengzhou 450002, China)
A series of molecular identification methods including 16S rDNA PCR-RFLP (Restriction Fragment Length Polymorphism), IGS PCR-RFLP, 16S rDNA gene sequencing and phylogenetic analysis were used to study the genetic diversity of clover rhizobia from Ruzhou. The results showed that a total of 21 clover rhizobia were obtained and they were of the same kind of 16S rDNA PCR-RFLP pattern and were divided into six IGS PCR-RFLP patterns. Through the 16S rDNA gene sequencing and phylogenetic alalysis of the representative strains, the representatives were with inner group similarities of 99.9-100% and were with 99.9%~100% similarities of the knownRhizobiumspp., such asR.anhuienseandR.leguminosarum. So the clover rhizobia isolated from Ruzhou can be identified asRhizobiumstrains.
white clover;Rhizobia; 16S rDNA; IGS; RFLP
2016-04-13
国家自然科学基金项目(31400008);郑州轻工业学院博士科研基金项目(2014bsjj006)。
张俊杰(1984-),男,河南郑州人,讲师,主要从事细菌分类与分子生态学方面的研究。
1000-2340(2017)01-0076-06
Q939.11+4
A

