牛IRS1基因3’UTR的克隆、鉴定及生物信息学分析
李文清, 李中举, 孟志鹏, 牛梦宇, 李万利
(1.河南农业大学生命科学学院,河南 郑州 450002; 2.河南省农业科学院畜牧兽医研究所,河南 郑州 450002)
牛IRS1基因3’UTR的克隆、鉴定及生物信息学分析
李文清1, 李中举1, 孟志鹏1, 牛梦宇1, 李万利2
(1.河南农业大学生命科学学院,河南 郑州 450002; 2.河南省农业科学院畜牧兽医研究所,河南 郑州 450002)
以奶牛乳腺组织为材料,采用3’RACE技术克隆到奶牛IRS1基因3’UTR的全长,进一步对测得的序列进行了验证,同时利用TargetScan 7.0和PicTar软件预测可能结合的microRNA,并对多个软件预测结合的microRNA,进行最小结合自由能分析。结果表明,成功克隆了1 327 bp的牛IRS1 3’UTR全长,与NCBI上的预测序列一致率达到99%;miR-128、miR-96、miR-27a为2个软件共同预测到的结合microRNA;3个结合位点的最小自由能分别为-89.58 、-87.91 、-100.88 kJ·mol-1,表明牛IRS1基因表达可能受到这3个microRNA的调控。
牛;胰岛素受体基质1;3’非翻译区;3’RACE;最小自由能
胰岛素受体基质1(Insulin Receptor Substrate 1,IRS1)是一种几乎在所有细胞和组织中都表达的蛋白,是类胰岛素生长因子1受体(Insulin like growth factor 1 receptor, IGF1R)和胰岛素受体(Insulin Receptor, IR)的停泊蛋白[1],属于胰岛素受体基质家族。这个家族由4个蛋白构成,分别是IRS1~IRS4,IRS1是这个家族中的主要成员。它能激活下游多个信号通路,主要包括PI3K/AKT和MAPK/ERK信号通路,通过这些信号通路IRS1可以调节代谢、促进生长及促进肿瘤恶性转化,在细胞信号网络中起着核心作用[2]。虽然牛的基因组序列在2009年就已经公布出来,但是很多基因都是预测序列,其3’UTR是未知的,牛的IRS1基因就是这样的。在NCBI上只有IRS1的预测序列,序列号为XM_003581871.2,而这段序列在Ensenble数据库中显示全部为内含子,因此不同数据库报道不一致。IRS1基因mRNA的3’非编码区(UTR)作为其mRNA的重要组成部分,可能与microRNA结合而被引导发生降解或翻译受阻,从而引起下游信号通路发生改变。miRNA是一类长度约23 nt的内源性的非编码RNA,它与奶牛乳腺泌乳和乳成份合成相关[3-5]。为研究奶牛IRS1基因在奶牛乳腺泌乳网络中所起的作用,找到可能对其调控的microRNA,阐明IRS1基因转录后调控对奶牛泌乳、促生长及代谢的影响,本研究根据GenBank中公布的牛IRS1预测序列设计引物,应用RACE(Rapid Amplification of cDNA Ends,cDNA末端快速扩增技术测序)法成功地克隆了牛IRS1 3’UTR序列,并对其序列进行生物信息学分析。
1 材料与方法
1.1 材料
1.1.1 试验材料 参照MOYES活体取样的方法[6]采集泌乳中期荷斯坦奶牛乳腺组织0.5 g,置于液氮中保存备用。
1.1.2 菌株和试剂 大肠杆菌感受态细胞JM109;克隆载体T-Vector p MDTM20;总RNA的提取采用RNAiso Plus;基因组DNA的去除采用Recombinant DNase I(RNase Free);3‘RACE合成试剂盒3′-Full RACE Core Set with PrimeScriptTMRtase;胶回收纯化试剂盒MiniBEST Agarose Gel DNA Extraction Kit Ver.3.0;PCR扩增试剂盒LATaq®with GC Buffer;连接酶DNA Ligation Kit Ver.2.1,以上产品均购自TaKaRa公司。
1.1.3 引物设计与合成 根据NCBI上发表的IRS1预测序列(GenBank号:XM_003581871.2),采用Oligo 6.0引物设计软件设计1对特异引物进行已知序列验证:上游引物F0: GGTCAAGGACTTCAAACAGC;下游引物R0: ATACCTCAGTTCCACATCCC。3’端序列同源获取序列设计的上游引物F4: CCACAAGAGAATGAGTTTACGC;下游引物R4: AAAAGTGACAATGTAGCCCTGGT。IRS1 3’RACE的引物设计,外侧上游引物F7: TTTGGAATGAATAATGTGGGTGTT;内侧上游引物F8: TTCATTTCCTGCATTGTCTCA,下游引物由TaKaRa 3’RACE试剂盒提供。对IRS1 3’RACE序列验证设计的上游引物F1: GAAGACCTAAATGACCTCAGCAAA;下游引物R5: TAATGTTACACTGGACGTTCTC。引物由上海生工合成。
1.2 方法
1.2.1 牛乳腺组织Total RNA 提取 使用RNAiso Plus(TaKaRa)提取奶牛乳腺组织Total RNA,具体操作按照试剂盒说明书进行。然后使用Recombinant DNase I(RNase Free)(TaKaRa)进行基因组DNA的去除。
1.2.2 牛IRS1基因3’UTR的扩增 具体操作参照3’RACE试剂盒说明书进行,即使用已知序列验证时的反转录反应液,分别以F7加RACE outer primer、F8加RACE inner primer进行套式PCR扩增。PCR扩增的反应程序为:94℃预变性2min;94℃,30s;55℃,30s;72℃,1min;35个循环;最后72℃延伸5min。
1.2.3 序列拼接 使用Sequencher 4.9软件对上述测得的序列进行拼接。
1.2.4 牛IRS1基因3’UTR的克隆、鉴定及分析 将3’RACE产物纯化回收后,克隆于M13-47载体中进行测序,将序列测通。后又对RACE序列进行PCR扩增、纯化、测序,以验证RACE序列是否是根据已知序列得到的。
1.2.5 序列BLAST比对分析 牛与其他哺乳动物IRS1基因的3’UTR的比较生物信息学分析 应用NCBI上的BLAST软件进行同源序列比对,确定测序基因所属的物种。
1.2.6 靶位点预测 利用miRanda软件预测牛IRS1基因 3’UTR区miRNA结合位点。
2 结果与分析
2.1 牛乳腺组织Total RNA提取结果
利用Trizol试剂从牛乳腺组织中提取到了完整的总RNA,经凝胶电泳呈现3条带(图1),其R260/280在1.8和2.0之间,纯度较好。
2.2 已知序列验证结果
取5 μL以F0和R0为引物扩增的PCR产物进行1%琼脂糖凝胶电泳,结果如图2。在750 bp和500 bp间出现单一的预期目的条带,经胶回收纯化PCR产物后,对其进行测序,测序结果与已知序列一致,为590 bp的序列。
2.3 3 ′端序列获取结果
通过设计F4和R4引物,取5 μL同源获取的PCR产物进行1%琼脂糖凝胶电泳,结果如图3。
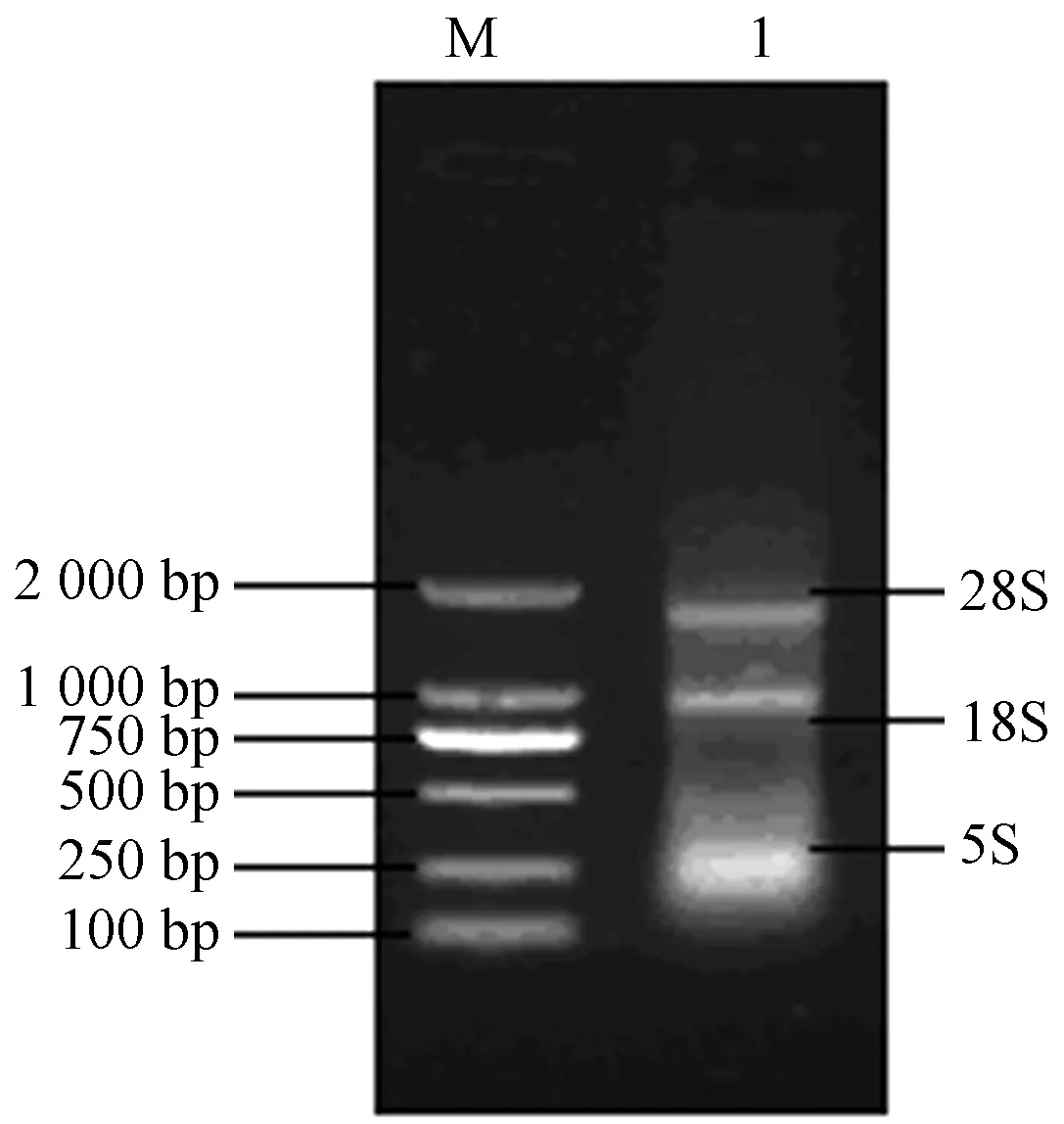
注:M为DL2000 DNA Marker;1为牛乳腺组织Total RNA。
Note: M indicates DL2000 DNA Marker; 1 indicates total RNA of bovine mammary gland.
图1 牛乳腺组织Total RNA电泳图
Fig.1 Total RNA eletrophoretogram of bovine mammary gland tissue

注:M为DL2000 DNA Marker;Lane 1为已知序列的PCR产物;Lane 2为未加反转录酶的阴性对照。
Note: M indicates DL2000 DNA Marker; Lane 1 indicates PCR production of known sequence; Lane 2 indicates negative control without reverse transcriptase.
图2 已知序列PCR电泳图
Fig.2 PCR electrophoretogram of known sequence
在1 000 bp处有单一条带。经胶回收纯化后再次进行测序分析表明其大小为937 bp。
取5 μL 3′RACE的套式PCR产物进行1%琼脂糖凝胶电泳,结果如图4。
切胶回收目的产物,约250 bp,命名为CTG111-3P1。直接CTG111-3P1测序,序列有套峰,需要克隆测序。克隆测序时使用M13-47对CTG111-3P1-1和CTG111-3P1-11质粒测序,将序列测通。
2.4 RACE序列验证结果
取5 μL CTG111 F1/R5为引物扩增的PCR产物进行1%琼脂糖凝胶电泳,结果如图5。图5表明在2 000 bp和1 000 bp间出现单一条带。对PCR产物进行纯化测序,测序结果表明得到的RACE序列是根据已知序列得到的。
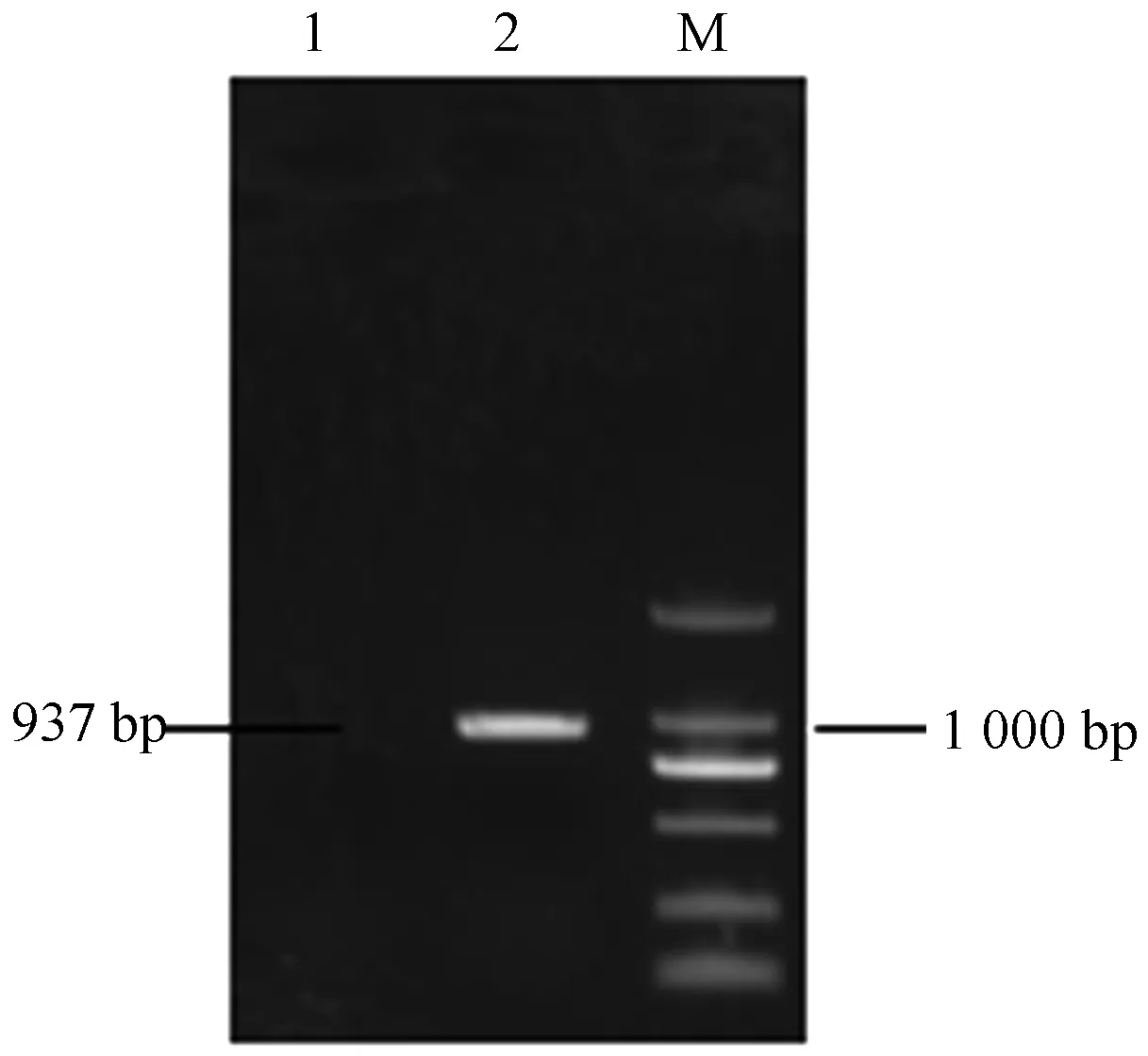
注:M为DL2000 DNA Marker;Lane 1为未加反转录酶的阴性对照;Lane 2为同源获取的PCR产物。
Note: M indicates DL2000 DNA Marker; Lane 1 indicates negative control without reverse transcriptase; Lane 2 indicates PCR production of homologous acquisition.
图3 同源获取的PCR产物电泳图
Fig.3 PCR eletrophoretogram of homology sequence
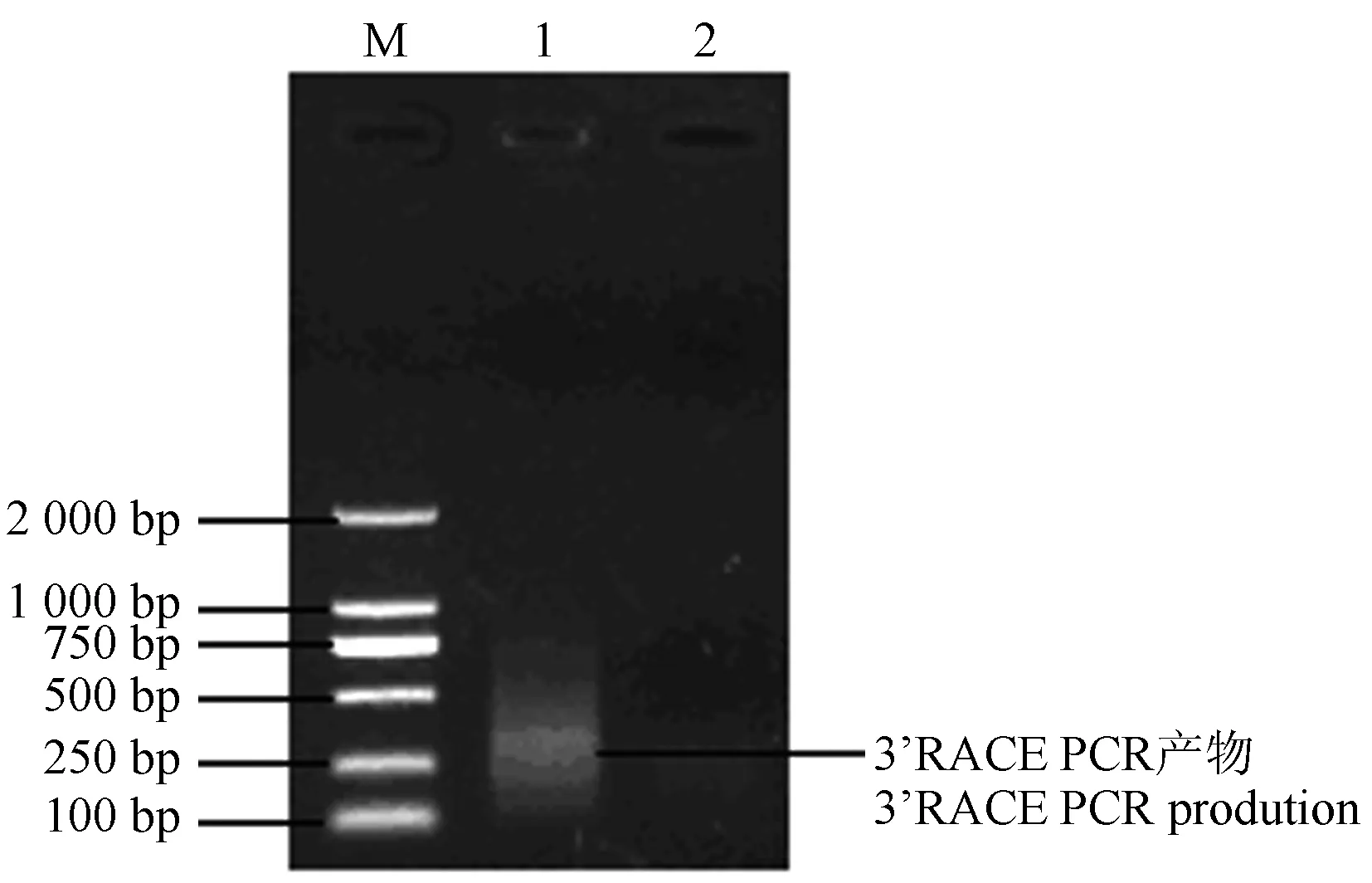
注:M为DL2000 DNA Marker;Lane 1为3’RACE PCR产物;Lane 2为未加反转录酶的阴性对照。
Note: M indicates DL2000 DNA Marker; Lane 1 indicates PCR production of 3’RACE; Lane 2 indicates negative control without reverse transcriptase.
图4IRS1基因3′RACE套式PCR电泳图
Fig.4 PCR electrophoretogram ofIRS1 3’RACE
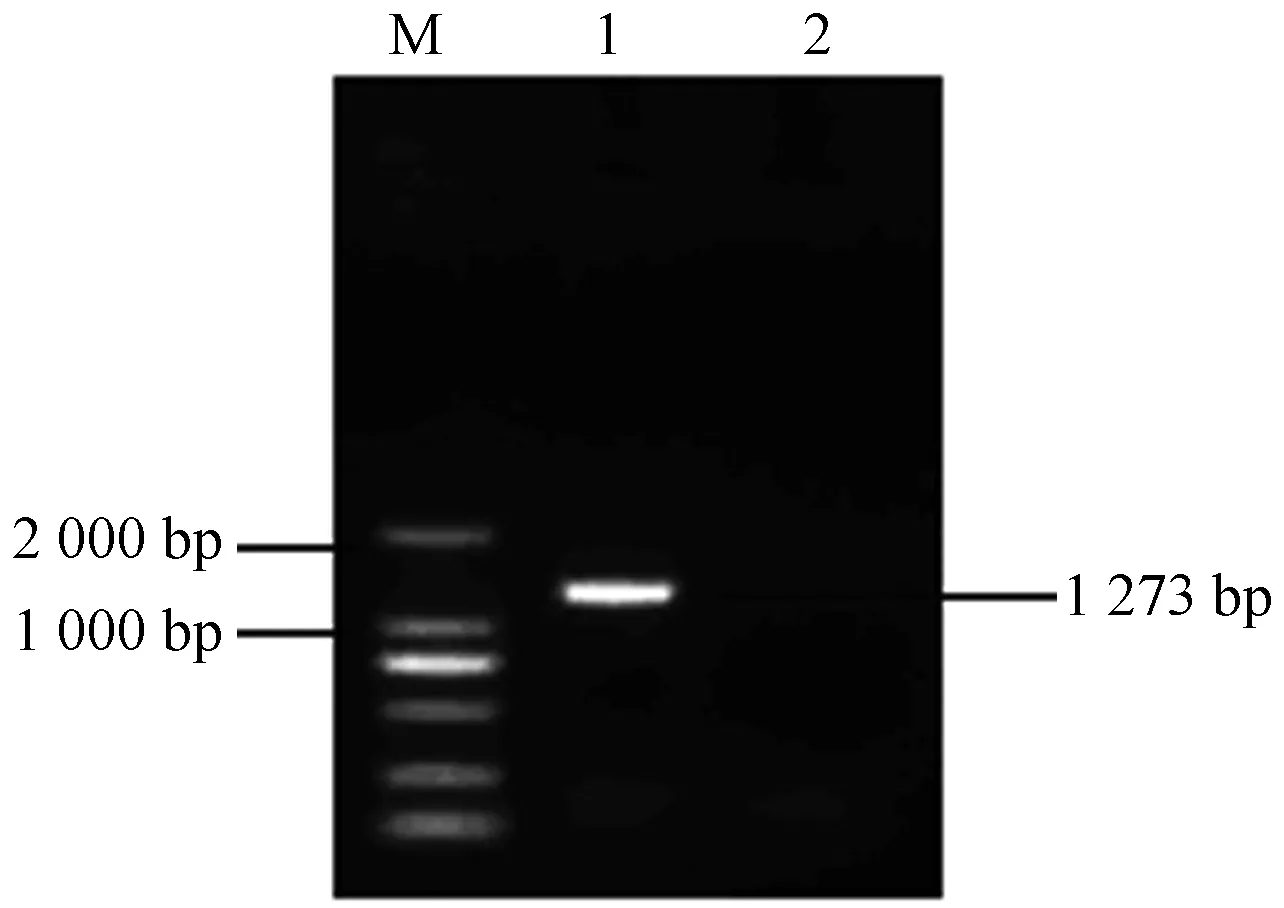
注:M为DL2000 DNA Marker,Lane 1为RACE序列验证PCR产物,Lane 2为未加反转录酶的阴性对照。
Note: M indicates DL2000 DNA Marker; Lane 1 indicates PCR production of RACE sequence; Lane 2 indicates negative control without reverse transcriptase.
图5 RACE序列验证的PCR产物电泳图
Fig.5 PCR electrophoretogram of verifing RACE sequence
2.5 序列拼接和同源比对结果
用Sequencher 4.9软件对上述测得的序列进行拼接,最终获得的3’端未知序列。测序、扩增加上已知序列共获得可确认的牛IRS1长度为1 516 bp,3’UTR序列长度为1 338 bp,其中包括11 bp polyA的尾巴,将polyA尾巴去掉后还剩下1 327 bp。对测得的序列进行同源比对,结果发现IRS1基因的3’RACE序列与NCBI上公布的牛IRS1的预测序列(XM 003581871.2)基本一致,一致性达到99%。与人的IRS1序列的一致性为87%,可见IRS1基因的3’UTR在不同物种间的保守性较高,然而是否存在microRNA的结合位点还要进行下一步的分析。
2.6 靶位点预测及同源性分析
利用TargetScan 7.0软件分析可能结合的microRNA,有11个microRNA靶向牛IRS1基因3’UTR的保守位点,包括miR-128、miR-96/1271、miR-302a、miR-27-3p、 miR-15/16/195/424-5P/497、miR-29-3p。利用PicTar软件,有11个microRNA靶向牛IRS1基因3’UTR的保守位点,包括miR-126、miR-96、miR-145、miR-183、 miR-27a、miR-128a/b miR-27b、miR-30a-5p、miR-30d、miR-30e。取2个软件共同预测到的microRNA(miR-128、miR-96、miR-27a)和实测的IRS1 3’UTR序列代入网址http://bibiserv.techfak.uni-bielefeld.de/rnahybrid,进行结合位点最小自由能分析,结果如表1所示。

表1 结合位点自由能和二级结构分析Table 1 Free energy and secondary structure analysis of binding sites
3 讨论
一些研究者认为在奶牛乳腺泌乳过程中的确有大量的microRNA参与到乳汁合成及调控的过程中[3-5],但microRNA调控奶牛乳腺具体基因的报道较少。IRS1蛋白是多个信号通路中的关键蛋白,主要激活下游PI3K/AKT和MAPK/ERK信号通路。IRS1基因的转录后表达调控对研究奶牛泌乳启动及乳成份合成是非常重要的。但是,牛的基因很多都是预测序列,其3’UTR是未知的,这给后续的一些研究带来困难。本试验采用3’RACE技术来获得牛IRS1的3’UTR,最后获得1 327 bp的片段长度,不包括末尾的Poly(A),与NCBI上公布的牛IRS1的预测序列(XM 003581871.2)基本一致,一致性达到99%。通过3’RACE测序还获得IRS1 11bp polyA的尾巴,这个polyA序列不是真实的长度。3’RACE接头序列polyT的长度是人为设定的,根据试剂盒的不同,polyA的长度也不相同,所以本研究得到这个polyA序列是预测值,不能反应它的真实长度。利用生物信息学软件对测得的牛IRS1 3’UTR序列进行microRNA靶位点预测,结果得到许多可能结合的microRNA。多个软件共同预测到的microRNA具有较高的可能性,本研究就进一步对2个软件共同预测到的microRNA进行结合位点最小自由能分析。分析发现bta-miR-128、bta-miR-96、bta-miR-27a与IRS1 3’UTR的结合位点最小自由能(△G)≤-62.79 kJ·mol-1,一般认为最小自由能(△G)≤-62.79 kJ·mol-1是可以结合的[7]。
[1] SHPAKOV A O, PERTSEVA M N. Structural and functional characterization of insulin receptor substrate proteins and the molecular mechanisms of their interaction with insulin superfamily tyrosine kinase receptors and effector proteins[J]. Membr Cell Biol, 2000, 13:455-484.
[2] CAO Z, LIU L Z, DIXON D A, et al. Insulin-like growth factor-I induces cyclooxygenase-2 expression via PI3K, MAPK and PKC signaling pathways in human ovarian cancer cells[J]. Cell Signal, 2007, 19:1542-1553.
[3] LI Z, LIU H Y, JIN X, et al. Expression profiles of microRNAs from lactating and non lactating bovine mammary glands and identification of miRNA related to lactation [J]. BMC Genomics, 2012, 13:731-746.
[4] GU Z, ELESWARAPU S, JIANG H. Identification and characterization of microRNAs from the bovine adipose tissue and mammary gland[J]. FEBS Lett, 2007, 581 (5):981-988.
[5] WANG M, MOISS, KHAN M J, et al. MicroRNA expression patterns in the bovine mammary gland are affected by stage of lactation[J]. J Dairy Sci, 2012,95(11): 6529-6535.
[6] MOYES K M, DRACKLEY J K, MORIN D E, et al.Gene network and pathway anaylsis of bovine mammary tissue challenged with streptococcus uberis reveals induction of cell proliferation and inhibition of PPARgamma signaling as potential mechanism for the negative relationships between immune response and lipid metabolism[J].BMC Genomics, 2009,19(10):542-552.
[7] HOFACKER I L. Vienna RNA secondary structure server[J]. Nucleic Acids Res, 2003, 31(13):3429-3431.
(责任编辑:蒋国良)
Cloning, identification and bioinformatics analysis of the 3’UTR of bovineIRS1
LI Wenqing1, LI Zhongju1, MENG Zhipeng1, NIU Mengyu1, LI Wanli2
(1.Life Science College, Henan Agricultural University, Zhengzhou 450002,China; 2 Institute of Animal Husbandry and Veterinary Science, Henan Academy of Agricultural Sciences, Zhengzhou 450002,China)
Using bovine mammary tissue as material, bovineIRS1 gene 3’UTR full length was cloned by 3’RACE technology. Measured sequence was further validated. At the same time, TargetScan 7.0 and PicTar sofeware were used to predict the possible binding microRNA. Minimal binding free energy of the multiple software conjunct predicting microRNAs was analyzed. The results showed that the 1327bp 3’UTR full length of bovineIRS1 was successfully cloned. The detected sequence was 99% consistent with the predicted sequences in NCBI. MiR-128,miR-96 and miR-27a were predicted by two authoritative software together. The minimum free energy of three binding sites was -89.58, -87.91 and -100.88 kJ·mol-1, respectively. These results suggested that the bovineIRS1 gene expression might be regulated by the three microRNAs.
bovine;IRS1; 3’ untranslated region; 3’RACE; minimum free energy
2016-05-23
国家自然科学基金项目(31501978);河南省高等学校重点科研项目(16A230010)
李文清(1979-),女,河南潢川人,副教授,主要从事奶牛营养与牛奶品质研究。
李万利(1979-),男,河南驻马店人,副研究员,博士。
1000-2340(2017)01-0048-05
S852.23
A

