中国青凤蝶3个亚种的遗传变异分析
杨斯琦 王亚楠 王方方 何娅 靳亚丽
摘要[目的]探讨中国青凤蝶(Graphium sarpedon Linnaeus)3个亚种的遗传变异。[方法]对青凤蝶3个亚种的COⅠ基因和EF-1α基因部分序列进行测定,并对序列的碱基组成、转换颠换数和遗传距离等进行分析。[结果]在测得的COⅠ基因(709 bp)和EF-1α(1 064 bp)基因中,有44个变异位点,6个简约信息位点,COⅠ基因A+T平均含量为69.3%,存在较强的含量偏向性,亚种间的遗传距离相差甚小。[结论]青凤蝶3个亚种之间没有明显序列差异,仍然适宜采用传统的基于形态学的青凤蝶亚种分类体系。
关键词青凤蝶;COⅠ基因;EF-1α基因;遗传变异
中图分类号Q969.42文献标识码A文章编号0517-6611(2017)31-0156-04
Abstract [Objective]To investigate the genetic variation in three subspecies of Graphium sarpedon Linnaeus in China.[Method] The COⅠ gene and the EF1α gene were partially sequenced.The nucleotide composition, the number of transition and transversion, genetic distance of the segment had been analyzed.[Result]There were 44 variation sites and 6 parsimonyinformative sites in combined sequence of the COⅠ gene(709 bp)and EF1α(1 064 bp) gene in the examined species. The average A+T content of COⅠ gene was 69.3%,which showed strong A+T bias. But genetic distance among subspecies were very small.[Conclusion]It has no significant sequence differences between the three subspecies, which is still suitable to use the traditional morphological classification system for Graphium sarpedon subspecies.
Key wordsGraphium sarpedon;COⅠ gene; EF1α gene;Genetic variation
青凤蝶(Graphium sarpedon Linnaeus),又名樟青凤蝶或青带凤蝶,属于鳞翅目(Lepidoptera),凤蝶科(Papilionidae),青凤蝶属(Graphium),分布于我国陕西、湖北、湖南、四川、云南、贵州、西藏、江西、浙江、福建、广西、广东、海南、台湾、香港等地区[1]。青凤蝶在我国有3个亚种,即指名亚种[G.sarpedon sarpedon(Linnaeus)]、蓝斑亚种[G. sarpedon connectens(Fruhstorfer)]、斑带亚种[Graphium sarpedon semifasciatum(Honrath)]。目前对青凤蝶斑带亚种的分类尚存有争议,魏忠民等[2]通过对中国青凤蝶标本的观察,归纳出6种中国青凤蝶的变异型,描述其主要形态特征,认为青凤蝶的斑带亚种是一种变型,而不是亚种。对于青凤蝶亚种的争议问题,以DNA序列为基础的分辨依据研究较少。
目前国内外学者结合线粒体基因和核基因等分子标记
对昆虫类群的系统学研究已有大量的文献报道。线粒体DNA具有结构简单、进化速度快、高拷贝数、易于分离纯化等特点,是研究系统发育、种群遗传变异和分化、区分近缘种的重要工具[3]。由于线粒体基因是母系遗传,其中所含的进化信息并不能完全反映双亲进化的历史,核基因也含有丰富的生物学信息,结合适当的核基因将更好地分析蝶类的系统发育[4]。
笔者对我国分布的青凤蝶3个亚种的COⅠ基因和EF-1α基因部分序列进行测定,研究它们在DNA序列上的差异,以期为我国青凤蝶3个亚种分类地位提供分子证据。
1材料与方法
1.1材料
青凤蝶3个亚种的标本来自我国广西、上海,包括作为外群的木兰青凤蝶,共 15只成虫标本(表1)。
1.2方法
1.2.1提取基因组DNA及PCR扩增。
选取部分腹部或足部组织作为试验材料,采用试剂盒insect kit(Omega)提取基因组DNA,样品保存在-20 ℃备用。試验采用的引物序列如下。
COⅠ上游引物:5′-GGTCAACAAATCATAAAGATATTG-3′;
下游引物:5′-TAAACTTCAGGGTGACCAAAAAAT-3′。
EF-1α上游引物:5′-TCGATATCGCTTTGTGGAAGTT-3′;
下游引物:5′-ACGCACGGCAAAACGACCGAGAG-3′。
PCR 反应体积为50 μL,反应体系包括1.25 U的 Taq DNA聚合酶,0.2 mmol/L dNTP,上下游引物分别为0.4 μmol/L,10×PCR Buffer 为5 μL,模板 DNA 溶液 2 μL(50~100 ng DNA)。扩增条件为95 ℃预变性3 min;95 ℃变性30 s,55 ℃复性30 s,72 ℃延伸1 min,30个循环;最后在72 ℃延伸7 min,4 ℃保温。用 Eppendorf 梯度PCR仪进行扩增。
1.2.2PCR产物纯化、测序。
用1%的琼脂糖凝胶电泳检测 PCR 产物,用DNA 凝胶回收试剂盒,对目的条带进行回收和纯化后,连接到T载体,经过转化挑克隆,提取质粒再委托上海赛默飞(Thermo Fisher)公司进行双向测序。
1.2.3序列分析及差异比较。
测定15只成虫标本的COⅠ和EF-1α基因部分序列,正反链序列拼接后,再Blast进行序列同源性比较,基于Kimura-2参数,采用MEGA6.0软件进行序列组成分析,如序列碱基组成(nucleotide composition)、保守位点(conservedsjtes)、变异位点(variable sjtes)、简约信息位点(parsimony information sites)、自裔位点(singletonsites)等。选择Tamurar Nei模型,计算各分类单元之间的遗传距离、转换数与颠换数比值(Ts/Tv)。
2结果与分析
2.1基因序列的确定
根据所选引物的位置,COⅠ基因和EF-1α基因预测扩增的片段分别是709 bp和1 064 bp,电泳检测的结果与预期相符(图1)。运用Blast对序列进行分
析,显示其与青凤蝶的COⅠ基因和EF-1α基因具有很高
的同源性。
2.2序列组成分析
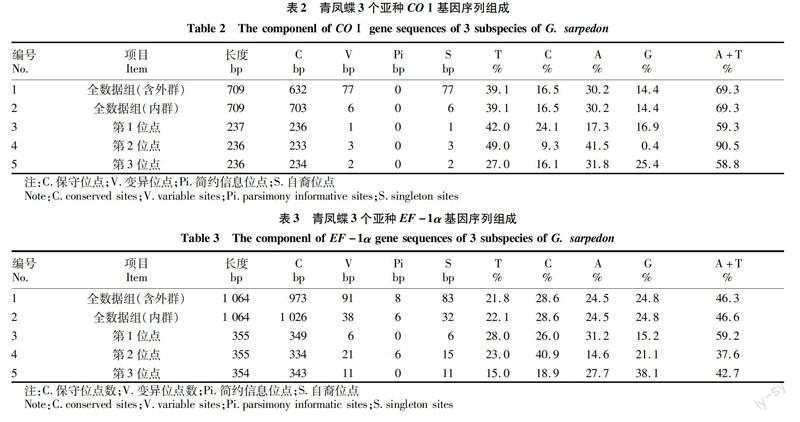
除外群外,COⅠ基因部分序列共709 bp,变异位点6 bp,无简约信息位点,自裔位点6 bp,碱基组成上 T、C、A、G的含量分别是39.1%、16.5%、30.2%、144%,A+T平均含量为69.3%,其中密码子第2位点A+T含量最高,达90.5%,第2位点G含量最低,平均为0.4%,T含量最高,达49.0%,这表明密码子的碱基使用频率存在明显的偏向性(表2)。
核EF-1α基因部分序列共1 064 bp,变异位点38 bp,简约信息位点6 bp,自裔位点32 bp,T、C、A、G的含量分别是22.1%、28.6%、24.5%、24.8%,A+T平均含量为46.6%,其中第1位点含量最高,达到59.2%,第1位点G的含量最低,平均为15.2%,T的含量最高,达到28%,表明密码子的碱基使用频率也存在一定的偏向性(表3)。
通过Paup4.0b10软件对COⅠ基因和EF-1α基因进行PHT检验(同质性检验partition homogeneity test),结果显示基因进化水平不具有显著差异(P=1.0>0.01),因而可以将2组数据整合在一起进行分析。利用MEGA6.0软件,基于Kimura-2参数对COⅠ和 EF-1α基因部分序列的整合数据进行分析,统计碱基转换数与颠换数比值(R)。
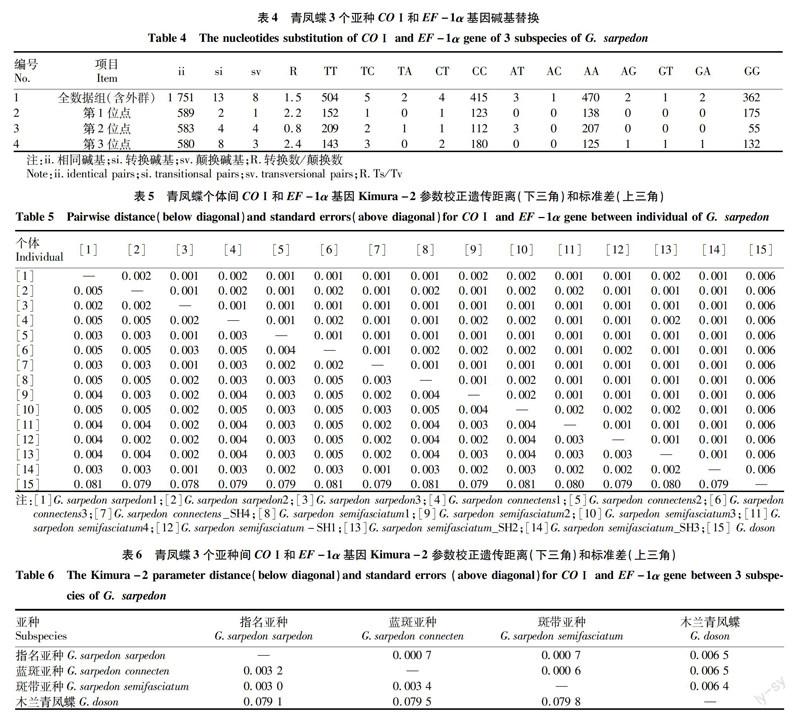
由表4可知,核苷酸替換发生率比较低,第3位点略高,转换多于颠换,第1位点最保守。转换数与颠换数比值(R)平均为1.5<2,表现出较为明显的替换饱和。
2.3遗传距离分析
基于Kimura-2参数,采用MEGA 6.0计算个体间及亚种间的遗传距离(表5)。不含外群的个体两两之间的遗传距离在0.1%~0.5%,平均遗传距离是03%,将3个亚种进行分组后计算遗传距离,并与外群对照(表6),我国青凤蝶3个亚种间遗传距离是0.30%~034%,两两之间的遗传距离相差很小,其中指名亚种[G.sarpedon sarpedon(Linnaeus)]与斑带亚种[G.sarpedon semifasciatum(Honrath)]差异相对更小,与蓝斑亚种[G.sarpedon connectens(Fruhstorfer)]的差异相对更大。
2.4系统发育信号检测
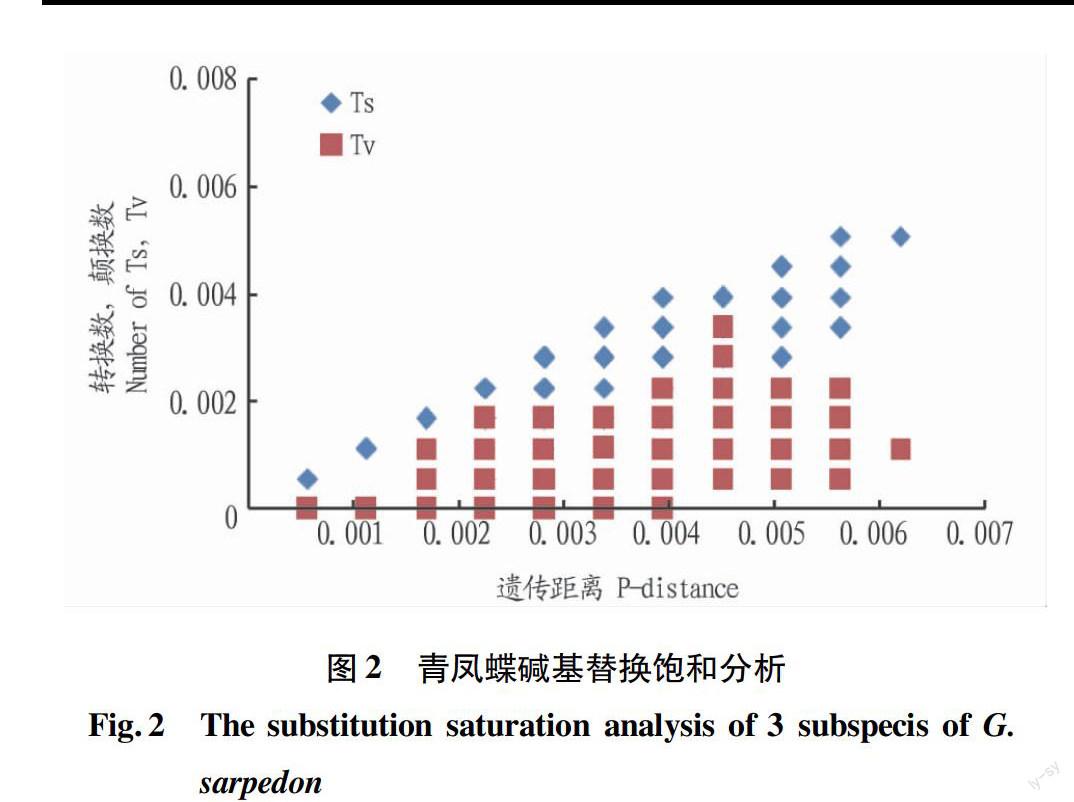
采用碱基替换饱和性分析,对数据进行系统发育信号强弱的检测,以遗传距离为横坐标,以碱基转换数和颠换数为纵坐标作散点图(图2)。
从图2可以看出,随着遗传距离的增大,Ts增加的速度大于Tv增加的速度,且与遗传距离之间有良好的线性关系,Tv则逐渐趋于稳定,达到饱和。
通过Paup4.0b10软件对数据做PTPtest序列信息检验,结果显示P为0.002,表明该数据具有较显著的系统发育信息,而非随机数据,可用来进行系统发育的推断[5-6]。
3讨论
我国青凤蝶分布有3个亚种,据《中国蝶类志》中描述,青凤蝶的形态特征为翅窄长,底色黑,无尾状突起,前后翅中央贯穿1列略呈方形的蓝绿色斑(后翅前面的1个为白色),后翅外缘有1列绿蓝色的新月斑。后翅反面近翅基有1条红色短线,翅中部至后缘处有数条红色斑纹。
3个亚种之间形态特征存在一些差异,青凤蝶蓝斑亚种的绿色斑偏蓝,前翅顶角有1个绿斑特别小;斑带亚种的后翅色带不全。有学者提出青凤蝶的斑带亚种是一种变型,而不是亚种。为此,该研究联合2个基因序列的整合数据对青凤蝶3亚种间遗传变异进行了分析。之所以选择COⅠ和EF-1α基因联合分析,一方面是增加系统发生分析的置信度[7-8],另一方面也与2个基因各自的特点有关。线粒体COⅠ是编码线粒体细胞色素氧化酶亚基Ⅰ的基因,是常用的分子标记之一,它既相对保守又存在高变区,高变区内遗传进化速率更快,种间的遗传差异也更明显,适合于种间和种内分类鉴定及系统发生研究[9]。EF-1α基因进化速度比较快,它也用于分析低阶元的系统发育,已有学者做了相关的研究,如Reed等[10]用线粒体COⅠ、COⅡ基因的全长和核基因的EF-1α基因联合分析了凤蝶科凤蝶属23个种和亚种的个体。
在鳞翅目同种个体之间,COⅠ基因的差异为025%[11],从试验结果来看,我国青凤蝶3个亚种间COⅠ基因相似
度为 99.15%,核苷酸差异很小,表明COⅠ基因在个体间变异率较低。3个亚种间EF-1α基因相似度96.43%,表明存在一定的变异率。3亚种青凤蝶在形态上的差异可能
是由于地理隔离形成,其自身的遗传变异性非常低[12]。
从系统发育树的拓扑结构看,其未显示3个亚种间有很好的单系性,而出现了并系性,且各支自举检验值较低(<50%),因而没有列出发育树,置信度低下可能是因为分子数据所提供的信息位点较少[13],或者是COⅠ基因和EF-1α基因序列突变达到饱和。结合对3个亚种基因序列两两距离进行P-distance计算,并按照亚种分组后计算遗传距离,可以看出指名亚种和斑带亚种的亲缘关系较近,但不足以认定斑带亚种是属于指名亚种的一种变型。
综上所述,结合目前已有的2个基因部分序列和各亚种之间形态特征的差异,仍然适宜采用传统的基于形态学的青凤蝶亚种分类体系。
参考文献
[1] 武春生.中国动物志:昆虫纲 第25卷 鳞翅目 凤蝶科[M].北京:科学出版社,2001.
[2] 魏忠民,武春生.中国青凤蝶变异型初步观察[J].昆虫知识,2006,43(3):431-432.
[3] 寿建新.国内外蝴蝶分类认识总结[J].西安文理学院学报(自然科学版),2014,17(4):21-27.
[4] 袁锋,袁向群.蝶类分子系统学研究进展[J].西北农业学报,2013,22(12):1-14.
[5] KIM K J,JANSEN R K.A chloroplast DNA phylogeny of lilacs(Syringa,Oleaceae):Plastome groups show a strong correlation with crossing groups[J].American J Bot,1998,85(9):1338-1351.
[6] AUSTIN C C,ZUG G R.Molecular and morphological evolution in the southcentral Pacific skink Emoia tongana(Reptilia:Squamata):Uniformity and humanmediated dispersal[J].Australian J Zool,1999,47(5):425-437.
[7] MONTEIRO A,PIERCE N E.Phylogeny of Bicyclus(Lepidoptera:Nymphalidae)inferred from COI,COII and EF1α gene sequences[J].Mol Phylogenet Evol,2001,18(2):264-281.
[8] NORMARK B B.Molecular systematics and evolution of the aphid family Lachnidae[J].Mol Phylogenet Evol,2000,14(1):131-140.
[9] 吳冬霞,朱国萍,陈娜,等.线蛱蝶亚科蝶类部分类群线粒体COI 基因的分子系统发生分析[J].生命科学研究,2007,11(1):64-71.
[10] REED R D,SPERLING F A.Interaction of process partitions in phylogenetic analysis:An example from the swallowtail butterfly genus Papilio[J].Mol Biol Evol,1999,16(2):286-297.
[11] HEBERT P D N,STOECKLE M Y,ZEMLAK T S,et al.Identification of birds through DNA barcodes[J].PLoS Biol,2004,2(10):1657-1663.
[12] 叶维萍,叶海燕,卢慧甍,等.基于线粒体12S rRNA和ND5基因序列的中国飞蝗属3亚种系统发育关系研究[J].昆虫分类学报,2005,27(1):5-12.
[13] 刘晓燕,吴孝兵,诸立新.中国黄粉蝶亚科六属间基于COⅡ和EF-1α基因部分序列的系统发育关系(鳞翅目:粉蝶科)[J].昆虫学报,2007,50(6):604-609.
——和田盘羊

