基于ISSR—PCR分子标记的滑菇遗传多样性分析
李红+张敏+肖千明++宋莹++曹君


摘要:以辽宁省主栽的16个滑菇菌株为研究对象,应用ISSR分子标记技术对其遗传多样性进行了分析,从20条引物中筛选出9条多态性丰富、谱带清晰且重复性好的引物进行了ISSR-PCR扩增。9条引物共扩增出101条带,其中多态性谱带92条,多态位点百分率为91%。遗传相似系数变化范围为0.59~0.90。在遺传相似系数为0.69时,可将16个滑菇菌株划为5大类群,为今后滑菇的分类鉴定及遗传育种亲本的选配提供了理论依据。
关键词:滑菇;ISSR;分子标记;遗传多样性
中图分类号: S646.1+60.3文献标志码: A文章编号:1002-1302(2017)06-0039-03
滑菇(Pholiota nameko Ito et Imai)别称光帽鳞伞、光帽黄伞、滑子蘑、光滑环锈伞、光盖环锈伞、光盖库恩菇、珍珠菇,是一种较为常见的低温型食用菌,具有丰富的营养成分和很强的药理活性。滑菇是世界上五大人工栽培的优质食用菌之一。我国是滑菇的主要生产和消费大国,产量居世界首位,每年大量对外出口,国内市场滑菇价格高于香菇和平菇等其他菌类价格。目前滑菇栽培所用的菌株同名异物和同物异名现象严重,因此对滑菇菌株进行有效的分类和亲缘关系鉴定是滑菇育种成功与否的首要因素。
ISSR是1994年由Zietkiewicz等创建的一种简单序列重复区间扩增多态性分子标记,它是在SSR基础上发展起来的技术,根据基因组广泛存在SSR的特点,利用SSR本身设计引物,无需预先克隆和测序[1]。ISSR通常为显性标记,呈孟德尔式遗传,具有很好的稳定性和多态性,DNA用量少、技术要求低、成本低廉、通用性好,已成功地运用于居群生物学研究、菌株鉴定、物种的分类系统学比较,并作为构建遗传图谱的工具[2]。
在食用菌方面,ISSR技术较多用于种群内、种群间、物种间的系统发育和遗传多样性研究。秦莲花等利用ISSR标记结合ITS分析,以豹皮香菇和虎皮香菇为外群,对12个香菇菌株进行遗传分析,结果认为此方法是鉴别香菇菌株的良好方法[3]。王子迎等用13个ISSR引物对26个安徽野生香菇和6个栽培菌株进行了遗传多样性分析,并根据ISSR分析选择遗传距离远近不同的亲本进行杂交,评价了其杂种优势[4]。马志刚等基于重复序列(TATG),设计了7条引物,对侧耳属的22个菌株和1株双抱蘑菇菌株的基因组DNA进行ISSR分析,验证了重复序列(TATG)4在侧耳属中的存在[5]。Zhang等设计2个锚定ISSR引物,将17个香菇菌株很好地鉴别开[6]。但在滑菇方面,尚无相关研究报道。本研究应用ISSR-PCR分子标记的方法对滑菇菌株遗传多样性进行分析,以期为滑菇的分类鉴定及遗传育种亲本的选配提供理论依据。
1材料与方法
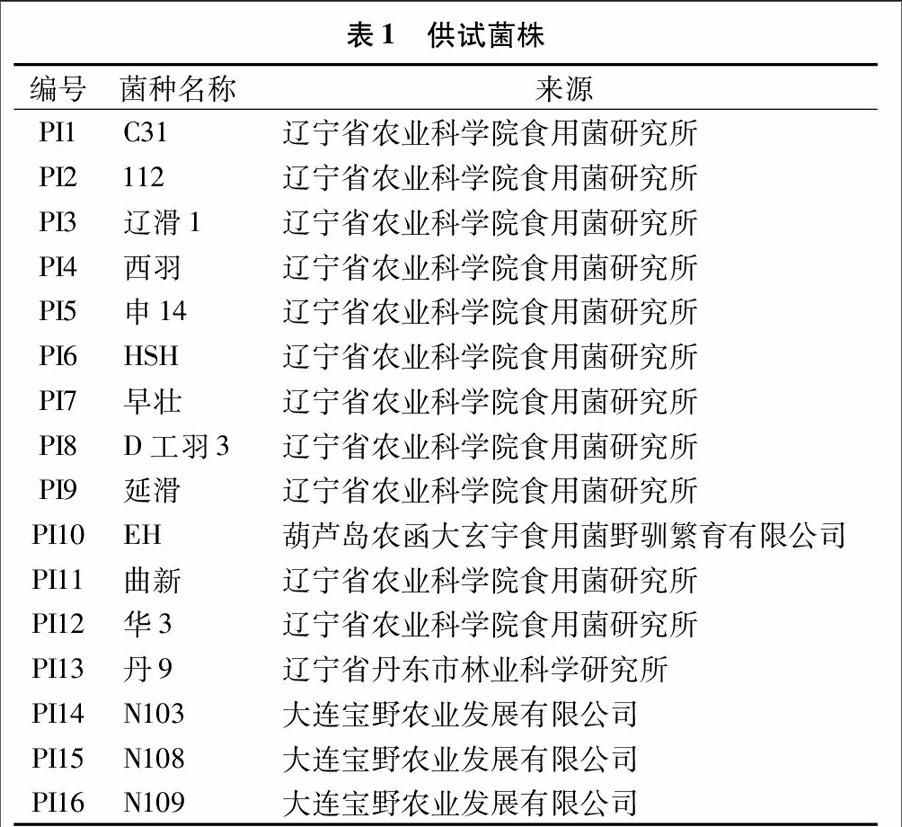
1.1供试菌株
本研究以辽宁省主栽的16个滑菇菌株为研究对象(表1)。
1.2培养基
PDB综合培养基用于总DNA提取的菌丝培养,配方:马铃薯400 g,葡萄糖40 g,磷酸二氢钾6 g,硫酸镁3 g,蛋白胨 6 g,水2 000 mL,pH值不需要调整。
1.3试剂和仪器
ISSR-PCR 反应所用的Taq DNA Polymerase、dNTP、10×buffer、Mg2+购自北京鼎国昌盛公司,DNA marker(DL5000)、引物购自 TaKaRa (大连)有限公司。PCR 仪为德国 Biometra 公司的 Thermocycler型。
1.4引物
本研究使用的20个引物见表2。
1.5菌丝体培
将活化的菌丝切下0.5 cm2菌块,接种于PDB综合培养基,23 ℃摇床培养,转数为150 r/min。将培养好的菌液,无菌水洗涤2次,汲干菌丝体表面的水分,-20 ℃保存备用。
1.6总DNA提取及检测
采用改良的CTAB法[7]提取基因组总DNA,0.2%琼脂糖凝胶电泳检测质量。
1.7PCR反应及电泳
引物参照加拿大哥伦比亚大学公布的ISSR引物序列[8],由TaKaRa 有限公司(大连)合成。
PCR反应体系(20 μL):模板DNA 200 ng/μL,dNTPs 200 μmol/L,引物0.75 μmol/L,Taq DNA聚合酶0.5 U,Mg2+ 2.5 mmol/L,10×buffer 2 μL,用ddH2O补至总体积20 μL。
扩增程序为:94 ℃预变性2 min;94 ℃变性 30 s,45~55 ℃ 退火1 min(退火温度因不同引物而定),72 ℃延伸 2 min,38个循环;最后72 ℃延伸10 min,4 ℃保存。
PCR产物在2.0%琼脂糖凝胶上电泳,用GelDoc-It型凝胶成像系统照相。
1.8数据处理
电泳结果采取 0/1 赋值记带,将在琼脂糖凝胶上出现DNA片段的记为 1,不出现的记为 0,统计后输入电脑,用聚类分析软件NTsys进行聚类分析,并计算遗传相似度。
2结果与分析
2.1基因组DNA提取
16个滑菇菌株基因组DNA图谱见图1。
2.2引物筛选
从20 条ISSR引物中筛选出9条多态性丰富、谱带清晰且重复性好的引物,对16个供试菌株进行了ISSR-PCR扩增。9条引物共扩增出101条带(图2至图5),其中多态性条带为92条,多态位点百分率为91%。每条引物扩增出大约11条带,DNA片段大小为250~2 000 bp。
2.2基于ISSR分析结果构建树状图
16个菌株间的遗传相似系数变化范围在 0.59~0.90之间。当遗传相似系数为 0.69时,供试菌株被聚类成5个类群:第1类为早壮、N109;第2类为N103、 N108、延滑;第3类
为丹9、申14、C31、西羽、EH、曲新、D工羽3、HSH,其中丹9和申14亲缘关系最近,遗传相似系数达到0.9;第4类为112、辽滑1;第5类为华3,与其他菌株亲缘关系最远,遗传距离为0.59(图6)。
3结论与讨论
选择杂交亲本是食用菌杂交育种中最为重要的环节之一。一直以来,育种工作者都是通过表型分析的DUS(distinctness,uniformity and stability,简称DUS)测试来鉴定和选择亲本。这种方法耗时费力,鉴定结果也易受环境的干扰。随着分子生物学技术的发展,在分子水平上进行亲本菌株的选择势在必行。目前,关于滑菇分类鉴定多局限于传统分类法和同工酶分析研究[9],应用分子标记鉴定的研究报道较少。
本研究应用ISSR-PCR分子标记的方法对滑菇菌株遗传多样性进行了分析,从20条引物中筛选出9条多态性丰富、谱带清晰且重复性好的引物进行了ISSR-PCR扩增。结果显示9条引物均能将供试菌株区分开,表明ISSR分子标记对滑菇菌株遗传多样性分析是可行的。16个滑菇菌株遗传相似性较高,遗传背景比较一致,部分品种间亲缘关系较近,可能与来源同一地区有关。
参考文献:
[1]闫可,尹勇刚,傅常娥,等. 桦褐孔菌菌株遗传多样性的ISSR分析[J]. 食用菌学报,2012,19(2):26-30.
[2]王春晖,尹永刚,胡汝晓,等. 基于ISSR和RAPD标记的八株灰树花栽培菌株遗传多样性分析[J]. 食用菌学报,2013,20(4):1-5.
[3]秦莲花,宋春艳,谭琦,等. 用ITS和ISSR分子標记技术鉴别香菇生产用种[J]. 菌物学报,2006,25(1):94-100.
[4]王子迎,王书通. 安徽野生香菇遗传多样性及杂种优势的ISSR分析[J]. 菌物学报,2006,25(2):211-216.
[5]马志刚,吕作舟,郑和斌,等. ISSR标记在侧耳属菌株分类学中的初步应用[J]. 华中农业大学学报,2006,25(1):55-59.
[6]Zhang Y,Molina F I. Strain typing of Lentinula edodes by random amplified polymorphic DNA assay[J]. FEMS Microbiol Lett,1995,131(1):17-20.
[7]张丹,宋春艳,章炉军,等. 基于全基因组序列的香菇商业菌种SSR 遗传多样性分析及多位点指纹图谱构建的研究[J]. 食用菌学报,2014,21(2):1-8.
[8]王守现,刘宇,张英春,等. 六个灰树花菌株遗传多样性分析[J]. 北方园艺,2010(1):201-204.
[9]张敏,肖千明,李红,等. 滑菇主要栽培品种间亲缘关系的同工酶研究[J]. 辽宁农业科学,2010(4):25-27.冯涛,刘娟,华夏雪. 利用SSR、SRAP分子标记鉴定桃早熟芽变[J]. 江苏农业科学,2017,45(6):42-44.
doi:10.15889/j.issn.1002-1302.2017.06.009

