凝结芽孢杆菌N-乙酰-β-D-氨基葡萄糖苷酶基因的克隆鉴定及酶学性质
夏美娟,宦才辉,姜婷,郑兆娟,欧阳嘉,2*
(1.南京林业大学化学工程学院;2.南京林业大学林木遗传与生物技术省部共建教育部重点实验室,南京210037)
凝结芽孢杆菌N-乙酰-β-D-氨基葡萄糖苷酶基因的克隆鉴定及酶学性质
夏美娟1,宦才辉1,姜婷1,郑兆娟1,欧阳嘉1,2*
(1.南京林业大学化学工程学院;2.南京林业大学林木遗传与生物技术省部共建教育部重点实验室,南京210037)
N-乙酰-β-D-氨基葡萄糖苷酶(N-acetyl-β-D-glucosaminidase,NAGase)是微生物几丁质酶系中不可缺少的一部分,近年来由于其在几丁质降解过程中的特殊作用而受到关注。笔者从凝结芽孢杆菌NL01基因组上克隆获得了1个假定的N-乙酰-β-D-氨基葡萄糖苷酶基因,长度为1 761 bp,编码586个氨基酸,蛋白质理论分子量为64.5 kDa,编码氨基酸序列与已报道的N-乙酰-β-D-氨基葡萄糖苷酶相似性仅为38%。将该基因克隆至表达载体pETDuet-1上,并在大肠杆菌 BL21(DE3)中进行重组表达,结果显示重粗酶具有N-乙酰-β-D-氨基葡萄糖苷酶活力,经镍柱纯化获得纯酶的比酶活为74.3 U/mg。对重组酶酶学性质研究发现,该酶是一种新型耐高温N-乙酰-β-D-氨基葡萄糖苷酶。最适pH为5.5,最适温度为75℃,pH和热稳定性较好,在pH 3.5~7.5范围内比较稳定,在不高于65℃条件下热处理30 min后,酶活力可以保持在70%以上。
凝结芽孢杆菌;N-乙酰-β-D-氨基葡萄糖苷酶;表达纯化;酶学性质
N-乙酰-β-D-氨基葡萄糖苷酶(N-acetyl-β-D-glucosaminidase,NAGase)是几丁质酶系中不可缺少的一部分,主要负责催化水解β-1,4-糖苷键生成N-乙酰-β-D-氨基葡萄糖苷。它的底物包括壳二糖及其聚合物和衍生物[1],多发现于糖苷水解酶家族GH20[2]。N-乙酰-β-D-氨基葡萄糖苷酶近年来由于应用意义显著,被广泛研究。已有研究表明,昆虫和甲壳动物体内的N-乙酰-β-D-氨基葡萄糖苷酶参与降解食物和周期性蜕壳[3];真菌中的N-乙酰-β-D-氨基葡萄糖苷酶与真菌细胞分裂和形态的形成相关[4];植物中的N-乙酰-β-D-氨基葡萄糖苷酶有抑菌防御作用[5];细菌中的N-乙酰-β-D-氨基葡萄糖苷酶可以降解几丁质作为碳源和能量来源[6]。目前,N-乙酰-β-D-氨基葡萄糖苷酶研究多集中于动物来源,在植物中玉米籽[7]真菌的粗糙脉孢菌[8]和里氏木霉[1]中也有发现,但来源于细菌的较少,主要存在于枯草芽孢杆菌[9]和乳酸乳球菌[10]。其中,鲤鱼血液来源的N-乙酰-β-D-氨基葡萄糖苷酶最适pH为6.5,在pH 7.0~11.0的范围内稳定性较好,是一种偏碱性的酶[11];南美白对虾来源的N-乙酰-β-D-氨基葡萄糖苷酶最适pH为5.2,在pH 4.2~10.0的范围内稳定性较好,在20~40℃酶活力稳定,是一种常温的酶[12]。
笔者对实验室保存的1株凝结芽孢杆菌NL01测序过程发现其上存在假定的N-乙酰-β-D-氨基葡萄糖苷酶基因,鉴于细菌来源的N-乙酰-β-D-氨基葡萄糖苷酶研究较少,本研究通过PCR技术扩增获得该假定的N-乙酰-β-D-氨基葡萄糖苷酶基因,将其构建到表达载体pETDuet-1上,在大肠杆菌表达宿主BL21(DE3)中进行诱导表达,并对重组蛋白的酶学性质进行分析,为进一步研究其对凝结芽孢杆菌的生长影响提供理论依据。
1 材料与方法
1.1 供试材料
目的基因来源菌BacilluscoagulansNL01、克隆宿主菌EscherichiacoliTrans1-T1、表达宿主菌EscherichiacoliBL21(DE3)由本实验室保存。基因克隆载体pEASY-Blunt Simple(氨苄青霉素抗性)、Fast Pfu DNA聚合酶和购自北京全式金生物技术有限公司;基因表达载体pETDuet-1(氨苄青霉素抗性)购自Novagen公司;限制性内切酶EcoR I和XhoI购自Takara公司;T4DNA连接酶购自New England Biolabs(北京)公司;DNA胶回收试剂盒购自Promega公司;质粒提取试剂盒和细菌基因组提取试剂盒购自上海捷瑞生物工程有限公司;Tryptone、Yeast Extract购自Oxoid公司; 4-硝基苯-β-D-葡萄糖苷(4-nitrophenyl-β-D-glucoside,pNPG),4-硝基苯-N-乙酰-β-D-氨基葡萄糖苷(4-nitrophenyl-N-acetyl-β-D-glucosaminide,pNPNAc),4-硝基苯酚(4-nitrophenol,pNP)均购自Sigma公司;镍柱购自GE Healthcare公司;其他生化试剂为国产分析纯产品。
1.2 引物设计
参考已完成全基因组测序的凝结芽孢杆菌NL01(GenBank登录号:NZ_LBMQ01000001.1)的全基因组DNA序列设计上下游引物,由北京六合华大基因科技股份有限公司合成。
上游引物ghf3.f:5′-GCGGAATTCGATGAA-AAGATCCGGAT-3′(EcoR I)和下游引物ghf3.r:5′-ACTCTCGAGTTAGTAATGAAGACCGTG-3′(XhoI)。
1.3 培养基及培养条件
用于培养凝结芽孢杆菌NL01的液体培养基及培养条件:葡萄糖20 g/L、Yeast Extract 10 g/L,在50℃、100 r/min条件下振荡培养。
用于培养大肠杆菌的LB培养基及培养条件:Tryptone 10 g/L、氯化钠10 g/L、Yeast Extract 5 g/L,在37℃、200 r/min条件下振荡培养。固体培养基为液体培养基中加入终浓度为15 g/L的琼脂粉。
1.4 目的基因的扩增及测序
提取凝结芽孢杆菌NL01的全基因组作为PCR反应的模板,以ghf3.f和ghf3.r为引物PCR扩增目的基因,PCR产物经1%琼脂糖凝胶电泳验证后,连接到pEASY-Blunt Simple载体上,连接产物转化至E.coliTrans1-T1,在含有氨苄青霉素抗性平板上挑选阳性转化子试管培养并提取重组质粒。将重组质粒命名为Blunt Simple-ghf3,经双酶切验证后送北京六合华大基因科技股份有限公司测序。
1.5 重组N-乙酰-β-D-氨基葡萄糖苷酶的表达与纯化
利用限制性内切酶EcoR I和XhoI同时双酶切重组质粒Blunt Simple-ghf3和pETDuet-1,胶回收相应片段后用T4DNA连接酶连接后转化E.coliBL21(DE3),在含氨苄青霉素抗性平板上挑选阳性转化子试管培养并提取重组质粒进行双酶切验证,将验证正确的重组菌株命名为E.coliBL21(pETDuet-ghf3)。培养重组菌株至D(600)达到0.8时加入终浓度为1 mmol/L的IPTG,25℃条件下诱导6 h,收集菌体,用超声波破碎菌体后离心,所得上清即为粗酶液。将粗酶液通过Ni2+亲和柱纯化,用不同梯度的洗脱缓冲液洗脱,收集目的蛋白组分,进行SDS-PAGE鉴定。
1.6 测定方法
N-乙酰-β-D-氨基葡萄糖苷酶酶活测定方法:反应体系总体积为1 mL,分别加入600 μL 0.1 mol/L柠檬酸和0.2 mol/L磷酸氢二钠配成的缓冲溶液(pH 5.5),300 μL 15 mmol/L的pNPNAc溶液以及100 μL适当稀释的酶液,混合均匀后在75℃条件下水浴10 min后,立即加入2 mL 1 mol/L的Na2CO3溶液终止反应。在400 nm波长处测定吸光值,根据pNP标准曲线确定pNP的生成量,1个酶活力单位定义为1 min内催化水解生成1 μmolpNP所需要的酶量[13]。
Bradford法测定蛋白浓度:取要测定的样品0.1 mL,加入3 mL Bradford,混合均匀,13 min后,以水溶液为空白对照,595 nm处测定其吸光度。将测得的吸光值代入标准曲线中得到样品的蛋白浓度。
1.7 酶学性质分析
为了测定N-乙酰-β-D-氨基葡萄糖苷酶的最适pH,在pH 3.0~8.0的范围内测定其酶活,所用缓冲液为柠檬酸-磷酸氢二钠缓冲液。为了测定N-乙酰-β-D-氨基葡萄糖苷酶的最适温度,在45~95℃的范围内测定其酶活。为了测定酶的pH稳定性,将酶液置于不同pH的缓冲液中,4℃下放置24 h后测定残留酶活。为了测定酶的温度稳定性,将酶液置于不同温度下孵育30 min,测定残留酶活力。
2 结果与分析
2.1 N-乙酰-β-D-氨基葡萄糖苷酶基因的克隆和测序
以凝结芽孢杆菌NL01的基因组为模板,PCR扩增得到假定的N-乙酰-β-D-氨基葡萄糖苷酶基因。将得到的PCR产物连接pEASY-Blunt Simple载体后转化至E.coliTrans1-T1中,从抗性筛选平板上选取转化子,试管培养并提取重组质粒Blunt Simple-ghf3,双酶切验证,结果如图1所示,重组质粒双酶切产物在3.9和1.7 kb附近出现条带,与预期一致,说明转化子含有正确的重组质粒,提取质粒送样测序。
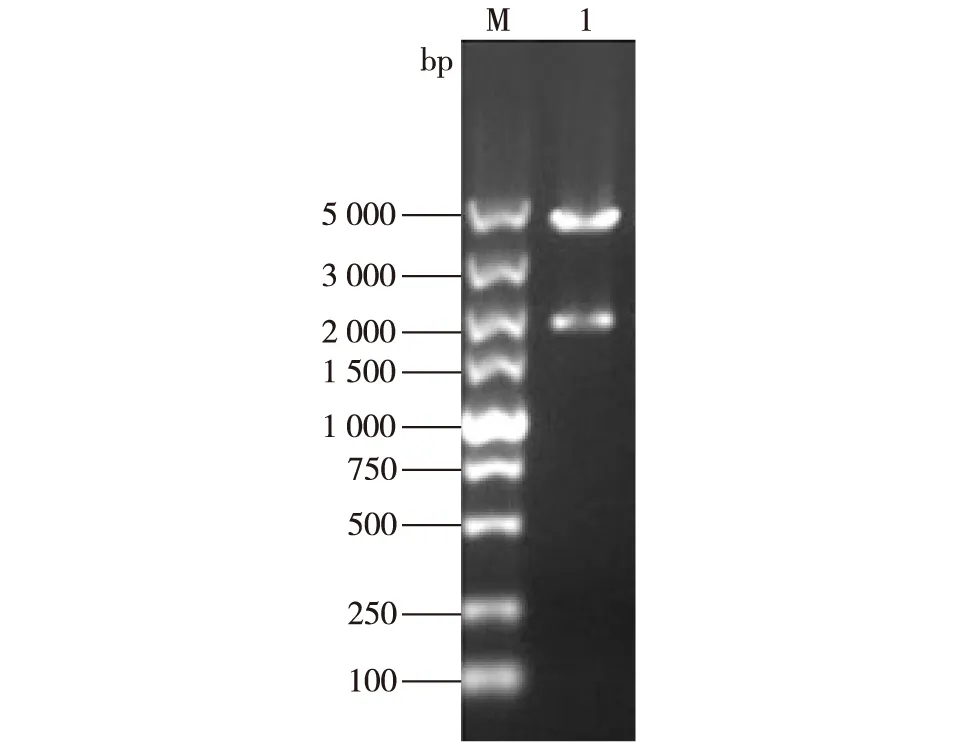
注:泳道1为来自转化子的重组质粒。图1 双酶切后的重组质粒Blunt-ghf3Fig. 1 Recombinant plasmid Blunt-ghf3 double digested by EcoR I and Xho I
2.2 N-乙酰-β-D-氨基葡萄糖苷酶序列分析
测序结果显示,来源于凝结芽孢杆菌NL01的假定N-乙酰-β-D-氨基葡萄糖苷酶基因序列全长1 761 bp,编码586个氨基酸。根据Henrissat建立的糖苷水解酶家族分类方法,属于GH-3家族,目前已经鉴定的N-乙酰-β-D-氨基葡萄糖苷酶大多属于GH-20家族[2],尚未见报道属于GH-3家族。将不同来源的N-乙酰-β-D-氨基葡萄糖苷酶氨基酸序列进行同源性比对,结果如图2所示,该基因编码蛋白与已报道的真核生物来源的N-乙酰-β-D-葡萄糖苷酶,如来源于Litopenaeusvannamei(GenBank登录号为ACR23316.1)[11]、Triboliumcastaneum(GenBank登录号为ABQ95982.1)[14]和TrichodermareeseiQM6a/Rutc30(GenBank登录号为EGR50812.1)[1]的相似性低于40%;与细菌来源的N-乙酰-β-D-氨基葡萄糖苷酶,如来源于Bacillusanthracis(GenBank登录号为AAT55709.1)[15]、Bacillussubtilis168(GenBank登录号为AAA67857.1)[7]的相似性低于20%。而与NCBI数据库里公布的凝结芽孢杆菌HM-08和S-lac基因组上注释的假定的N-乙酰-β-D-葡萄糖苷酶同源相似性达到95%。比对数据表明,本研究中凝结芽孢杆菌NL01来源的N-乙酰-β-D-葡萄糖苷酶可能是一种新型的N-乙酰-β-D-葡萄糖苷酶,该酶在不同凝结芽孢杆菌菌株中普遍存在。

注:1.凝结芽孢杆菌NL01. Bacillus coagulans NL01;2.南美白对虾 Litopenaeus vannamei (ACR23316.1);3.拟谷盗属 Tribolium castaneum (ABQ95982.1);4.里氏木霉 Trichoderma reesei (EGR50812.1)。图2 假定编码的氨基酸序列与其他N-乙酰-β-D-氨基葡萄糖苷酶序列同源性分析(催化残基以△标记)Fig. 2 Homology analysis of deduced amino acid sequences from Bacillus coagulans NL01 and the other N-acetyl-β-D-glucosaminidase
2.3 重组质粒pETDuet-ghf3的构建和鉴定
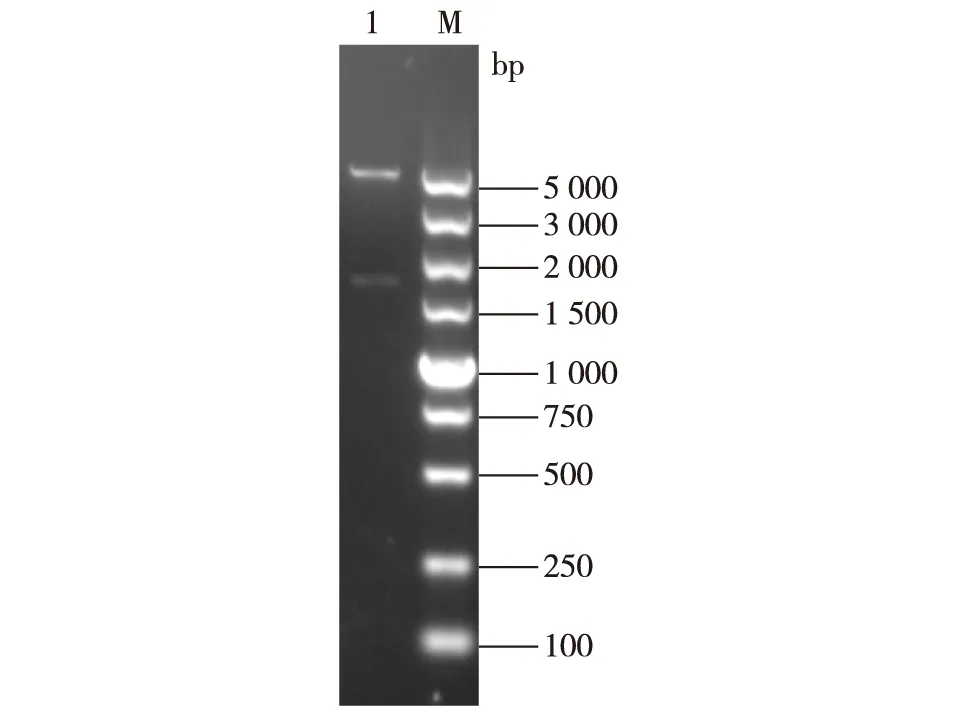
注:泳道1为来自转化子的重组质粒。图3 双酶切后的重组质粒pETDuet-ghf3Fig. 3 Recombinant plasmid pETDuet-ghf3 double digested by EcoR I and Xho I
用EcoR I和XhoI双酶切pETDuet-1和Blunt Simple-ghf3,胶回收相应片段后连接并转化入E.coliBL21(DE3)中,从抗生素平板上选取转化子,试管培养并提取重组质粒pETDuet-ghf3,进行双酶切验证,结果如图3所示,重组质粒双酶切产物在5.4和1.7 kb附近出现条带,说明转化子含有正确质粒。
2.4 重组N-乙酰-β-D-氨基葡萄糖苷酶的诱导表达与纯化

注:泳道M为标准蛋白分子量,kDa;泳道1为重组大肠杆菌BL21(pETDuet-ghf3)诱导后的破碎液;泳道2~5分别为以0%,20%,60%和100%洗脱液洗脱的重组蛋白。图4 重组蛋白的SDS-PAGE电泳Fig. 4 SDS-PAGE electrophoresis of recombinant protein
将重组菌E.coliBL21(pETDuet-ghf3) 诱导表达后进行超声波破碎,利用镍柱对重组蛋白进行纯化,结果如图4所示,目的蛋白分子质量约为66.0 kDa。分别以pNPG和pNPNAc为底物,利用1.6测定方法检测重组酶酶活力,结果发现,以pNPG为底物没有酶活,而以pNPNAc为底物测得粗酶液比酶活5.0 U/mg,经镍柱纯化获得的纯酶的比酶活74.3 U/mg,纯化倍数为14.9倍。
2.5 重组N-乙酰-β-D-氨基葡萄糖苷酶酶学性质分析
以pNPNAc为底物来确定重组N-乙酰-β-D-氨基葡萄糖苷酶的最适pH。在pH 5.5条件下测得最高的酶活力,并且在pH 4.5~6.5范围内可以得到的酶活力在60%以上。将不同pH的缓冲液与酶液等量混合后,4℃条件下静置24 h,将处理过的酶液取出适量在pH 5.5、75℃条件下检测残余酶活力,残余酶活力以初始酶活力的相对百分数表示。结果显示,在pH 3.5~7.5范围内,重组酶的稳定性非常好,残余酶活力在80%以上(图5a)。
同时,本研究还探讨了温度对酶活力的影响。将酶液在40~75℃温度范围内热处理30 min后迅速冷却至室温,然后取出适量酶液在pH 5.5、75℃条件下检测酶的残余酶活力,残余酶活力以初始酶活力的相对百分数表示。试验结果显示,酶在65℃以下处理30 min,酶活力基本保持在70%以上,在温度为75℃的条件下测得最高的酶活力。(图5b)。
此外,本研究还探讨了不同金属离子对酶活力的影响。在酶活测定体系中分别添加终浓度为2 mmol/L的Mg2+、Ca2+、Co2+、Mn2+、Cu2+、Ni2+和Fe2+。在最适温度75℃和最适pH 5.5的条件下测定,同时设置空白对照。结果显示,不同的金属离子对酶活力影响有差异,其中,Mn2+和Fe2+对酶有轻微的促进作用,Cu2+和Ca2+有轻微的抑制作用,Mg2+、Co2+和Ni2+对酶活力影响不大(图5c)。
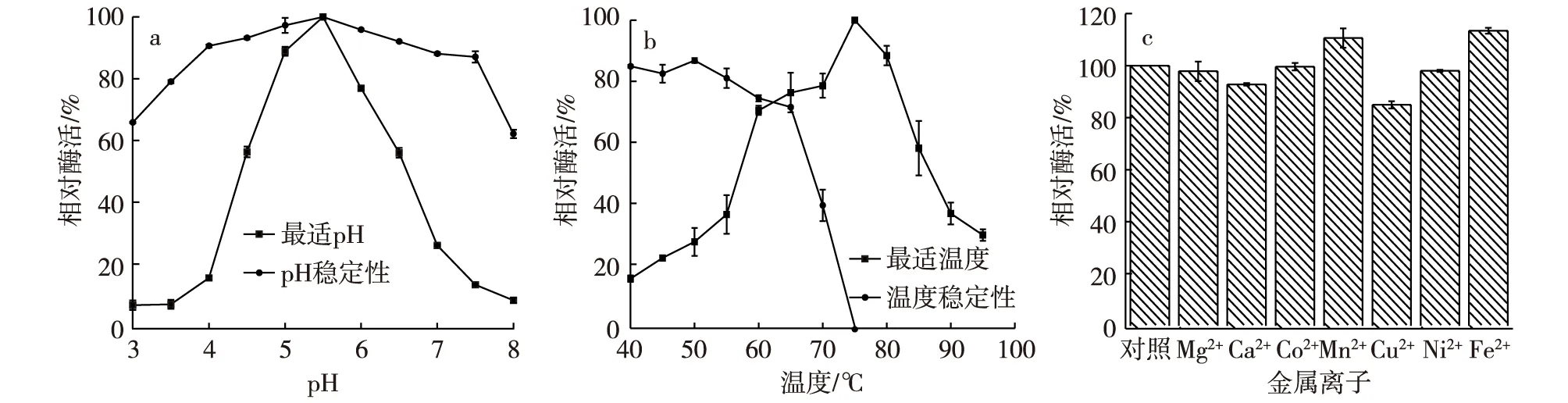
图5 重组N-乙酰-β-D-葡萄糖苷酶的酶学分析Fig. 5 The enzymological characteristics of N-acetyl-β-D-glucosaminidase
在报道的细菌来源的N-乙酰-β-D-氨基葡萄糖苷酶当中,来源于乳酸乳球菌IL1403的N-乙酰-β-D-氨基葡萄糖苷酶的最适pH为5.5,酸碱稳定区域pH为5.5~8.0,酶在pH低于5.5的条件下不稳定;最适温度为37℃,酶在50℃条件下热处理30 min后的酶活性完全丧失[10]。来源于芽孢杆菌NCIM 5120的N-乙酰-β-D-氨基葡萄糖苷酶的最适pH为6.0,在pH 5.5~10.0范围内稳定性较好,最适温度为70℃[16]。本研究中来源于凝结芽孢杆菌的N-乙酰-β-D-氨基葡萄糖苷酶最适pH为5.5,在pH 3.5~7.5范围内稳定性较好,与上述来源于芽孢杆菌NCIM 5120的N-乙酰-β-D-氨基葡萄糖苷酶相比耐酸性更好;本研究中N-乙酰-β-D-氨基葡萄糖苷酶最适温度为75℃,酶在65℃以下热处理30 min后的稳定性较好,与上述来源于乳酸乳球菌IL1403的N-乙酰-β-D-氨基葡萄糖苷酶相比耐热性更高。
3 结 论
1)鉴定了来自于凝结芽孢杆菌NL01的一种新型N-乙酰-β-D-氨基葡萄糖苷酶基因,该基因全长1 761 bp,其编码的N-乙酰-β-D-氨基葡萄糖苷酶属于糖苷水解酶GH-3家族,与已报道的其他微生物来源的N-乙酰-β-D-氨基葡萄糖苷酶相似性极低。
2)以大肠杆菌为表达宿主菌,将N-乙酰-β-D-氨基葡萄糖苷酶基因连接到表达载体pETDuet-1上,构建重组菌株后进行异源表达,并利用镍柱纯化获得重组蛋白,结果显示重组酶是一种新型耐高温N-乙酰-β-D-氨基葡萄糖苷酶,纯酶比酶活为74.3 U/mg;该酶在65℃以下热处理30 min后稳定性较好,在pH 3.5~7.5范围内稳定性较好。
[1]CHEN F, CHEN X Z, QIN L N, et al. Characterization and homologous overexpression of an N-acetylglucosaminidase Nag1 fromTrichodermareesei[J]. Biochemical and Biophysical Research Communications, 2015, 459(2):184-188.
[2]TZELEPIS G D, MELIN P, JENSEN D F, et al. Functional analysis of glycoside hydrolase family 18 and 20 genes inNeurosporacrassa[J]. Fungal Genetics and Biology, 2012, 49(9):717-730.
[3]KEYHANI N O, WANG L X, LEE Y C, et al. The chitin catabolic cascade in the marine bacteriumVibriofurnissii[J]. The Journal of Biological Chemistry, 1996, 271(52):33409-33413.
[4]PERA L M, BAIGORM D, CALLIERI D. Influence of environmental conditions on hyphal morphology in pellets ofAspergillusniger:role ofβ-N-acetyl-D-glucosaminidase[J]. Current Microbiology, 1999, 39(2):65-67.
[5]GRAHAM L S, STICKLEN M B. Plant chitinases[J]. Canadian Journal of Botany, 1994, 72(8):1057-1083.
[6]LI D C. Review of fungal chitinases[J]. Mycopathologia, 2006, 161(6):345-360.
[7]OIKAWA A, ITOH E, ISHIHARA A, et al. Purification and characterization ofβ-N-acetylhexosaminidase from maize seedlings[J]. Journal of Plant Physiology, 2003, 160(9):991-999.
[8]TZELEPIS G D, MELIN P, JENSEN D F, et al. Functional analysis of glycoside hydrolase family 18 and 20 genes in Neurospora crassa[J]. Fungal Genetics and Biology, 2012, 49(9):717-730.
[9]MARGOT P, MAUЁL C, KARAMATA D. The gene of theN-acetylglucosaminidase, aBacillussubtilis168 cell wall hydrolase not involved in vegetative cell autolysis[J]. Molecular Microbiology, 1994, 12(4):535-545.
[11]UENO R, YUAN C S. Purification and properties of neutralβ-N-acetylglucosaminidase from carp blood [J]. Biochimica et Biophysica Acta, 1991, 1074:79-84.
[12]XIE X L, CHEN Q X, LIN J C, et al. Purification and some properties ofβ-N-acetyl-D-glucosaminidase from prawn (Penaeusvannamei)[J]. Marine Biology, 2004, 146(1):143-148.
[13]姚卫蓉, 丁霄霖. pNPG法测定纤维素酶系中β-葡萄糖苷酶 [J]. 微生物学通报, 1998, 25(3):182-183. YAO W R, DING X L. Determination ofβ-glucosidases from cellulose system by pNPG [J]. Journal of Microbiology, 1998, 25(3):182-183.
[14]HOGENKAMP D G, ARAKANE Y, KRAMER K J, et al. Characterization and expression of theβ-N-acetylhexosaminidase gene family ofTriboliumcastaneum[J]. Insect Biochemistry and Molecular Biology, 2008, 38(4):478-489.
[15]CHEN Y, FUKUOKA S, MAKINO S. A novel spore peptidoglycan hydrolase ofBacilluscereus:biochemical characterization and nucleotide sequence of the corresponding gene,sleL[J]. Journal of Bacteriology, 2000, 182(6):1499-1506.
[16]AMUTHA B, KHIRE J M, KHAN M I. Characterization of a novel exo-N-acetyl-β-D-glucosaminidase from the thermotolerantBacillussp. NCIM 5120[J]. Biochimica et Biophysica Acta (BBA)-General Subjects, 1998, 1425(2):300-310.
Molecular cloning and enzymological characteristics of a novelN-acetyl-β-D-glucosaminidase fromBacilluscoagulans
XIA Meijuan1, HUAN Caihui1, JIANG Ting1, ZHENG Zhaojuan1, OUYANG Jia1,2*
(1. College of Chemical Engineering, Nanjing Forestry University;2. Key Laboratory of Forest Genetics and Biotechnology of Ministry of Education, Nanjing Forestry University, Nanjing 210037, China)
Chitin, an unbranched homopolymer of 1,4-β-linked N-acetyl-D-glucosamine (GlcNAc), is widely distributed and believed to be the second most abundant and renewable source in nature, next to cellulose. Chitinases, chitin-degrading enzymes are mainly composed of chitobiosidase, N-acetyl-β-D-glucosaminidase,β-D-glucosaminidase and chitosanase. As an indispensable member of the chitinolytic system and involved in chitin degradation, N-acetyl-β-D-glucosaminidase (NAGase) catalyzes the hydrolysis ofβ(1-4) linked N-acetylglucosaminyl residues from glycol-conjugates at the non-reducing end, producing different lengths of GlcNAc. The NAGase is widely distributed among most types of living organisms and has numerous biological functions. Bacterial NAGase has a crucial physiological role in cell wall recycling, even in altering the function of the host cell. In recent years, NAGase has been extensively studied due to its significance in many fields. In this study, theBacilluscoagulansgenome was used as the template to amplify the putative N-acetyl-β-D-glucosaminidase gene, whose length is 1 761 bp. It encodes 586 amino acids and the theoretical molecular weight mass is 64.5 kDa. The results of amino acid sequence analysis showed that the enzyme had a very low similarity with the reported N-acetyl-β-D-glucosaminidase, only 38%. The gene was cloned into pETDuet-1 and expressed inEscherichiacoliBL21(DE3). The crude enzyme had N-acetyl-β-D-glucosaminidase activity and the purified specific activity was 74.3 U/mg after Ni-NTA purification. The optimal pH and temperature is 5.5 and 75℃, respectively. Enzymological characteristics showed that the enzyme was stable in a range of pH from 3.5 to 7.5. The enzyme activity remained more than 70% after incubation for 30 min below 65℃. A low-level inhibition of N-acetyl-β-D-glucosaminidase (15.1%) was observed with Cu2+(2 mmol/L), and a low-level promotion of N-acetyl-β-D-glucosaminidase (10.7%) and (13.5%) was observed with Mn2+and Fe2+(2 mmol/L), resepectively. These results indicated that the N-acetyl-β-D-glucosaminidase is a new type of N-acetyl-β-D-glucosaminidase with thermo stability.
Bacilluscoagulans; N-acetyl-β-D-glucosaminidase; cloning and expression; enzymological characteristics
2016-06-27
2016-09-30
国家自然科学基金(51561145015,31300487);江苏省自然科学基金(BK20130970)。
夏美娟,女,研究方向为生物化工。通信作者:欧阳嘉,女,教授。E-mail:hgouyj@njfu.edu.cn
Q812
A
2096-1359(2017)02-0070-06

