梭梭14-3-3蛋白基因 HaFT-1和 HaFT-2克隆及表达分析
李亚婕,张 桦,蒋圆圆,麻 浩,姚正培,任燕萍,王 泽,马 林
(1.新疆农业大学 农学院,乌鲁木齐 830052;2.哈密职业技术学院,新疆哈密 839000; 3.南京农业大学 农学院,南京 210095;4.新疆农业大学 干旱区荒漠研究所,乌鲁木齐 830052)
梭梭14-3-3蛋白基因 HaFT-1和 HaFT-2克隆及表达分析
李亚婕1,张 桦1,蒋圆圆2,麻 浩3,4,姚正培1,任燕萍1,王 泽4,马 林1
(1.新疆农业大学 农学院,乌鲁木齐 830052;2.哈密职业技术学院,新疆哈密 839000; 3.南京农业大学 农学院,南京 210095;4.新疆农业大学 干旱区荒漠研究所,乌鲁木齐 830052)
根据前期梭梭干旱转录组测序得到的Unigene序列,成功克隆2个梭梭14-3-3蛋白基因,分别命名为 HaFT-1和 HaFT-2,并对其进行基因表达分析。结果表明: HaFT-1基因在梭梭的根、种子中均可表达,且根中表达最强; HaFT-2基因在梭梭同化枝中表达最强,根的表达量次之。干旱胁迫后 HaFT-1和 HaFT-2基因的表达量均下调。本研究初步分析 HaFT-1、 HaFT-2基因的表达模式,为进一步研究该基因功能奠定基础。
梭梭;14-3-3蛋白;基因克隆;表达
梭梭(Haloxylonammodendron)为梭梭属(Haloxylon Bunge),藜科(Chenopodiaceae)超旱生小乔木,呈高大灌丛状[1]。其主要生长在中国的西北部,是重要的固沙植物,在沙地、固定和半固定沙丘,荒漠上广泛分布[2]。梭梭也是中国荒漠半荒漠区固沙造林面积最大的建群树种。具有耐旱、耐高温、耐盐碱、耐风蚀等优良品性[3]。在防沙固沙、减缓荒漠化、维护生态安全方面有着不可或缺的作用。
14-3-3蛋白是一类高度保守且功能多样的调节蛋白,其在真核生物中普遍存在,分子量为28~33 ku[4]。该蛋白是由Moore等[5]1967年首次从牛脑组织中发现的一种酸性、可溶异源二聚体蛋白,根据该蛋白层析后分离组分的片段数和在淀粉凝胶电泳中的迁移率,命名为14-3-3蛋白。目前,已有20多种植物的14-3-3蛋白基因被克隆和鉴定,在番茄中已有12个14-3-3蛋白基因被发现,分别命名为 TFT1-TFT12[6]。余梅等[7]在茶树中发现14-3-3蛋白基因,且该基因在花蕾发育晚期起到调控作用。赵秀玲等[8]通过RT-PCR技术,首次从菠菜中发现并克隆14-3-3蛋白基因,且命名为 So14-3-3,对该基因半定量,结果发现硝酸盐处理后, So14-3-3基因在根和叶的表达增强。研究表明,14-3-3蛋白可调控基因的表达、调节转录因子的核定位和参与多种酶活性的调控,并且在细胞信号传导、细胞周期调节、细胞凋亡、胁迫应答等多种细胞活动中起到重要作用[9]。不同的14-3-3蛋白具有不同的组织特异性,调控植物的发育、应答不同外界环境的刺激[10]。14-3-3蛋白参与的各种代谢调控、物质运输和植物激素转导等调控,是通过与其他蛋白相互作用而进行的[11]。因此,研究14-3-3蛋白在植物中的功能和调控机理对理解植物响应外界信号的分子机制十分重要。但有关梭梭14-3-3蛋白基因克隆和功能特征鲜见报道,本研究采用RT-PCR方法从梭梭的同化枝中提取并克隆14-3-3蛋白基因 HaFT-1和 HaFT-2,并且对这2个基因在梭梭的不同部位的表达以及在干旱胁迫下梭梭同化枝的表达进行分析,为研究14-3-3蛋白在梭梭抗逆分子机制中的作用奠定基础。
1 材料与方法
1.1 材 料
1.1.1 试验材料 以新疆吐鲁番荒漠植物园采集的梭梭种子为试验材料。种子用蒸馏水浸泡5 min用φ=75%酒精消毒30 s后转入蒸馏水中冲洗3遍,待酒精洗净后用次氯酸钠溶液浸泡10 min,再用蒸馏水冲洗4遍,接种于MS培养基中待发芽后培养至3周左右,取其同化枝,提取RNA用于基因克隆。
1.1.2 试剂 总RNA提取试剂盒(Trizol法)、反转录试剂盒、琼脂糖凝胶DNA回收试剂盒、DNA marker、dNTPs、Buffer(Mg2+)、Taq酶分别购自Life Technologies Corporation、北京全式金生物技术有限公司、北京天根生化科技有限公司和TakaRa公司。氨苄青霉素(Amp+)和卡那霉素(Kan+)由植物分子生物学研究室提供。其他试剂为国产分析纯。
1.2 方 法
1.2.1 HaFT-1和 HaFT-2的克隆 RNA的提取和反转录操作方法参照Trizol说明书和First Srtand cDNA Synthesis kit反转录试剂盒相关内容。
根据前期梭梭干旱转录组测序结果拼接得到的18个类似14-3-3蛋白基因相关序列,挑选2个拼接出全部读码框的Unigene,并进行同源序列比对有较高相似性,推测可能属于14-3-3蛋白。根据这2个序列设计2对特异性引物(见表1) HaFT-1基因引物为编号1和2, HaFT-2基因引物为编号3和4。以梭梭cDNA为模版,进行PCR反应。其反应体系为Buffer(Mg2+) 2.5 μL、dNTPs 1.0 μL、上下游引物各1.0 μL、cDNA模板1.0 μL、Taq酶0.5 μL、最后加入ddH2O至总体积25 μL,扩增程序为94 ℃ 预扩增5 min;94 ℃ 30 s,52 ℃ 30 s,72 ℃ 1 min, 35个循环;72 ℃延伸10 min,4 ℃保存。反应结束后,取扩增产物在10 g/L琼脂糖凝胶上电泳检测PCR产物。将回收的PCR产物与Peasy-T1载体(全式金公司)连接,转入DH5α中,验证其中阳性克隆,送上海生工测序。

表1 PCR引物序列Table 1 Primer sequence of PCR
1.2.2 HaFT-1和 HaFT-2的序列分析 分别利用Protparam在线软件、ProtScale的Hphob./Kyte &Doolittle算法、TMHMM Server v.2.0软件及SOPMA在线软件,对 HaFT-1、 HaFT-2蛋白的一级结构、氨基酸组成、分子量等理化性质,编码的氨基酸序列疏水性/亲水性,蛋白的跨膜结构,蛋白的二级结构进行预测分析。通过NCBI网站Blast对氨基酸序列的同源性进行搜索,并进行同源序列比对, MEGA 4.0软件对其进行系统进化树构建。
1.2.3 HaFT-1和 HaFT-2在梭梭不同组织中的表达分析 取野生梭梭的根、同化枝、花和种子,每个组织取3个平行样,于液氮中速冻,于-80 ℃保存。提取野生梭梭的根、同化枝、花和种子的RNA,反转录,以18S rRNA (GenBank登录号:AJ577394)为内参基因,进行RT-PCR反应。 HaFT-1和 HaFT-2半定量RT-PCR反应引物见表1中的5、6、7和8编号。其反应体系为94 ℃ 5 min,94 ℃ 30 s,60 ℃ 30 s,72 ℃ 45 s,72 ℃ 8 min,28个循环。
1.2.3 HaFT-1和 HaFT-2干旱胁迫后表达分析 实时荧光定量PCR试验采用荧光染料 SYBR Green I[Roche FastStart Univorsal SYBR Green Master(ROX)],选取18S rRNA基因作为内参基因,检测 HaFT-1和 HaFT-2这2个14-3-3蛋白基因在干旱胁迫下的转录表达水平。以正常生长的1 a生梭梭同化枝(土壤含水量20%)作为对照,以土壤含水量在15%、12%、9%、6%及3%时的梭梭同化枝为干旱胁迫样品,比较编码 HaFT-1和 HaFT-2这2个14-3-3蛋白基因的表达差异。根据目的基因和内参基因的CT值,并采用Livak等的2-△△CT法[12],对 HaFT-1和 HaFT-2这2个14-3-3蛋白基因进行相对定量分析。 HaFT-1和 HaFT-2实时荧光定量反应引物见表1中的11、12、13和14编号 。反应条件为95 ℃ 10 s,95 ℃ 5 s,60 ℃ 10 s,72 ℃ 20 s,循环45次。生成熔解曲线步骤为95 ℃ 0 s,20 ℃/s;65 ℃ 15 s,20 ℃/s;95 ℃ 0 s,0.1 ℃/s,40 ℃保存。每次试验的每个反应样品均设置3个技术性重复,按上述反应程序在ABI 7500型实时荧光定量PCR仪上进行试验。
2 结果与分析
2.1 HaFT-1和 HaFT-2基因的RT-PCR反应及目的基因获得
以梭梭cDNA为模板进行PCR反应,得到与目的片段大小相符的特异性条带如图1中1、2泳道所示,分别为 HaFT-1、 HaFT-2片段大小为856 bp和1 003 bp。送上海生工测序后,获得 HaFT-1及 HaFT-2基因序列。GenBank登录号分别是KU565486和KU565487。
2.2 梭梭 HaFT-1和 HaFT-2基因生物信息学分析
2.2.1 序列特征分析 通过NCBI的ORF finder分析表明, HaFT-1和 HaFT-2 基因的ORF为795 bp和792 bp,编码氨基酸个数分别为264和263,通过Blastp分析得知该基因编码的蛋白质均属于14-3-3家族(如图2)。

M.DL 2 000 Marker;1. HaFT-1PCR扩增产物 HaFT-1 PCR amplification products;2. HaFT-2 PCR扩增产物 HaFT-2 PCR amplification products
通过Protparam、ProtScale、TMHMM Server v.2.0软件分别对 HaFT-1和 HaFT-2基因编码的蛋白质序列进行预测,结果表明该蛋白的分子量分别为29 985.5、29 762.3,等电点为4.79、4.84,分子式分别为C1310H2068N358O429S9、C1304H2067N353O426S8。不稳定系数为41.89、51.43,均为不稳定蛋白。应用SignalP 3.0对 HaFT-1和 HaFT-2基因编码的蛋白序列进行信号肽预测,未发现信号肽。
2.2.2 梭梭 HaFT-1和 HaFT-2基因系统进化分析 将 HaFT-1和 HaFT-2基因序列在 NCBI 数据库中进行在线分析和 BLAST 比对,通过与NCBI数据库其他物种 14-3-3蛋白基因的编码区全序列同源性比较发现,梭梭 HaFT-1与甜菜(Betavulgaris)有较高的同源性(92%),其次与蚕豆(Viciafaba)的同源性达86%,与陆地棉(Gossypiumhirsutum)、菜豆(Phaseolusvulgaris)、大戟属(Euphorbia)的氨基酸的同源性分别达85%、84%、82%。 HaFT-2与冰菜(Mesembryanthemumcrystallinum)和甜菜(Betavulgaris)有较高的同源性,达92%。其次是木薯(Manihotesculentacrantz),同源性达88%,与虎眼万年青(Ornithogalumcaudatum)、大豆(Glycinemax)、鹰嘴豆(Cicerarietinum)、橡胶树(Heveabrasiliensis)同源性分别达87%、84%、84%、83%。

A.基因 HaFT-1的保守结构域 Conserved domains of HaFT-1 gene;B.基因 HaFT-2的保守结构域 Conserved domains of HaFT-2 gene
采用DNAMAN软件对甜菜(Betavulgaris)、陆地棉(Gossypiumhirsutum)、大戟属(Euphorbia)、毛果杨(Populustrichocarpa)、木薯(Manihotesculentacrantz)、冰菜(Mesembryanthemumcrystallinum)、虎眼万年青(Ornithogalumcaudatum)的14-3-3蛋白与梭梭的14-3-3蛋白进行多重序列比对结果见图3。 HaFT-1 和 HaFT-2基因氨基酸序列与其他物种14-3-3蛋白序列在5个区域高度保守(标红线区域)。除保守区域外,氨基末端和羧基末端有明显差异,相似结构证明14-3-3蛋白在功能上具有保守性,但是局部的差异结构表明可能存在功能上的差异。
使用MEGA 5.0中邻接法构建系统进化树,进化树中物种及其登录号见表2,由图4可知,植物14-3-3蛋白共聚为两大类,其中, HaFT-1与甜菜(XP_010689898)聚为一类,说明 HaFT-1与甜菜的亲缘关系较近。 HaFT-2编码的氨基酸序列与甜菜(XP_010692757)和冰菜(P93259)聚为一类。
2.3 HaFT-1和 HaFT-2组织特异性分析
用野生梭梭的根、同化枝、花和种子的cDNA为模板,进行RT-PCR半定量。结果显示, HaFT-1和 HaFT-2在不同组织中的表达水平不相同,其中 HaFT-1在根和种子中有明显的表达,在根中表达量比种子的表达量高,而 HaFT-2在梭梭的根和同化枝中有明显的表达,在同化枝中表达高于根的表达量。
2.4 梭梭 HaFT-1和 HaFT-2干旱胁迫后表达分析
以18S rRNA基因为内参基因,特异性引物进行荧光定量PCR分析 HaFT-1和 HaFT-2基因在PEG6000模拟干旱胁迫下的表达量,采用的是相对定量2- △△Ct法计算2个基因在盐胁迫下的表达量(图6)。结果显示, HaFT-1在模拟干旱胁迫处理土壤含水量为12%时,表达量明显升高。到处理后含水量为9%时,基因的表达量达到处理前的5.9倍, HaFT-1基因在处理后含水量6%恢复至处理前的水平,在含水量为3%时恢复至处理前的1.5倍。 HaFT-2在处理后含水量为12%时,表达量达到处理前的1.5倍,之后略有下降,并达到处理前表达量水平。说明 HaFT-1基因受到干旱胁迫诱导。 HaFT-2在模拟干旱胁迫前后基本没有变化,因而 HaFT-2与抗旱性关系不明显。
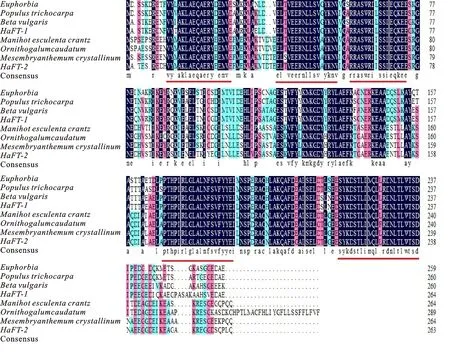
图3 HaFT-1和 HaFT-2氨基酸序列比对Fig.3 HaFT-1 and HaFT-2 amino acid sequence alignments
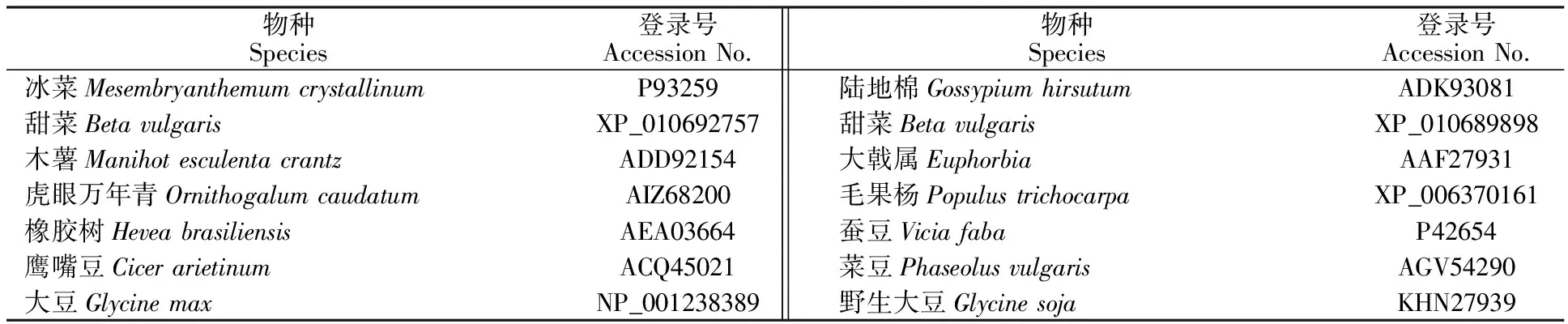
表2 相关物种 14-3-3蛋白基因及其登录号Table 2 14-3-3 gene related species and their GenBank accession numbers
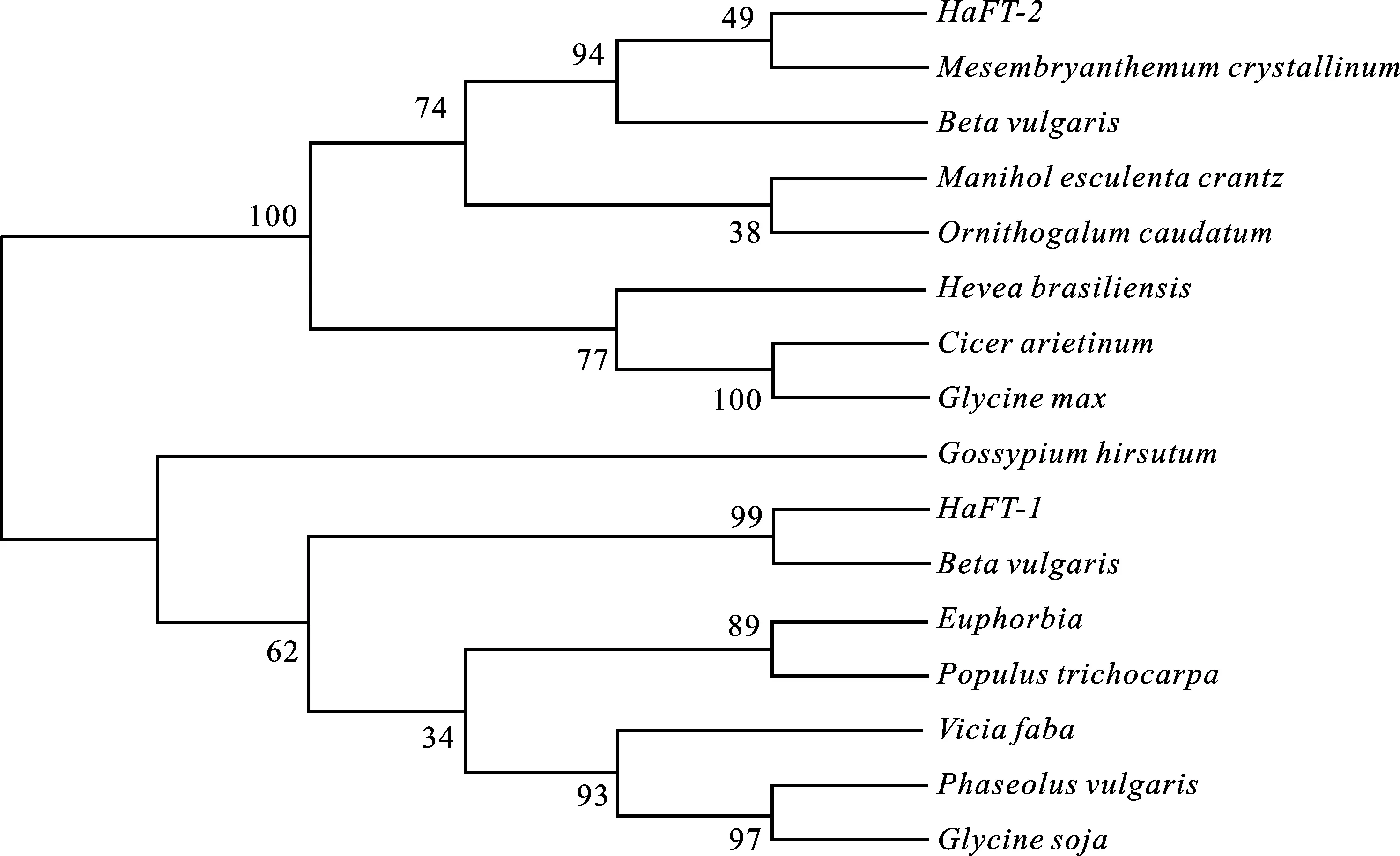
图4 不同植物14-3-3蛋白( HaFT-1和 HaFT-2)序列系统进化树Fig.4 Phylogentic tree of 14-3-3 ( HaFT-1, HaFT-2) sequence based on different plant species

A.梭梭的根 Roots of Haloxylon ammodendron;B.梭梭的同化枝 Assimilation sticks of Haloxylon ammodendron;C.梭梭的种子 Seeds of Haloxylon ammodendron;D.梭梭的花 Flowers of Haloxylon ammodendron
3 讨论与结论
14-3-3蛋白具有组织特异性,在调控植物的发育表达和应答外界环境刺激信号转导中处于中心地位[10],在逆境生理反应中起到重要作用[5]。目前,拟南芥(Arabidopsisthaliana)中有13个成员[13]、烟草(Nicotianatabacum)中有11个成员[14]、大豆(Glycinemax)中有16个成员[15]、水稻(Oryzasativa)中有8个成员被成功鉴定[16]。本研究以梭梭为研究材料,通过RT-PCR方法从中克隆出2个与逆境相关的14-3-3蛋白基因,分别命名为 HaFT-1和 HaFT-2,并对其进行生物信息学分析。这2个14-3-3蛋白基因ORF大小为795 bp和792 bp,编码的氨基酸个数分别为264和263。同时,分别对基因 HaFT-1和 HaFT-2编码的氨基酸序列进行分析和预测,结果发现,2个蛋白的理化性质相似,二级结构均是α螺旋所占比例最大,并且都是亲水性、不稳定蛋白,不具有跨膜结构。氨基酸序列同源性比对发现,2个14-3-3蛋白均与甜菜同源性最高,均为92%。各个物种的14-3-3蛋白在保守区域外,氨基酸序列差异相对较大。分子进化树显示, HaFT-1与甜菜聚为一枝; HaFT-2与甜菜、冰菜聚为一枝,二者亲缘关系最近。

图6 梭梭14-3-3蛋白基因在干旱胁迫下的表达Fig.6 Expression patterns of Haloxylon ammodendron 14-3-3 protein gene under condition of drought stress
对14-3-3蛋白相关特异性表达研究表明,茶树14-3-3蛋白基因在其他组织不表达,仅在晚期花蕾中特异性表达[7]。甘蔗14-3-3蛋白基因有较高并且较丰富的表达信号在其根、茎、叶中,其中叶具有最少含量,茎具有显著上调的表达活性[4]。研究短柄草14-3-3基因家族表达模式发现,短柄草的7个14-3-3蛋白基因在根、茎、未成熟叶、成熟叶和叶鞘等组织中均有表达,暗示这7个14-3-3蛋白基因可能是组成型表达基因[17]。本研究明确14-3-3蛋白基因在梭梭根、同化枝、花和种子中的表达均不相同,其中基因 HaFT-1在根及种子中表达,在根中表达量比种子的表达量高,基因 HaFT-2在同化枝和根中表达明显,在同化枝中表达高于根的表达量。综合2个14-3-3蛋白基因在不同组织中的表达情况,可以看出,2个基因在花中的表达量均不明显。梭梭14-3-3蛋白基因在不同组织中的表达说明其与不同组织的功能有密切关系,且表达具有特异性。
研究杉木14-3-3蛋白基因发现,杉木14-3-3蛋白基因可能参与杉木应答干旱逆境反应[18]。部分研究者通过研究14-3-3蛋白与质膜H+-ATP酶的互作来探讨14-3-3蛋白应答干旱胁迫的分子机理[19-20]。梭梭是旱生植物中的典型代表,为深入研究梭梭14-3-3蛋白抵御干旱逆境的机理,利用荧光定量qRT-PCR方法探究 HaFT-1和 HaFT-2在干旱胁迫下的表达情况。在干旱胁迫下, HaFT-1和 HaFT-2表达量均有所上升,但变化趋势却有着明显的不同。 HaFT-1在模拟干旱胁迫处理土壤含水量为12%时,表达量明显升高,到处理后含水量为9%时,基因的表达量达到处理前的5.9倍, HaFT-1基因在处理后含水量6%恢复至处理前的水平,在含水量为3%时恢复至处理前的1.5倍。 HaFT-2在处理后含水量为12%时,表达量达到处理前的1.5倍,之后略有下降,并达到处理前表达量水平。说明 HaFT-1基因受到干旱胁迫诱导。 HaFT-2在模拟干旱胁迫前后基本没有变化,因而 HaFT-2与抗旱性关系不明显。这表示 HaFT-1和 HaFT-2 这2个基因对应答干旱胁迫表现出不同的调控机制。目前,对于梭梭14-3-3蛋白的遗传转化体系尚不清楚,有待于以后进一步深入研究。
Reference:
[1] 王成云.梭梭属植物对大气干旱的季节性生理生化响应[D].乌鲁木齐:新疆农业大学,2006.
WANG CH Y,Seasonal physiological and biochemical response ofHaloxylonBunge to atmosphere drought[D].Urumqi: Xinjiang Agricultural University,2006(in Chinese with English abstract).
[2] 郭泉水,郭志华,阎 洪,等.我国以梭梭属植物为优势的潜在荒漠植被分布[J].生态学报,2005,25(4):848-853.
GUO Q SH,GUO ZH H,YAN H,etal.Study on potential distribution ofHaloxylonplants dominated desert vegetation in China[J].ActaEcologicaSinica,2005,25(4):848-853(in Chinese with English abstract).
[3] 董占元,姚云峰,赵金仁,等.梭梭光合枝细胞组织学观察及其抗逆性特征[J].干旱区资源与环境,2000,14(5):78-83.
DONG ZH Y,YAO Y F,ZHAO J R,etal.Anatomical observations on the plotosynthatic branch ofHaloxylonammodendrom(C.A.Mey) Bunge and it is the character of drought and salt resistance[J].JournalofAridLandResourcesandEnvironment,2000,14(5):78-83(in Chinese with English abstract).
[4] 罗炼芳,孔 冉,苏俊波.甘蔗14-3-3基因克隆及表达分析[J].南方农业学报,2013,44(11):1757-1764.
LUO L F,KONG R,SU J B.Cloning and expression analysis of 14-3-3 gene fromSaccharumspp.Hybrids[J].JournalofSouthernAgriculture,2013,44(11):1757-1764(in Chinese with English abstract).
[5] MOORE B W,PEREZ V J.Specific acidic proteins of the nervous system[J].InPhysiologicalandBiochemicalAspectsofNervousIntegration,1967:343-359.
[6] XU W,SHI W.Expression profiling of the 14-3-3 gene family in response to salt stress and potassium and iron deficiencies in young tomato (Solanumlycopersicum) roots:analysis by real-time RT-PCR[J].AnnalsofBotany,2006,98(5):965-974.
[7] 余 梅,江昌俊,房婉萍,等.茶树花蕾14-3-3蛋白基因的分子克隆及差异表达分析[J].中国农业科学,2008,41(10):2983-2991.
YU M,JIANG CH J,FANG W P,etal.Cloning and analysis of differential expression of a 14-3-3 protein gene from tea flower bud[J].ScientiaAgriculturaSinica,2008,41(10):2983-2991(in Chinese with English abstract).
[8] 赵秀玲,徐慧妮,郭传龙,等.菠菜14-3-3蛋白基因的克隆及硝酸盐胁迫下的表达分析[J].西北植物学报,2013,33(3):450-457.
ZHAO X L,XU H N,GUO CH L,etal.Cloning and expression analysis of 14-3-3 protein gene ofSpinachunder nitrate stress[J].ActaBotanicaBoreali-OccidentaliaSinica,2013,33(3):450-457(in Chinese with English abstract).
[9] 周 颖,李冰樱,李学宝.14-3-3蛋白对植物发育的调节作用[J].植物学报,2012,47(1):55-64.
ZHOU Y,LI B Y,LI X B.Roles of 14-3-3 proteins in regulating plant development[J].ChineseBulletinofBotany,2012,47(1):55-64(in Chinese with English abstract).
[10] OKSVOLD M P,HUITFELDT H S,LANGDON W Y.Identification of 14-3-3 zeta as an EGF receptor interacting protein[J].FebsLetters,2004,569 (1-3):207-210.
[11] 崔 娜,于志海,韩明利,等.植物14-3-3蛋白研究进展[J].西北植物学报,2012,32 (4):843-845.
CUI N,YU ZH H,HAN M L,etal.Research advancement of 14-3-3 proteins in plant[J].ActaBotanicaBoreali-OccidentaliaSinica,2012,32(4):843-845(in Chinese with English abstract).
[12] LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) method[J].Methods,2001,25(4):402-408.
[13] ROSENQUIST M,ALSTERFJORD M,LARSSON C,etal.Data mining theArabidopsisgenome reveals fifteen 14-3-3 genes.Expression is demonstrated for two out of five novel genes[J].PlantPhysiology,2001,127(1):142-149.
[14] COTELLE V,MEEK S E,PROVAN F,etal.14-3-3s regulate global cleavage of their diverse binding partners in sugar-starvedArabidopsiscells[J].EmboJournal,2001,19(12):2869-2876.
[15] LI X,DHAUBHADEL S.Soybean 14-3-3 gene family:identification and molecular characterization[J].Planta,2010,233(3):569-582.
[16] WANG C,MA Q H,LIN Z B,etal.Cloning and characterization of a cDNA encoding 14-3-3 protein with leaf and stem-specific expression from wheat[J].DnaSequencetheJournalofDnaSequencing&Mapping,2008,19(2):130-136.
[17] 唐振鑫,杨海灵.短柄草14-3-3基因家族的生物信息学及表达模式研究[J].广东农业科学,2013(7):139-142.
TANG ZH X,YANG H L.Bioinformatic and expression pattern analysis ofBrachypodium14-3-3 gene family[J].GuangdongAgriculturalSciences,2013(7):139-142(in Chinese with English abstract).
[18] 倪点乐.杉木14-3-3蛋白基因的克隆和表达分析[D].福州:福建农林大学,2013.
NI D L.Cloing and expression analysis of 14-3-3 protein genes inCunninghamialanceolate(Lamb.) Hook[D].Fuzhou: Fujian Agriculture and Forestry University,2013(in Chinese with English abstract).
[19] 周 冰.蚕豆14-3-3蛋白与质膜H+-ATP酶应答干旱胁迫的分子机理研究[D].昆明:昆明理工大学,2013.
ZHOU B.Molecular mechanisms of response to drought stress for broad bean 14-3-3 proteins and plasma membrane H+-ATPase[D].Kunming: Kunming University of Science and Technology,2013(in Chinese with English abstract).
[20] 李 松.玉米14-3-3蛋白和质膜H+-ATP酶应答干旱胁迫的分子机制研究[D].昆明:昆明理工大学,2013.
LI S.Molecular mechanisms of 14-3-3 proteins and plasma membrane H+-ATPase enzyme of maize response to drought stress[D].Kunming: Kunming University of Science and Technology,2013(in Chinese with English abstract).
(责任编辑:史亚歌 Responsible editor:SHI Yage)
Cloning and Expression Analysis of HaFT-1 and HaFT-2 Genes inHaloxylonammodendron
LI Yajie1,ZHANG Hua1,JIANG Yuanyuan2,MA Hao3,4,YAO Zhengpei1,REN Yanping1,WANG Ze4and MA Lin1
(1.College of Agronomy,Xinjiang Agricultural University,Urumqi 830052,China; 2.Hami Vocational Technical College,Hami Xinjiang 839000,China; 3.College of Agronomy,Nanjing Agricultural University,Nanjing 210095,China; 4.Desert Institute in Drought Areas,Xinjiang Agricultural University,Urumqi 830052,China )
Based on Unigene sequences fromHaloxylonammodendronarid transcriptome database,twoHaloxylonammodendrongenes HaFT-1 and HaFT-2 of 14-3-3 protein were successfully cloned,and it was analyzed and gene was expressed.The results showed that the HaFT-1 gene expression was both in roots and seeds ofHaloxylonammodendron,and showed the strongest expression in root;the HaFT-2 gene showed the highest expression in assimilation sticks ofH.ammodendron,and then in the roots.After drought stress,HaloxylonHaFT-1 and HaFT-2 gene expressions were adjusted downward.This study preliminarily analyzed the expression pattern of HaFT-2 and HaFT-1 gene,and laid foundation for further research on function of this gene.
Haloxylonammodendron;14-3-3 protein;Gene clone;Expression
LI Yajie,female,master student.Research area:resistance of plant breeding.E-mail:631680263@qq.com
MA Lin,male,professor.Research area:agriculture in arid areas and conservation tillage.E-mail: malin5738@126.com
日期:2017-03-03
网络出版地址:http://kns.cnki.net/kcms/detail/61.1220.S.20170303.0833.050.html
2016-03-02
2016-03-31
国家自然科学基金(31260181)。
李亚婕,女,硕士生,研究方向为抗逆植物育种学。E-mail:631680263@qq.com
马 林,男,教授,研究方向为干旱区农业和保护性耕作。E-mail:malin5738@126.com
Q943.2
A
1004-1389(2017)03-0455-08
Received 2016-03-02 Returned 2016-03-31
Foundation item The National Natural Science Foundation of China (No.31260181).

