陆地棉幼苗NaCl胁迫下转录因子的转录组学分析
彭 振何守朴龚文芳潘兆娥贾银华卢艳丽杜雄明,*
1中国农业科学院棉花研究所 / 棉花生物学国家重点实验室, 河南安阳455000;2四川农业大学玉米研究所, 四川温江 611130
陆地棉幼苗NaCl胁迫下转录因子的转录组学分析
彭 振1,2何守朴1龚文芳1潘兆娥1贾银华1卢艳丽2,*杜雄明1,*
1中国农业科学院棉花研究所 / 棉花生物学国家重点实验室, 河南安阳455000;2四川农业大学玉米研究所, 四川温江 611130
以棉花耐盐种质早熟长绒7号和感盐种质南丹巴地大花为材料, 利用转录组测序(RNA-Seq)技术分析2个种质在200 mmol L–1NaCl胁迫4 h和24 h后幼苗叶片的转录因子家族及转录因子的表达情况。结果表明, 从样本中鉴定出54个转录因子家族的2815个转录因子。盐胁迫4 h后, 长绒7号有249个转录因子基因表达发生变化; 南丹巴地大花中有261个转录因子基因表达发生变化。在胁迫24 h后, 2个种质对盐响应的转录因子数量均剧增。只在耐盐种质长绒7号特异表达的有106个(4 h)和184个(24 h)转录因子基因, 对于2个种质共同表达的转录因子有143个(4 h)和282个(24 h)。通过筛选, 鉴定出26个与耐盐相关的转录因子家族124个转录因子基因, 并采用荧光定量验证转录组数据的准确性。推测高表达的11个转录因子的基因CotAD_66280 (HD-ZIP)、CotAD_47058 (ERF)、CotAD_18472 (G2-like)、CotAD_04289 (C2H2)、CotAD_57763 (HSF)、CotAD_23656 (TCP)、CotAD_02221 (ERF)、CotAD_23975 (WRKY)、CotAD_54036 (MYB)、CotAD_27788 (NAC)和CotAD_23815 (HSF)有可能与陆地棉抗盐性密切相关, 可作为陆地棉耐盐育种的候选基因源。
陆地棉; NaCl胁迫; 转录组分析; 转录因子
Keywords:Upland cotton; NaCl stress; Transcriptome analysis, Transcription factor
近年来, 全球气候变化和土壤盐渍化加重, 可耕地越来越少, 已经成为全球性的生态问题, 人类面临的形势日趋严峻。在我国, 盐碱地面积多达3.6亿公顷, 占全国可利用面积的4.88%, 另有近2.0亿公顷盐碱荒地资源未被开发利用, 这些盐碱地主要分布在华北、西北、东北和东部滨海地带[1]。随着耕地面积减少, 棉花种植逐渐向盐碱地集中, 因此这些盐碱地蕴藏着巨大的开发潜力。棉花是比较耐盐碱的作物之一, 但是从目前研究来看盐碱地高浓度的盐分仍然是制约棉花产量的主要因子[2]。所以要在盐碱地广泛植棉, 必须进一步提高棉花的抗盐能力, 其中通过转基因技术提高棉花耐盐性是解决盐碱地植棉的重要途径之一。近年来, 随着基因工程技术的迅速发展, 棉花耐盐性研究已逐步转向分子水平。尽管在其他作物如水稻、小麦中已经克隆鉴定了大量盐胁迫应答基因, 但棉花耐盐基因的克隆鉴定研究并不多, 转基因功能验证效果明显的例子更少。而且这些基因大多是通过同源克隆得到, 如GhNHX1[3]、GhDREB1[4]、GhMKK1[5]、GhWRKY34[6]、GhAnn1[7]等。近年来随着棉花基因组的测序完成[8]以及高通量测序技术的发展, 将为棉花耐盐分子机制和功能基因研究提供极大便利。
植物抗逆性方面受多个信号传导途径的调控,而多基因协同表达又受转录因子调控[9]。因此通过克隆转化植物转录因子, 利用关键转录因子的调控作用促使多个功能基因表达来提高作物的综合抗性, 无疑是一个更为有效的方法和途径。近年来,棉花中大量不同类型的转录因子基因相继被分离出, 如AP2/DREBP[4]、bZIP[10]、C2H2[11]、C3H[12]、ERF[13]、NAC[14]、WRKY[6,15]等。已有报道称棉花转录组中发现大量转录因子在盐胁迫条件下表达发生变化。如 Guo等[16]利用基因芯片分析盐胁迫条件下陆地棉根转录组的变化, 发现有123个转录因子基因受盐胁迫诱导表达。Zhang等[17]通过分析亚洲棉-石系亚 1号盐胁迫早期(3 h)转录组鉴定了4002个转录因子基因, 其中差异表达转录因子基因 1598个(占 48.93%)。尽管如此在棉花耐盐性基因工程应用上还是有限的, 而且以上研究仅发掘盐胁迫响应的转录因子, 是否与耐盐性密切有关还需进一步验证。
虽然植物根系是识别和应答盐胁迫的感知部位,很多研究也是基于根系, 但是陆地棉不同, 早期已有研究证明陆地棉体内积累的盐分95%在地上部分[18]。孙小芳等[3]研究表明棉花地上部盐分器官水平上的区域化分布特征明显。本实验室前期生理试验也证明盐胁迫下耐盐种质比感盐种质叶片保持更低的Na+和 Na+/K+比, 有助于保持相对较高的光合特性,更好地适应不利环境[19]。因此, 叶片对棉花耐盐性的贡献也不可忽视。棉花种质间耐盐性存在较大差异并且耐盐机制复杂, 不同棉花种质在盐胁迫条件下其转录因子的时空表达模式也不尽相同。本研究通过比较转录组(RNA-seq)分析来探讨不同抗感盐陆地棉种质叶片盐胁迫响应转录因子的表达特性,以期获得与耐盐相关的转录因子, 更好地理解陆地棉对盐胁迫响应的分子机制, 增加对棉花耐盐机制的认识, 并为转基因耐盐种质提供功能基因源参考。
1 材料与方法
1.1 材料及处理方法
所用陆地棉耐盐种质为早熟长绒 7号(Earlistaple 7, 简称“E”), 盐敏感种质为南丹巴地大花(简称“N”), 均由国家棉花种质资源中期库近 10年的盐池和盐碱地抗逆鉴定中挑选出来[8]。选择籽粒饱满均匀一致的种子若干, 休眠的种子先经太阳暴晒1~2 d。种子经15%双氧水消毒4 h, 无菌水冲洗4次, 在25℃下灭菌水浸泡24 h。将浸泡过的种子埋在装满无菌石英砂的发芽盒中, 置 28℃培养箱3 d, 待子叶刚刚伸展开时, 将幼苗移至 1/2 Hoagland水培营养液的人工气候室培养槽中, 人工气候室光照强度> 450 μmol m−2; 昼夜温度28/22℃;光照时间14 h/10 h; CO2浓度>350 mg L–1; 空气湿度60%~80%。为了保证幼苗正常生长, 每7 d更换一次培养液, 在幼苗长至三叶期进行盐处理, 盐处理的前一天更换培养液。用200 mmol L–1NaCl溶液处理幼苗, 分别取盐处理4 h和24 h以及未进行盐处理(对照, 4 h)幼苗的3片真叶, 迅速用液氮冷冻, 存于–80℃冰箱备用。
1.2 叶片总RNA提取、文库构建及转录组测序
用植物总RNA提取试剂盒(RNA Prep Pure Plant Kit, TIANGEN)分别提取6个样本的总RNA, 并用NanoDrop-2000分光光度计测定其A260、A280值, 计算样品浓度, 然后以 15%的变性聚丙稀酰胺凝胶电泳, 检测质量合格后送测序公司进行 cDNA文库的构建, 在Illumina HiSeq 2000测序仪上进行双端测序(Paired-end)。 初步分析测序数据交由深圳华大基因研究院。后续分析以下 4个比对方案, N4/N0和N24/N0分别表示南丹巴地大花 (N) 受NaCl胁迫后4 h和24 h与0 h (对照)比较; E4/E0和E24/E0分别表示早熟长绒7号 (E) 受NaCl胁迫后4 h和24 h与0 h (对照)比较。
1.3 生物信息学分析
由转录组测序所得的数据称为原始读段(raw reads), 首先对raw reads进行质控(QC), 以确定测序数据是否适用于后续分析。质控后, 经过滤得到干净读段(clean reads), 用SOAPaligner/SOAP2软件[20]将6个样本获得的clean reads比对到陆地棉(BGI-AD1)公布的参考基因组和参考基因序列(https://www. cottongen.org/data/download/genome), 最多允许5个错配。统计 reads在参考序列上的分布情况及覆盖度。经检验合格后, 则运用RPKM法(Reads per kb per million reads)计算基因表达量[21], 计算得到的基因表达量可直接用于比较不同样品间的基因差异表达。在本试验中, 以2个种质无盐处理(E0和N0)作为对照, 4个NaCl处理样本(E4, N4, E24, N24)与各自对照样本比较其基因表达水平。参照 Audic等[22]描述的方法, 对4个比对方案(E4/E0, E24/E0, N4/N0, N24/N0)中每个基因进行显著性分析。只有符合错配率[23](false discovery rate, FDR) ≤0.001且倍数差异(Ratio)在 2倍以上的基因被定义为样品间差异表达的基因。最后基于差异表达基因, 进行GO功能富集分 析 (WEGO: http://wego.genomics.org.cn/cgi-bin/ wego/index.pl)。本研究测得的转录组原始数据可以从 NCBI/GEO数据库获取, 登录号为 GSE58655 (http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc= GSE58655)。
1.4 转录因子家族基因的鉴定和耐盐相关转录因子的筛选
将6个转录组测序鉴定到的所有基因合并为一个基因集数据库。从北京大学的植物转录因子数据库PlantTFDB (http://planttfdb.cbi.pku.edu.cn/)公布的陆 地 棉 (Gossypium hirsutum)和 雷 蒙 德 氏 棉(Gossypium raimondii)转录因子家族信息库中分别下载对应转录因子蛋白序列作为总库。然后利用Blast工具BlastX模块将转录组中鉴定的所有基因核酸序列和2个物种转录因子家族蛋白序列同源比对,筛选条件为匹配率(identify)≥90%且 E-value≤1E–15。最后通过基因ID列表获得不同比对方案差异表达的转录因子基因信息进行后续分析。
参考 Chao等[24]和 Guo等[25]的研究方法, 依据抗感种质棉花响应盐胁迫的基因表达量差异, 我们定义与耐盐相关转录因子基因必须符合以下参数之一: (1)耐盐种质中特异的差异表达转录因子且|log2Ratio (Ex/E0)|≥2; (2)抗感盐种质中共有的差异表达转录因子的log2Ratio(Ex/E0) – log2Ratio(Nx/N0)≥1 (上调基因)或 log2Ratio(Ex/E0) – log2Ratio(Nx/N0)≤ –1 (下调基因), x = 4或24。
1.5 实时荧光定量PCR验证
选择筛选的耐盐相关的差异表达的转录因子基因15个, 利用Primer-NCBI在线设计软件(http://www. ncbi.nlm.nih.gov/tools/primer-blast/)设计引物(表 1),进行实时荧光定量PCR分析。将各样品3次生物学重复的RNA采用第1链cDNA合成试剂盒(TaKaRa,日本)反转录为 cDNA, 以反转录产物为模板, 以Actin为内参, 采用 QIAGEN公司的 SYBR Green RT-PCR试剂盒, 利用ABI 7500FAST荧光定量PCR系统(ABI公司, 美国)进行荧光定量检测。各样品每个基因3次生物学重复, 按2–ΔΔCT相对定量法计算相对表达量。
2 结果与分析
2.1 全转录组转录因子的鉴定与定量分析
参考陆地棉(AD1)基因组信息, 本试验6个转录组文库共鉴定了42 234个基因。基于BlastX比对筛选条件, 鉴定到基因总库与陆地棉转录因子家族(50个家族1151个成员)同源序列比对后获得可信度高的转录因子基因1208个; 与雷蒙德氏棉转录因子家族(58个家族4895个成员)比对获得转录因子基因2744个。由于植物转录因子数据库(PlantTFDB)公布的陆地棉转录因子是基于以前早期短序列转录本或蛋白序列预测的, 所以数量有限。二倍体雷蒙德氏棉是四倍体陆地棉祖先种, 基因组组装注释相对完善, 大部分基因与陆地棉同源, 转录因子数据库比较系统完整, 比对结果更为可靠。因此将二倍体雷蒙德氏棉和四倍体陆地棉2个物种转录因子整合取并集, 共获得2815个转录因子(图1), 隶属于54个转录因子家族。

表1 实时荧光定量PCR所用的引物Table 1 Primers for real-time quantitative PCR
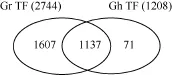
图1 本研究鉴定基因总库与雷蒙德氏棉转录因子和陆地棉转录因比对结果Fig. 1 BlastX results of transcription factors number of G. raimondii homologous and G. hirsutum in response to salt stress
2.2 不同耐盐种质响应 NaCl胁迫的转录因子家族及转录因子表达模式
在NaCl胁迫4 h后, 与对照相比, 早熟长绒7号中有 249个转录因子差异表达, 其中183个基因上调, 含倍数大于10 (指的是log2Ratio值, 下同)的转录因子9个, 除了HSF (CotAD_71942)、HD-ZIP (CotAD_71942) 和MYB-related (CotAD_02694)表达倍数在2个种质中相差不大外, 其余6个均大于南丹巴地大花的2.4~13.1倍(表2); 下调转录因子基因66个, 下调倍数最大的是编码 bZIP转录因子基因(CotAD_28150)。 感盐种质南丹巴地大花中有 261个转录因子差异表达(158上调, 103下调)(图2)。表达倍数大于10的转录因子有4个; 下调倍数最大的也是编码 bZIP转录因子基因(CotAD_13899)。随着盐胁迫时间延长至 24 h, 诱导表达的转录因子基因表达倍数增加了1.5~1.8倍。在耐盐种质中有466个转录因子诱导差异表达(347上调, 119下调), 倍数大于10有17个转录因子(主要隶属NAC、WRKY和 ERF家族), 其中有14个表达倍数均大于南丹巴地大花的2倍以上(表2)。下调基因表达倍数大于四倍的转录因子有 9个; 在感盐种质中差异转录因子数目表现略少, 涉及到 277个上调和 116个下调共 393个转录因子(图2)。上调转录因子中有11个基因表达倍数大于10, 5个下调转录因子表达倍数大于6。
进一步分析 4个比对方案转录因子家族数目发现, 早期盐胁迫阶段(4 h), 2个种质中转录因子分别隶属于42个和40个家族, 其中bHLH、bZIP、ERF、NAC和GRAS家族转录因子最多, 且大部分上调表达 (图3)。在24 h, 2个种质中转录因子隶属于43个和41个转录因子家族, 而且主要发现在耐盐种质中上调的bHLH、bZIP、ERF、NAC和WRKY家族转录因子数目均多于感盐种质。同时发现下调的转录因子家族中, C2H2和MYB-related家族转录因子数目在感盐种质均多于耐盐种质(图3)。

图2 4个比较方案中差异表达的上下调转录因子基因数目Fig. 2 Number of up and down-regulated transcription factor genes in four compared schemes.

表2 早熟长绒7号和南丹巴地大花NaCl胁迫下表达倍数大于10和小于–4的差异表达的转录因子基因Table 2 Differentially expressed transcription factor (TF) genes in response to NaCl stress expressed at more than 10-folds and less than –4 in Earlistaple 7 and Nandanbadidahua

(续表2)
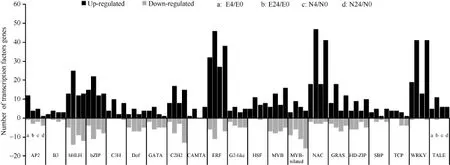
图3 四种比对方案里上下调转录因子所属家族的基因数目比较Fig. 3 Number of differentially expressed transcription factor genes belonged to different families from four comparisons横坐标表示不同转录因子家族, 纵坐标轴表示4种比对方案里对应转录因子家族上下调基因数目。字母a, b, c, d分别表示N4/N0、N24/N0、E4/E0和E24/E0四个比对方案。The x-axis indicates the different transcription factor families in the four comparisons, and the y-axis represents the number of differentially expressed (up- or down-regulated) transcription factor genes. The letters a, b, c, d, indicate the four comparisons (N4/N0, N24/N0, E4/E0, and E24/E0), respectively.
2.3 不同耐盐种质在 NaCl胁迫响应的共有和特有转录因子表达分析
比较分析抗、感盐不同的 2个种质共同差异表达的转录因子表明, NaCl处理4 h后, 只在早熟长绒7号特异表达的转录因子共有106个, 包括79个上调和27个下调。上调倍数最大的转录因子是G2-like (CotAD_18472, 12.3); 下调倍数最小的是转录因子bZIP (CotAD_28150, –4.55)。而在南丹巴地大花中下调转录因子(64) 略多于上调转录因子(54) (图4-A)。相比4 h, NaCl胁迫24 h后早熟长绒7号特异转录因子增加至 184, 尤其是上调转录因子(133), 表达倍数大于 10的转录因子有 4个, 即 TCP (CotAD_ 23656), HSF (CotAD_57763)、ERF (CotAD_47058)、NAC (CotAD_27788)。相反, 南丹巴地大花下调转录因子减少(图4-B)。而且发现, 在早熟长绒7号中共表达的74个转录因子(占71%)倍数均高于南丹巴地大花, 而下调表达转录因子中没有。同时发现2个种质间对于同一转录因子只在一个种质上调(下调)表达或者两者表达趋势相同, 没有发现相反情况。
早熟长绒7号在NaCl胁迫4 h和24 h间差异表达转录因子中, 68个(37.5%)只在早期特异表达, 其中涉及上调转录因子家族的23个; 285个(61.2%)只在后期特异表达; 涉及上调转录因子家族达 33个(图4-C); 南丹巴地大花种质情况类似(图4-D)。这些在特定时期差异表达的转录因子基因被认为在渗透胁迫(4 h)和离子胁迫(24 h) 2个盐胁迫阶段发挥不同的作用, 而且离子胁迫阶段参与的转录因子数目和家族更多。还有一特点需要说明, 尽管存在共同的差异表达基因, 但是其表达趋势在 2个时间点是一致的(即都是上调/下调), 而且在24 h表达量相对更高(极个别低)。说明这些基因参与对逆境胁迫基础调节响应的整个过程。

图4 两个棉花种质在盐胁迫不同时间点差异转录因子数目比较Fig. 4 Comparison of the number of differentially expressed transcription factor genes under salt stress at different time points in two cotton varietiesA和B图分别表示盐胁迫4 h和24 h后2个种质间共有和特有的上下调转录因子及家族数目。C和 D图分别表示早熟长绒 7号和南丹巴地大花在盐胁迫4 h和24 h共有和特有的上下调转录因子及家族数目。箭头朝上表示上调基因, 朝下为下调基因, 括号里显示所属转录因子家族数目。Panels A and B show the numbers of common/specific transcription factor genes at 4 and 24 hours, respectively, that were differentially expressed under NaCl stress between two cotton varieties. Panels C and D show the numbers of common/specific transcription factor genes in Earlistaple 7 and Nandanbadidahua, respectively, that were differentially expressed between 4 and 24 hours of NaCl stress. Upward and downward arrows indicate up-regulated and downregulated genes, respectively. The figure in parentheses is the number of transcript factor families.
2.4 耐盐相关转录因子基因的筛选
分别分析 2个种质共同差异表达转录因子家族表明, 在 4 h只在耐盐种质上调表达的转录因子家族是FAR1、BBR-BPC、NF-X1、SBP、ZF-HD、GRF和CAMTA, 这与24 h特异表达转录因子家族完全不同(AP2和BES1)。比较盐胁迫后2个时间点2个种质特异和共同的转录因子家族, 24 h表达的转录因子隶属家族占15个, 其中包含了4 h表达的转录因子家族(7个)(图5)。
通过严格的筛选条件, 分别筛选出 31个(4 h,长绒7号特异)、22个(4 h, 2个种质共同差异表达)、66个(24 h, 早熟长绒7号特异)和40个(24 h, 2个种质共同差异表达) 耐盐相关转录因子, 去除在4 h和24 h重叠的转录因子, 最后获得124个与耐盐可能相关的转录因子(附表1)。按照这些转录因子基因在早熟长绒7号中的表达模式, 可以分为早期诱导(只在4 h差异表达)、持续诱导(同时4 h和24 h差异表达)、后期诱导(只在24 h差异表达) 3种类型。其独特的转录因子家族有: 早期4 h的BBR-BPC、FAR1、G2-like和DBB; 持续诱导中有M-type; 后期诱导转录因子家族AP2、C3H、GeBP、GRF、TALE、YABBY 6种。其中, CotAD_66280 (HD-ZIP)、CotAD_ 47058 (ERF)、 CotAD_18472 (G2-like)、 CotAD_04289 (C2H2)、CotAD_57763 (HSF)、CotAD_23656 (TCP)、CotAD_02221 (ERF)、CotAD_23975 (WRKY)、CotAD_54036 (MYB)、CotAD_27788 (NAC)、CotAD_ 23815 (HSF)共11个转录因子基因在抗、感种质中差异表达倍数相差很大, 达到 1000倍以上(log2Ratio值>10)。而且除了CotAD_66280 (HD-ZIP)基因诱导后下调外, 其他10个都表现出盐诱导上调高表达。对124个转录因子基因进行GO功能富集分析表明,除了56个基因没有找到GO注释, 68个转录因子基因按照参与的生物过程分类设计了21类(图6)。表达类别最多是大分子代谢过程(24个), 其次是生物合成过程(20个)和细胞代谢过程(20个), 氮化合物代谢过程(18个), 初级代谢过程(18个)以及各种胁迫响应(21个)。没有找到GO注释的56个转录因子基因被暗示为在陆地棉中新鉴定的未知功能转录因子。附表 1中这些经过筛选的候选转录因子基因可以为将来抗盐转基因工程所需的基因源提供参考。
2.5 实时定量荧光PCR验证
从筛选到的 124个耐盐相关转录因子中选取ERF、HSF、NAC、WRKY等家族的 15个基因(表1), 利用实时荧光定量 PCR对其盐处理前后表达变化分析, 发现荧光定量结果与转录组结果显著相关, 4 h和24 h时间点的相关系数分别是0.7374和0.8340 (图7), 证明了转录组测序数据结果的可靠性。
3 讨论
植物应对逆境响应过程涉及到渗透物质的合成与保护, 离子平衡调节, 活性氧的清除和质膜的保护等多方面。从分子水平看, 这些胁迫相关蛋白编码的基因离不开各类转录因子的转录调控, 同时在盐胁迫不同的阶段呈现的不同表达模式[26]。本研究表明, 盐胁迫后无论耐盐种质还是感盐种质, 有大量的转录因子家族及其转录因子受到诱导表达, 表达量也发生剧烈变化(图3和附表1)。这均表明不同类型的转录因子参与调控不同的生物过程, 也可以解释为什么2个种质在NaCl胁迫下, 有的转录因子既有转录上调表达的, 也有下调表达的。这种表达的差异可能是棉花重新组织和调节生理生化活动,提高某些抗性蛋白的数量, 或者抑制某些耗能活动相关的表达, 以此调节体内各种生理代谢活动, 以适应不利环境。

图5 盐胁迫4 h和24 h后耐盐种质特异表达和两个种质共同表达的上下调转录因子所在家族交集维恩图Fig. 5 Venn diagram of transcript factor families for specific differentially expressed in Earlistaple 7 and overlapped between two cotton varietys after 4 h and 24 h under NaCl stress
转录因子在其转录水平表达的具有种质特异性。早熟长绒7号在盐胁迫4 h和24 h后分别有106个和184个差异表达转录因子是NaCl特异响应转录因子, 南丹巴地大花中则分别有118个和111个的差异表达转录因子为特异响应因子(图4)。这2个种质转录表达的差异性表明其转录水平调控方式存在差异, 甚至可以说对盐害的忍受机制不相同, 这可能是它们耐盐性不同的根本原因。转录因子在同一种质不同胁迫阶段其转录水平具表达的时期特异性,本研究中2个种质均经历盐胁迫4 h和24 h。通过比较发现这 2个时期均有其特异性和共同性(图 5),表明这些转录因子在盐胁迫条件下无论耐盐种质还是盐敏感种质转录水平调控方式存在不同时期差异,结合Peng等[19]生理实验结果, 可以认为是对早期的渗透胁迫(4 h)和后期离子胁迫(24 h)响应差异造成的。

图6 68个耐盐相关转录因子基因的GO功能富集分析Fig. 6 GO functional annotation of 68 salt-tolerance-related TF genesAll genes were assigned to at least one GO term and were grouped into three main GO categories and 31 sub-categories. TF: transcription factor.
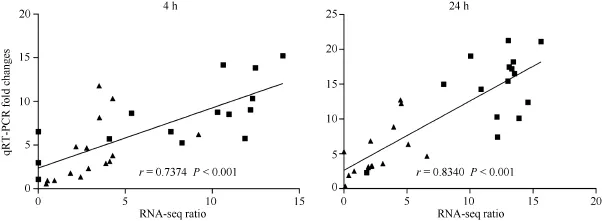
图7 15个转录因子基因的实时荧光定量PCR及转录组表达量的相关性分析Fig. 7 Correlation analysis of 15 differentially expressed TF genes under salt stress for 4 h and 24 h based on qRT-PCR and RNA-seq datar值代表皮尔逊相关系数, 方块代表早熟长绒7号基因, 三角形代表南丹巴地大花基因。Pearson’s correlation coefficients (r) are 0.7374 and 0.8340, respectively (P < 0.001). Box menans Earlistaple 7 gene and triangle means Nandanbadidahua gene.
对于盐敏感种质和耐盐种质在盐胁迫条件下所差异表达的转录因子基因部分相同, 其共性表明不同耐盐性种质在盐胁迫时有某种相同的响应机制,与棉花基础抗性有关。同时, 我们也发现这些共同的差异表达基因在2个种质中诱导或抑制程度是不同的(表2和附表1), 而且还有在耐盐种质中诱导的基因反而在感盐种质是抑制的, 例如CotAD_23815、CotAD_41797、CotAD_19813等。这些基因的抑制可能是在感盐种质中受到其他机制调控, 这些基因的鉴定有助于进一步理解感盐种质对盐敏感的原因。
目前与耐盐相关的转录因子家族主要有 AP2、ERF、bHLH、bZIP、C2H2、MYB、NAC、WRKY等[27]。例如在植物中广泛存在的, 逆境胁迫响应较多的转录因子家族是ERF[28]。通过比较基因在耐盐性不同的种质之间差异变化, 可以筛选出潜在的与耐盐性相关的基因。我们的研究发现在筛选的 124个与耐盐相关转录因子中ERF占有25个(附表1)。关于ERF基因在育种中的应用, Guo等[29]将番茄中OPBP1基因转入烟草提高植株的耐盐能力; Zhang等[30]将 GmERF3转入烟草也提高了转基因烟草抗旱、耐盐和抗病能力。此外, 陆地棉 ERF家族的GhERF1、GhERF2、GhERF3和GhERF6基因的克隆与表达分析表明其响应盐、低温和干旱胁迫及乙烯和ABA处理, 对提高非生物逆境抗性具有较大的潜力[13,31]。棉花中该家族抗盐胁迫的研究目前较少,需要继续筛选潜在的抗性基因, 而我们筛选的 ERF基因可以作为转基因的候选参考。
在植物中特有的NAC (NAM、ATAF和CUC2)转录因子家族, 参与逆境胁迫的应答[32]。本研究分析发现2个种质4 h均有18个NAC基因上调, 而在24 h后, 分别有47个(早熟长绒7号)和48个(南丹巴地大花) NAC转录因子基因上调表达。下调表达基因均较少(图 2), 表明 NAC家族转录因子主要通过诱导表达起调控作用。最后通过筛选获得了15个候选耐盐相关NAC基因(附表1)。Meng等[14]首次从陆地棉各种组织中分离到6个全长的NAC基因家族新成员(GhNAC1~GhNAC6)。发现这6个转录因子基因在叶片中表达量最高。其中GhNAC4和GhNAC6同时被干旱、高盐、低温和 ABA处理强烈诱导。Huang等[33]随后报道了GhNAC7~GhNAC13, 并发现它们在根部表达最多, 受低温、脱落酸、干旱和高盐的诱导。本研究从15个耐盐相关NAC转录因子Nr注释发现有 9个是已测序的陆地棉基因, 即GhNAC1 (CotAD_28258)、GhNAC4 (CotAD_29738)、GhNAC5 (CotAD_30367)、GhNAC7 (CotAD_52619)、GhNAC9 (CotAD_65778)、GhNAC11 (CotAD_28325; CotAD_53191)、GhNAC13 (CotAD_38495; CotAD_ 37052)。这些基因在盐胁迫条件下要么持续表达要么在24 h差异表达。基于前人的实验结果, 植物中同时对不同逆境高水平应答的NAC转录因子暗示着它们能使转基因植物增强几种胁迫耐受性[34]。如果进一步深入研究这些鉴定筛选的基因功能, 对陆地棉耐盐基因工程发展将具有重要意义。
在植物中特有的一种锌指蛋白家族是WRKY。已发现的 WRKY类转录因子主要参与植物形态发育, 对病原体的防卫反应, 对逆境胁迫如干旱和低温等响应[35]。本研究中发现大量的WRKY转录因子基因受盐胁迫诱导表达, 而且随着胁迫时间延长,上调表达基因数目剧增(图3)。胁迫24 h后2个种质差异表达的WRKY基因共22个基因(表2), 它是陆地棉对盐胁迫应答反应中潜在的重要调控因子。目前已克隆了若干个陆地棉WRKY基因, 并证明其超表达能提高植株的耐盐性[6,15,36-38]。
除了以上三大家族转录因子以外, 在各个比对方案里的其他转录因子家族在植物中作用同等重要。bZIP (basic region/leucine zipper碱性亮氨酸拉链)转录因子识别的核心区域基本都是在受 ABA诱导的基因启动子中, 而干旱, 高盐和ABA处理均诱导ABA生物合成, 因此 bZIP必然参与这些逆境胁迫响应[39]。本研究中, 4 h和24 h盐胁迫2个种质bZIP转录因子基因占有比例较多, 而且上调基因居多(图3)。从注释上看基本也是预测的蛋白, 说明对这类蛋白基因研究的不多。早期从拟南芥中分离出逆境诱导的4种bZIP蛋白, 受干旱、高盐或外源ABA诱导, 调控下游的耐盐和耐旱等诸多抗性相关基因的表达[40]。但还没有发现在棉花上的研究。对于植物bHLH转录因子家族来说, 其蛋白功能也研究得较少。初步研究表明, 植物 bHLH转录因子广泛参与了所有生长和发育过程的调控作用[41]。而在陆地棉逆境胁迫研究中仅克隆到 1个 bHLH转录因子。Meng等[42]首次从陆地棉克隆了GhbHLH1基因全长,这个基因能够迅速被ABA处理和干旱胁迫诱导, 但是在叶片中却不受高盐和低温的诱导表达。本研究筛选出来的5个bHLH基因均在耐盐种质叶片中差异表达, 可以作为下一步研究对象。除了 ERF和NAC之外, MYB类转录因子也是植物中数量最大,功能多样化的转录因子家族之一[43]。目前, 棉花上对 MYB的研究集中在棉胚珠和纤维发育阶段[44-46],另一方面 MYB虽然参与植物逆境胁迫的调控, 但是对陆地棉相关的研究并不多[47-50]。我们研究发现,陆地棉受盐胁迫差异表达的 MYB转录因子数目占有不小的比例。这些转录因子的结构、功能仍需后续的大量试验研究。另外, 在植物中数量不多且逆境研究中不常见的的一些转录因子家族如BBR-BPC、FAR1、G2-like、GATA、GeBP、M-type、TALE、TCP和YABBY等在比较转录组分析结果中发现大量相关转录因子显著上调表达(附表1)。这部分基因不应被忽视, 可以作为后续研究的重点参考,进一步分析抗逆表达特性和功能。
4 结论
本研究从棉花幼苗中鉴定出3467个转录因子。发现NaCl胁迫后4 h, 耐盐种质早熟长绒7号有249个转录因子基因表达发生变化; 在感盐种质南丹巴地大花中则有261个转录因子基因表达发生变化。随着胁迫时间延长至24 h, 2个种质对盐响应的转录因子数量均剧增。只在耐盐种质特异表达的有 106个(4 h)和184个(24 h)个转录因子基因, 对于2个抗感盐种质共同表达的转录因子有143个(4 h)和282个(24 h)。筛出与耐盐相关的26个转录因子家族的124个转录因子基因, 可作为陆地棉耐盐育种的候选基因。
[1] 王佳丽, 黄贤金, 钟太洋, 陈志刚. 盐碱地可持续利用研究综述. 地理学报, 2011, 66: 673–684 Wang J L, Huang X J, Zhong T Y, Chen Z G. Review on sustainable utilization of salt-affected land. Acta Geograph Sin, 2011, 66: 673–684 (in Chinese with English abstract)
[2] 张国伟, 路海玲, 张雷, 陈兵林, 周治国. 棉花萌发期和幼苗耐盐性评价及耐盐指标筛选. 应用生态学报, 2011, 22: 2045–2053 Zhang G W, Lu H L, Zhang L, Chen B L, Zhou Z G. Salt tolerance evaluation of cotton (Gossypium hirsutum L.) at its germinating and seedling stages and selection of related indices. Chin J Appl Ecol, 2011, 22: 2045–2053 (in Chinese with English abstract)
[3] Wu C A, Yang G D, Meng Q W, Zheng C C. The cotton GhNHX1 gene encoding a novel putative tonoplast Na+/H+antiporter plays an important role in salt stress. Plant Cell Physiol, 2004, 45: 600–607
[4] Gao S Q, Chen M, Xia L Q, Xu H H, Xu Z S, Li L C, Zhao C P, Cheng X G, Ma Y Z. A cotton (Gossypium hirsutum) DRE-binding transcription factor gene, GhDREB, confers enhanced tolerance to drought, high salt, and freezing stresses in transgenic wheat. Plant Cell Rep, 2009, 28: 301–311
[5] Lu W, Chu X, Li Y, Wang C, Guo X. Cotton GhMKK1 induces the tolerance of salt and drought stress, and mediates defence responses to pathogen infection in transgenic Nicotiana benthamiana. PLoS One, 2013, 8: e68503
[6] Zhou L, Wang N N, Gong S Y, Lu R, Li Y, Li X B. Overexpression of a cotton (Gossypium hirsutum) WRKY gene, GhWRKY34, in Arabidopsis enhances salt-tolerance of the transgenic plants. Plant Physiol Biochem, 2015, 96: 311–320
[7] Zhang F, Li S, Yang S, Wang L, Guo W. Overexpression of a cotton annexin gene, GhAnn1, enhances drought and salt stress tolerance in transgenic cotton. Plant Mol Biol, 2015, 87: 47–67
[8] Li F G, Fan Y, Lu C R, Xiao G H, Zou C S, Kohel R J, Ma Z Y, Shang H H, Ma X F, Wu J Y, Liang X M, Huang G, Percy R G, Liu K, Yang W H, Chen W B, Du X M, Shi C C, Yuan Y L, Ye W W, Liu X, Zhang X Y, Liu W Q, Wei H L, Wei S J, Huang G D, Zhang X L, Zhu S J, Zhang H, Sun F M, Wang X F, Liang J, Wang J H, He Q, Huang L H, Wang J, Cui J J, Song G L, Wang K B, Xu X, Yu J Z, Zhu Y X, Yu S X. Genome sequence of cultivated Upland cotton (Gossypium hirsutum TM-1) provides insights into genome evolution. Nat Biotechnol, 2015, 33: 524–530
[9] Winicov I. New molecular approaches to improving salt tolerance in crop plants. Ann Bot, 1998, 82: 703–710
[10] 李月, 许朋斐, 陈全家, 代培红, 刘超, 曲延英, 刘晓东. 棉花bZIP转录因子基因 GhbZIP15的克隆与表达分析. 棉花学报, 2015, 27: 515–523 Li Y, Xu P F, Chen Q J, Dai P H, Liu C, Qu Y Y, Liu X D. Molecular cloning and expression of a bZIP transcription factor gene GhbZIP15 in cotton (Gossypium hirsutum L.). Cotton Sci, 2015, 27: 515–523 (in Chinese with English abstract)
[11] 张新宇, 林书岱, 张涛, 裴柳玲, 唐清, 刘峰, 刘永昌. 棉花C2H2类型锌指蛋白基因GhSIZ1的克隆及表达分析. 棉花学报, 2015, 27: 189–197 Zhang X Y, Lin S D, Zhang T, Pei L L,Tang Q, Liu F, Liu Y C. Cloning and expression analysis of GhSIZ1, encoding a C2H2 zinc finger protein in cotton (Gossypium hirsutum). Cotton Sci, 2015, 27: 189–197 (in Chinese with English abstract)
[12] Guo Y H, Yu Y P, Wang D, Wu C A, Yang G D, Huang J G, Zheng C C. GhZFP1, a novel CCCH-type zinc finger protein from cotton, enhances salt stress tolerance and fungal disease resistance in transgenic tobacco by interacting with GZIRD21A and GZIPR5. New Phytol, 2009, 183: 62–75
[13] Jin L G, Li H, Liu J Y. Molecular characterization of three ethylene responsive element binding factor genes from cotton. J Integr Plant Biol, 2010, 52: 485–495
[14] Meng C, Cai C, Zhang T, Guo W Z. Characterization of six novel NAC genes and their responses to abiotic stresses in Gossypiumhirsutum L. Plant Sci, 2009, 176: 352–359
[15] Yan H, Jia H, Chen X, Hao L, An H, Guo X. The cotton WRKY transcription factor GhWRKY17 functions in drought and salt stress in transgenic Nicotiana benthamiana through ABA signaling and the modulation of reactive oxygen species production. Plant Cell Physiol, 2014, 55: 2060–2076
[16] Guo J, Shi G, Guo X, Zhang L, Xu W, Wang Y, Su Z. Transcriptome analysis reveals that distinct metabolic pathways operate in salt-tolerant and salt-sensitive upland cotton varieties subjected to salinity stress. Plant Sci, 2015, 238: 33–45
[17] Zhang X Y, Yao D X, Wang Q H, Xu W Y, Wei Q, Wang C C, Liu C L, Zhang C J, Yan H, Ling Y, Su Z, Li F G. mRNA-seq analysis of the Gossypium arboreum transcriptome reveals tissue selective signaling in response to water stress during seedling stage. PLoS One, 2013, 8: e54762
[18] Gouia H, Ghorbal M H, Touraine B. Effects of NaCl on flows of N and mineral ions and on NO3-reduction rate within whole plants of salt-sensitive bean and salt-tolerant cotton. Plant Physiol, 1994, 105: 1409–1418
[19] Peng Z, He S P, Gong W F, Sun J L, Pan Z E, Xu F F, Lu Y L, Du X M, Comprehensive analysis of differentially expressed genes and transcriptional regulation induced by salt stress in two contrasting cotton genotypes. BMC Genom, 2014, 15: 760
[20] Lam T W, Yiu S M, Kristiansen K, Wang J. SOAP2: an improved ultrafast tool for short read alignment. Bioinformatics, 2009, 25: 1966–1967
[21] Mortazavi A, Williams B A, McCue K, Schaeffer L, Wold B: Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods, 2008, 5: 621–628
[22] Audic S, Claverie J M. The significance of digital gene expression profiles. Genome Res, 1997, 7: 986–995
[23] Benjamini Y, Yekutieli D. The control of the false discovery rate in multiple testing under dependency. Ann Statist, 2001, 29: 1165–1188
[24] Chao D Y, Luo Y H, Shi M, Luo D, Ling H X. Salt-responsive genes in rice revealed by cDNA microarray analysis. Cell Res, 2005, 15: 796–810
[25] Guo P G, Baum M, Grando S, Ceccarelli S, Bai G H, Li R H, von Korff M, Varshney R K, Graner A, Valkoun J. Differentially expressed genes between drought-tolerant and drought-sensitive barley genotypes in response to drought stress during the reproductive stage. J Exp Bot, 2009, 60: 3531–3544
[26] 李田, 孙景宽, 刘京涛. 植物转录因子家族在耐盐抗旱调控网络中的作用. 生命科学, 2015, 27: 217–227 Li T, Sun J K, Liu J T. Role of different transcription factor families in the regulatory networks of drought and salinity tolerance in plants. Chin Bull Life Sci, 2015, 27: 217–227 (in Chinese with English abstract)
[27] 朱冬梅, 贾媛, 崔继哲, 付畅. 植物对盐胁迫应答的转录因子及其生物学特性. 生物技术通报, 2010, (4): 16–21 Zhu D M, Jia Y, Cui J Z, Fu C. Plant transcription factors in response to salt stress and its biological characteristics. Biotechnol Bull, 2010, (4): 16–21 (in Chinese with English abstract)
[28] 莫纪波, 李大勇, 张慧娟, 宋凤鸣. ERF转录因子在植物对生物和非生物胁迫反应中的作用. 植物生理学报, 2001, 47: 1145–1154 Mo J B, Li D Y, Zhang H J, Song F M. Roles of ERF transcription factors in biotic and abiotic stress response in plants. Plant Physiol J, 2011, 47: 1145–1154 (in Chinese with English abstract)
[29] Guo Z J, Chen X J, Wu X L, Lin J Q, Xu P. Overexpression of the AP2/EREBP transcription factor OPBP1 enhances disease resistance and salt tolerance in tobacco. Plant Mol Biol, 2004, 55: 607–618
[30] Zhang G, Chen M, Li L, Xu Z, Chen X, Guo J, Ma Y. Over-expression of the soybean GmERF3 gene, an AP2/ERF type transcription factor for increased tolerances to salt, drought and diseases in transgenic tobacco. J Exp Bot, 2009, 60: 3781–3796
[31] Qiao Z X, Huang B, Liu J Y. Molecular cloning and functional analysis of an ERF gene from cotton (Gossypium hirsutum). Biochim Biophys Acta, 2008, 1779: 122–127
[32] Puranik S, Sahu P P, Srivastava P S, Prasad M. NAC proteins: regulation and role in stress tolerance. Trends Plant Sci, 2012, 17: 369–381
[33] Huang G Q, Li W, Zhou W, Zhang J M, Li D D, Gong S Y, Li X B. Seven cotton genes encoding putative NAC domain proteins are preferentially expressed in roots and in responses to abiotic stress during root development. Plant Growth Regul, 2013, 71: 101–112.
[34] 李小兰, 胡玉鑫, 杨星, 于晓东, 李秋莉. 非生物胁迫相关 NAC转录因子的结构及功能. 植物生理学报, 2013, 49: 1009–1017 Li X L, Hu Y X, Yang X, Yu X D, Li Q L. Structure and functions of NAC transcription factors involved in abiotic stress. Plant Physiol J, 2013, 49: 1009–1017 (in Chinese with English abstract)
[35] 李蕾, 谢丙炎, 戴小枫, 杨宇红. WRKY转录因子及其在植物防御反应中的作用. 分子植物育种, 2005, 3: 401–408 Li L, Xie B Y, Dai X F, Yang Y H. WRKY transcription factors and their roles in plant defense responses. Mol Plant Breed, 2005, 3: 401–408 (in Chinese with English abstract)
[36] Shi W, Hao L, Li J, Liu D, Guo X, Li H. The Gossypium hirsutum WRKY gene GhWRKY39-1 promotes pathogen infection defense responses and mediates salt stress tolerance in transgenic Nicotiana benthamiana. Plant Cell Rep, 2014, 33: 483–498
[37] Shi W, Liu D, Hao L, Wu C A, Guo X, Li H. GhWRKY39, a member of the WRKY transcription factor family in cotton, has a positive role in disease resistance and salt stress tolerance. Plant Cell Tiss Organ Cul, 2014, 118: 17–32
[38] Chu X, Wang C, Chen X, Lu W, Li H, Wang X, Guo X. The cotton WRKY gene GhWRKY41 positively regulates salt and drought stress tolerance in transgenic Nicotiana benthamiana. PLoS One, 2015, 10: e0143022
[39] Choi H I, Hong J H, Ha J O, Kang J Y, Kim S Y. ABFs, a family of ABA-responsive element binding factors. J Biol Chem, 2000, 275: 1723–1730
[40] Bailey D, O’Hare P. Transmembrane bZIP transcription factors in ER stress signaling and the unfolded protein response. Antioxid Redox Signal, 2007, 9: 2305–2322
[41] Toledo-Ortiz G, Huq E, Quail P H. The Arabidopsis basic/helixloop-helix transcription factor family. Plant Cell, 2003, 15: 1749–1770
[42] Meng C M, Zhang T Z, Guo W Z. Molecular cloning and characterization of a novel Gossypium hirsutum L. bHLH gene in re-sponse to ABA and drought stresses. Plant Mol Biol Rep, 2009, 27: 381–387
[43] Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15: 573–581
[44] Loguercio L L, Zhang J Q, Wilkins T A. Differential regulation of six novel MYB-domain genes defines two distinct expression patterns in allotetraploid cotton (Gossypium hirsutum L.). Mol Gen Genet, 1999, 261: 660–671
[45] Suo J, Liang X, Pu L, Zhang Y, Xue Y. Identification of GhMYB109 encoding a R2R3 MYB transcription factor that expressed specifically in fiber initials and elongating fibers of cotton (Gossypium hirsutum L.). Biochim Biophys Acta, 2003, 1630: 25–34
[46] 王诺菡, 于霁雯, 嫚吴 , 马启峰, 李兴丽, 裴文锋, 李海晶, 黄双领, 张金发, 喻树迅. 棉花GhMYB0基因的克隆、表达分析及功能鉴定. 作物学报, 2014, 40: 1540–1548 Wang N H,Yu J W, Wu M, Ma Q F, Li X L, Pei W F, Li H J, Huang S L, Zhang J F, Yu S X. Cloning, expression, and functional analysis of GhMYB0 gene from cotton (Gossypium hirsumtum L.). Acta Agron Sin, 2014, 40: 1540–1548 (in Chinese with English abstract)
[47] 王雅琴, 石淼, 张新宇, 刘永昌, 薛飞, 孙杰, 李艳军. 棉花GhMYB113基因的克隆与表达分析. 西北植物学报, 2013, 33: 878–884 Wang Y Q, Shi M, Zhang X Y, Liu Y C, Xue F, Sun J, Li Y J. Cloning and expression analysis of GhMYB113 gene in Gossypium hirsutum. Acta Bot Boreali-Occident Sin, 2013, 33: 878–884 (in Chinese with English abstract)
[48] 于月华, 倪志勇, 梁小莉, 刘真芳, 陈全家, 高文伟. 棉花转录因子基因GhMYB的克隆及特征分析. 棉花学报, 2015, 27: 31–38 Yu Y H, Ni Z Y, Liang X L, Liu Z F, Chen Q J, Gao W W. Cloning and characterization of a transcription factor gene GhMYB from Gossypium hirsuturm L. Cotton Sci, 2015, 27: 31–38 (in Chinese with English abstract)
[49] 李菲, 柳展基, 王立国, 刘勤红, 刘任重. 棉花转录因子基因(GhMYB11)的克隆与表达分析. 农业生物技术学报, 2015, 23: 161–169 Li F, Liu Z J, Wang L G, Liu Q H, Liu R Z. Cloning and expression analysis of the transcription factor gene (GhMYB11) in Gossypium hirsutum L. J Agric Biotechnol, 2015, 23: 161–169 (in Chinese with English abstract)
[50] 丁震乾, 陈天子, 刘廷利, 刘小双, 张保龙, 周兴根. 棉花干旱诱导MYB类转录因子GhRAX3的功能分析. 中国农业科学, 2015, 18: 3569–3579 Ding Z Q, Chen T Z, Liu T L, Liu X S, Zhang B L, Zhou X G. Function analysis of a drought stress induced MYB transcription factor GhRAX3 in cotton. Sci Agric Sin, 2015, 48: 3569–3579 (in Chinese with English abstract)
Transcriptome Analysis of Transcription Factors in Upland Cotton Seedlings under NaCl Stress
PENG Zhen1,2, HE Shou-Pu1, GONG Wen-Fang1, PAN Zhao-E1, JIA Yin-Hua1, LU Yan-Li2,*, and DU Xiong-Ming1,*
1Institute of Cotton Research, Chinese Academy of Agricultural Sciences / State Key Laboratory of Cotton Biology, Anyang 455000, China;2Maize Research Institute, Sichuan Agricultural University, Wenjiang 611130, China
A comparative transcriptomics was used to analyze the expression changes of transcription factors and their families in leaves of the two cotton (Gossypium hirsutum L.) varieties (Earlistaple 7 with salt-tolerance and Nandanbadidahua with salt-sensitivity) under 200 mmol L–1NaCl stress for 4 and 24 hours. We have identified and quantified 2815 transcription factors from 54 transcription factor families of the samples. A total of 249 differentially expressed transcription factor genes of ‘Earlistaple 7’ and 261 transcription factor genes of ‘Nandanbadidahua’ were identified, after 4 hours of NaCl stress. With prolonging stress time (24 hours), the number of differentially expressed transcription factors in two cotton varieties responsive to salt was increased. It was found that the 106 (4 hours) and 184 (24 hours) transcription factor genes were specially expressed in salt-tolerant variety, and the 143 (4 hours) and 282 (24 hours) were exhibited commonly in both cotton lines. We have obtained 124 salt-tolerance-related transcription factor genes from 26 transcription factor families through screening, and further validated the data accuracy by quantitative Real-time PCR. Total 11 highly expressed transcription factors genes by salt induction, including CotAD_66280 (HD-ZIP), CotAD_47058 (ERF), CotAD_18472 (G2-like), CotAD_04289 (C2H2), CotAD_57763 (HSF), CotAD_23656 (TCP), CotAD_02221 (ERF), CotAD_23975 (WRKY), CotAD_54036 (MYB), CotAD_27788 (NAC), and CotAD_23815 (HSF) were induced to be closely related to salt tolerance in upland cotton, and might be used as a source of candidate tolerant genes for upland cotton breeding.



10.3724/SP.J.1006.2017.00354
本研究由“十二五”国家科技支撑计划项目(2013BAD01B03)和国家自然科学基金项目(31301365)资助。
This study was supported by the National Key Technology Support Program of China (2013BAD01B03) and the National Natural Science Foundation of China (31301365).
*通讯作者(Corresponding authors): 杜雄明, E-mail: dxm630723@163.com; 卢艳丽, E-mail: yanli.lu82@hotmail.com
联系方式: E-mail: cripengzhen09@126.com
稿日期): 2016-05-12; Accepted(接受日期): 2016-09-18; Published online(
日期): 2016-09-28.
URL: http://www.cnki.net/kcms/detail/11.1809.S.20160928.0948.008.html

