大熊猫嗅觉受体基因家族的生物信息学分析
简佐义, 李午佼, 张修月, 岳碧松, 范振鑫
(四川大学生命科学学院,成都610064)
大熊猫嗅觉受体基因家族的生物信息学分析
简佐义, 李午佼, 张修月, 岳碧松, 范振鑫*
(四川大学生命科学学院,成都610064)
嗅觉受体(OR)在嗅觉系统识别气味的过程中起关键作用,哺乳动物OR蛋白由其最大的多基因家族编码。本研究深入分析了大熊猫Ailuropodamelanoleuca的OR基因家族,结果显示大熊猫有1 048个OR基因,包括645个完整基因、219个假基因和184个片段基因。大熊猫的OR基因根据序列相似性可划分为22个家族、240个亚家族,平均每个亚家族的成员不足5个,表明大熊猫的OR基因家族具有丰富的序列多样性。基于大熊猫、北极熊Ursusmaritimus、家犬Canislupusfamiliaris和小鼠Musmusculus的OR蛋白序列的多物种聚类分析显示它们具有267个直系同源基因簇,而大熊猫没有特有的OR基因,表明其嗅觉特异性不明显。大熊猫OR基因的数量明显比其近亲北极熊的少,显示其在进化过程中丢失了大量OR基因,这可能与其食性的显著改变有关。
大熊猫;嗅觉受体;基因家族;生物信息学
嗅觉受体(olfactory receptor,OR)是一类能够与气味分子特异性结合的G蛋白偶联受体(G protein-coupled receptor,GPCR),通常含有310~340个氨基酸且具有7个跨膜结构域,在动物的嗅觉系统中起关键作用(Buck & Axel,1991;Freitagetal.,1998;Hasin-Brumshteinetal.,2009;Niimura,2012)。OR基因家族是哺乳动物最大的多基因家族,能编码多种OR蛋白(Hughesetal.,2014)。哺乳动物的OR基因通常表现出单外显子特征,因其编码区没有内含子,但在编码区上游存在不翻译的外显子,因此不管在表达过程中被剪接成多少种转录本,翻译后都会得到同样的蛋白(Youngetal.,2003)。研究表明,严重假基因化是OR基因家族的显著特征,如人类有51.8%的OR基因是假基因(Niimura,2012)。多种因素可能导致OR基因的假基因化:移码突变、无义突变等破坏了原来的编码区;部分保守位点发生删除导致蛋白功能的失活;单核苷酸多态性可能改变某个关键位点的氨基酸,造成OR与特定气味分子的结合能力丧失;启动子遭破坏,导致基因失活等(Laietal.,2008)。
气味感知是一个复杂的过程:气味分子与嗅神经元(olfactory sensory neuron,OSN)表面的OR蛋白特异性结合,使细胞膜去极化并在膜内产生第二信使,从而启动级联的酶促反应,开启嗅觉系统识别通路(Sicard & Holley,1984)。OSN是一类高度专业的化学感受器细胞,特定气味分子刺激一组OSN细胞,部分细胞会产生特异性反应(Shouetal.,2005)。研究发现,一种OR能够识别多种结构相似的气味分子,而一种气味分子也能够被多种OR识别,这一机制被称为“组合编码”(Mombaerts,2004)。
大熊猫Ailuropodamelanoleuca是世界生物多样性保护的旗舰物种,偏爱食竹,其99%的食物为竹类,所食竹类达50余种(胡锦矗,1995)。进入发情期的大熊猫存在交互模态信号通讯现象,即借助化学通讯信号做出对异性声音信号刺激的行为反应(徐蒙等,2011)。大熊猫将肛周腺分泌物或尿液涂抹在乔木树干上形成嗅味标记,而嗅味标记集中的地方通常是它们的发情场(刘国琪等,2005)。采食时大熊猫主要依靠嗅觉判定食物的性质,在夜间更是如此(汤纯香,1992)。Li等(2010)从大熊猫基因组中鉴定出659个OR基因,但没有深入分析。本研究全面分析了大熊猫的OR基因家族,并与其近亲北极熊Ursusmaritimus进行比较,以期深化对熊科Ursidae这一基因家族的认识。本研究能够为后续进一步研究大熊猫的嗅觉系统提供基础信息。
1 材料与方法
1.1 数据
大熊猫全基因组序列(ailMel1,BGI-Shenzhen AilMel 1.0 Dec. 2009)下载自UCSC网站(http://genome.ucsc.edu/);北极熊全基因组序列(Accession ID:PRJNA210951)下载自EMBL-EBI网站(http://www.ebi.ac.uk/)。从UniProt数据库(http://www.uniprot.org/)下载获得1 210个经鉴定的OR蛋白序列。从GenBank数据库(https://www.ncbi.nlm.nih.gov/genbank/)下载获得9个与OR同属GPCR超家族的非OR蛋白序列,登录号分别是:NP_000671、NP_000675、NP_000667、NP_071640、NP_000854、NP_000857、NP_000862、NP_001471和NP_001043。
1.2 OR基因的鉴定
orfam(https://github.com/jianzuoyi/orfam)是本实验室开发的哺乳动物OR基因自动注释程序,大熊猫和北极熊OR基因的鉴定由orfam完成。主要步骤如下:首先进行数据预处理,用cd-hit(Fuetal.,2012)对下载获得的1 210个OR蛋白序列聚类分析,序列一致性大于95%的蛋白只保留1条,结果得到608条非冗余的已知OR蛋白序列集;然后为orfam提供3个FASTA格式的序列文件:上一步得到的608条非冗余的OR蛋白序列集、下载获得的9个非OR蛋白序列以及大熊猫基因组序列,由此鉴定出大熊猫的OR基因。orfam鉴定得到的OR基因分为3类:完整基因 (Intact)、片段基因(Truncated)和假基因(Pseudo)。Intact是具有完整编码序列,有编码功能蛋白潜力的基因;Truncated则是完整OR基因的一部分,其缺失C端或N端,甚至C、N两端都缺失;Pseudo是指在进化过程中发生突变从而丧失了编码功能蛋白能力的基因,在编码序列上表现为含有提前的终止密码子、移码突变以及在蛋白的保守区域发生了插入或删除等。值得注意的是,Truncated可能是由于基因组测序深度不够造成的,这类基因在未来有更加完善的基因组版本可用时可能被鉴定成完整的OR基因,因此计算假基因的比例时,将Truncated当作Intact对待。
1.3 系统进化树的构建
大熊猫完整的OR基因翻译获得的蛋白序列与下载获得的作为外群的9个非OR蛋白序列合并成1个FASTA文件,经MAFFT工具包(Katohetal.,2005)中的E-INS-i进行多序列比对,结果由MEGACC(Kumaretal.,2012)构建系统进化树:采用邻接法(neighbor-joining),bootstrap值为1 000。
1.4 单拷贝OR基因和特有OR基因的鉴定
利用OrthoMCL(Lietal.,2003)对大熊猫、北极熊、小鼠Musmusculus和家犬Canislupusfamiliaris的完整OR基因翻译的蛋白序列进行聚类,序列相似性大于60%的蛋白被聚集成1个簇。若某一簇中4个物种分别只有1个OR,则该簇的基因被认为是单拷贝基因;若某一簇仅含有大熊猫的OR基因,则认为是大熊猫特有的OR基因。
2 结果与分析
2.1 大熊猫OR基因家族的组成
大熊猫OR基因家族共有1 048个成员,其中,完整基因645个、片段基因184个、假基因219个(表1)。大熊猫有20.9%的OR基因发生了假基因化,这些基因在进化过程中丧失了编码功能蛋白的能力,是一种进化的遗迹。但有研究表明(Zhangetal.,2007),部分OR假基因依然具有表达活性,其产物可能参与了功能OR基因的表达调控。比较发现,大熊猫OR基因家族成员的数量及假基因的比例与家犬的相近(Quignonetal.,2005)。大熊猫绝大部分基因编码310~330个氨基酸(图1:左)。大熊猫OR基因的总数和完整OR基因的数量都明显比北极熊的少,表明大熊猫在进化过程中丢失了大量OR基因,这可能导致它们嗅觉严重退化。
OR基因通常成簇地分布在染色体上。分析大熊猫OR基因在基因组中的分布情况,显示绝大多数scaffold上都只有不足5个OR基因(图1:右),这是由于测序深度的限制,大熊猫基因组没有拼装到染色体,OR基因分散在大量scaffold上,未体现出成簇分布的规律。

表1 大熊猫和北极熊嗅觉受体基因的数量
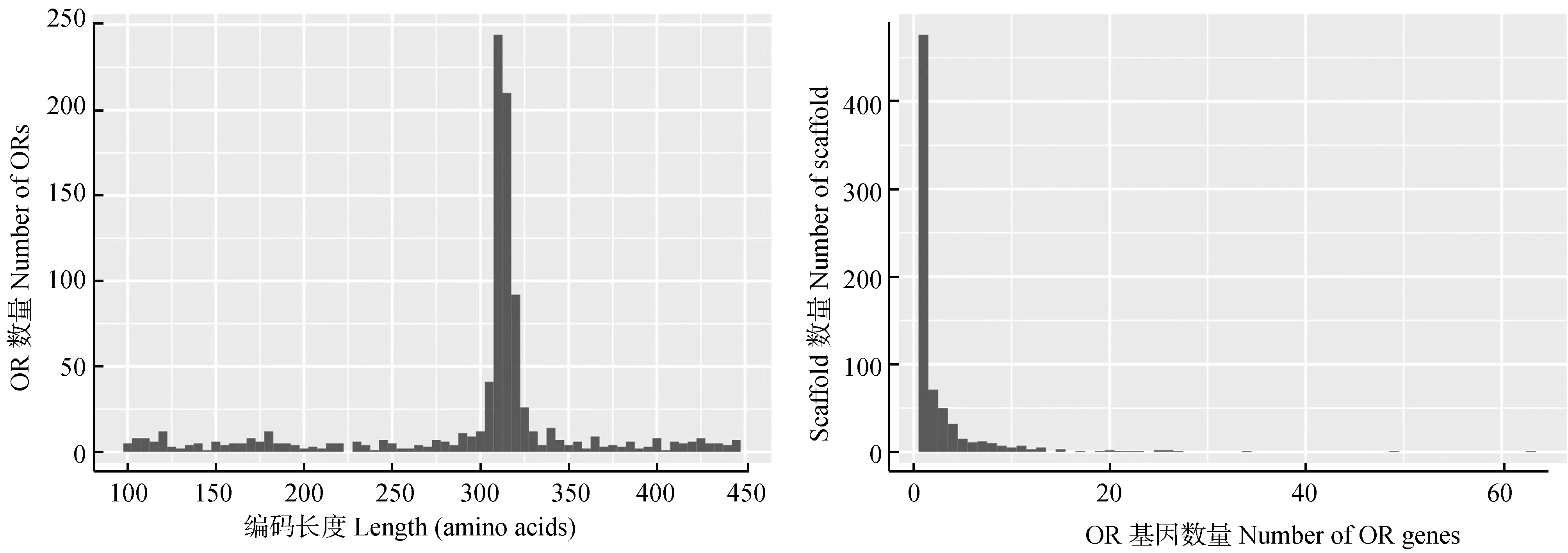
图1 大熊猫嗅觉受体蛋白序列长度分布(左)和scaffold上嗅觉受体基因数量(右)
2.2 大熊猫OR基因家族和亚家族的分类
哺乳动物有2类OR基因:Class Ⅰ和Class Ⅱ,它们在系统进化树上通常聚为2个独立的分支,这2类基因可能具有不同的起源(Niimura,2012)。水生动物基因组中通常只存在Class Ⅰ类基因,而陆生动物基因组中2类基因都存在,并且Class Ⅰ类基因大部分具有特定的功能。基于邻接法构建的系统进化树中,蓝色分支是外群基因,红色分支和黑色分支是2个清晰的独立分支,分别是Class Ⅰ和Class Ⅱ(图2)。从数量上看,Class Ⅱ类基因明显多于Class Ⅰ类,这一现象存在于绝大多数哺乳动物中。2类OR基因可根据序列相似性继续划分为家族和亚家族,参考Glusman等(2000)的方法:序列相似性大于40%或60%的蛋白被划分为家族或亚家族。结果显示,大熊猫OR基因可划分为22个家族、240个亚家族,平均每个亚家族的基因数不足5个,说明大熊猫OR基因家族具有丰富的序列多样性。
基因家族由基因重复和趋异形成。为进一步探索大熊猫OR基因家族的趋异程度,将所有完整的OR基因和假基因翻译的蛋白序列进行两两比对,结果显示大熊猫OR蛋白之间的序列相似性为19.5%~100%,表明大熊猫OR基因家族的部分成员趋异程度非常大,仅根据序列相似性已经不能判断它们是否同源。此外有2个完整的OR蛋白具有相同的序列,有6个OR假基因翻译的蛋白分别具有1个相同的拷贝,这是OR基因家族通过基因重复进行扩张的一种表现,然而不排除这些相同拷贝是由于基因组拼装错误造成的。
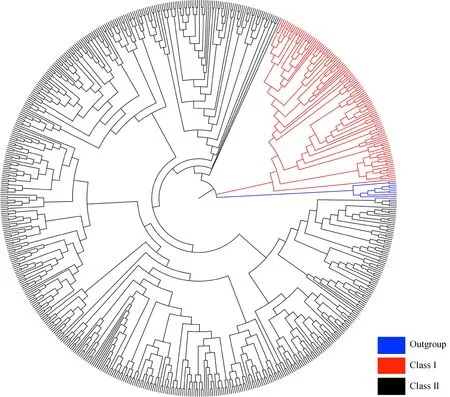

图2 645个大熊猫嗅觉受体蛋白构建的系统进化树
2.3 大熊猫OR基因家族的气味识别潜能
图3显示:大熊猫、北极熊、家犬和小鼠的3 779个功能OR蛋白由OrthoMCL聚成590个簇;大熊猫没有特有的OR基因,北极熊、家犬和小鼠特有的OR基因簇分别为9个、1个和46个;除小鼠外,另外3个物种的OR基因家族的特异性并不明显,它们都只保留了从祖先继承而来的基础嗅觉遗传信息,在进化过程中,它们的OR基因家族并没有显著地增加基因序列的多样性;小鼠拥有较多特异的OR基因,可能由于昼伏夜出,其生存严重依赖于嗅觉。4个物种具有267个直系同源OR基因簇,这些同源基因在进化过程中相对保守,它们可能负责一些常见气味的识别,以维持动物基本的嗅觉能力。

图3 维恩图显示4种哺乳动物之间的直系同源基因簇
选择大熊猫的单拷贝OR基因来研究其嗅觉潜能。单拷贝基因通常是管家基因(house-keeping genes),它们的表达维持着生物体的基本功能。通过研究单拷贝OR基因所编码蛋白的气味识别潜能,能够阐明对哪些气味的识别能力是动物共有的。为此,从OrthoMCL的聚类结果中获得4种哺乳动物的54个单拷贝OR基因簇,并提取出大熊猫的单拷贝OR基因。根据Nguyen等(2012)的方法鉴定这些基因表达的蛋白的气味识别潜能,该方法的基本依据是:序列相似性达到60%以上的OR蛋白通常具有相似的气味识别潜能。表2显示,大熊猫的单拷贝OR基因参与了环境中常见气味的识别,如花香、木香、柠檬味、甜味、脂肪味以及其他一些刺激性气味,表明哺乳动物对这些气味的识别是它们基本的生存本领。此外,大熊猫3个单拷贝OR基因(AmOR247、AmOR84、AmOR360)表达的蛋白都与小鼠识别花香和木香潜能的OL151_MOUSE蛋白具有较高的序列相似性。大熊猫对木香的识别较为敏感可能与它们的食性有关,它们习惯于啃食坚硬的木材,而对柔软的食物比较排斥。
3 讨论
长期以来,人们对大熊猫嗅觉的了解仅停留在观察记录层面,没有从遗传进化的角度去深入了解它们的嗅觉系统。OR在动物的嗅觉系统中发挥关键作用,本研究采用生物信息学方法对大熊猫的OR基因家族进行了深入分析,以期深化对其嗅觉系统的认识。结果显示,大熊猫在进化过程中丢失了大量OR基因,其OR基因总数和功能OR基因数量都明显少于北极熊,它们的嗅觉相对于其近亲北极熊来说可能发生了严重退化。基于序列相似性的聚类分析表明,大熊猫没有特有的OR基因,显示其可能并不具有特殊的嗅觉能力,仅保留了来自祖先的部分嗅觉。
比较大熊猫和北极熊的OR基因家族,发现两者的嗅觉能力与各自生存对嗅觉的依赖程度有关。大熊猫虽然属食肉目Carnivora动物,但它们现在99%的食物为竹类(胡锦矗,1995),单一的食物结构使大熊猫在觅食时对嗅觉的依赖大为降低。北极熊生活在极地,依靠捕食其他动物为生,其猎物常生活在水下或被冰雪覆盖,这就要求它们拥有灵敏的嗅觉来判定猎物的位置。嗅觉能力一方面取决于OR基因的数量,另一方面取决于OR基因的序列多样性。如家犬的OR基因数量在哺乳动物中并不占优势,但它们却拥有敏锐的嗅觉,是因其OR基因丰富的序列多样性弥补了数量的不足(Niimuraetal.,2014)。因此,尽管大熊猫在进化过程中丢失了大量OR基因,但其仍然拥有一个庞大的OR基因家族,并且其OR基因的序列多样性也非常丰富,这显示大熊猫可能依然具有强大的嗅觉能力。事实上,嗅觉是大熊猫重要的感觉之一,因为它们昼夜活动,并且夜间活动比白天多,而夜间视力不能充分发挥,敏锐的嗅觉对于它们来说极为重要。大熊猫还通过嗅觉标记来回应求偶信号(徐蒙等,2011),通过嗅觉来判断食物的性质(汤纯香,1992;胡锦矗,1995)等。
本研究使用的大熊猫基因组序列为2.25 Gb,约覆盖其全基因组的94%(Lietal.,2010),相信本研究较全面地鉴定出了大熊猫的OR基因,当然由于现有序列中还存在0.05 Gb的间隙以及基因组的拼接质量问题,部分OR基因也可能没有被鉴定出来。OR基因家族是脊椎动物最大的多基因家族,它们通常成簇地分布在染色体上,由于大熊猫的基因组只拼接到scaffold水平,因而没能分析其OR基因在染色体上的分布情况。未来如果有更加完善的大熊猫基因组版本可用,对这一基因家族的分析将更加全面。由于目前已知气味识别潜能的OR蛋白数量较少,本研究只搜集到22个,因此只能确定部分大熊猫OR基因的气味识别潜能。随着越来越多物种的基因组被测序并公开,多物种OR基因家族的比较分析将为深入了解这一重要家族提供新的视角。本研究获得了大熊猫OR基因家族的基础信息,将有助于进一步研究这一濒危物种的嗅觉系统。
胡锦矗. 1995. 大熊猫的摄食行为[J]. 生物学通报, 30(9): 14-18.
刘国琪, 王昊, 尹玉峰. 2005. 王朗自然保护区中大熊猫发情场的嗅味树和嗅味标记调查[J]. 生物多样性, 13(5): 445-450.
汤纯香. 1992. 大熊猫采食行为的研究[J]. 动物学杂志, 27(4): 46-49.
徐蒙, 王智鹏, 刘定震, 等. 2011. 发情期大熊猫(Ailuropodamelanoleuca)交互模态信号通讯[J]. 科学通报, 56(36): 3073-3077.
Buck L, Axel R. 1991. A novel multigene family may encode odorant receptors: a molecular basis for odor recognition[J]. Cell, 65(1): 175-187.
Freitag J, Ludwig G, Andreini I,etal. 1998. Olfactory receptors in aquatic and terrestrial vertebrates[J]. Journal of Comparative Physiology, 183(5): 635-650.
Fu L, Niu B, Zhu Z,etal. 2012. CD-HIT: accelerated for clustering the next-generation sequencing data[J]. Bioinformatics, 28(23): 3150-3152.
Glusman G, Bahar A, Sharon D,etal. 2000. The olfactory receptor gene superfamily: data mining, classification, and nomenclature[J]. Mammalian Genome, 11(11): 1016-1023.
Hasin-Brumshtein Y, Lancet D, Olender T. 2009. Human olfaction: from genomic variation to phenotypic diversity[J]. Trends in Genetics, 25(25): 178-184.
Hughes GM, Teeling EC, Higgins DG. 2014. Loss of olfactory receptor function in hominin evolution[J]. PLoS ONE, 9(1): e84714. DOI:10.1371/journal.pone.0084714.
Katoh K, Kuma K, Toh H,etal. 2005. MAFFT version 5: improvement in accuracy of multiple sequence alignment[J]. Nucleic Acids Research, 33(2): 511-518.
Kumar S, Stecher G, Peterson D,etal. 2012. MEGA-CC: computing core of molecular evolutionary genetics analysis program for automated and iterative data analysis[J]. Bioinformatics, 28(20): 2685-2686.
Lai PC, Bahl G, Gremigni M,etal. 2008. An olfactory receptor pseudogene whose function emerged in humans: a case study in the evolution of structure-function in GPCRs[J]. Journal of Structural and Functional Genomics, 9(1): 29-40.
Li L, Stoeckert CJ, Roos DS. 2003. OrthoMCL: identification of ortholog groups for eukaryotic genomes[J]. Genome Research, 13(9): 2178-2189.
Li R, Fan W, Tian G,etal. 2010. The sequence anddenovoassembly of the giant panda genome[J]. Nature, 463(7284): 1106.
Mombaerts P. 2004. Genes and ligands for odorant, vomeronasal and taste receptors[J]. Nature Reviews Neuroscience, 5(4): 263-278.
Nguyen DT, Lee K, Choi H,etal. 2012. The complete swine olfactory subgenome: expansion of the olfactory gene repertoire in the pig genome[J]. BMC Genomics, 13(1): 584.
Niimura Y. 2012. Olfactory receptor multigene family in vertebrates: from the viewpoint of evolutionary genomics[J]. Current Genomics, 13(13): 103-114.
Niimura Y, Matsui A, Touhara K. 2014. Extreme expansion of the olfactory receptor gene repertoire in African elephants and evolutionary dynamics of orthologous gene groups in 13 placental mammals[J]. Genome Research, 24(9): 91-92.
Quignon P, Giraud M, Rimbault M,etal. 2005. The dog and rat olfactory receptor repertoires[J]. Genome Biology, 6(10): R83.
Shou S, Miyamichi K, Sakano H. 2005. One neuron-one receptor rule in the mouse olfactory system[J]. Trends in Genetics, 20(12): 648-653.
Sicard G, Holley A. 1984. Receptor cell responses to odorants: similarities and differences among odorants[J]. Brain Research, 292(2): 283-296.
Young JM, Shykind BM, Lane RP,etal. 2003. Odorant receptor expressed sequence tags demonstrate olfactory expression of over 400 genes, extensive alternate splicing and unequal expression levels[J]. Genome Biology, 4(11): R71.
Zhang X, De la Cruz O, Pinto JM,etal. 2007. Characterizing the expression of the human olfactory receptor gene family using a novel DNA microarray[J]. Genome Biology, 8(5): R86.
Bioinformatics Analysis of the Giant Panda Olfactory Receptor Gene Family
JIAN Zuoyi, LI Wujiao, ZHANG Xiuyue, YUE Bisong, FAN Zhenxin*
(College of Life Sciences, Sichuan University, Chengdu 610064, China)
Olfactory receptors (ORs) play a critical role in recognizing odorant molecules in the olfactory sensory system. OR proteins are encoded by the largest multigene family in the mammalian genome. In this study, the OR gene family of giant panda (Ailuropodamelanoleuca) was analyzed in depth. The results revealed that the giant panda had 1 048 OR genes, including 645 intact OR genes, 219 pseudogenes and 184 truncated OR genes. The OR genes of giant panda could be classified into 22 families and 240 subfamilies according to the protein sequence similarity. The average number of OR genes in each subfamily was less than 5, and this indicated that the OR gene family of giant panda had high sequence diversity. Multispecies OR gene clustering analysis was performed with OR protein sequences fromA.melanoleuca, polar bears (Ursusmaritimus), dogs (Canislupusfamiliaris) and mice (Musmusculus), the results showed that these species shared 267 orthologous OR gene clusters. No specific OR genes were identified in giant panda, indicating that the giant panda had no special olfactory capacity. Moreover, we found that the polar bear was a close relative of the giant panda, and the number of OR genes from giant panda was significantly less than that of polar bear, demonstrating that the giant panda had lost a great number of OR genes during evolution, which may be caused by the great change in feeding habits.
Ailuropodamelanoleuca; olfactory receptor; gene family; bioinformatics
2016-05-13 接受日期:2016-10-31
简佐义, 男, 主要研究方向为基因组学, E-mail:jianzuoyi@qq.com
*通信作者Corresponding author, E-mail:zxfan@scu.edu.cn
10.11984/j.issn.1000-7083.201600124
Q75; Q959.8
A
1000-7083(2017)01-0001-06

