小麦相关功能基因的全基因组电子定位及基因注释
王长彪,韩 斌,刘 江,崔 婷,任永康,赵兴华,董艳辉,李亚丽,唐朝晖
(1.山西省农业科学院生物技术研究中心,山西太原030031;2.山西大学生物工程学院,山西太原030006;3.山西省农业科学院作物科学研究所,山西太原030031)
小麦相关功能基因的全基因组电子定位及基因注释
王长彪1,韩 斌2,刘 江1,崔 婷2,任永康3,赵兴华1,董艳辉1,李亚丽1,唐朝晖1
(1.山西省农业科学院生物技术研究中心,山西太原030031;2.山西大学生物工程学院,山西太原030006;3.山西省农业科学院作物科学研究所,山西太原030031)
电子克隆技术是通过对基因组或EST序列的组装和拼接快速获得功能基因的方法。运用电子克隆技术,以小麦功能标记为探针,在小麦全基因组数据库中克隆与之相对应的小麦功能基因,并对其进行生物信息学分析。结果表明,运用UMN19,bx7_f_r,ZSBy9F2_R2,PPO33,Sus2_SNP_227_589L2,Pin_b_1,YP7A,cssfr7这些功能标记,可以克隆出与之相对应的功能基因。研究有助于下一步对小麦相关功能基因进行精细作图、图位克隆及分子标记辅助育种等工作。
生物信息学;小麦;功能基因;电子克隆
小麦是世界上最重要的农作物之一。过去几十年中,运用SSR,RFLP,AFLP,RAPD和DArT等分子标记技术对小麦重要基因进行了染色体区域的分子定位,然而这些遗传标记通常与目标基因具有一定的距离并且受群体的影响较大。因此,小麦功能基因的挖掘和克隆受到很大的限制[1]。
2014年《Science》杂志上,2个研究组分别公布了中国春小麦品种的每条染色体臂的基因组序列[2]以及3B[3]染色体基因组序列,另外2个研究小组[4-5]整理了现代普通小麦的系统发育历史,这些成果将为加速小麦功能基因的研究提供有力的基因资源。同时,小麦纹枯病[6]、白粉病[7-8]、叶锈病[8]、叶枯病[8]、赤霉病[9]、禾谷孢囊线虫病[10]和条锈病[11]等病害已经经过田间鉴定筛选,为进行基因克隆奠定了有力的资源基础。
电子克隆技术是继图位克隆、转座子标签法克隆、mRNA差异显示技术、抑制差减杂交技术和基因表达系列分析技术后发展起来的基因克隆新方法[5]。它依据丰富的基因组和EST遗传资源[12],借助日新月异的生物信息学技术,依托电子计算机强大的云计算能力,通过对基因组或EST序列的组装和拼接快速获得功能基因[13]。随着作物基因组计划的逐步推进,该方法具有成本低、速度快、针对性强的特点,其优势将日益凸显。到目前为止,大约有97个与性状相关的小麦功能标记被开发出来。
本研究运用电子克隆技术,以小麦功能标记为探针,在小麦全基因组数据库中克隆与之相对应的小麦功能基因,并对其进行生物信息学分析,为小麦功能基因的进一步研究奠定了基础。
1 材料和方法
1.1 材料
本试验中小麦全基因组序列从URGI(https:// urgi.versailles.inra.fr/download/iwgsc/)以FASTA的格式下载,小麦功能分子标记来源于文献[1]。
1.2 方法
1.2.1 小麦功能基因的定位 在Linux平台上,采用re-PCR策略以97个功能标记为引物将其定位到小麦全基因组序列中,并对定位结果进行统计。使用Fgenesh程序对能与功能标记联配的序列进行功能基因的预测。
1.2.2 小麦功能基因的序列分析 使用blast2go软件对获得的功能基因进行基因的GO[14]注释及基于KEGG[15]的生物通路富集分析。
2 结果与分析
2.1 小麦功能基因的序列分析
2.1.1 功能基因的GO注释 大量全基因组数据的增多,使得关于基因、基因产物以及生物学通路的数据也越来越多。利用计算机程序提供的结构化标准生物学模型,在基因组范围内描述蛋白质功能,成为从整体水平系统研究基因及其产物的系统方法。GO注释系统包含生物学过程、分子功能和细胞组成3个分支[16]。
本研究对功能基因所做的GO注释结果中,涉及生物学过程的功能基因有18个。生物学过程二级水平中包含的6种反应类型有单器官进程(single-organismprocess)、应激反应(response tostimulus)、定位(localization)、多器官进程(multi-organism process)、细胞进程(cellular process)及代谢进程(metabolic process)(表1)。其中,参与单器官进程的功能基因有1种,应激反应4种,定位1种,多器官进程2种,细胞进程10种,代谢进程15种(图1)。
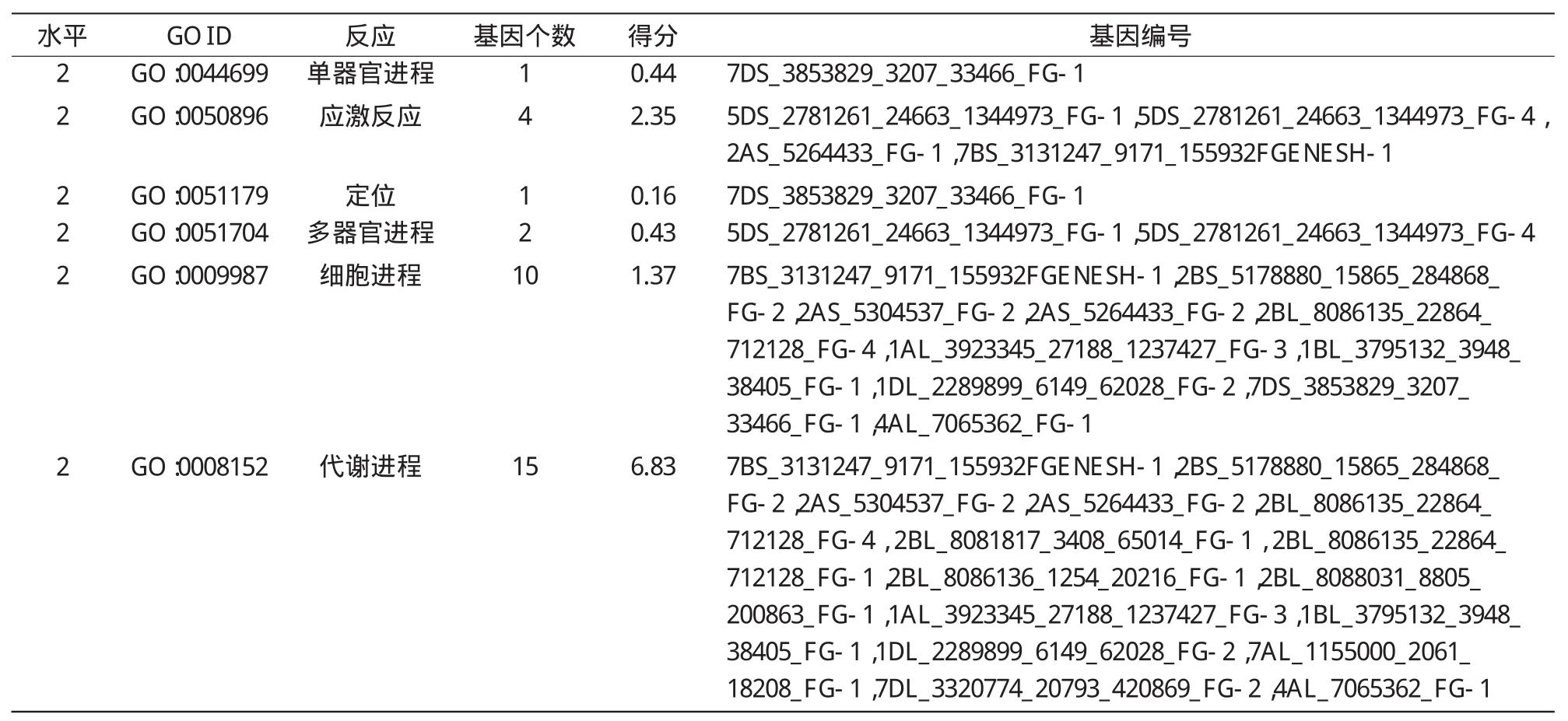
表1 小麦功能基因生物学过程二级水平的GO注释
涉及分子功能的功能基因有22个(表2)。分子功能二级水平包含5种类型:营养库活性类(nutrientreservoiractivity)功能基因3个,结合类(binding)功能基因12个,酶活性调节类(enzymeregulator activity)功能基因1个,转运活性类(transporter activity)功能基因1个,催化活性类(catalytic activity)功能基因15个(图2)。
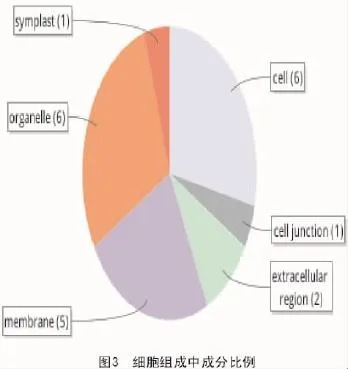
与细胞组成相关的功能基因共10个(表3)。细胞组分二级水平包含6种类型(表3):细胞器(organelle)功能基因6个,共质体(symplast)功能基因1个,细胞联结(cell junction)功能基因1个,细胞(cell)功能基因6个,胞外区(extracellular region)功能基因2个,膜(membrane)功能基因5个(图3)。
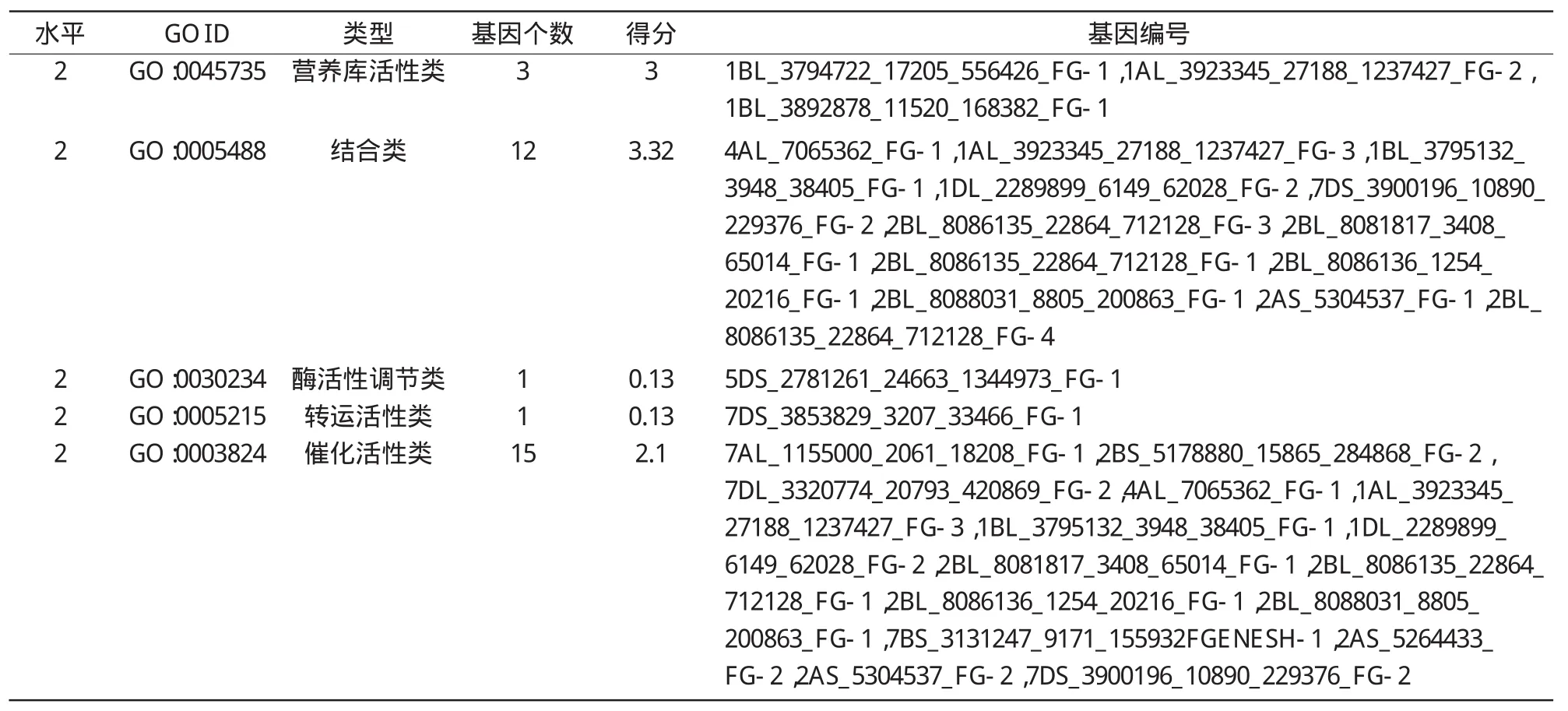
表2 小麦功能基因分子功能二级水平的GO注释


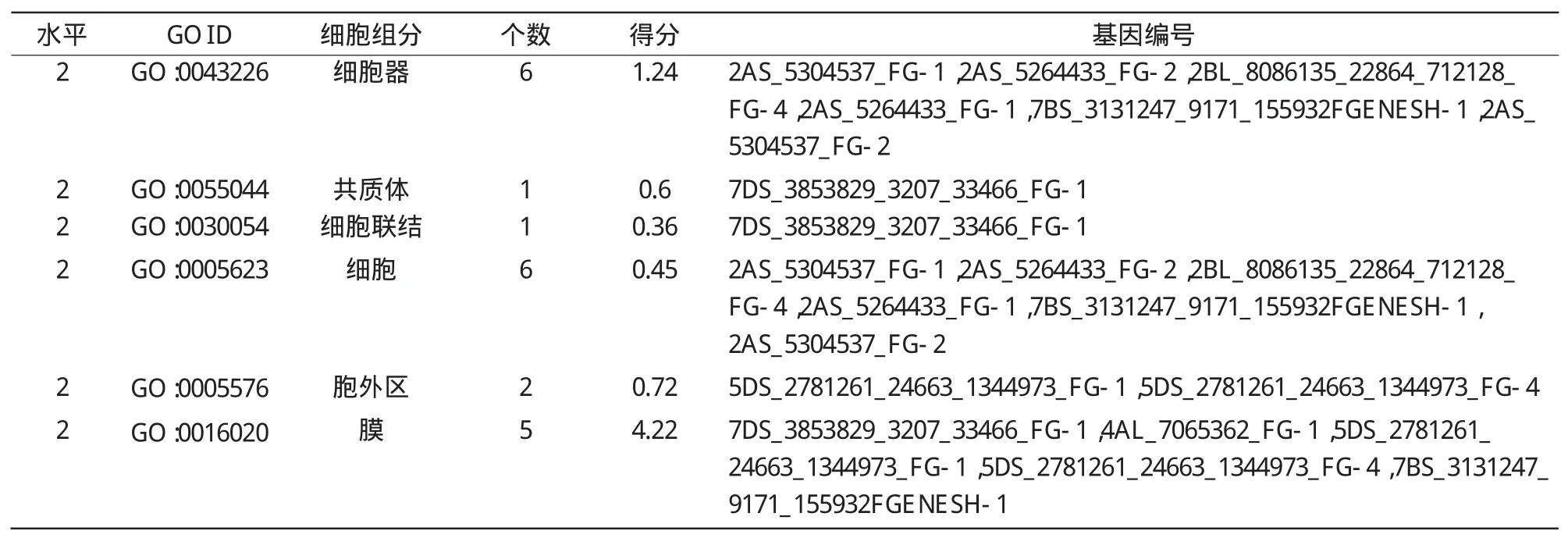
表3 小麦功能基因细胞组成二级水平的GO注释
2.1.2 基于KEGG的生物通路富集分析 运用KEGG数据库对获得的功能基因进行生物通路富集分析[17-18],共有20个功能基因编码的12种酶涉及到11个生物化学途径:萜类和类固醇的生物合成,丙酮酸代谢,糖酵解途径,类胡萝卜素的生物合成,抗生素生物合成,酪氨酸代谢,淀粉和蔗糖代谢,异喹啉类生物碱的生物合成,柠檬酸循环(TCA循环),嘌呤代谢,硫胺素代谢。这些酶包括合酶、乙酰转移脱氢酶、氧化酶、腺嘌呤焦磷酸酶、磷酸酶(表4)。

表4 小麦功能基因基于KEGG的生物通路富集分析
2.2 小麦功能基因的定位
将97个小麦功能标记与小麦全基因组序列库进行电子定位和功能注释后,得到与小麦功能标记对应的小麦功能基因及功能预测(表5)。将所得基因的功能与之相应的小麦功能标记的功能进行比对,结果表明,运用UMN19,bx7_f_r,ZSBy9F2_R2,PPO33,Sus2_SNP_227_589L2,Pin_b_1,YP7A,cssfr7这些功能标记可以克隆出其相应的功能基因;运用其余的功能标记所克隆出的功能基因与之在功能上有一定差异。
3 结论
随着小麦基因组序列测序的完成,越来越多的基因组序列信息能够帮助人们了解基因结构及其功能[19]。电子克隆速度快,成本低,适宜小实验室操作的优点可以使研究者将更多的精力和资金投入到后续基因功能的研究中去。运用生物信息学技术进行小麦功能基因的电子克隆将起到不可替代的作用。它将大大加速小麦基因结构、功能的研究进程,同时它也会推动比较基因组学的发展以及小麦起源、进化和发育方面的研究。
本研究利用电子克隆技术成功克隆到小麦的功能基因,证明该技术的有效性[20]。然而,电子克隆也存在一定的缺点,因种内多态性的存在,电子克隆所得序列与真实的序列可能存在差异,其准确性有待进一步的验证。
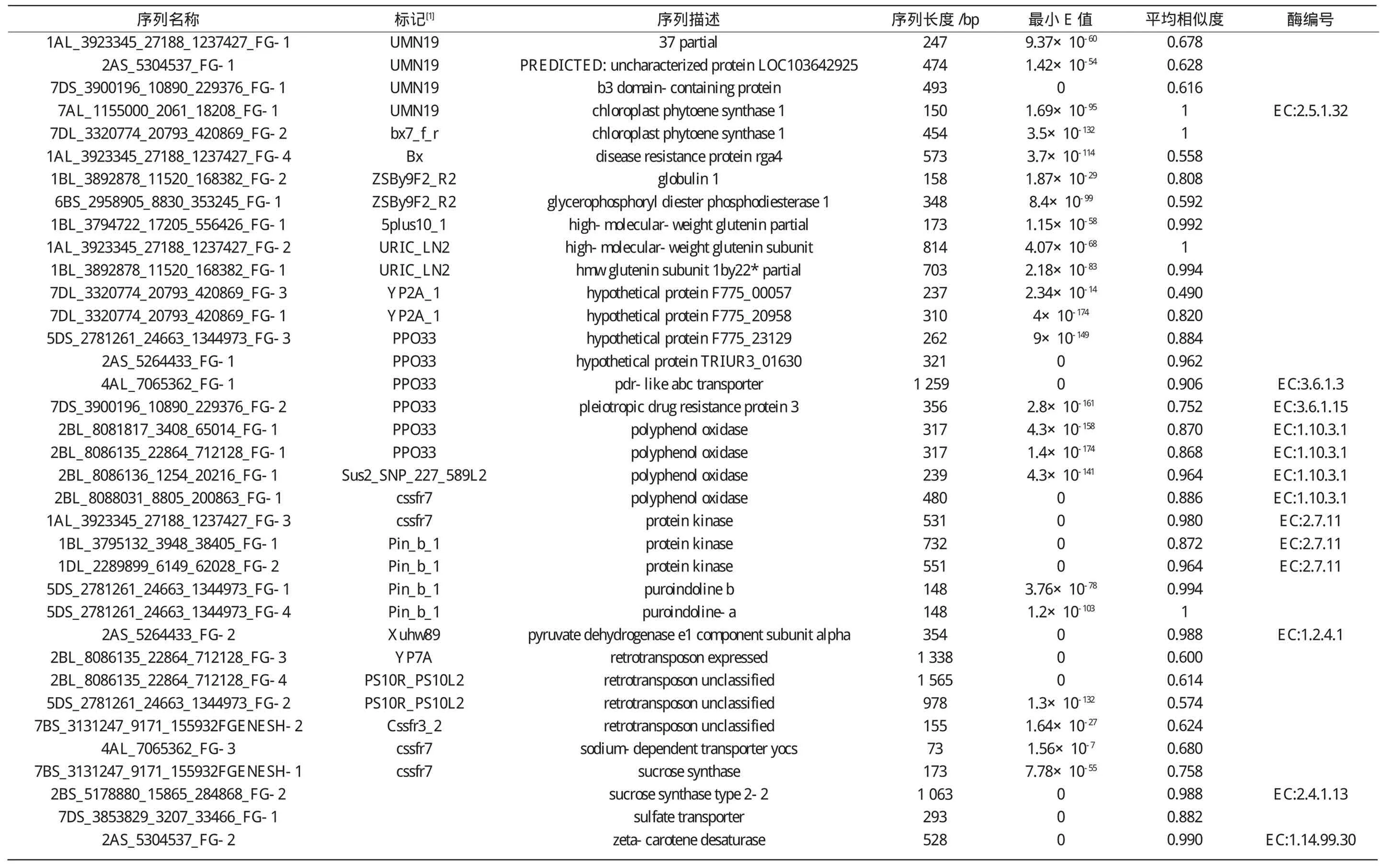
表5 小麦功能基因的特征
参考文献:
[1]Liu Y N,HeZ H,Rudi Appels,et al.Functional markers in wheat:current status and future prospects[J].Theoretical and Applied Genetics,2012,125:1-10.
[2]Mayer K F,Rogers J,Doleěel J,et al.A chromosome-based draft sequenceofthehexaploidbreadwheat(Triticum aestivum)genome [J].Science,2014,345:1251788-1251799.
[3]Frédéric C,Adriana A,Sébastien T,et al.Structural and functional partitioningofbreadwheatchromosome3B[J].Science,2014,345:1249721-1249727.
[4]Thomas M,SandveS R,LiseH,et al.Ancient hybridizations among the ancestral genomes of bread wheat[J].Science,2014,345:1250092-1250095.
[5]Matthias P,Kugler K G,Sandve S R,et al.Genome interplay in the grain transcriptome of hexaploid bread wheat[J].Science,2014,345:1250091-1250097.
[6]邢小萍,袁虹霞,孙炳剑,等.小麦品种对纹枯病的抗性鉴定及评价[J].河南农业科学,2007(6):77-78.
[7]胡锐,邢彩云,吴营昌,等.11个优质小麦品种对小麦白粉病抗性的初步鉴定[J].河南农业科学,2011,40(5):108-110.
[8]邢小萍,刘春元,袁虹霞,等.35个优质小麦品种(系)对叶部主要病害的抗性鉴定及评价[J].河南农业科学,2005(10):49-52.
[9]夏金初.小麦品种资源抗赤霉病性鉴定研究[J].湖南农业科学,1987(S1):43-45.
[10]王振跃,高书峰,李洪连,等.不同小麦品种(系)对禾谷孢囊线虫病的抗性鉴定[J].河南农业科学,2006(5):50-52.
[11]何文兰,宋玉立,杨共强.离体叶段鉴定小麦白粉病、条锈病和叶锈病抗性方法的研究[J].河南农业科学,1999(10):20-22.
[12]黄骥,张红生,曹雅君,等.水稻功能基因的电子克隆策略[J].中国水稻科学,2002,16(4):295-298.
[13]王冬冬,朱延明,李勇,等.电子克隆技术及其在植物基因工程中的应用[J].东北农业大学学报,2006,37(3):403-408.
[14]刘武艺.基因本体论的蟾蜍bHLH转录因子功能富集分布[J].生物信息学,2011,9(4):292-302.
[15]袁媛,唐东芹,杨柳燕,等.郁金香‘上农早霞’花色变异相关基因的信息分析 [J].上海交通大学学报:农业科学版,2015,33(2):9-17.
[16]胡文静.棉纤维起始发育优势基因表达谱和三个新基因的克隆与功能初步分析[D].南京:南京农业大学,2010.
[17]Masaaki Kotera,Yuki Moriya,Toshiaki Tokimatsu,et al.KEGG and genome net,new developments,metagenomic analysis[J]. EncyclopediaofMetagenomics,2015:329-339.
[18] Kiyoko F,Aoki-Kinoshita,Minoru Kanehisa,et al.Glycomic analysis using kEGG GLYCAN glycoinformatics[J].Methods in MolecularBiology,2015,1273:97-107.
[19]韩斌,王长彪,任永康,等.小麦分子育种研究进展IV.小麦相关分子标记[J].山西农业科学,2015,43(4):489-492.
[20]罗火林,熊冬金,罗丽萍,等.辣椒CaArcA基因的电子克隆与生物信息学分析[J].生物技术,2013,23(6):17-21.
In Silico Mapping and Annotation of Whole Genome Related Functional Genes in Wheat
WANG Changbiao1,HANBin2,LIU Jiang1,CUI Ting2,RENYongkang3,ZHAOXinghua1,DONG Yanhui1,LI Yali1,TANG Zhaohui1
(1.BiotechnologyResearchCenter,Shanxi AcademyofAgricultural Sciences,Taiyuan030031,China;2.CollegeofBio-engineering,Shanxi University,Taiyuan030006,China;3.InstituteofCropSciences,Shanxi AcademyofAgricultural Sciences,Taiyuan030031,China)
In silicocloning was a newway of gene-cloning by bioinfomatics,which developed with genome enforcementand EST project.Thisstudyclonedwheatfunctional geneinsilicocloningtechnology,labeledwithwheatfunctional markersasprobesinthewheat genome database.Furthermore,bioinformatics analysis were made on these functional genes.The results showed that the functional markers of bx7_f_r,ZSBy9F2_R2,PPO33,Sus2_SNP_227_589L2,UMN19,Pin_b_1,YP7A and cssfr7 could be cloned the corresponding functional genes.This study will contribute to the next step of wheat gene cloning,fine mapping and marker assisted breedingwork.
bioinformatics;wheat;functional gene;insilicocloning
S512.1
A
1002-2481(2016)07-0894-06
10.3969/j.issn.1002-2481.2016.07.02
2016-12-16
山西省科技攻关项目(20130311001-8);山西省农业科学院育种工程项目(11yzgc028);山西省农业科学院育种基础项目(yzjc1201);山西省农业科学院科技攻关项目(2013GG51)
王长彪(1975-),男,山西祁县人,助理研究员,主要从事分子标记辅助育种研究工作。唐朝晖为通信作者。

