新疆维吾尔族群体19个STR基因座遗传多态性
陈鹏宇,王 斌,韩岩岩,谢成明,余 舰
(1.遵义医学院附属医院 司法医学鉴定中心,贵州 遵义 563099;2.天津市公安局 物证鉴定中心,天津 300384;3.遵义医学院公共卫生学院 营养与食品卫生教研室, 贵州 遵义 563099)
临床医学研究
新疆维吾尔族群体19个STR基因座遗传多态性
陈鹏宇1,王 斌2,韩岩岩3,谢成明1,余 舰1
(1.遵义医学院附属医院 司法医学鉴定中心,贵州 遵义 563099;2.天津市公安局 物证鉴定中心,天津 300384;3.遵义医学院公共卫生学院 营养与食品卫生教研室, 贵州 遵义 563099)
目的 对新疆地区维吾尔族人群19个短串联重复序列(Short tandem repeats, STR)基因座的遗传多态性进行研究,评价其在法医学个体识别、亲子鉴定中的应用价值,并分析与其它不同群体的遗传关系。方法 应用GoldeneyeTM20A试剂盒对来自新疆维吾尔族的101例无关个体进行19个STR基因座的复合扩增,ABI 3130XL 全自动测序仪对扩增产物进行电泳分离,GeneMapper v3.2 软件进行基因分型,统计各基因座等位基因频率及法医学参数,并结合已公开报道的其他17个群体遗传多态性数据,计算群体间遗传距离,构建系统发生树。结果 GoldeneyeTM20A系统的19个STR基因座在新疆维吾尔族人群的基因型分布均符合Hardy-Weinberg平衡,且各基因座之间均不存在连锁现象,19个STR累积的个人识别率为0.99999999999999999999993,累积的非父排除率为0.999999997481278。群体间遗传关系分析显示,新疆维吾尔族与新疆哈萨克族首先聚类,其它14个汉族群体及云南白族,辽宁满族聚为一大类。结论 19个STR组成的复合检测系统在新疆维吾尔族人群中具有高度的多态性和鉴别能力,可用于该群体法医学个体识别、亲权鉴定及群体遗传学研究。
短串联重复序列;遗传多态性;新疆维吾尔族;群体遗传
短串联重复序列(Short tandem repeats, STR)是一类广泛存在于人类基因组中具有高度多态性的DNA 序列,具有分型简便快速、易于标准化和自动化等特点,是法医物证鉴定的主要目标序列之一[1],目前国内外已有多样化的商品化STR复合检测试剂盒用于法医个体识别和亲子鉴定[2]。GoldeneyeTM20A复合STR检测系统的19个STR基因座包含了中国国家DNA数据库所选用其它试剂盒中的全部STR 基因座,具有更好的数据库检索兼容性和准确性,以及更高的多态性和法医学鉴别能力[3]。我国维吾尔族为新疆地区世居的少数民族,具有其独特的历史、地理、文化以及群体遗传学特征,对其进行STR基因座遗传多态性研究,建立相应群体遗传学数据,对涉及该群体法医学个体识别和亲权鉴定案件的准确评估,以及群体遗传学研究具有重要意义[4]。本研究采用GoldeneyeTM20A复合STR检测系统,对新疆维吾尔族人群19个STR基因座(D8S1179,D21S11,D7S820,CSFIPO,D3S1358,TH01,D13S317,D16S539,D2S1338,D19S433,vWA,TPOX,D18S51,D5S818,FGA,Penta D,Penta E,D6S1043,D12S391)遗传多态性进行了调查,评价其在法医学个体识别、亲子鉴定中的应用价值,并分析了与其它不同群体的遗传关系。
1 对象与方法
1.1 研究对象 经知情同意,随机采集新疆地区维吾尔族101名健康、无血缘关系个体(男 68名,女33名)外周血1 mL,EDTA抗凝,Chelex-100 法提取基因组DNA[5]。
1.2 PCR扩增 按照产品指南,采用GoldeneyeTM20A PCR试剂盒(北京基点认知公司)对19个STR基因座(D8S1179, D21S11,D7S820,CSF1PO,D3S1358,TH01,D13S317,D16S539,D2S1338,D19S433,vWA,TPOX,D18S51,D5S818,FGA,PentaD,Penta E, D6S1043和D12S391)及牙釉基因进行复合扩增,PCR反应在ABI 9700型扩增仪上进行。
1.3 分型和命名 扩增产物采用ABI 3130XL型遗传分析仪电泳分离,使用GeneMapper ID Version 3.2软件包[6]进行等位基因分型。在对每一批次样本检测中,以DNA 9947A作为阳性对照,去离子水作为阴性对照。等位基因命名通过与厂家提供的等位基因分型标准物比较,并按照国际法医遗传学会(International Society for Forensic Genetics,ISFG)推荐的原则进行命名。
1.4 统计学分析 采用Powerstats V1.2[7]软件对各基因座进行Hardy-Weinberg平衡检验,采用SHEsis[8]软件包进行19个STR基因座的配对连锁不平衡检验。采用Powerstats V1.2软件计算等位基因频率、杂合度(heterozygosity,He)、多态性信息量(polymorphism information content,PIC)、个人识别率(power of discrimination,PD)以及非父排除率(probability of exclusion,PE)。应用公式[9]计算累积个人识别率(cumulative power of discrimination,CDP)和累积非父排除率(cumulative probability of exclusion,CPE)。
通过文献检索获得来自不同地区14个汉族和3个少数民族共17个群体的等位基因频率数据[2-7],基于共有的19个基因座,应用Phylip 3.695软件包[10]计算18个群体间的Nei’s遗传距离,应用Mega 7.0软件包[11]构建相邻连接(Neighbour-Joining, NJ)系统发生树。
2 结果
2.1 等位基因频率分布 在受检的101例新疆维吾尔族无关个体中,19个STR基因座上共检出190个等位基因,等位基因频率分布在0.005 0~0.535 0(见表1)。
表1 新疆维吾尔族群体19个STR基因座等位基因频率分布(n=101)
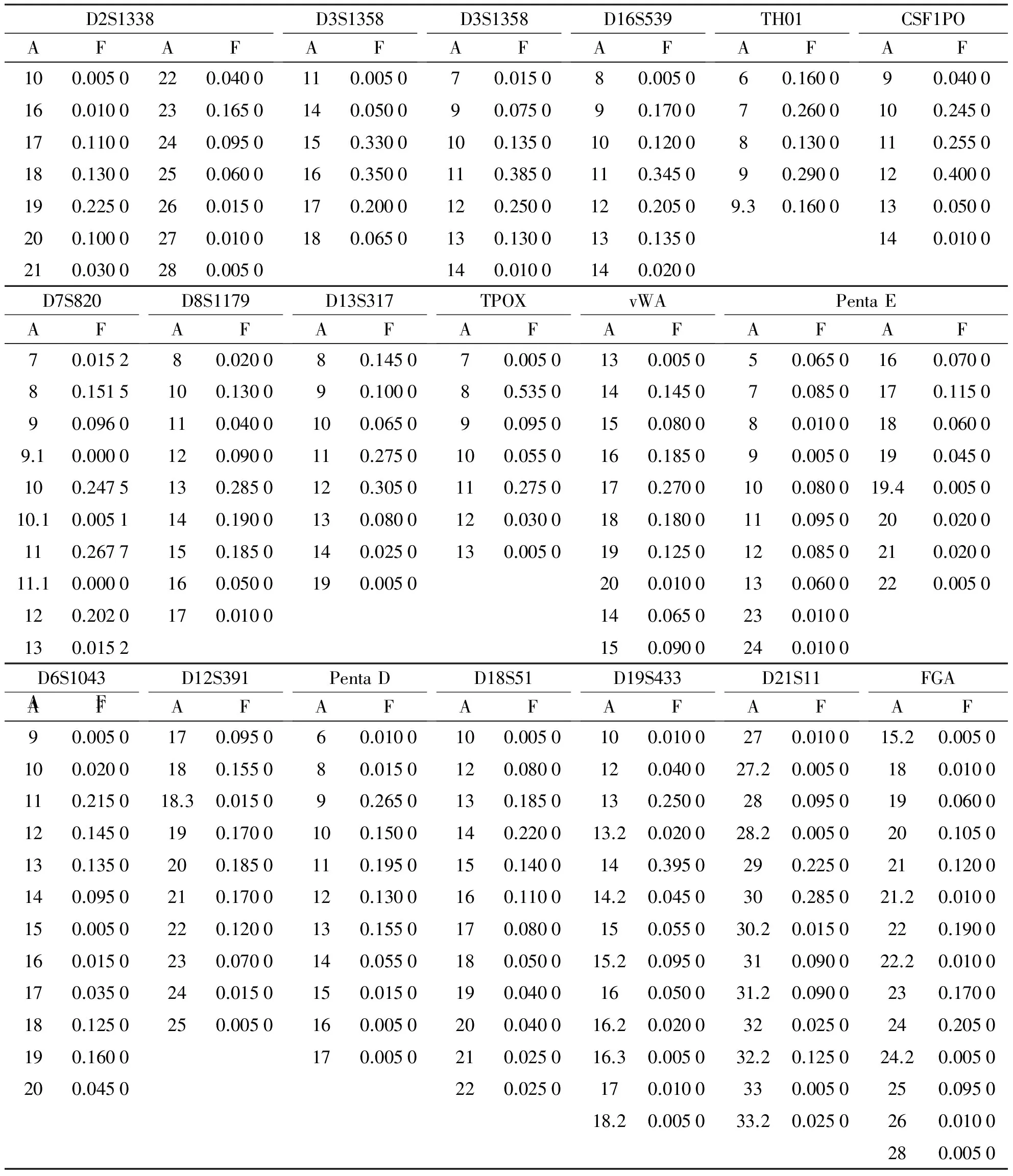
D2S1338AFAFD3S1358AFD3S1358AFD16S539AFTH01AFCSF1POAF100.0050220.0400110.005070.015080.005060.160090.0400160.0100230.1650140.050090.075090.170070.2600100.2450170.1100240.0950150.3300100.1350100.120080.1300110.2550180.1300250.0600160.3500110.3850110.345090.2900120.4000190.2250260.0150170.2000120.2500120.20509.30.1600130.0500200.1000270.0100180.0650130.1300130.1350140.0100210.0300280.0050140.0100140.0200D7S820AFD8S1179AFD13S317AFTPOXAFvWAAFPentaEAFAF70.015280.020080.145070.0050130.005050.0650160.070080.1515100.130090.100080.5350140.145070.0850170.115090.0960110.0400100.065090.0950150.080080.0100180.06009.10.0000120.0900110.2750100.0550160.185090.0050190.0450100.2475130.2850120.3050110.2750170.2700100.080019.40.005010.10.0051140.1900130.0800120.0300180.1800110.0950200.0200110.2677150.1850140.0250130.0050190.1250120.0850210.020011.10.0000160.0500190.0050200.0100130.0600220.0050120.2020170.0100140.0650230.0100130.0152150.0900240.0100D6S1043AFAFD12S391AFPentaDAFD18S51AFD19S433AFD21S11AFFGAAF90.0050170.095060.0100100.0050100.0100270.010015.20.0050100.0200180.155080.0150120.0800120.040027.20.0050180.0100110.215018.30.015090.2650130.1850130.2500280.0950190.0600120.1450190.1700100.1500140.220013.20.020028.20.0050200.1050130.1350200.1850110.1950150.1400140.3950290.2250210.1200140.0950210.1700120.1300160.110014.20.0450300.285021.20.0100150.0050220.1200130.1550170.0800150.055030.20.0150220.1900160.0150230.0700140.0550180.050015.20.0950310.090022.20.0100170.0350240.0150150.0150190.0400160.050031.20.0900230.1700180.1250250.0050160.0050200.040016.20.0200320.0250240.2050190.1600170.0050210.025016.30.005032.20.125024.20.0050200.0450220.0250170.0100330.0050250.095018.20.005033.20.0250260.0100280.0050
A:等位基因;F:等位基因频率
2.2 法医学参数 对19个STR进行Hardy-Weinberg平衡检验,P值为0.090 8~0.994 0(见表2),各基因座基因型分布符合Hardy-Weinberg衡。对19个基因座两两之间的连锁不平衡检验,r2为0.005~0.030,所有基因座之间不存在连锁不平衡现象。
新疆维吾尔族群体中19个STR基因座的He值为0.070 0~0.440 0;PIC值为0.572 4~0.920 0;DP值为0.805 8~0.979 2,PE值为0.245 6~0.857 0,其中多态性最低的基因座为TPOX,多态性最高的基因座为Pent E。19个STR累积的个人识别率为0.99999999999999999999993,累积的非父排除率为 0.999999997481278。
表2 新疆维吾尔族群体19个STR基因座法医学参数

基因座HePICDPPEHWE基因座HePICDPPEHWED2S13380.87000.85390.95780.73460.9544D19S4330.76000.73410.90320.52700.8858D3S13580.65000.67300.87380.35520.0908D21S110.79000.80490.94160.58060.2896D5S8180.72000.71130.90220.45990.4593PentaD0.82000.80170.93960.63670.8144D6S10430.88000.84470.95640.75480.6541PentaE0.93000.92000.97920.85700.9906D7S8200.85860.76220.91130.71190.1314CSF1PO0.77000.66050.84200.54460.2177D8S11790.82000.79550.93640.63670.9381TH010.85000.74510.90200.69490.1093D12S3910.84000.83790.94540.67530.5758TPOX0.56000.57240.80580.24560.1576D13S3170.76000.75950.92560.52700.4151FGA0.88000.83760.95360.75480.5417D16S5390.80000.74460.91620.59900.6443vWA0.88000.79220.93060.75480.1261D18S510.87000.85210.96120.73460.9940
He:杂合度;PIC: 多态性信息量;DP:个人识别率;PE:非父排除率;HWE:Hardy-Weinberg平衡检验的P值,P< 0.05 有统计学意义。
2.3 18个群体间遗传距离及系统发生树 根据19个STR基因座计算得到18个群体间的Nei’s遗传距离(见表3)可见,新疆维吾尔族与新疆哈萨克族[12]的遗传距离最近(0.0292),其次为辽宁回族[13](0.0388),与其它群体距离均较远(0.0414~0.0664)。14个汉族群体[14-16]间的遗传距离均较近,而且集中于一个较小的数值范围(0.0008~0.0237)。
根据18个群体Nei’s遗传距离构建的相邻连接系统发生树(见图1)可见,新疆维吾尔族与新疆哈萨克族[12]首先聚类,其它14个汉族群体及云南白族[17],辽宁回族[13]聚为一大类。在14个汉族群体中,山东汉族、河北汉族 ,河南汉族、安徽汉族,山西汉族、甘肃汉族、江苏汉族聚集为一个亚类,而山东汉族-河北汉族、河南汉族-安徽汉族、山西汉族-甘肃汉族分别形成三个独立的分支;云南汉族、四川汉族、江西汉族、闽南汉族、闽西汉族、广东汉族聚集成另一个亚类。
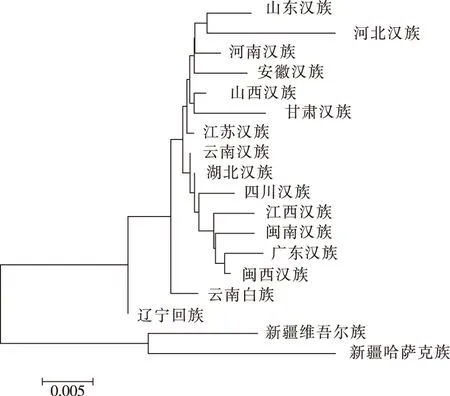
图1 基于Nei’s遗传距离构建的新疆维吾尔族和其它17个中国人群的相邻连接系统发生树
表3 新疆维吾尔族和17个群体之间的Nei’s遗传距离

Nei’s新疆维吾尔族新疆哈萨克族辽宁回族云南白族云南汉族河南汉族湖北汉族山东汉族四川汉族山西汉族甘肃汉族河北汉族江西汉族闽南汉族闽西汉族广东汉族江苏汉族安徽汉族新疆维吾尔族0.0000新疆哈萨克族0.02920.0000辽宁回族0.03880.04370.0000云南白族0.04300.04870.00910.0000云南汉族0.04390.05130.00610.00460.0000河南汉族0.04140.05310.00860.00750.00490.0000湖北汉族0.04440.05250.00630.00490.00080.00510.0000山东汉族0.05620.06100.01010.01040.00660.00850.00720.0000四川汉族0.04610.05410.01070.00810.00400.00980.00430.01050.0000山西汉族0.04320.05050.00680.00590.00380.00430.00460.00710.00920.0000甘肃汉族0.04740.05760.01220.01150.00790.01070.00890.01380.01380.00820.0000河北汉族0.06640.07100.01980.01990.01470.01590.01480.01700.01790.01520.02370.0000江西汉族0.05630.06120.00990.00920.00550.01210.00520.01170.00990.00950.01610.01970.0000闽南汉族0.04960.06040.01130.01160.00590.01050.00610.01220.00920.00990.01430.01940.00800.0000闽西汉族0.04690.05460.00950.00810.00330.00950.00320.01050.00510.00910.01260.01630.00650.00630.0000广东汉族0.04930.05750.01290.01010.00530.01400.00580.01480.00680.01230.01680.02240.00790.00740.00430.0000江苏汉族0.04440.05160.00640.00500.00180.00430.00190.00620.00610.00350.00820.01520.00730.00790.00590.00920.0000安徽汉族0.04810.05910.01180.01210.00690.00780.00690.01110.01010.00720.01290.01880.01460.01510.01140.01680.00640.0000
3 讨论
维吾尔族是我国主要少数民族之一,据2010年人口普查数据,其人口总数达10,069,346,居少数民族人口第4位[18]。约80%的维吾尔族聚居在新疆维吾尔自治区天山以南的喀什、和田一带、阿克苏和库尔勒地区,为地理上相对封闭隔离的群体。维吾尔族在不同的历史时期,经历了与不同民族、语言的复杂融合,现代维吾尔语为维吾尔族的共同语言,属阿尔泰语系突厥语族。特殊的地理、历史和文化背景,意味着新疆维吾尔族独特的群体遗传结构特征,对其进行STR基因座遗传多态性研究,建立适合新疆维吾尔族人群的群体遗传学基础数据,对涉及该群体个体识别和亲权鉴定案件的准确评估,以及群体遗传学研究具有重要意义[4]。
目前许多商品化的试剂盒应用于中国国家DNA数据库(National DNA database,NDNAD),如 Identifiler、Sinofiler、PowerPlex 16、PowerPlex 18D、Goldeneye 16A、AGCU 17+1等,这些试剂盒包含了15~17个基因座,但是不同试剂盒之间仅有11个共有的STR基因座,GoldeneyeTM20A 复合STR检测系统由19个STR基因座和1个判定性别的牙釉基因构成,包含了中国国家DNA数据库所选用的这些试剂盒中的全部 STR 基因座,在数据库信息检索时,可实现对同一样本数据中更多STR分型信息的比对,提高了数据库检索的兼容性和准确性[3]。同时,使用更多的STR基因座,也意味着更高的多态性和法医学鉴别能力[19]。
本研究采用GoldeneyeTM20A试剂盒19个基因座对新疆维吾尔族群体进行遗传多态性调查,在19个基因座上都达到了Hardy-Weinberg平衡,说明群体样本采集符合统计学要求,群体分型数据可信。19个STR配对连锁不平衡检验均符合连锁平衡,说明基因座之间无连锁关系,因此可以直接应用乘积定律,计算代表19个STR组成的复合系统总体鉴别能力的累积个人识别率和累积非父排除率,以及该复合系统在具体个案分析中相应的匹配概率和父权指数。
STR 多态性程度及其应用价值一般用杂合度、多态信息量、个体识别力和非父排除率等法医学参数进行评估。杂合度和多态性客观地反映了一个遗传标记的多态性水平,个人识别率和非父排除率则分别反映了遗传标记在法医学个人识别和亲子鉴定中的鉴别能力。 一个遗传标记的多态性越高,应用于法医的鉴别能力也会越高。Shriver等[9]认为当一个基因座DP>0.80、PE>0.50 时,属高度多态性遗传标记,具有较好的应用价值。在本研究的19个基因座中,除与其它试剂盒共有的D3S1358、D5S818、TPOX 3个基因座外,其余16个STR基因座均具有高度多态性。在法医学个人识别、亲子鉴定及群体遗传研究中具有较高的应用价值。按照乘积定律计算,19个STR累积的个人识别率和非父排除率分别高达0.99999999999999999999993和0.999999997481278,说明由该19个STR 组成的复合检测系统 GoldeneyeTM20A 在新疆维吾尔族人群中具有高度的多态性和鉴别能力,适用于该地区人群法医学个人识别和亲子鉴定。
遗传距离计算和聚类分析显示新疆维吾尔族和新疆哈萨克族的遗传关系接近,与辽宁回族的遗传关系相对较近,与14个不同地区汉族的遗传关系均较远。18个群体中,新疆维吾尔族和新疆哈萨克族属于阿尔泰语系,其它均属汉藏语系。提示不同语系民族之间具有遗传差异,同一语系的民族具有相近的遗传关系,该结果与以往对其它民族群体遗传关系分析得出的规律一致[10]。14个汉族群体的遗传关系较为接近,并呈现出一定的南北方地理分布的差异及连续过渡的特征,同时在地理位置上接近的群体,如山东汉族-河北汉族,河南汉族-安徽汉族,山西汉族-甘肃汉族分别聚集为不同的分支,说明地理上越接近的群体,遗传关系越近。
综上所述,本研究对新疆维吾尔族人群19个STR基因座遗传多态性进行调查,建立了该群体的遗传多态性数据,19个STR基因座组成的复合检测系统在新疆维吾尔族具有高度的多态性和鉴别能力,可用于该群体法医学个体识别、亲权鉴定及群体遗传学研究。
[1] Edwards A, Civitello A, Hammond H A, et al. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats[J]. Am J Hum Genet,1991,49(4): 746-756.
[2] 阮修艳, 王伟妮, 杨雅冉,等. 北京汉族群体39 个短串联重复序列基因座多态性及其遗传关系[J]. 遗传, 2015,37(7): 683-691.
[3] 王洁, 黄艳梅, 张庆霞,等. 国产GoldeneyeTM20A试剂盒性能指标验证[J]. 中国法医学杂志,2012,27(1): 12-15.
[4] Hering S, Nixdorf R, Dressier J, et al. Identification of more sequence variations in the D8S1179 locus [J]. Forensic Sci Int, 2005,149(2): 275-278.
[5] Walsh P S, Metzger D A, Higuchi R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material[J]. Biotechniques, 1991,10(4): 506-513.
[6] Gene MapperTMID Software v3.2 User Guide[EB/OL]. (2006). http://www.appliedbiosystems.com.
[7] Tereba A. Tools for analysis of population statistics Profiles, PowerStats Version 1.2, Promega Corporation[EB/OL]. (1999). http://www.promega.com/geneticidtools/powerstats/.
[8] Shi Y Y, He L. SHEsis, a powerful software platform for analyses of linkage disequilibrium, haplotype construction, and genetic association at polymorphism loci[J]. Cell Res, 2005,15(2):97-98.
[9] 侯一平, 王宝捷, 郭大玮. 法医物证学[M]. 第3版. 北京: 人民卫生出版社, 2009: 22-23.
[10] Felsenstein J. PHYLIP, the PHYLogeny Inference Package, Version 3.695, Department of Genome Sciences and Department of Biology, University of Washington[EB/OL]. (2013). http://evolution.gs.washington.edu/phylip.html.
[11] Sudhir K, Glen S, Koichiro T. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0[EB/OL]. (2015). http://www.kumarlab.net/publications.
[12] Zhang L P, Xu C B, Chen H J, et al. Genetic polymorphism of 19 STR loci in Xinjiang Barkol Kazakh population[J]. J Cent South Univ (Med Sci),2012,37(9): 934-938.
[13] 沈红缨, 喻少波, 郭飞, 等. 辽宁回族人群19个STR基因座遗传多态性[J]. 中国法医学杂志,2013,28(6): 504-505.
[14] Wang H D, Wu D, Feng Z Q, et al. Genetic polymorphisms of 20 short tandem repeat loci from the Han population in Henan, China[J]. Electrophoresis,2014,35(10): 1509-1514.
[15] Yin C Y, Ji Q, Li K, et al. Analysis of 19 STR loci reveals genetic characteristic of eastern Chinese Han population[J]. Forensic Sci Int Genet, 2015,14: 108-109.
[16] Zhang M X, Han S Y, Gao H M, et al. Genetic Polymorphisms of 19 STR Loci in Shandong Han Population[J]. J Forensic Med, 2013,29(6): 440-443, 446.
[17] Li Y, Hong Y, Li X J, et al. Allele frequency of 19 autosomal STR loci in the Bai population from the southwestern region of mainland China[J]. Electrophoresis,2015,36(19): 2498-2503.
[18] 国务院人口普查办公室, 国家统计局人口和就业统计司. 中国2010年人口普查资料[EB/OL]. (2012 )[2016.5.31]. http://www.stats.gov.cn/tjsj/pcsj/rkpc/6rp/indexch.htm.
[19] Hares D R. Expanding the CODIS core loci in the United States[J]. Forensic Sci Int Genet ,2012,6(1): e52-54.
[收稿2016-06-13;修回2016-09-01]
(编辑:王福军)
Study of genetic polymorphism of 19 STR loci in Xinjiang Uyghur population
ChenPengyu1,WangBin2,HanYanyan3,XieChengming1,YuJian1
(1. Department of Forensic Judicial Authentication Center, Affiliated Hospital of Zunyi Medical University, Zunyi Guizhou 563099, China; 2. Institute of Forensic Science, Tianjin Public Security Bureau, Tianjin 300384, China;3. Department of Nutrition and Food Hygiene, School of Public Health, Zunyi Medical University, Zunyi Guizhou 563099, China)
Objective To study the genetic polymorphism of 19 short tandem repeats (STR) loci in Xinjiang Uyghur population, evaluate the application value in forensic identification and kinship testing, and analyze the population genetic relationship with previously published population data. Methods 101 unrelated Uyghur individuals from Xinjiang were screened, amplification of 19 STR loci were performed using the GoldeneyeTM20A multiple STR kits. PCR products were separated by ABI 3130XL sequencer and genotyped with GeneMapper v3.2 software. Allele frequencies and forensic parameters were calculated with diverse software. To analyze genetic relationship with other 17 previously reported population data, genetic distance between populations were calculated and phylogenetic tree was also constructed. Results At all 19 STR loci, no significant deviation from Hardy-Weinberg equilibrium (HWE) were observed (P> 0.05), and no significant deviations from LD between pairwise STR loci were found. The combined power of discrimination (CPD) and the combined power of exclusion (CPE) was 0.99999999999999999999993 and 0.999999997481278, separately. Two main clusters existed within the 18 populations. The first main cluster consisted of Uyghur and Kazakh populations in Xinjiang. The second main cluster included 14 Han groups from different regions of China, Yunnan Bai and Liaoning Hui populations. Conclusion The 19 STR multiple system afford high genetic polymorphism and discrimination power in Xinjiang Uyghur population, and has important significance in forensic identification, paternity testing, and population genetic study.
short tandem repeats; genetic polymorphism; Xinjiang Uyghur; population genetics
遵义医学院博士启动基金资助项目(NO:院字[2015]01)。
余舰,男,教授,硕士生导师,研究方向:法医学,E-mail:783065265@qq.com。
DF795.2
A
1000-2715(2016)04-491-06

