一种改进的时延动态贝叶斯网络构建算法研究
刘飞
(宝鸡文理学院 物理与光电技术学院,陕西 宝鸡 721016)
一种改进的时延动态贝叶斯网络构建算法研究
刘飞
(宝鸡文理学院 物理与光电技术学院,陕西 宝鸡 721016)
从大规模实验数据中构建的网络可以反向发掘网络结点之间潜在的相互作用关系,可以更深层次解释网络结点间复杂的作用机理,因此产生了很多网络构建的理论建模方法。在一些实验数据中某个时间点的样本值表达减少或者被敲除,这会影响网络构建的精度。为了克服这个问题提出了相对变化率的策略来识别结点间潜在的作用关系。在时序实验数据中,用策略融合时延动态贝叶斯方法来进行网络构建。可以缩减贝叶斯的搜索空间,以此来获得较高的网络构建性能和精度。DREAM(Dialogue for Reverse Engineering Assessments and Methods)竞赛项目最早被提出用来严格检测结点间网络构建模型方法的性能和优劣。在这个数据集上推测出来的网络在AUROC(area under the ROC curve)和AUPR (area under the precision recall curve)指标上和其它方法进行了比较,实验结果验证算法在网络构建过程中总体性能略胜一筹。
复杂网络;网络构建;相对变化率;时延;贝叶斯网络
0 引 言
随着高通量数据的产生和计算机技术的发展,对这些大数据的处理和挖掘已经成为研究的热点。一个网络可以抽象为由结点和边组成的一个图,结点表示现实网络中的一些研究对象,边可以理解为结点之间潜在的相互作用关系。如在疾病基因网络中,一些疾病基因可以看成网络中的结点,基因之间的相互作用关系可以理解为网络中的边,那么就可以通过网络的手段知道哪些基因作用关系密切,可能会成为致病因素,这就对药物设计和疾病治疗起到一定的理论意义。因此构建的网络可以系统地研究网络中结点之间的调配关系,为深入挖掘结点之间的复杂关系及作用机制提供理论模型。
现今已经诞生了很多数学模型和计算机方法来推断网络构建,这些模型方法主要包括布尔网络模型(Boolean networks)[1],常微分方程模型[2],信息论模型[3-4],线性规划[5]模型和贝叶斯网络模型[6-7]。这些方法能够对网络系统动态行为提供更深层次的理解,但是构建方法的精度对模型中参数的依赖性非常大,且算法的时间复杂度非常大,这就使得构建大规模网络变得异常困难[8]。由于基因调控过程的复杂性、调控的方向性、网络的规模性以及基因表达数据的噪声等因素的影响,这些都会影响基因调控网络构建性能。如基于互信息的方法不能检测网络的方向,不能区分直接和间接调控;基于优化模型的方法很难解决高维低样本数据;基于贝叶斯网络的方法具有较高的计算复杂度,且很难处理大规模网络等。如何克服这些模型和方法中的缺点,是基因调控网络构建方法提高其性能的关键所在。
贝叶斯网络模型是一种图形概率模型,利用贝叶斯规则建立结点间的联合概率分布来构造调控网络的。贝叶斯网络模型是基于统计理论的,并且能够应用到各种类型的生物数据中,因此能够捕捉到表达数据中固有的噪声,而且准许在决定结点间相互作用关系的过程中可以利用贝叶斯规则来结合一些先验生物学知识。动态贝叶斯网络(DBN)是贝叶斯网络的扩展版,既可以使用稳态数据,也可以应用到基因表达时间序列数据中。当构建的调控网络包含大量的基因结点时,DBN需要花费大量时间来学习条件依赖关系。为此我们提出了一种数据相对变化率的策略来预处理随机结点数据,以此来识别出网络中一些关键的结点,在这个基础之上我们再采用时延的动态贝叶斯方法和一些先验知识来构建网络,所构建网络的优劣用性能指标AUPR和AUROC值来评估。试验结果验证我们的方法取得了良好的网络构建性能,在网络的构建准确率和精度方面都取得了较好的结果。
1 理论方法
1.1 相对变化率
网络中的边表示结点之间潜在的相互作用关系,在给定的网络中,一个结点的表达水平值发生变化会影响那些和它有相互作用关系的结点的表达值。如果结点在整个网络中扮演了一个非常重要的角色,那么它的变化会引起其周围近邻结点的变化,我们也称这些点为中心结点或者关键结点(Hub Node)。因此一些结点值的表达类型不同(如原态数据,表达缺失数据,表达值减小数据和带有噪声的数据等)就会影响网络构建的精度。这里我们引入相对变化率的数据预处理策略来识别出一些中心结点,为进一步网络构建减小时间复杂度。
对于给定实验数据集中的每一个结点数据,我们以它的原态表达数据作为参照点,然后计算每个数据结点的相对变化率,数据的相对变化率公式定义如下:
i=1,…,n;j=1,…,n
其中Ri,j表示结点j在结点i表达值敲除情况下的相对变化率,Gi,j表示结点j在结点i表达值敲除情况下的表达值,Wj表示结点j原态表达值,Max(G:,j)-Min(G:,j)表示结点j对所有结点表达值变化范围,如果i=j,则相对变化率Ri,j为零。
如果结点的变化率大于给定的一个阈值,我们就认为这个结点是关键结点,在整个网络中扮演非常重要的角色。当结点的表达值相对于参照结点的变化小于给定的一个阈值时,我们认为它是噪声,把它从潜在的调控结点表单中剔除。如图1所示,计算结点1到10对于结点1的相对变化率,其中结点2,4,5,7,8明显高出阈值,那么我们就认为这些节点潜在的被结点1调控,结点1在整个网络中是一个关键结点。

图1 相对变率的事例框图
1.2 动态贝叶斯网络
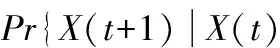
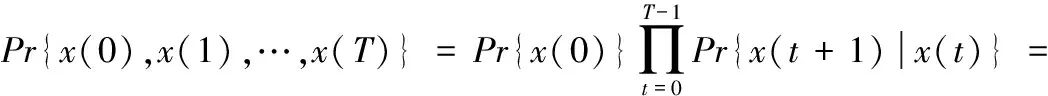

图2 动态贝叶斯网络
1.3 时延动态贝叶斯网络
K Murphy[11-12]提出了动态贝叶斯的表示方法,网络推断及结构学习,但是这个模型有两个主要的缺陷。第一,这个算法的时间复杂度非常大,对于网络中某结点,其父节点可能是剩余结点的一个或者多个,其算法的时间复杂度会随着网络规模成指数级增长。由于计算的时间太长一般认为一个结点的父节点数量限制在30个以内。第二,这个算法没有考虑相关性的延时问题,这会降低网络构建的精度。
为了克服这个缺陷,Conzen[7]74提出了基于时序数据的带有时延的动态贝叶斯网络构建算法,这种方法能够有效地降低算法的时间复杂度和提高算法的准确性。在网络中相互作用的两个结点,它们的时序表达值都有一个相应的延时趋势或者同时发生变化,那么用这种方法就可以限制一个结点的父节点的数量,以此来降低算法的时间复杂度。还发现在生物分子网络中,调控结点和被调控结点之间都会存在一个转录时延周期,那么结点表达值的延迟就代表了生物相对延时周期。这种方法考虑了结点变量间的相对关系,借助表达值的变化来估计调控结点和被调控结点之间的关系。那么用这种带有延迟的动态贝叶斯方法就可以直接从时序的基因表达数量识别出其对应的网络。
在我们的方法中,首先用相对变化率从时序数据中找出那些关键的网络结点,这些结点比其它结点更可能成为其它节点的父节点,在生成的网络中这些结点会扮演非常重要的角色,用相关性给这些关键结点先构建出一个调控网络。其次用时延的动态贝叶斯网络方法再从这些时序数据中构建另一个调控网络,把两个网络中都存在的那些边保留成为我们构建的最终网络,用DREAM[13]人工合成数据和真实网络数据来对我们的算法进行检测,证明我们方法的有效性和准确性。
2 结果分析讨论
2.1 构建的网络和标准网络比对结果
网络预测的性能可以用如下参数来评估,用真阳性(true positive, TP),假阳性(false positiv, FP),真阴性(true negative, TN),假阴性(flase negative, FN),召回率(Recall)也叫真阳性率(true positive rate, TPR),假阳性率(flase positive rate, FPR),精确度(Precision)也叫查准率(Positive Predictive Value, PPV)和准确率(accuracy, ACC)来对构建的网络进行检测,它们的公式定义如下:TPR=TP/(TP+FN),FPR=FP/(FP+TN),PPV=TP/(TP+FP),ACC=(TP+TN)/(TP+FP+TN+FN)。除了上述指标外还有受试者工作特性曲线(Receiver Operation Characteristic Curve, ROC曲线)和其曲线下的面积(Area under ROC, AUROC),精度召回率曲线(Precision and Recall Curve, PR曲线)和其曲线下的面积(Area under PR, AUPR)来衡量构建算法的优劣性。
我们的方法成功地应用到了人工合成数据集上[14-15],在E.coli数据集上,真实网络有10个结点,10条边。我们预测出来的网络共有11条边,其中7条正确,精度达到70%。在另外一个酵母(yeast)数据中,有50个结点,77条边,我们的方法预测的带有方向的网络如图3所示,其中预测正确的边有52条,准确率达到了68%。实验验证了我们的方法取得了较好的性能。
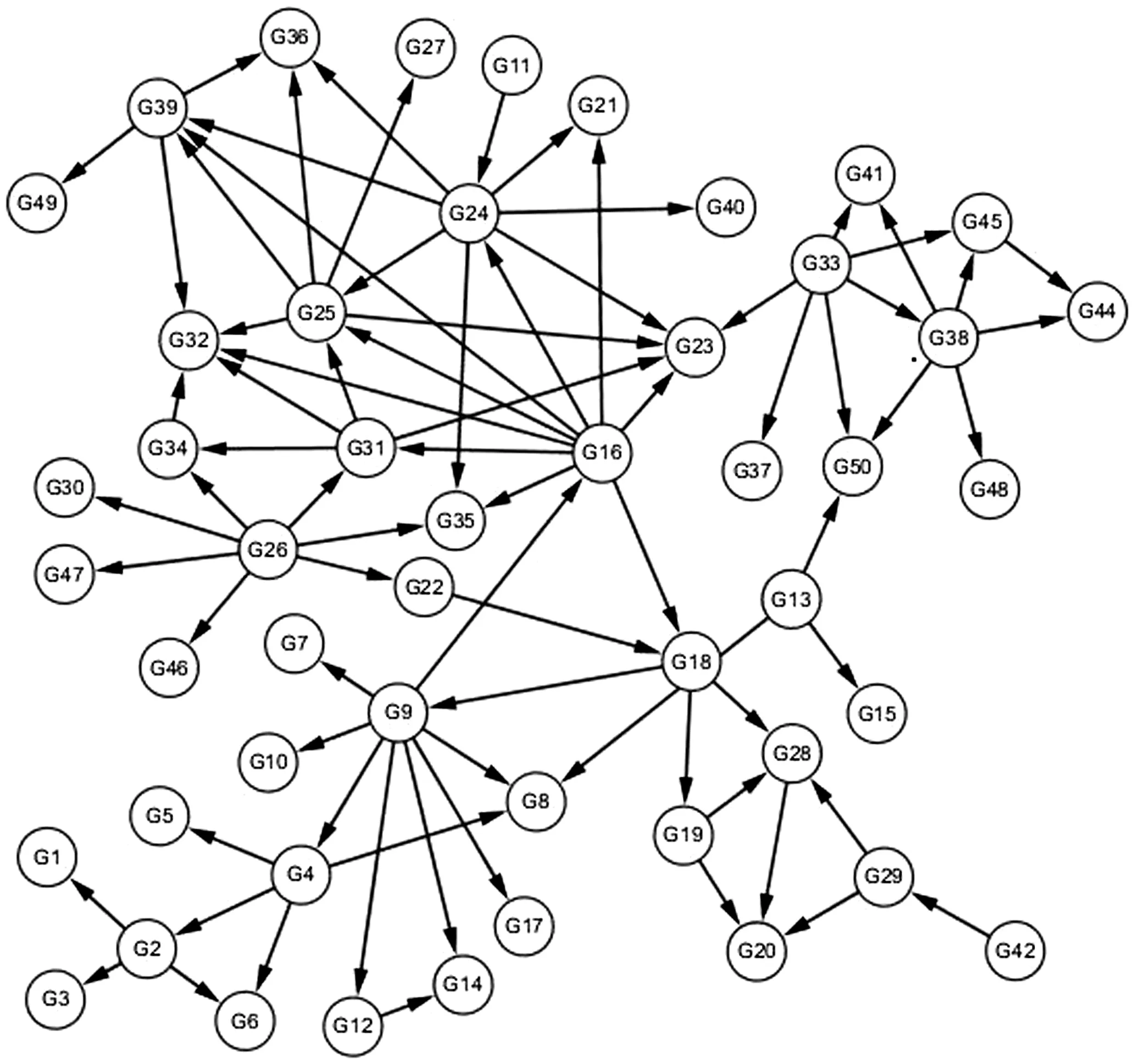
图3 50个结点的预测网络
2.2 相对变化率在网络构建中的作用
在三个不同的实验数据集上用我们的方法来构建网络,在10个结点的实验数据集上,只用相对变化率的方法来构建网络;在50个结点的实验数据集上,用相对变化率结合时延动态贝叶斯的方法来构建网络;在100个结点的实验数据集上,只用动态贝叶斯网络的方法来构建网络,以此来测试这三种网络构建方法的性能。
结果显示在10个结点和50个结点的网络构建中效果明显好于100个节点的网络构建,在E.coli数据集上构建网络的效果明显好于在yeast数据集上,具体参数如表1所示。结果显示采用了相对变化率策略可以比单独使用延迟动态贝叶斯方法取得更高的网络构建性能,这就可以解释为什么在100个结点的网络构建中,AUPR和AUROC值都相对偏低,在10个结点和50个结点的网络构建中,AUPR和AUROC值都相对偏高。这是因为在100个结点的网络构建中只采用了延迟的动态贝叶斯方法,在10个和50个结点的网络构建中都有相对变率策略的干预,由此可见,相对变化率的策略能提高网络构建的精度。
为了进一步说明相对变化率策略在网络构建中所起的作用,在10个结点的数据集上分别采用延迟动态贝叶斯方法,相对变化率策略方法和它们二者结合的方法来构建网络,这三种网络构建方法所获得的AUPR值如图4所示。实验结果说明采用相对变化率策略的方法和它们二者相结合的方法性能都优于单独使用时延动态贝叶斯方法,这充分说明相对变化率策略在网络构建中起着比较重要的作用。动态时延贝叶斯网络构建方法在计算结点潜在调控的父节点时,选取范围偏大导致网络中有较高的假阳性边,以致降低了网络构建的精度,如何把相对变化率策略和时延动态贝叶斯网络更好的结合,以提高网络构建的精度将是我们将来继续研究的问题。

表1 预测三种网络取得的参数性能

图4 三种不同方法在三个网络上的AUPR值比较图
3 结束语
文中提出了一种数据相对变化率的策略来预处理随机结点数据,以此来识别出网络中一些关键的结点,为后续的网络构建提供帮助,这些关键结点在生成的网络中会扮演一个非常重要的角色,它们会潜在地调控网络中的其它结点。在这个基础之上我们再采用时延的动态贝叶斯方法和一些先验知识来构建网络,所构建网络的优劣用性能指标AUPR和AUROC值来评估。试验结果验证我们的方法取得了良好的网络构建性能,在网络的构建准确率和精度都取得了较好的结果。如何把相对变化率策略和时延动态贝叶斯网络更好地结合,以提高网络构建的精度将是我们将来继续研究的问题。
[1] SHMULEVICH I,DOUGHERTY E R, KIM S, et al. Probabilistic Boolean networks: a rule-based uncertainty model for gene regulatory networks[J]. Bioinformatics, 2002, 18(2): 261-274.
[2] CASTELO R, ROVERATO A. Reverse engineering molecular regulatory networks from microarray data with qp-graphs[J]. Journal of Computational Biology, 2009, 16(2): 213-227.
[3] MEYER P E, LAFITTE F,BONTEMPI G. AR/bioconductor package for inferring large transcriptional networks using mutual information[J]. BMC bioinformatics, 2008, 9(1): 461.
[4] ZHANG X, ZHAO X M, HE K, et al. Inferring gene regulatory networks from gene expression data by path consistency algorithm based on conditional mutual information[J]. Bioinformatics, 2012, 28(1): 98-104.
[5] SAKAMOTO E, IBA H. Inferring a system of differential equations for a gene regulatory network by using genetic programming[C]. Evolutionary Computation, 2001. Proceedings of the 2001 Congress on. IEEE, 2001, 1: 720-726.
[6] YOUNG W C, RAFTERY A E, YEUNG K Y. Fast bayesian inference for gene regulatory networks using ScanBMA[J]. BMC Systems Biology, 2014, 8(1): 47.
[7] ZOU M, CONZEN S D. A new dynamic bayesian network (DBN) approach for identifying gene regulatory networks from time course microarray data[J]. Bioinformatics, 2005, 21(1): 71-79.
[8] MADHAMSHETTIWAR P B, MAETSCHKE S R, DAVIS M J, et al. Gene regulatory network inference: evaluation and application to ovarian cancer allows the prioritization of drug targets[J]. Genome Medicine, 2012, 4(5): 1-16.
[10] FRIEDMAN N, MURPHY K,RUSSELL S. Learning the structure of dynamic probabilistic networks[C]. Proceedings of the Fourteenth Conference on Uncertainty in Artificial Intelligence. Morgan Kaufmann Publishers Inc., 1998: 139-147.
[11] MURPHY K P. Dynamic bayesian networks: representation, inference and learning[D].University of California, Berkeley, 2002.
[12] MURPHY K, MIAN S. Modelling gene expression data using dynamic Bayesian networks[R]. Technical Report, Computer Science Division,University of California, Berkeley, CA, 1999.
[13] MARBACH D, SCHAFFTER T, MATTIUSSI C, et al. Generating realistic in silico gene networks for performance assessment of reverse engineering methods[J]. Journal of Computational Biology, 2009, 16(2): 229-239.
[14] MARBACH D, PRILL R J, SCHAFFTER T, et al. Revealing strengths and weaknesses of methods for gene network inference[J]. Proceedings of the National Academy of Sciences, 2010, 107(14): 6286-6291.
[15] PRILL R J, MARBACH D, SAEZ-RODRIGUEZ J, et al. Towards a rigorous assessment of systems biology models: the DREAM3 challenges[J]. PloS One, 2010, 5(2):9202.
The Study on an Improved Time-delayed Dynamic Bayesian Network Construction Algorithm
Liu Fei
(Institute of Physics and Optoelectronics Technology, Baoji University of Arts and Science, Baoji Shaanxi 721016, China)
Networks constructed from large-scale experimental data can reversely explore potential interaction among network nodes and help us understand at a deeper level the complex functional mechanism among these nodes. In this context, many theoretical approaches have been presented for network construction. The reduction or knockout of sample values at a certain time point in some experimental data would affect the accuracy of network construction. To solve this problem, we present the strategy of relative variation ratio to recognize potential interaction among the nodes. In the time sequence experimental data, this strategy and time-delayed dynamic Bayesian method are integrated for network construction. Our approach can reduce Bayesian searching space so as to obtain better performance and accuracy in network construction. The Dialogue for Reverse Engineering Assessments and Methods (DREAM) competitive event was first proposed for rigorous assessment of the performance and advantages/disadvantages of modeling methods for inter-node network construction. The network speculated with our data set is compared with other methods in two metrics: area under the ROC curve (AUROC) and area under the precision-recall curve (AUPR). Experimental results show that overall performance of our algorithm is slightly better than others in the process of network construction.
complex network; network construction; relative variation ratio; time delay; Bayesian network
国家自然科学基金(11547247,11105003);宝鸡市科技计划项目 (15RKX-1-5-18,14GYGG-5-2);宝鸡文理学院科研项目(ZK16009,ZK16028)资助;陕西省科技攻关计划项目(2014K08-17);陕西省教育厅科研计划项目(15JK1043)。
10.3969/j.issn.1000-3886.2016.04.011
TP391
A
1000-3886(2016)04-0033-03
刘飞,男(1981-),陕西榆林人,硕士,讲师,主要研究方向为复杂网络和数据挖掘。
定稿日期: 2015-12-28

