双吡啶吡咯Cu(Ⅱ)配合物与DNA相互作用研究
马 立, 易小艺, 张寿春
(中南大学 化学化工学院, 长沙 410083)
双吡啶吡咯Cu(Ⅱ)配合物与DNA相互作用研究
马 立, 易小艺, 张寿春*
(中南大学 化学化工学院, 长沙 410083)
合成了1种单核双吡啶吡咯Cu(Ⅱ)配合物[Cu(PDPH)2] (1),其中配体HPDPH为2,5-二(2′-吡啶基)吡咯.该配合物晶体属于单斜晶系,空间群为C2/c,a=39.1242(16) Å,b=8.6574(5) Å,c=33.7687(14) Å,β=123.0305(16)°,中心离子Cu2+处于变形八面体配位环境.采用紫外-可见光谱、荧光光谱及圆二色谱等方法,分别研究了单核配合物[Cu(PDPH)2] (1)、双核配合物[Cu2(PDPH)2(NO3)2] (2)和多聚配合物{[Cu2(PDPH)2(N3)2]}n(3)与DNA之间的相互作用.结果表明,3个Cu(Ⅱ)配合物均以沟面键合方式与CT-DNA结合,其结合强弱顺序为3 > 2 > 1.此外,采用凝胶电泳法研究了3种Cu(Ⅱ)配合物对超螺旋pBR322 DNA的切割作用.结果表明,3种配合物均能将pBR322 DNA切割为开口缺刻型或者线型DNA,表现出良好的切割活性.
铜配合物; 双吡啶吡咯配体; DNA键合; DNA切割
自上世纪70年代顺铂被发现具有抗肿瘤活性并成功应用于临床以来,小分子金属配合物作为抗癌药研究成为经久不衰的热门研究课题[1-2].DNA是生物的基本遗传物质和遗传信息的载体,在前药研究中,DNA常作为抗肿瘤药、抗菌药、抗病毒药等研究的重要靶点.小分子过渡金属配合物与DNA的相互作用研究是无机化学和生物学交叉的研究领域,通过它们相互作用研究有助于从分子水平上理解某些疾病的发病机理,也可以通过分子设计来寻找新的有效的治疗药物.
铜是人体一种必需的元素,在人体许多生理过程中发挥着重要作用.研究表明,许多铜配合物能够与DNA之间发生键合,并能够有效地切割DNA[3-7].研究表明,具有大环芳香结构的配合物可以更好地插入DNA碱基对,从而使配合物与DNA的结合能力增强[8-9].本课题组报道了多种含大环芳香结构配体菲咯啉或其衍生物的铜配合物具有很好的DNA结合能力与切割活性[10-12].此外,有课题组研究了系列三联吡啶铜配合物(Cu-terpy)可通过嵌入或沟面键合的方式与DNA结合[13-15].
本文利用紫外-可见光谱、荧光光谱及圆二色谱等方法,考察了与三联吡啶结构相似的双吡啶吡咯化合物为配体的几种铜配合物与DNA之间的相互作用,并通过凝胶电泳法研究了配合物对超螺旋DNA的切割能力.
1 实验部分
1.1试剂与仪器
小牛胸腺DNA(CT-DNA)、琼脂糖(agarose)、溴化乙锭(EB)、三羟甲基氨基甲烷(Tris)和抗坏血酸(H2A)均购自Sigma,超螺旋pBR322 DNA购自宝生物工程(大连)有限公司.其它试剂均为分析纯,用前未作进一步处理.紫外-可见光谱用UV-2450紫外-可见分光光度计测定,荧光光谱用Hitachi F4600荧光光谱仪测定,圆二色谱(CD)用Jasco J-815圆二色谱仪测定,凝胶电泳实验用北京六一DYY6C电泳仪测定.
1.2配合物的合成
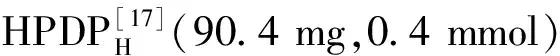
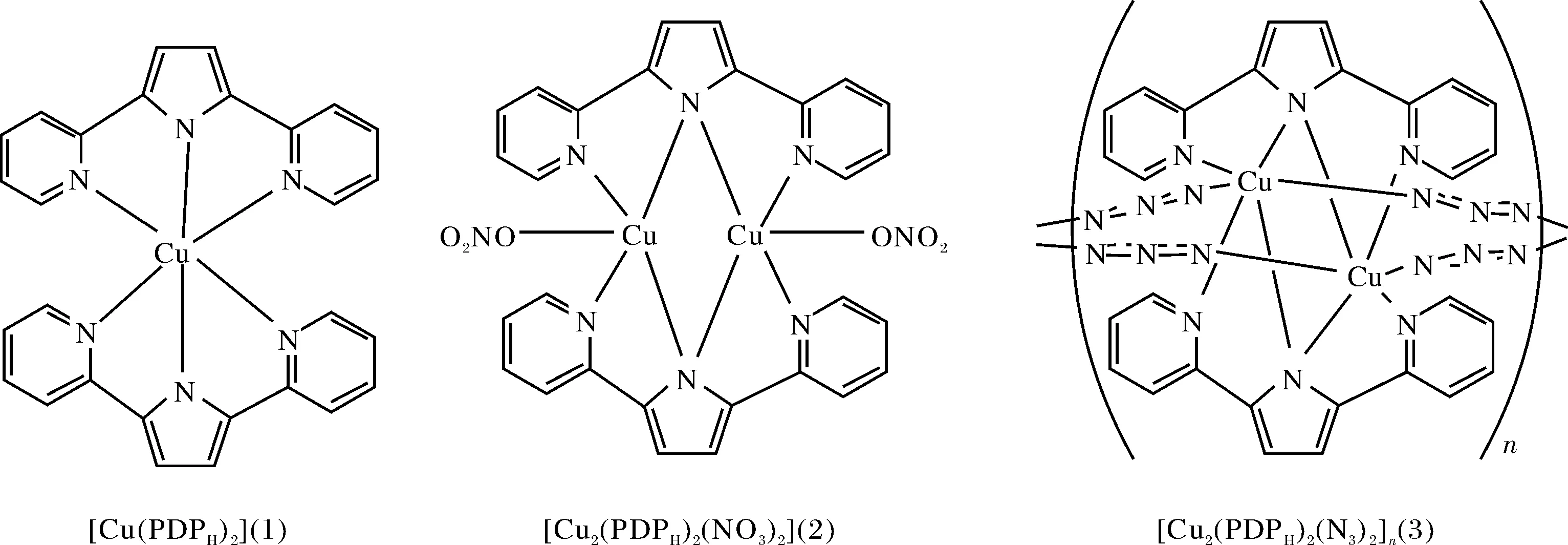
图1 双吡啶吡咯铜(Ⅱ)配合物1~3 的结构图Fig.1 Structures of the dipyridylpyrrole copper(Ⅱ) complexes 1~3
配合物2和配合物3均根据本课题组以前发表的论文中的方法合成[18].
1.3晶体结构测定
配合物1的晶体结构是用Bruker SMART APEX 1000 CCD单晶衍射仪进行测量.将测试样品置于X射线单晶衍射仪上,使用石墨单色器单色化的Mo-Kα射线(λ=0.71073 Å),在296 K的温度下,扫描收集配合物的晶体衍射数据.收集到的数据通过SAINT软件处理,SADABS进行吸收矫正处理,由SHELXTL-97软件利用直接法解出,从而得到确定的晶体结构,所有非氢原子坐标通过各向异性参数确定,有机配体的氢原子位置使用理论加氢得到,配合物结构的各向异性参数进行全矩阵最小二乘法修正.配合物的详细晶体学数据列于表1中.剑桥晶体数据中心(CCDC)数据编号:1474009.
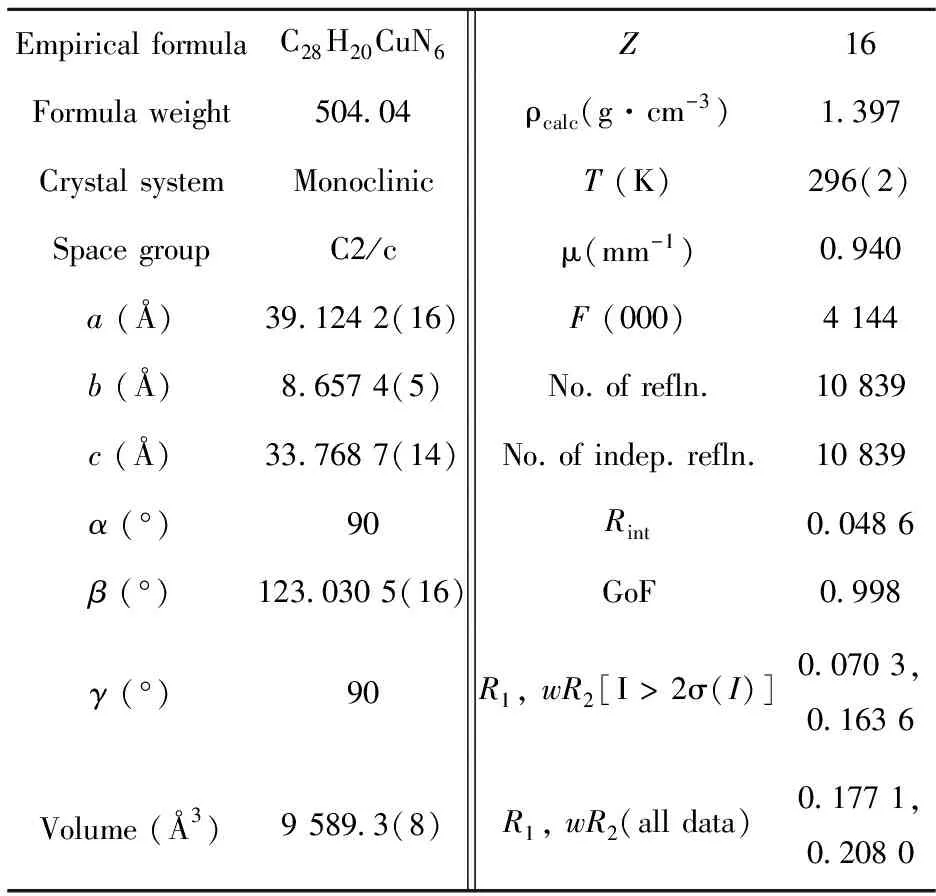
表1 配合物[Cu(PDPH)2] (1)的晶体学数据
1.4配合物与DNA相互作用
1.4.1紫外-可见光谱 用pH 为7.2的5.0 mmol·L-1Tris-HCl/50 mmol·L-1NaCl 的缓冲溶液溶解一定量的CT-DNA,测定该DNA溶液的紫外吸收光谱,并计算其在260 nm和280 nm处的吸光度比值为1.82,表明该CT-DNA中不含蛋白质[19].DNA溶液的浓度通过测定在260 nm处的吸光度来确定(DNA在260 nm处的摩尔吸光系数为6 600 L·mol-1·cm-1).配制3.0×10-5mol·L-1的3种铜(Ⅱ)配合物溶液,将DNA溶液按一定比例(r=0.00,0.05,0.10,0.15,0.20,0.25,0.30,0.35,r为DNA与配合物的物质的量之比)加入其中,在紫外-可见分光光度计上测定配合物溶液的紫外-可见光谱变化.通过以下公式计算出配合物与DNA的结合常数Kb[20].
[DNA]/(εa-εf)=[DNA]/(εb-εf)+
1/kb(εb-εf),
其中,εa表示配合物的表观摩尔吸光系数,其值为Aobsd/ccomplex,εb表示配合物与DNA完全结合后的摩尔吸光系数,εf为纯配合物的摩尔吸光系数.以[DNA]/(εb-εf)对[DNA]作图,得到的直线斜率和截距的比值即为结合常数Kb.1.4.2荧光光谱 用pH为7.2的5.0 mmol·L-1Tris-HCl/50 mmol·L-1NaCl 的缓冲溶液溶解配合物,配制成浓度为6.0×10-4mol·L-1的溶液,然后将配合物溶液慢慢滴加到含有1.0×10-5mol·L-1EB和1.0×10-4mol·L-1CT-DNA的混合溶液中,在室温下,分别测定每次加样后的荧光光谱(激发波长为530 nm,发射波长为600 nm).
1.4.3CD光谱 取3 mL 1.0×10-4mol·L-1的CT-DNA溶液,按r=0.00,0.05,0.10,0.15(r为配合物和DNA的物质量之比)的比例分别加入一定量的3.0×10-5mol·L-1的配合物溶液,在圆二色谱仪上设定波长220~320 nm,扫描速度10 nm/min,数据记录间隔0.1 nm,分别测定这几组溶液的CD曲线,样品测试前需要先拿Tris缓冲液作为参比液扫描基线.
1.4.4凝胶电泳实验 将配合物按照不同的浓度与pBR322 DNA混合,然后在配好的样品中分别加入1 μL抗坏血酸(H2A),在37 ℃、pH为7.4条件下恒温水浴1 h.加入2 μL的上样缓冲液(30 mmol·L-1EDTA,36%丙三醇,0.05%溴酚蓝)终止反应.利用EB染色的1.0%琼脂糖电泳分析切割结果.电泳液为TAE缓冲液(40 mol·L-1Tris-HAc/1 mmol·L-1EDTA ),电泳图用UVP凝胶成像系统分析.
2 结果与讨论
2.1晶体结构
配合物[Cu(PDPH)2]的晶体结构如图2所示,其主要键长和键角列于表2.该配合物分子由1个金属中心Cu2+离子和2个PDPH配体键合而成,Cu2+中心处于六配位的畸变八面体构型的配位环境中,其中2个PDPH配体吡啶环上氮原子N(1)、N(3)、N(5)、N(6)位于赤道平面,键角分别为
N(3)-Cu(1)-N(1) (150.16(17)°)和N(6)-Cu(1)-N(5) (150.43(16)°)均小于180°,2个PDPH配体吡咯环上氮原子N(2)和N(4)占据2个极轴上的顶点,与中心原子Cu构成的键角N(4)-Cu(1)-N(2)为176.5(2)°,也偏离180°,这与已经报道的三联吡啶铜配合物[Cu(terpy)2]2+的结构非常相似[21].两个吡咯N原子与中心Cu2+离子间的键长比四个吡啶N原子与中心Cu2+离子间的键长明显更短,说明在该配合物中,中心Cu2+离子与吡咯N原子的相互作用能力强于吡啶N原子.
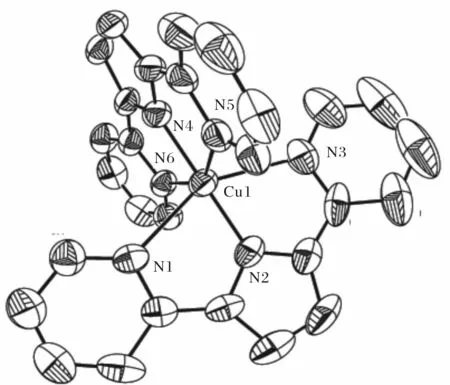
图2 配合物[Cu(PDPH)2](1)的分子结构图(为清晰可见,分子中氢原子已被省略掉)Fig.2 The molecular structure of complex[Cu(PDPH)2] (1)

Cu(1)-N(1)2349(4)Cu(1)-N(2)1861(4)Cu(1)-N(3)2315(4)Cu(1)-N(4)1874(4)Cu(1)-N(5)2365(5)Cu(1)-N(6)2268(5)N(1)-Cu(1)-N(5)9028(15)N(2)-Cu(1)-N(1)7466(19)N(2)-Cu(1)-N(3)7554(19)N(2)-Cu(1)-N(4)1765(2)N(2)-Cu(1)-N(5)10913(18)N(2)-Cu(1)-N(6)10044(18)N(3)-Cu(1)-N(1)15016(17)N(3)-Cu(1)-N(5)9791(15)N(4)-Cu(1)-N(1)10704(18)N(4)-Cu(1)-N(3)10280(17)N(4)-Cu(1)-N(5)7404(19)N(4)-Cu(1)-N(6)764(2)N(6)-Cu(1)-N(1)9804(16)N(6)-Cu(1)-N(3)8888(15)N(6)-Cu(1)-N(5)15043(16)
2.2紫外-可见光谱
金属配合物与DNA作用前后紫外-可见吸收谱带的位置及强弱会发生一定程度的变化,不同的作用方式对应着特定的谱带变化规律,据此可以判断二者的相互作用方式.研究表明,金属配合物中加入DNA后,配合物与DNA分子发生嵌插作用会造成其紫外-可见光谱的吸收峰强度减小(减色效应),且吸收峰位置显著红移[22-23].本实验中的3个铜(Ⅱ)配合物中芳香环之间的π-π跃迁以及金属中心与配体之间的电荷迁移跃迁能够在紫外可见区域形成强烈的电子吸收谱带.配合物与CT-DNA相互作用的紫外-可见光谱如图3所示.随着配合物中CT-DNA浓度的逐渐升高,配合物的吸收强度均逐渐减弱(减色效应).配合物1~配合物3在388 nm处的减色率分别为20.48%,22.77%和27.31%,且未见明显的红移,说明该3个铜(Ⅱ)配合物不是通过经典的插入方式与DNA发生相互作用.配合物1~配合物3与CT-DNA的结合常数Kb分别为4.8×104L·mol-1,5.01×104L·mol-1和5.61×104L·mol-1,均低于经典插入试剂EB的结合常数1.0×106L·mol-1[24].说明3个铜(Ⅱ)配合物与DNA的键合作用较EB要弱,可能是通过非经典嵌入方式,如沟槽键合等方式与DNA相互作用.根据键合常数(Kb)比较,3个配合物与DNA的作用强弱顺序为3>2>1.
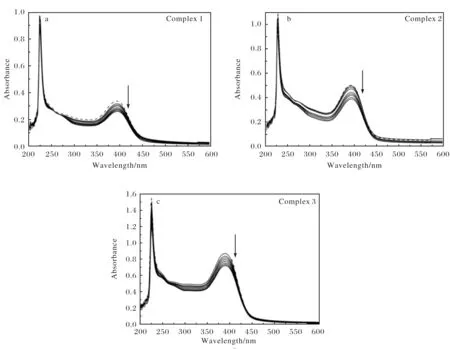
图3 配合物1~配合物3在不同CT-DNA浓度下的紫外-可见光谱图(图中箭头表示CT-DNA浓度逐渐增大时的吸光度的变化趋势)Fig.3 UV-Vis absorption spectra of complex 1~ complex 3 in the absence (---) and presence (-) of increasing concentration of CT-DNA
2.3荧光光谱
进一步通过荧光光谱法研究配合物与CT-DNA之间的相互作用及键合强弱.EB是研究小分子化合物与DNA相互作用最常用的荧光探针.DNA分子本身的荧光强度很弱,EB在溶液中的荧光强度也不高,但当EB插入到DNA的碱基对之间后,其荧光强度大大增强;当EB从DNA双螺旋碱基对间出来时,荧光强度则显著降低.因此,可以根据化合物对EB-DNA复合物荧光的影响来研究两者间的相互作用.
配合物1~配合物3与DNA相互作用的荧光光谱如图4所示.随着配合物的不断加入,EB-DNA复合物的荧光强度逐步减弱(荧光强度减弱率分别为18.35%,19.16%和23.66%),表明这3个铜(Ⅱ)配合物与DNA有较弱的键合作用,并可能是通过沟槽模式与CT-DNA相结合[25].配合物作用在双螺旋DNA的大沟或者小沟区,竞争了EB与DNA的结合,引起了DNA结构微环境的变化,使得部分EB从DNA复合物中游离出来,从而引起其荧光强度的降低.配合物与DNA的结合强弱还可通过如下Stern-Volmer方程式,计算淬灭常数进行比较[26].
I0/I=1+KSV.[Complex],
其中,KSV为淬灭常数,I0/I为加入和未加入配合物时EB-DNA体系的荧光强度的比值,[Complex]为体系中配合物的浓度.以加入和未加入配合物时体系的荧光强度的比值I0/I为纵坐标,[Complex]为横坐标,作出Stern-Volmer曲线,如图4(d)所示,得到配合物1~配合物3的淬灭常数KSV分别为6 422,7 398和13 977 L·mol-1.淬灭常数的大小反映了配合物与DNA的结合能力,即淬灭常数越大,结合力越大.因此,3个铜(Ⅱ)配合物与DNA的结合能力的大小顺序为3>2>1,该结论与紫外光谱所得结论一致.
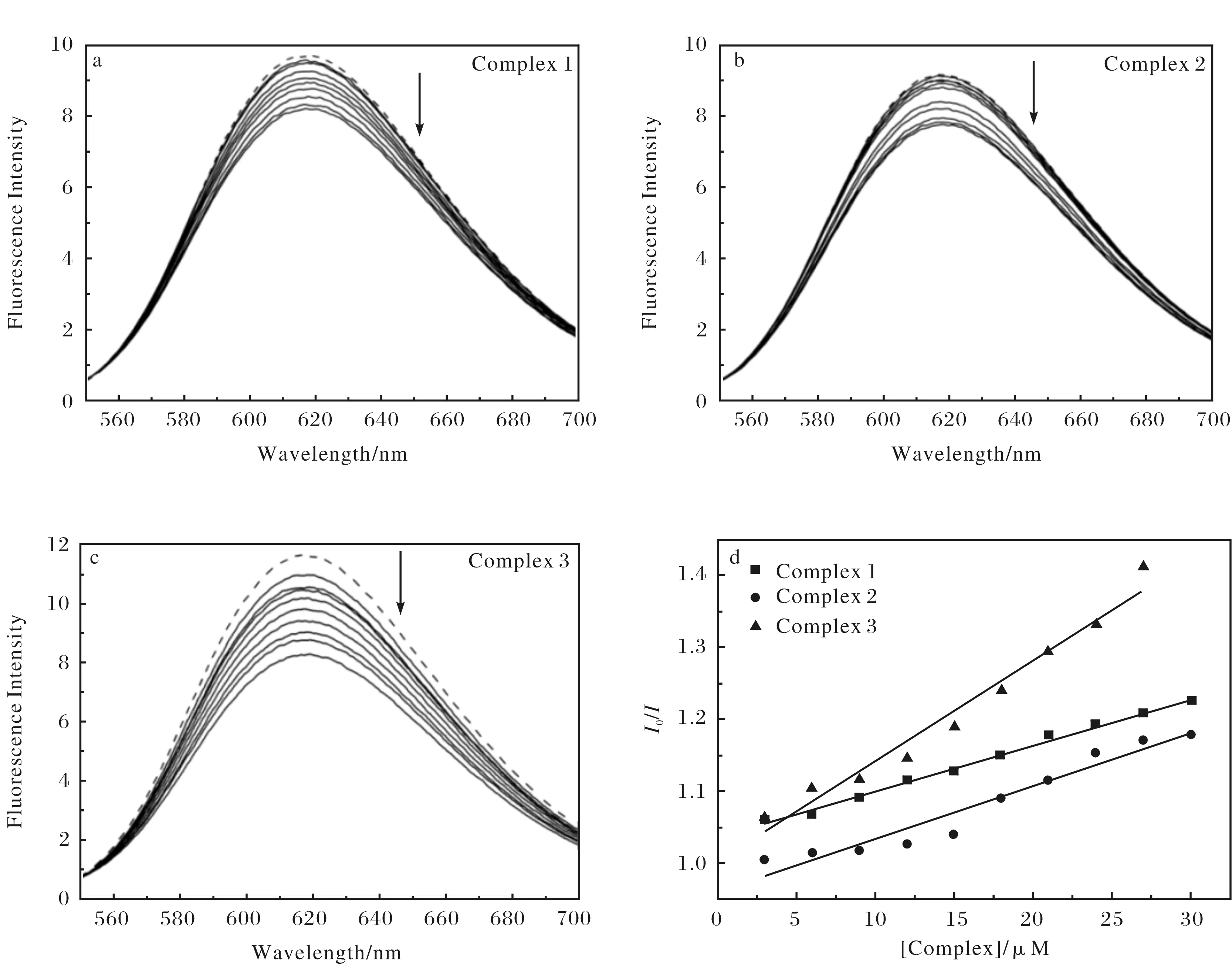
图4 EB-DNA体系在不同配合物浓度下的荧光光谱图(图中箭头表示配合物浓度逐渐增大时的变化趋势)(a~c)和配合物1~配合物3对EB-DNA 体系的荧光淬灭图(d)Fig.4 (a~c)Fluorescence emission spectra of EB-DNA system in the absence (---) and presence (-) of increasing amounts of complex 1 ~ complex 3; (d) Fluorescence quenching of EB-DNA system by complex 1 ~ complex 3
2.4圆二色谱
将不同浓度的配合物溶液分别加入到CT-DNA溶液中,测量其圆二色谱(CD)的变化,其结果如图5所示.随着配合物浓度的增加,DNA的CD谱图发生了明显的变化.245 nm处的负峰与275 nm处的正峰的强度均有明显的降低,但是没有明显的位移.说明铜(Ⅱ)配合物的加入使DNA双螺旋结构部分被破坏,双螺旋结构变得松散.结果表明,铜(Ⅱ)配合物1~3对DNA均有键合作用.
2.5DNA切割活性
在pH 7.4和37 °C,并以抗坏血酸(H2A)还原剂条件下,配合物1~配合物3均可将超螺旋pBR322 DNA(Form I,I型)切割成缺刻型(Form Ⅱ,Ⅱ型)和线型(Form Ⅲ,Ⅲ型)DNA.随着配合物浓度的增大,DNA的切割活性显著增强,I型DNA逐渐减少甚至是最终完全转变为Ⅱ型和Ⅲ型DNA,其结果如图6所示.配合物1当浓度达到40 μmol·L-1时,开始有Ⅲ型DNA产生,浓度达到48 μmol·L-1时,Ⅱ型DNA和Ⅲ型DNA同时存在,而I型DNA基本消失.配合物2在32 μmol·L-1时开始出现Ⅲ型 DNA;当浓度为40 μmol·L-1时,I型DNA完全变为Ⅱ型DNA和Ⅲ型DNA.配合物3在28 μmol·L-1时开始出现Ⅲ型 DNA;当浓度为32 μmol·L-1时,I型DNA即完全降解为Ⅱ型及Ⅲ型DNA;而当其浓度增大到48 μmol·L-1或更大时,DNA已被切割成碎片.以上结果表明,配合物1~配合物3均有良好的DNA切割活性,且3个配合物的切割活性大小顺序为3 > 2 > 1.与单核配合物1相比,双核配合物2和多聚配合物3具有更强的DNA结合能力及更好的DNA切割活性,这可能归因于在双核及多聚配合物中,两个或多个中心金属离子之间存在协同效应[27-28].一个配合物的金属中心离子数目愈多,这种协同效应可能愈明显,从而可能呈现愈强的DNA结合能力及DNA切割活性.

图5 CT-DNA在不同配合物浓度下的圆二色谱图(图中箭头表示配合物浓度逐渐增大时的变化趋势)Fig.5 CD spectra of CT-DNA in the absence (---) and presence (-) of increasing amounts of complex 1~ complex 3
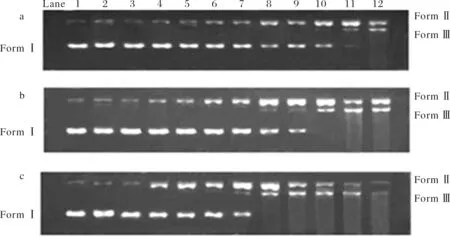
(a) 泳道1,DNA;泳道2,DNA+H2A;泳道3,DNA+配合物1 (44 μmol·L-1);泳道4~12,DNA+H2A+配合物1 (8,16,24,28,32,36,40,44,48 μmol·L-1);(b) 泳道1,DNA;泳道2,DNA+H2A;泳道3,DNA+配合物2 (44 μmol·L-1);泳道4~12,DNA+H2A+配合物2 (8,16,24,28,32,36,40,44,48μmol·L-1);(c) 泳道1,DNA;泳道2,DNA+H2A;泳道3,DNA+配合物3 (44 μmol·L-1) ;泳道4~12,DNA+H2A+配合物3 (8,16,24,28,32,36,40,44,48 μmol·L-1).图6 配合物(1~3)浓度对超螺旋pBR332DNA切割活性的影响Fig.6 Gel eletrophoresis diagram showing the cleavage of pBR322 DNA
3 结论
本文采用多种方法研究了3个不同双吡啶吡咯Cu(Ⅱ)配合物与DNA之间的相互作用.光谱研究结果表明,配合物与CT-DNA相互作用的主要模式是沟槽结合,即配合物作用在双螺旋DNA的大沟或者小沟区,引起DNA结构微环境的变化,配合物与DNA作用的强弱顺序3>2>1.3个配合物均能有效地切割超螺旋pBR322 DNA为缺刻型或者线型DNA,且随着配合物浓度的增大,切割活性显著增强.
[1] 王晓勇, 郭子建. 金属抗癌药物设计的新策略和新趋势[J]. 化学进展, 2009, 21(5): 845-855.
[2] MUHAMMAD N, GUO Z J. Metal-based anticancer chemotherapeutic agents[J]. Curr Opin Chem Biol, 2014, 19:144-153.
[3] SIGMAN D S, GRAHAM D R, AURORA V D, et al. Oxygen-dependent cleavage of DNA by the 1,10-phenanthroline. cuprous complex. Inhibition of Escherichia coli DNA polymerase I [J]. J Biol Chem, 1979, 254(24): 12269-12272.
[4] BALDINI M, BELICCHI-FERRARI M, BISCEGLIE F, et al. Copper(Ⅱ) complexes with substituted thiosemicarbazones of α-ketoglutaric acid: Synthesis, X-ray structures, DNA binding studies, and nuclease and biological activity [J]. Inorg Chem, 2004, 43(22): 7170-7179.
[5] 王 芳, 张万举, 徐志花, 等. 单核铜(Ⅱ)配合物[Cu(phen)(C2O4)]·H2O的合成、晶体结构及与DNA相互作用研究[J]. 华中师范大学学报(自然科学版),2011, 45(3): 435-438.
[6] JAYAMANI A, THAMILARASAN V, SENGOTTUVELAN N, et al. Synthesis of mononuclear Copper(Ⅱ) complexes of acyclic Schiff’s base ligands: Spectral, structural, electrochemical, antibacterial, DNA binding and cleavage activity [J]. Spectrochimica Acta Part A, 2014, 122C: 365-374.
[7] MA T L, XU J, WANG Y, et al. Ternary Copper(Ⅱ) complexes with amino acid chains and heterocyclic bases: DNA binding, cytotoxic and cell apoptosis induction properties[J]. J Inorg Biochem, 2015, 144: 38-46.
[8] LIU H K, SADLER P J. Metal complexes as DNA intercalators[J]. Accounts Chem Res, 2011, 44(5): 349-359.
[9] MANIKANDAMATHAVAN V M, UNNI N B. DNA binding and cytotoxicity of copper(Ⅱ) imidazole terpyridine complexes: Role of oxyanion, hydrogen bonding and π-π interaction[J]. Eur J Med Chem, 2013, 68: 244-252.
[10] ZHANG S C, ZHU Y G, TU C, et al. A novel cytotoxic ternary Copper(Ⅱ) complex of 1,10-phenanthroline and L-threonine with DNA nuclease activity[J]. J Inorg Biochem, 2004, 98(12): 2099-2106.
[11] ZHANG S C, ZHOU J L. Ternary Copper(Ⅱ) complex of 1,10-phenanthroline and L-glycine: crystal structure and interaction with DNA[J]. J Coord Chem, 2008, 61(15): 2488-2498.
[12] ZHANG S C, CHUN X G, CHEN Y, et al. Synthesis,crystal structure and DNA cleavage activity of a ternary Copper(Ⅱ) complex of dipyrido-quinoxaline and glycine[J]. Chin J Chem, 2011, 29(1): 65-71.
[13] PATEL M N, DOSI P A, BHATT B S. Antibacterial,DNA interaction and superoxide dismutase activity of drug based Copper(Ⅱ) coordination compounds[J]. Polyhedron, 2010, 29(17): 3238-3245.
[14] MANIKANDAMATHAVAN V M, RAJAPANDIAN V, FREDDY A J, et al. Effect of coordinated ligands on antiproliferative activity and DNA cleavage property of three mononuclear Cu(Ⅱ)-terpyridine complexes[J]. Eur J Med Chem, 2012, 57: 449-458.
[15] ALVAREZ N, VEIGA N, IGLESIAS S, et al. Synthesis,structural characterization and DNA interaction of new Copper-terpyridine complexes[J]. Polyhedron, 2014, 68: 295-302.
[16] MIN R, HU X, YI X, et al. Synthesis, structure, DNA binding and cleavage activity of a new copper(Ⅱ) complex of bispyridylpyrrolide[J]. Journal of Central South University, 2015, 22(5):1619-1625.
[17] JONES R A, KARATZA M, VORO T N, et al. Cheminform abstract: Extended heterocyclic systems. Part 1. The synthesis and characterization of pyrrolylpyridines, alternating pyrrole:Pyridine oligomers and polymers, and related systems[J]. Tetrahedron, 1996, 27(43):8707-8724.
[18] FANG W Z, WANG Y P, WANG Y F, et al. Anion-directed assembly of helical Copper(Ⅱ) complexes based on a bispyridylpyrrole ligand: synthesis,structural and magnetic properties[J]. Rsc Advances, 2015, 5: 8996-9001.
[19] MARMUR J. A procedure for the isolation of deoxyribonucleic acid from microorganisms[J]. J Mol Biol, 1961, 3: 208-218.
[20] WOLFE A, SHIMER G H, MEEHAN T. Polycyclic aromatic hydrocarbons physically intercalate into duplex regions of denatured DNA[J]. Biochemistry, 1987, 26(20): 6392-6396.
[21] ALLMANN R, HENKE W, REINEN D. Presence of a static Jahn-Teller distortion in copper(Ⅱ) terpyridine complexes 1. Crystal structure of diterpyridinecopper(Ⅱ) nitrate[J]. Inorg Chem, 1978, 17(2): 378-382.
[22] BARTON J K, DANISHEFSKY A, GOLDBERG J. Tris(phenanthroline) ruthenium(Ⅱ):stereoselectivity in binding to DNA[J]. J Am Chem Soc, 1984, 106(7): 2172-2176.
[23] NAIR R B, TENG E S, KIRKLAND S L, et al. Synthesis and DNA-binding properties of [Ru(NH3)4dppz]2+[J]. Inorg Chem, 1998, 37(1): 139-141.
[24] WARING M J. Complex formation between ethidium bromide and nucleic acids[J]. J Mol Biol, 1965, 13(1): 269-282.
[25] BOGER D L, FINK B E, BRUNETTE S R, et al. A simple,high-resolution method for establishing DNA binding affinity and sequence selectivity[J]. J Am Chem Soc, 2001, 123(25): 5878-5891.
[26] LAKOWICZ J R, WEBER G. Quenching of fluorescence by oxygen. Probe for structural fluctuations in macromolecules[J]. Biochemistry, 1973, 12(21): 4161-4170.
[27] ANBU S, KANDASWAMY M. Electrochemical, magnetic, catalytic, DNA binding and cleavage studies of new mono and binuclear copper(Ⅱ) complexes[J]. Polyhedron, 2011, 30 (1): 123-131.
[28] 张寿春, 邵 颖. 双核铜(Ⅱ)配合物在H2O2存在下对DNA的氧化切割活性[J].无机化学学报,2006, 20(9): 1733-1739.
Interaction betweeen dipyridylpyrrole copper(Ⅱ) complexes and DNA
MA Li, YI Xiaoyi, ZHANG Shouchun
(School of Chemistry and Chemical Engineering, Central South University, Changsha 410083)
A new mononuclear copper complex, [Cu(PDPH)2](1) was synthesized and characterized. The complex was crystallized in a monoclinic system with space group C2/c,a=39.1242(16) Å,b=8.6574(5) Å,c=33.7687 (14) Å, β=123.0305(16)°. The interaction of [Cu(PDPH)2] (1), dinuclear [Cu2(PDPH)2(NO3)2] (2) and polymer {[Cu2(PDPH)2(N3)2]}n(3) with CT-DNA was investigated respectively by UV-Vis absorption spectra, fluorescence spectra and circular dichroism (CD) spectra. The results indicated that these complexes bind to CT-DNA via groove binding mode, and the binding ability followed an order of 3 > 2 > 1. In addition, these copper(Ⅱ) complexes exhibited efficient DNA cleavage property and were able to promote the conversion of supercoiled DNA to nicked or linear form in the presence of ascorbate as a reducing agent.
copper(Ⅱ) complex; dipyridylpyrrole ligand; DNA-binding; DNA cleavage
2016-03-19.
国家自然科学基金项目(21571190).
1000-1190(2016)04-0551-08
O614.121
A
*通讯联系人. E-mail: zhang_shch@sina.cn.

