草鱼钠磷协同转运载体Slc34a2基因的克隆及组织表达分析
陈 沛 黄艳青 谢从新 王春芳
(1. 华中农业大学水产学院,淡水水产健康养殖湖北省协同创新中心,武汉 430070; 2. 中国水产科学研究院东海水产研究所,农业部东海与远洋渔业资源开发利用重点实验室,上海 200090)
草鱼钠磷协同转运载体Slc34a2基因的克隆及组织表达分析
陈沛1黄艳青2谢从新1王春芳1
(1. 华中农业大学水产学院,淡水水产健康养殖湖北省协同创新中心,武汉 430070; 2. 中国水产科学研究院东海水产研究所,农业部东海与远洋渔业资源开发利用重点实验室,上海 200090)
为了揭示草鱼对磷的吸收机制,运用RT-PCR和快速扩增cDNA末端方法,从草鱼(Ctenopharyngodon idella)肠中克隆获得钠磷协同转运载体基因Slc34a2,该基因全长为2446 bp,包含了1938 bp的开放阅读框,47 bp的5′非编码区(Untranslated region,UTR)和461 bp的3′UTR,编码645个氨基酸。草鱼SLC34A2蛋白的分子式为C3215H5125N801O902S30,分子量大小为70.39 kD,等电点为5.68,总平均疏水指数为0.458。对草鱼SLC34A2蛋白结构和功能预测分析,发现SLC34A2蛋白有11个跨膜域,1个半胱氨酸富集区,且N-端在胞外而C-端在胞内,也在第二个细胞外环中发现4个N-糖基化位点。用邻接法构建系统进化树,发现草鱼Slc34a2基因与硬骨鱼类聚类为一支,且草鱼SLC34A2蛋白与鲤(Cyprinus carpio)和斑马鱼(Danio rerio) SLC34A2的相似性最高,分别为90.3%和87.0%。实验采用了实时荧光定量PCR对草鱼Slc34a2 mRNA进行组织表达分析,结果表明Slc34a2 mRNA在组织中广谱表达,且在肠中表达最高,其次是肝脏、鳃、肾脏、脾脏、皮肤、肌肉、脑和头肾。实验为以后研究提高鱼对磷的利用和减少磷的排放奠定分子基础。
草鱼;Slc34a2;分子克隆;基因表达
磷是核酸(ATP和GTP)、中间代谢(如:蛋白质磷酸化、高能转运载体和血液缓冲)、细胞内信号通路、细胞增殖(DNA和RNA)、细胞膜的磷脂成分和骨骼结构所必须的一种矿物质[1]。目前在商业饲料中无机磷的添加量普遍较高,大约每吨饲料含有20—25 kg的Ca(H2PO4)2·H2O,且在投喂的饲料中约有10%—20%直接进入水环境而未被摄取,而在被摄食的饲料中约有40%磷被鱼吸收和存储,剩下60%磷会排入水环境[2,3]。由于存在鱼类对饲料利用率较低、残饵量较高的情况,这将导致大量的磷排入养殖水体[2,4]。当水域环境中磷含量过高时,水体很容易发生富营养化,结果导致环境严重的污染。
现在很多研究表明通过饲料配方的优化和合适的投喂策略可以提高鱼体对磷的利用率和减少磷的排放[5—9]。然而,由于饲料成分、水产养殖操作和鱼类物种的差异致使传统的策略对提高磷的利用率有一定的局限性。因此,本文运用RT-PCR和快速扩增cDNA末端(Rapid amplification of cDNA ends,RACE)技术,从草鱼(Ctenopharyngodon idella)中获得了钠磷协同转运载体基因Slc34a2,为揭示草鱼对磷的吸收机制提供一定的基础。
鱼吸收磷是通过被动扩散和主动转运[2],维持体内磷的平衡是通过肠的吸收和肾脏的重吸收[10]。Brichon[11]最早在欧洲鳗鲡(Anguilla anguilla L.)的肠中观察到了磷的主动转运,并表明了这一转运符合米氏(Michaelis-Menten)动力学。随后,一个主动协同转运系统在鲤(Cyprinus carpio)中被发现[12]。早期研究表明,在肾脏近端小管和小肠,机体对磷的主动吸收主要依靠IIb型钠磷协同转运载体(NaPi-IIb,SLC34A2)[13,14]。在哺乳动物中,II型钠磷协同转运载体家族(SLC34)有3个成员:NaPi-IIa(SLC34A1)、NaPi-IIb和NaPi-IIc(SLC34A3),其中SLC34A1和SLC34A2分别在肾近端小管和肠道上皮细胞中被发现[15]。而在鱼类中只有1种SLC34,即SLC34A2[16]。但是,到目前为止关于鱼类Slc34a2基因的研究还不多,只在斑马鱼(Danio rerio)[17,18]、虹鳟(Oncorhynchus mykiss)[19]、比目鱼(Pseudopleuronectes americanus)[16]、黄颡鱼(Pelteobagrus fulvidraco)[20]和其他鱼类[21]中获得了几条Slc34a2的cDNA全长或部分序列。此外,在这些鱼类中都没有Slc34a2基因详细的功能描述。草鱼作为我国重要的淡水鱼类养殖品种之一,具有生长快、饲料蛋白需求低和肉质好等优点。本文克隆草鱼Slc34a2基因全长,研究了其结构和功能的特征,组织表达的分布,为以后研究提高鱼对磷的利用和减少磷的排放奠定分子基础。
1 材料与方法
1.1实验材料
健康的草鱼(体重约35—50 g)购自湖北省武汉市白沙洲大市场,试验前暂养于华中农业大学水产实验创新中心实验室内1周。实验期间,每天于8:30 和16:30投喂商品颗粒饲料,并在投喂前换去1/3的水。1周后,随机抽取5尾草鱼,麻醉后分别取皮肤、肌肉、鳃、肝脏、脑、脾脏、肾脏、肠和头肾组织,放入装有Trizol(Invitrogen,美国)的2 mL EP管中,并迅速转移到液氮罐中冻存备用。
1.2试验方法
总RNA提取和cDNA合成试验用的器具经过0.1% DEPC(Diethypyro-carbonate,焦碳酸二乙酯)水浸泡过夜、双蒸水冲洗、高温高压灭菌。按照Trizol试剂说明书进行各组织样品总RNA的提取,分别用Nanodrop 2000C(Thermo Fisher Scientific,美国) 分光光度计和1.5%(w/v) 琼脂糖凝胶电泳检测RNA的浓度和完整性。利用SMARTTMRACE cDNA Amplification Kit Advantage®PCR Kit & Polymerase Mix试剂盒(Clontech,美国)合成RACE模板,具体方法参考试剂盒说明书。合成的cDNA都在-20℃保存。
草鱼Slc34a2 cDNA全长克隆根据发表在NCBI上已知的鲤(Cyprinus carpio,AAG35803.1)、鳑鲏(Tanakia limbata,EU647761.1)和斑马鱼(NP_878297.1)的Slc34a2基因序列,用ClustalW法比对序列并在其保守区用 Primer Premier 6.0和Oligo7.0软件设计1对兼并引物Slc34a2-F和Slc34a2-R(表1)。以草鱼肠cDNA为模板,Slc34a2-F和Slc34a2-R为引物进行PCR扩增。25 μL PCR反应体系中含有:2 μLcDNA模板,12.5 μL Premix TaqTM(0.05 U/μL)(TaKaRa,日本)酶,上下游引物各1 μL(10 μmol/L)和8.5 μL双蒸水。轻弹PCR管底,混匀样品,瞬时离心。PCR扩增程序:94℃预变性3min;94℃变性30s,56℃退火30s,72℃延伸30s,35个循环;延伸7min。扩增完成后,取5 μL反应产物,在 1.5%(w/v)的琼脂糖凝胶上以 200 V恒定电压电泳检测,对照 2000 bp DNA Marker(TaKaRa)验证扩增结果。参照DNA胶回收试剂盒(Invitrogen)说明书回收、纯化扩增产物,并连接至pMD19-T载体(TaKaRa),重组质粒转化至E.coli DH5α感受态(TaKaRa),经菌落PCR初步鉴定为阳性克隆后由上海生工生物有限公司双向测序。以草鱼肠5′和3′RACEcDNA为模板,根据RACE 扩增试剂盒说明分别扩增5′端和3′端序列,然后对所得序列进行拼接,获得其全长cDNA序列。

表1 本文中所用引物序列Tab. 1 Sequences of primers used in the present study
生物信息学分析利用DNAStar中的EdiSeq程序进行开放阅读框(ORF)的预测与氨基酸翻译,SeqMan程序进行cDNA的拼接; 应用ProtParam tool(http://www.expasy.org/)对其编码蛋白质的分子量、等电点等基本性质进行理论预测; 应用SignalP 4.1 Server(http://www.cbs.dtu.dk/services/ SignalP/)对其进行信号肽预测; 应用TMHMM Server v.2.0(http://www.cbs.dtu.dk/services/ TMHMM/)对蛋白质的跨膜区进行预测; 应用ClustalW软件对其同源蛋白质进行比对分析; 应用MEGA 6.0软件,分别采用邻接法(Neighbor-joining)对同源蛋白质进行系统进化树构建,利用自展法(Bootstrap)进行1000次重复,对系统树进行可信度评估,其余参数为默认。
Slc34a2 mRNA在不同组织中表达分析根据Slc34a2基因保守区域设计特异的定量引物为Slc34a2-qF和Slc34a2-qR(表1),而选取草鱼β-actin(DQ211096.1)作为内参基因,引物为β-actin-qF和βactin-qR(表1)。cDNA模板利用EASY Dilution(TaKaRa)稀释10倍后,取其2 μL作为实时荧光定量PCR(qPCR)的模板,用Rotor-Gene Q(QIAGEN,德国)平台进行实验。用目的基因和β-actin的cDNA作为模板分别来制备相应的标准曲线[22],然后进行目的基因的定量实验。20 μL的反应体系包括2 μL cDNA、上,下游引物各0.5 μL(10 μmol/L)、10 μL的SYBR®Premix Ex TaqTMII(2×)(TaKaRa)和7 μL双蒸水。反应条件为95℃预变性30s; 95℃变性5s,60℃退火20s,72℃延伸20s,35次循环; 然后进行熔解曲线的扩增。标准曲线结果显示目的基因和内参基因的R2值分别为0.999和0.998,相应的扩增效率(E)为92%和93%。
Slc34a2基因相对表达量的数据运用2-ΔΔCt方法[23]计算,其数值用平均值±标准误(Mean ± SEM)表示。数据分别经过正态性检验和方差齐性检验后,采用SPSS 21.0 软件进行One-Way方差分析(ANOVA),使用Duncan's multiple-range test进行相对表达分析,P<0.05表示存在显著性差异。
2 结果
2.1草鱼Slc34a2基因全长cDNA的克隆与序列分析
草鱼Slc34a2的cDNA全长为2446 bp(登录号为KM111528.1),包括1938 bp的开放阅读框,47 bp的 5′非编码区(Untranslated region,UTR),461 bp的3′UTR,poly(A)(多聚腺苷酸)尾巴,1个加尾信号(AATAAA)和1个mRNA不稳定基序(ATTTA)。推导草鱼SLC34A2蛋白的分子式为C3215H5125N801O902S30、分子量大小为70.39 kD、等电点为5.68、总平均疏水指数为0.458。通过TMHMM软件对该蛋白结构域进行预测,结果发现了11个跨膜域(Transmembrane domains,TMDs),分别为M1(aa 83—105)、M2(aa 118—140)、M3(aa160—182)、M4(aa 202—224)、M5(aa 342—364)、M6(aa 387—409),M7(aa 414—431)、M8(aa 438—455)、M9(aa 487—465)、M10(aa 508—530)和M11(aa 534—453)(图1); SLC34A2蛋白的N-端在胞外而C-端在胞内(图1)。
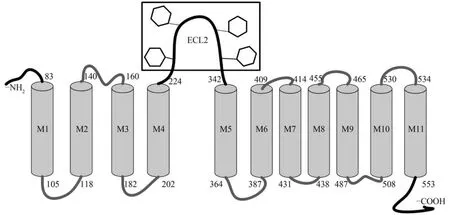
图1 预测的草鱼SLC34A2蛋白的二级结构模型Fig. 1 Predicted secondary structure model of grass carp SLC34A2在第二个细胞外环中的六边形表示N-糖基化位点Hexagon in second extracellular loop indicates N-glycosylation sites
2.2SLC34A2氨基酸序列比较分析
通过BLAST 比对10条硬骨鱼类的SLC34A2氨基酸序列,结果发现草鱼SLC34A2氨基酸序列与鲤相似度最高为90.3%、与斑马鱼的相似度为87%,与青鳉(Oryzias latipes,XP_004068374.1)和比目鱼(AAB16821.1)相似度分别为72.1%和70.2%,与伯氏朴丽鱼(Haplochromis burtoni,XP_005946750.1)、尼罗罗非鱼(Oreochromis niloticus,XP_0034479 62.2)、斑马宫丽鱼(Maylandia zebra,XP_004567 970.1)、墨西哥脂鲤(Astyanax mexicanus,XP_ 007239743.1)、黄颡鱼(ADM18964.2)的相似度分别为67.7%、67.1%、67.1%、66.5%和64.5%。
多重序列比较发现,在10个硬骨鱼类SLC34A2中第一个细胞内环(First intracellular loop,ICL-1)和第三个细胞外环(Third extracellular loop,ECL-3)非常的保守,而最大的不同是在ECL-2,N-端和C-端的区域。在草鱼SLC34A2蛋白最大的细胞外环ECL-2中发现有4个N-连接糖基化位点(N-X-S/T位于N-275,293,301和320); 其中3个N-连接糖基化位点(N-275,293和320)在10个物种间非常的保守,而N-301位点仅仅没有在青鳉SLC34A2蛋白中发现。除此之外,在C-端发现了一段非常保守的半胱氨酸富集区和PDZ结合基序X-T-X-L/F。
2.3草鱼SLC34A2系统进化树的构建
在NCBI中选取了17种已知或预测的SLC34A2蛋白序列来构建系统进化树,结果发现这些SLC34A2蛋白在进化上可分为三大支,在分类地位上分别属于软体动物类(Mmolluscs)、哺乳类(Mammals)和硬骨鱼类(Bony fishes)(图2)。草鱼SLC 34A2在进化上与鲤亲缘关系最近,与斑马鱼亲缘关系次之。
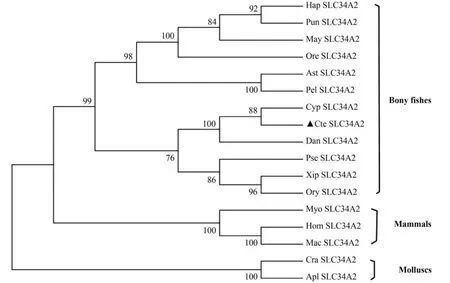
图2 SLC34A2氨基酸序列的系统进化树Fig. 2 The phylogenetic tree of SLC34A2 amino acid sequences Hap. Haplochromis burtoni(XP_005946750.1); Pun. Pundamilia nyererei(XP_005747132.1); May. Maylandia zebra(XP_004567970.1);Ore. Oreochromis niloticus(XP_003447962.2); Ast. Astyanax mexicanus(XP_007239743.1); Pel. Pelteobagrus fulvidraco(ADM18964.2);Cte. Ctenopharyngodon idellus(AIS39798.1); Da. Danio rerio(NP_878297.1); Ps. Pseudopleuronectes americanus(AAB16821.1); Xip. Xiphophorus maculatus(XP_005812800.1); Ory. Oryzias latipes(XP_004068374.1); Myo. Myotis davidii(ELK24687.1); Hom. Homo sapiens(XP_007239743.1); Cyp. Cyprinus carpio(AAG35803.1); Mac. Macaca fascicularis(EHH53597.1); Cr. Crassostrea gigas(EKC36780.1); Apl. Aplysia californica(XP_005107621.1)
2.4草鱼Slc34a2 mRNA在不同组织中的表达
采用了qPCR方法分析了Slc34a2 mRNA在草鱼组织中的表达,结果表明Slc34a2 mRNA在组织中广谱表达,且在各种组织中有显著性差异(P<0.05);Slc34a2 mRNA在肠中表达量最高,其次是肝脏、鳃、肾脏、脾脏、皮肤、肌肉、脑和头肾(图3)。
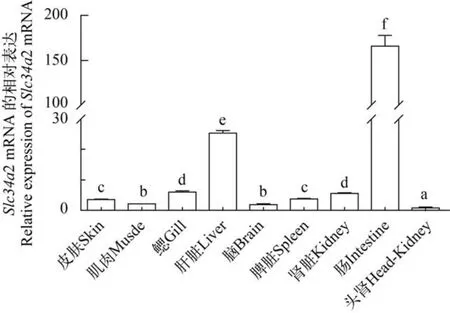
图3 草鱼Slc34a2 mRNA在组织中的分布Fig. 3 The expression of Slc34a2 in tissues of grass carp数据用平均值±标准误表示,不同的小写字母表示有显著性差异(P<0.05)Values are means ± SEM(n = 3). Bars with different lowercase letters are significantly different(P<0.05)
3 讨论
在哺乳动物中II型钠磷协同转运载体家族(SLC34)有3个成员:NaPi-IIa(SLC34A1)、NaPi-IIb(SLC34A2)和NaPi-IIc(SLC34A3),在鱼类中SLC34家族只有一个亚型为NaPi-IIb(SLC34A2)[14,15],而在本研究中只获得了一条编码645个氨基酸的草鱼Slc34a2基因。
3.1草鱼SLC34A2蛋白结构分析
先前的研究提出了真核生物SLC34蛋白的二级拓扑学结构应该由8个TMDs组成[13]。然而,Forster等[24]和Lambert等[25]表明哺乳动物的NaPi-IIa蛋白拓扑学结构最可能由8—12个TMDs组成,随后Radanovic等[10]提出了一个由12个TMDs组成的小鼠(Rattus norvegicus) NaPi-IIa拓扑模型。与哺乳动物不同的是,草鱼SLC34A2蛋白是由11个TMDs组成,这点与黄颡鱼肠SLC34A2A2蛋白结构相似[20]。草鱼SLC34A2蛋白的N-端和C-端分别在胞外和胞内,且在ECL-2有4个N-糖基化位点。在当前的研究中,N-连接的糖基化位点已经被视为SLC34A2蛋白的特征之一[10],并参与顶端膜中Slc34a2基因表达的调控[26]。也有研究表明,小鼠肠道SLC34A2是一种醇蛋白,且其糖基化与鼠的年龄有一定的关系[27,28]。
3.2SLC34A2氨基酸序列比较分析
通过序列比较分析了10种硬骨鱼的SLC34A2氨基酸序列,结果发现,ICL-1和ECL-3非常的保守,而主要区别是位于ECL-2,N-端和C-端区域。Murer等[15]发现ICL-1和ECL-3可能是运输通道的重要功能区域。SLC34A2蛋白的C-端对调控膜的运输和定位有重要的作用[29]。预测草鱼SLC34A2氨基酸序列发现在C-端有一个保守PDZ结合基序A-T-HL,该基序通过与PDZ蛋白质的交互作用来影响膜顶端转运蛋白的表达[30,31]。Forster等[24]在NaPi-IIb蛋白中发现了半胱氨酸富集区,而在NaPi-IIa和NaPi-IIc中没有发现。在NaPi-IIb类型的转运蛋白中,半胱氨酸残基是非常保守的,且在其他膜蛋白中不存在[32]。氨基酸序列比较分析也表明在硬骨鱼NaPi-IIb中半胱氨酸残基是非常很保守[20]。在非洲爪蟾蜍(Xenopus laevis)的卵母细胞中,半胱氨酸残基对NaPi-II转运蛋白的功能表达是必须的[32]。Hernando等[33,34]表明NaPi-II转运载体的C-端区域存在许多能识别不同细胞背景的结合位点。有趣的是,NaPi-IIb亚型在Madin-Darby狗肾细胞(MDCK)是严格限于刷状缘,而NaPi-IIa分布在顶端和基底膜[34]。为了研究半胱氨酸残基的作用,McHaffie等[32]通过小鼠NaPi-IIb构造体瞬变地和稳定地转染到MDCK的实验,表明了NaPi-IIb构造体的半胱氨酸残基具有棕榈酰化的作用和影响基底膜的传递和分拣的功能,这可能会导致细胞内部或顶端膜上物质的滞留。综上所述,我们可以推测在C-端的半胱氨酸富集区可以作为肠SLC34A2蛋白结构的标志性特征之一。
3.3草鱼SLC34A2组织表达
不同种类鱼的其他Slc34a2亚型也被报道。通过Northern和qPCR技术,Slc34a2的组织分布在不同鱼中展现出不同的结果。例如,比目鱼肠的Slc34a2 mRNA最初观察到是在大脑、性腺、心脏或肾脏组织中[16,35]。在斑马鱼中,两种Slc34a2(NaPi-IIb1和NaPi-IIb2)被克隆,NaPi-IIb1在肠、肾脏和眼睛中表达[17],而NaPi-IIb2的表达在其他组织中普遍存在[18]。虽然NaPi-IIb1和NaPi-IIb2的结构相似,但是功能却不同; NaPi-IIb2对电压敏感,对Na+和有较高的亲和力,但对pH不敏感[17]。在黄颡鱼的肠和肾脏中也分别克隆到了不同亚型的Slc34a2,即肠Slc34a2a1和Slc34a2a2,肾Slc34a2b,肠Slc34a2a1和Slc34a2a2主要在肠中表达,其他的组织中表达很低,而肾Slc34a2b主要分布在肾脏中,其次是肌肉和皮肤[20]。在虹鳟中,3个亚型Slc34a2分别在小肠、幽门盲和肾脏中被发现[36]。而本文通过qPCR表明Slc34a2在多种组织中广谱表达,且在肠中表达最高,其次是肝脏。Slc34a2在皮肤中表达表明鱼可能通过皮肤来吸收水中的无机磷[21,37]。综上所述,鱼类Slc34a2基因(NaPi-IIb)有几个亚型,而其在组织中的广泛表达表明了Slc34a2对调控机体磷酸盐平衡有着非常重要的作用。
[1]National Research Council(NRC). Nutrient Require-ments of Fish and Shrimp [M]. Washington,DC:National Academy Press. 2011,168—169
[2]Wang C F,Tang Q,Duan M M,et al. Phosphorus absorption and homeostasis regulation in fish [J]. Freshwater Fisheries,2014,44(1):106—111 [王春芳,唐琴,段鸣鸣,等. 鱼类磷吸收和磷平衡调节的机制及影响因素. 淡水渔业,2014,44(1):106—111]
[3]Sugiura S H,Marchant D D,Kelsey K,et al. Effluent profile of commercially used low-phosphorus fish feeds [J]. Environmental Pollution,2006,140(1):95—101
[4]Tang Q,Wang C,Xie C,et al. Dietary available phosphorus affected growth performance,body composition,and hepatic antioxidant property of juvenile yellow catfish Pelteobagrus fulvidraco [J]. Scientific World Journal,2012,2012(7):987570—987570
[5]Yang Y H,Wang Y Y,Lu Y,et al. Effect of replacing fish meal with soybean meal on growth,feed utilization and nitrogen and phosphorus excretion on rainbow trout(Oncorhynchus mykiss) [J]. Aquaculture International,2011,19(3):405—419
[6]Cho C Y,Bureau D P. A review of diet formulation strategies and feeding systems to reduce excretory and feed wastes in aquaculture [J]. Aquaculture Research,2001,32(s1):349—360
[7]Talbot C,Hole R. Fish diets and the control of eutrophication resulting from aquaculture [J]. Journal of Applied Ichthyology,1994,10(4):258—270
[8]Rodehutscord M. Response of rainbow trout(Oncorhynchus mykiss) growing from 50 to 200 g to supplements of dibasic sodium phosphate in a semi-purified diet[J]. Journal of Nutrition,1996,126(1):324—331
[9]Hernández A,Satoh S,Kiron V. Effect of monocalcium phosphate supplementation in a low fish meal diet for rainbow trout based on growth,feed utilization,and total phosphorus loading [J]. Fisheries Science,2005,71(71):817—822
[10]Radanovic T,Gisler S M,Biber J,et al. Topology of the type Ⅱa Na+/P(i) cotransporter [J]. The Journal of Membrane Biology,2006,212(1):41—49
[11]Brichon G. Phosphorus absorption by the intestine of the eel(Anguilla anguillaL). 1. Demonstration and characteristics of in vitro phosphate ion transport in the fresh water eel [J]. Comptes Rendus Des Séances De La Société De Biologie Et De SesFiliales,1973,167(8):1142—1145
[12]Nakamura Y. Sodium-dependent absorption of inorganic phosphate by the carp intestine [J]. Comparative Biochemistry & Physiology A Comparative Physiology,1985,80(3):437—439
[13]Wagner C A,Hernando N,Forster I C,et al. The SLC34 family of sodium-dependent phosphate transporters [J]. PflügersArchiv-European Journal of Physiology,2014,466(1):139—153
[14]Werner A,Kinne R K. Evolution of the Na-P(i) cotransport systems [J]. American Journal of Physiology-Regulatory Integrative and Comparative Physiology,2001,280(2):301—312
[15]Murer H,Forster I,Biber J. The sodium phosphate cotransporter family SLC34 [J]. PflügersArchiv-European Journal of Physiology,2004,447(5):763—767
[16]Werner A,Murer H,Kinne R K. Cloning and expression of a renal Na-Pi cotransport system from flounder [J]. American Journal of Physiology-Renal Physiology,1994,267(2):311—317
[17]Nalbant P,Boehmer C,Dehmelt L,et al. Functional characterization of a Na+-phosphate cotransporter(NaPi-Ⅱ)from zebrafish and identification of related transcripts [J]. Journal of Physiology,1999,520(1):79—89
[18]Graham C,Nalbant P,Scholermann B,et al. Characterization of a type Ⅱbsodium-phosphate cotransporter from zebra fish(Danio rerio) kidney [J]. American Journal of Physiology Renal Physiology,2003,284(4):727—736
[19]Sugiura S H,Ferraris R P. Contributions of different NaPi cotransporter isoforms to dietary regulation of P transport in the pyloric caeca and intestine of rainbow trout [J]. Journal of Experimental Biology,2004,207(12):2055—2064
[20]Chen P,Tang Q,Wang C. Characterizing and evaluating the expression of the type Ⅱb sodium-dependent phosphate cotransporter(slc34a2) gene and its potential influence on phosphorus utilization efficiency in yellow catfish(Pelteobagrus fulvidraco) [J]. Fish Physiology & Biochemistry,2015,42(1):1—14
[21]Sugiura S. Identification of intestinal phosphate transporters in fishes and shellfishes [J]. Fisheries Science,2009,75(1):99—108
[22]Deer D M,Lampel K A,González-Escalona N. A versatile internal control for use as DNA in real-time PCR and as RNA in real-time reverse transcription PCR assays [J]. Letters in Applied Microbiology,2010,50(4):366—372
[23]Pfaffl M W. A new mathematical model for relative quantification in real-time RT-PCR [J]. Nucleic Acids Research,2001,29(9):2002—2007
[24]Forster I C,Köhler K,Biber J,et al. Forging the link between structure and function of electrogenic cotransporters:the renal type Ⅱa Na+/Pi cotransporter as a case study [J]. Progress in Biophysics & Molecular Biology,2002,80(3):69—108
[25]Lambert G,Traebert M,Hernando N,et al. Studies on the topology of the renal type Ⅱ NaPi-cotransporter [J]. Pflügers Archiv-European Journal of Physiology,1999,437(6):972—978
[26]Hayes G,Busch A,Lötscher M,et al. Role of N-linked glycosylation in rat renal Na/Pi-cotransport [J]. Journal of Biological Chemistry,1994,269(39):24143—24149
[27]Arima K,Collins J F,Hines E R,et al. Molecular cloning of murine sodium-phosphate cotransporter type Ⅱb(Na/Pi-Ⅱb) gene promoter and characterization of gene structure 1 [J]. Biochimica et Biophysica Acta-Gene Structure and Expression,2000,1494:149—154
[28]Arima K,Hines E R,Kiela P R,et al. Glucocorticoid regulation and glycosylation of mouse intestinal type Ⅱb Na-Pi cotransporter during ontogeny [J].American Journal of Physiology Gastrointestinal and Liver Physiology,2002,283(2):426—434
[29]Murer H,Hernando N,Forster L,et al. Molecular mechanisms in proximal tubular and small intestinal phosphate reabsorption(Plenary Lecture) [J]. Molecular Membrane Biology,2001,18(1):3—11
[30]Fanning A S,Anderson J M. Protein-protein interactions:PDZ domain networks [J]. Current Biology,1996,6(11):1385—1388
[31]Gisler S M,Stagljar I,Traebert M,et al. Interaction of the type Ⅱa Na/Pi cotransporter with PDZ proteins [J]. Journal of Biological Chemistry,2001,276(12):9206—9213
[32]Mchaffie G S,Graham C,Kohl B,et al. The role of an intracellular cysteine stretch in the sorting of the type ⅡNa/phosphate cotransporter [J]. Biochimica et Biophysica Acta-Biomembranes,2007,1768(9):2099—2106
[33]Hernando N,Karim-Jimenez Z,Biber J,et al. Molecular determinants for apical expression and regulatory membrane retrieval of the type Ⅱa Na/Pi cotransporter [J]. Kidney International,2001,60(2):431—435
[34]Hernando N,Sheikh S,Karim-Jimenez Z,et al. Asymmetrical targeting of type Ⅱ Na-P(i) cotransporters in renal and intestinal epithelial cell lines [J]. American Journal of Physiology Renal Physiology,2000,278(3):361—368
[35]Kohl B,Herter P,Hülseweh B,et al. Na-Pi cotransport in flounder:same transport system in kidney and intestine[J]. American Journal of Physiology Renal Physiology,1996,270(6):937—944
[36]Sugiura S H,Kelsey K,Ferraris R P. Molecular and conventional responses of large rainbow trout to dietary phosphorus restriction [J]. Journal of Comparative Physiology B,2007,177(4):461—472
[37]Møbjerg N,Werner A,Hansen S M,et al. Physiological and molecular mechanisms of inorganic phosphate handling in the toad Bufobufo [J]. PflügersArchiv-European Journal of Physiology,2007,454(1):101—113
MOLECULAR CHARACTERIZATION AND TISSUE DISTRIBUTION OF SODIUM-DEPENDENT PHOSPHATE COTRANSPORTER GENE(SLC34A2) IN GRASS CARP CTENOPHARYNGODON IDELLA
CHEN Pei1,HUANG Yan-Qing2,XIE Cong-Xin1and WANG Chun-Fang1
(1. Freshwater Aquaculture Collaborative Innovation Center of Hubei Province,the College of Fisheries,Huazhong Agricultural University,Wuhan 430070,China; 2. Key Laboratory of East China Sea and Oceanic Fishery Resources Exploitation and Utilization,Ministry of Agriculture,East China Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Shanghai 200090,China)
To reveal the mechanism of grass carp(Ctenopharyngodon idella) on absorbing phosphate,a sodium-dependent phosphate cotransporter gene(Slc34a2) was cloned from grass carp intestine. The full-length cDNA of Slc34a2 was consisted of 2446 bp with a 1938 bp open reading frame,a 47 bp 5′UTR(untranslated region) and a 461 bp 3′UTR,which encoded 645 amino acids with an estimated formula of C3215H5125N801O902S30that has 70.39 kD molecular weight,a 5.68 isoelectric point,and 0.458 grand average of hydropathicity. The putative grass carp SLC34A2 protein had eleven membrane-spanning domains with the extracellular N-termini and intracellular C-termini and acysteinerich region in C-termini domain as well as four N-glycosylation sites in second extracellular loop. The phylogenetic tree based onneighbor-joining method revealed that the SLC34A2 of grass carp was clustered with other teleost fish. The deduced amino acid sequence showed 90.3% and 87.0% sequence identity to Cyprinus carpio and Danio rerio,respectively. The highest level of Slc34a2 mRNA was in the intestine,followed by the liver,gill,kidney,spleen,skin,muscle,brain and head-kidney. Through cloning the full-length of Slc34a2 grass carp,this paper studied the characteristics of SLC34A2 structure,function and tissues distribution for laying the molecular foundation for further efforts to improve phosphate utilization and minimize the excretion of phosphorus.
Grass carp; Slc34a2; Molecular characterization; Gene expression
S965.1;Q344+.1
A
1000-3207(2016)05-0879-07
10.7541/2016.113
2015-08-31;
2016-01-06
国家自然科学基金(31172421); 现代农业产业技术体系建设专项基金(CARS-46)资助 [Supported by the National Natural Science Foundation of China(31172421); the Earmarked Fund for China Agriculture Research System(CARS-46)]
陈沛(1990—),男,湖北监利人; 硕士研究生; 主要研究方向为分子营养学。E-mail:chenpei879368301@126.com
王春芳(1975—),主要研究方向为鱼类营养与环境。E-mail:cfwang@mail.hzau.edu.cn

