芋头过氧化物酶基因克隆
莫俊杰,梁钾贤,胡汉桥,黄 红,陈 妤
(广东海洋大学农学院,广东 湛江 524088)
芋头过氧化物酶基因克隆
莫俊杰,梁钾贤,胡汉桥,黄 红,陈 妤
(广东海洋大学农学院,广东 湛江524088)
根据芋头的密码子使用频率,用 iCODEHOP 设计简并引物扩增芋头过氧化物酶基因序列,待芋头过氧化物酶基因非侧翼序列扩增成功后,依据其序列合成 hiTAIL-PCR 引物扩增芋头过氧化物酶基因的两侧序列。结果从芋头基因组DNA中克隆出 1 165 bp序列,经同源性分析为过氧化物酶基因的部分序列;再利用hiTAIL-PCR技术可以扩增出芋头过氧化物酶基因的两侧序列,扩增序列为 689 bp,测序结果能与1 165 bp序列拼接到一起,长1 854 bp。与已知的过氧化物酶进行序列比对,发现两侧还有氨基酸的编码序列,因此进行第2次hiTAIL-PCR,扩增出165 bp芋头过氧化物酶基因的侧翼序列,拼接后得到2 019 bp。用Softberry上的基因预测软件分析后,发现包含过氧化物酶4个外显子,同源性分析表明,此过氧化物酶基因为含血红素的第三类过氧化物酶。
芋头;过氧化物酶基因;hiTAIL-PCR;克隆
莫俊杰,梁钾贤,胡汉桥,等. 芋头过氧化物酶基因克隆[J].广东农业科学,2016,43(9):37-43.
芋头(Colocasia esculenta)主要种植于热带地区,在东南亚和太平洋地区是一种重要的经济作物。2016年国际粮农组织报告,2004—2014年期间,我国的芋头年均生产量居全球第2位,仅次于尼日利亚。然而,芋疫病是芋头生产中的重要病害[1-2]。该病流行性强,破坏性大,主要为害叶片,也为害叶柄及块茎。大量研究表明,过氧化物酶活性与植物的抗病性具有正相关关系[3]。因此,揭示芋头耐逆的分子机理、克隆与逆境胁迫相关的重要功能基因对芋头耐逆性和广适应性的基因工程改良及扩大芋头生产具有重要意义。
获得一个新基因的方法很多,多数是采用建立文库的方法来筛选,但从文库中筛选一个特定的基因比较困难。从细菌中构建的DNA文库至少有数万个克隆,如何从众多克隆中获得目的基因,始终是DNA文库筛选工作的一个难点。虽然有许多有效的筛选方法,但仍未能真正克服文库筛选工作量大的问题。现代生物信息学飞速发展,很好地解决了这一难题[4]。通过在GenBank中查询与目的基因相关的序列,找出其保守区。据此设计出简并引物,从而快速地克隆出目的基因片段。事实证明,CODEHOPs(Consensus-degenerate hybrid oligonucleotide primers)是鉴定新基因的非常有效的方法[5-6],而且利用iCODEHOP在线设计简并引物非常方便[7-8]。
TAIL-PCR(thermal asymmetric interlaced PCR)是建立在PCR技术基础上的染色体步移技术,为扩增已知序列旁侧的未知DNA片段提供了捷径[9]。该技术以基因组DNA为模板,使用高退火温度的长特异引物和低退火温度的短简并引物,通过特殊的热不对称(高严谨性PCR和低严谨性PCR交替)循环程序,有效扩增特异产物。该技术具有简单快速、特异性高、分离出的DNA序列可以用于图位克隆、遗传图谱绘制和直接测序等优点,近年来,被分子生物学研究者广泛应用,成为分子生物学研究中非常实用的基因侧翼序列克隆技术[10-11],同时在植物基因克隆方面也取得了很大进展。
hiTAIL-PCR(high-efficiency thermal asymmetric interlaced PCR)是 TAIL-PCR进一步改良的方法,它能够阻止非目标产物的扩增和抑制小目标产物,但是允许大目标序列的有效扩增。经测试,反应的成功率超过90%,在大多数情况下获得的主要产物片段为1~3 kb[12]。hiTAIL-PCR技术非常实用[13-16]。例如,郭翠等[14]通过一次hiTAIL-PCR和一次长链PCR扩增方法获得了转抗草甘膦基因玉米品系D-3的外源DNA插入片段的全DNA序列及两端侧翼序列,并建立了转化体特异性PCR检测方法;赵静等[15]以海州香薷基因组DNA为模板,通过hiTAIL-PCR和walking技术扩增得到其细胞壁转化酶基因启动子片段,长度为1 727 bp;廖小芳等[16]利用hiTAIL-PCR技术扩增红麻cox1全长及侧翼序列,结果表明,在红麻不育系UG93A和保持系UG93B中分别获得长度为2 149、3 244 bp的序列,包含cox1基因CDS全长及其5′和3′部分侧翼序列。然而,Zhou等[17]认为,hiTAIL-PCR还需改进,因为在hiTAIL-PCR中扩增出许多非特异性产物,而没有为下游分析生成足够的产物。由此他们修改了hiTAIL-PCR程序和重新设计了随机引物,结果发现修改后的hiTAIL-PCR反应产物的产量和特异性都得到了提高,用来鉴定由 phiC31整合酶调控的基因组整合部位更加精确和有效。
采用hiTAIL-PCR技术,已有大量植物功能基因被成功克隆。然而与芋头逆境胁迫相关的重要功能基因的克隆却鲜有报道。目前芋头过氧化物酶在NCBI中只有1个300 bp左右的EST(expressed sequence tags)序列,同源分析表明它与其他物种的过氧化物酶基因序列最大的一致性只有80%。因此不能用其他物种的过氧化物酶序列作为参考直接设计引物扩增。本研究根据芋头的密码子使用频率采用iCODEHOP设计简并引物扩增芋头过氧化物酶基因序列,然后采用hiTAIL-PCR技术扩增该基因的两侧序列,为进一步了解芋头过氧化物酶基因家族的功能及其分子机制提供理论依据,为进一步研究芋头抗疫病的机理奠定基础。
1 材料与方法
1.1试验材料
芋头抗疫病品种湛江-5、感病品种湛江-4来源于广东省湛江市农家栽培品种。DNA回收试剂盒(TAKARA公司TaKaRa AgaroseGelDNAPurification Kit Ver.2.0)、pMD-18T 载体试剂盒(TAKARA公司)、PCR仪(美国BIORAD公司)、电泳仪(北京六一仪器厂)、凝胶成像系统(美国BIO-RAD公司)。
1.2试验方法
1.2.1芋头过氧化物酶基因非侧翼序列扩增 采用改进的CTAB法提取芋头基因组DNA,调整DNA浓度至20 ng/μL。根据芋头的密码子使用频率用iCODEHOP 设计扩增芋头过氧化物酶基因序列的简并引物(表1)。
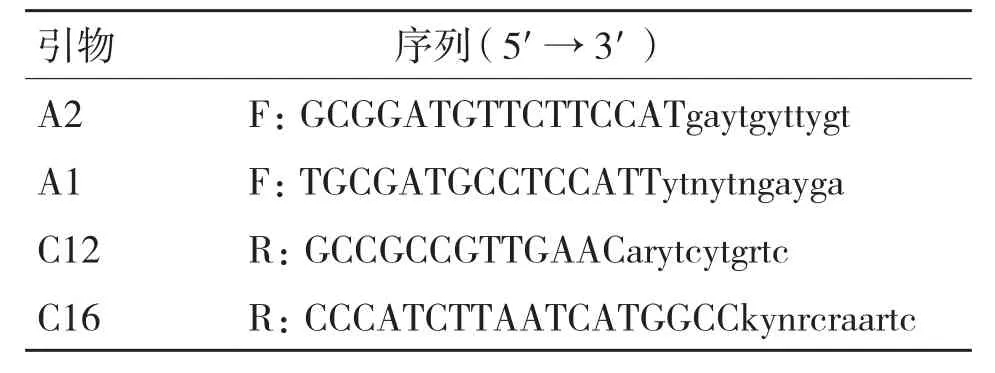
表1 扩增芋头过氧化物酶基因非侧翼序列引物
PCR反应体系(60 μL)如下:19.5 μL灭菌双蒸水,30 μL 2×MightyAmp 缓冲液,1.5 μL MightyAmpDNA聚合酶,6.0 μL引物混合液(按等体积混合 10 μmol/L A1和C12、A1和C16、A2 和C12、A2和C16 前导引物和随后引物),3.0 μL芋头DNA模板。PCR反应条件如下:98℃ 2 min;98℃ 10 s、55℃ 15 s、68℃ 2 min,40 个循环;68℃ 10 min。取 5 μL PCR反应产物点样进行琼脂糖凝胶电泳检测,然后取剩余的扩增阳性的反应液用DNA回收试剂盒回收DNA。再取回收的DNA用 pMD-18T 载体试剂盒进行连接,连接过夜后用 CaCl2转化方法进行转化。将涂抹转化菌液后的平皿倒置于37℃培养 12~16 h,挑选白色菌落(含重组质粒)进行扩增并进行重组子鉴定。最后,将扩增阳性的反应液送上海英骏生物技术有限公司测序。
1.2.2芋头过氧化物酶基因两侧序列扩增 待芋头过氧化物酶基因序列扩增成功后,依据其序列合成 hiTAIL-PCR引物(表2)。hiTAILPCR分 3 轮进行。第 1 轮反应用 20 μL反应体系:5.9 μL灭菌双蒸水,10.0 μL 2×MightyAmp缓冲液,2.0 μL LAD1-1(LAD1-2、LAD1-3、LAD1-4)引物,0.6 μL POD-0a(POD-0b)引物,0.5 μL MightyAmpDNA聚合酶,1.0 μL芋头DNA模板。第 2 轮反应用25 μL反应体系:18.18 μL灭菌双蒸水,2.50 μL 10×ExTaq 缓冲液,2.00 μL dNTP 混合液,0.60 μL AC1 引物,0.60 μL POD-1a(POD-1b)引物,0.12 μL ExTaq 酶,1.00 μL稀释 40 倍的第 1 轮PCR产物。第 3 轮反应用 25 μL反应体系:18.2 μL灭菌双蒸水,2.5 μL 10×ExTaq 缓冲液,2.0 μL dNTP 混合液,0.6 μL AC1 引物,0.6 μL POD-2a(POD-2b)引物,0.1 μL ExTaq酶,1.0 μL稀释10倍的第2轮PCR产物。hiTAIL-PCR反应条件参照文献[12]的方法并作出相应修改(表3)。3轮PCR反应完后,其产物同1.2.1进行检测、回收、连接、转化、再检测、送公司测序。

表2 hiTAIL-PCR引物
2 结果与分析
2.1芋头过氧化物酶基因非侧翼序列扩增
利用 iCODEHOP 设计的简并引物可以从芋头抗疫病品种湛江-5和感病品种湛江-4的基因组DNA中扩增出芋头过氧化物酶基因序列,其中以芋头抗疫病品种湛江-5 DNA为模板的扩增效果更明显,扩增所得序列约1 000 bp(图1)。利用DNA回收试剂盒回收,得到的芋头过氧化物酶基因序列为100 ng/μL,可以进行下一步连接、转化。经重组子鉴定后,将扩增阳性的反应液送公司测序,结果表明成功地从芋头基因组DNA中克隆出 1 165 bp序列,经同源性分析为过氧化物酶基因的部分序列。
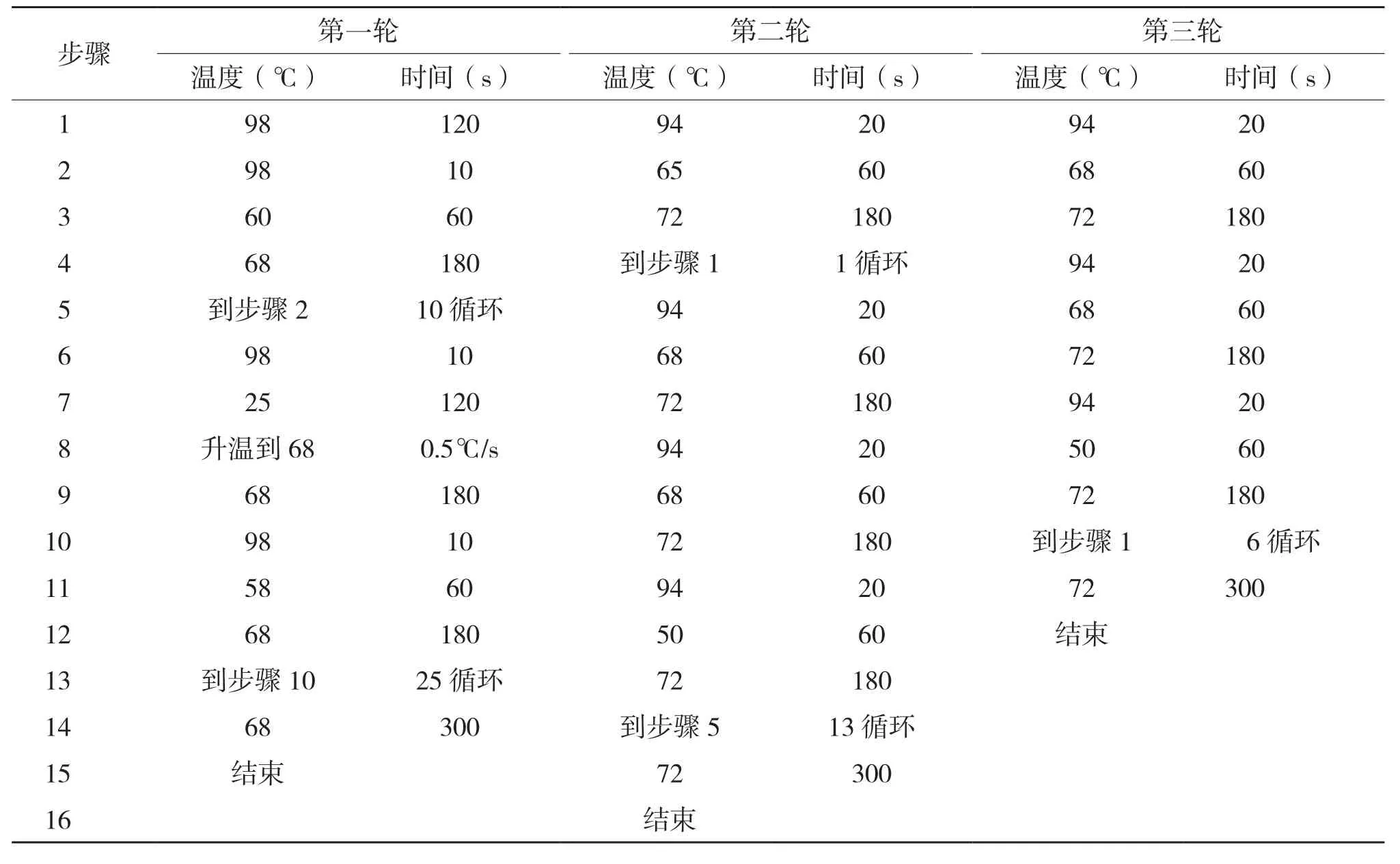
表3 hiTAIL-PCR反应条件
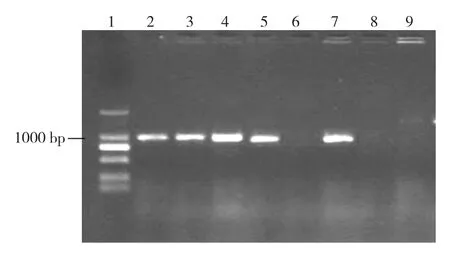
图1 芋头过氧化物酶基因非侧翼序列扩增检测

图2 hiTAIL-PCR扩增检测
2.2芋头过氧化物酶基因两侧序列扩增
利用hiTAIL-PCR技术可以扩增出芋头过氧化物酶基因的两侧序列(图2)。用DNA回收试剂盒回收扩增产物后,进行连接、转化,于37℃培养 12~16 h,挑选白色菌落(含重组质粒)进行扩增并进行重组子鉴定,确定转化的菌落里含有目的基因(图3)。经测序并删减与上述1 165 bp片段重复的序列后,确定所扩增出来的芋头过氧化物酶基因的两侧序列为689 bp。芋头过氧化物酶基因1 165 bp序列加上两侧序列后为1 854 bp。与已知的过氧化物酶进行序列比对,发现两侧还有氨基酸的编码序列,因此进行了第2次hiTAIL-PCR。而第2次hiTAIL-PCR又扩增出来 165 bp芋头过氧化物酶基因的侧翼序列,与之前得到的1 854 bp序列拼接后为2 019 bp。用Softberry上的基因预测软件分析后,发现包含过氧化物酶4个外显子(图4)。
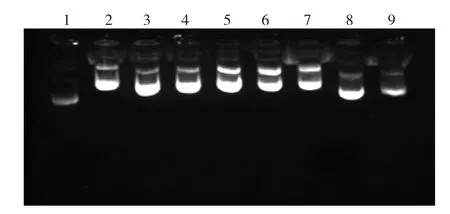
图3 菌落PCR结果
同源性分析表明,此过氧化物酶基因为含血红素的第三类过氧化物酶。再次与已知的过氧化物酶进行序列比对发现,两侧还有氨基酸的编码序列。因此进行第3次hiTAIL-PCR,扩增出了DNA片段,但在克隆时遇到了一定困难,多次试验后仍未成功。

图4 拼接分析结果
3 讨论
PCR最初是为扩增已知序列设计的,后来发展的简并PCR技术却能扩增未知序列。当待研究的基因序列不明,仅知其一部分氨基酸序列,即可根据氨基酸序列合成一组简并引物进行PCR扩增,对基因家族中的未知成员(基因)进行分离。简并PCR与常规PCR之间的一个重大差别,是用简并引物代替具有特定序列的特异PCR引物。简并引物的出现是由于遗传密码具有简并性,这样可以根据氨基酸的保守序列反推到DNA水平设计引物。由于大多数氨基酸的遗传密码不止一种,因此引物的部分碱基不能确定。可以根据各种不同的遗传密码规律设定DNA和氨基酸之间的相互转换,这样设计出来的引物是将可能编码一个给定氨基酸序列的核苷酸组合设计为一组,实际上是多种序列的混合物,序列的大部分是相同的,但在某些位点有所变化,称之为简并引物[18]。其实,简并PCR一直是寻找和发现“新”基因或基因家族新成员的一种非常有用的工具[4]。李运合等[19]利用 CODEHOP 和 iCODEHOP 设计了8对简并引物,从芒果子叶中扩增到了8条不同长度的 cDNA片段,测序结果表明,这些片段包含4类不同DNA序列。经过在 NCBI 上进行BLASTx分析,这些序列与其他植物的 LAX 基因具有高度同源性。研究结果表明,CODEHOP和 iCODEHOP设计的简并引物可信性强并具有高效性。王龙等[20]通过 iCODEHOP 在线设计细菌通透酶的简并引物,以发酵黄芪菌 FGM 基因组DNA为模板进行扩增,得到740 bp PCR产物。序列通过 BLASTx 检索与 GenBank 进行同源性比对后,结果表明,此DNA产物序列与其他菌属来源的通透酶蛋白序列具有相似性,所克隆的序列即为 FGM 通透酶基因片段。由此认为,用 iCODEHOP 在线设计的简并引物可信性强,阳性率高。崔文明等[21]利用 ICO-DEHOP和 CODEHOP 在线简并引物设计软件设计β-Hexcase 简并引物,以 LJJ 基因组DNA为模板经PCR扩增得到 614 bp 产物。该DNA序列经BLASTx比对发现与其他已知 β-Hexcase 基因具有相似性,表明所克隆的序列即为 LJJβ-Hexcase基因片段。
本试验用 iCODEHOP 设计简并引物扩增芋头过氧化物酶基因序列,并用 hiTAIL-PCR技术进一步对芋头过氧化物酶基因的两侧序列进行了扩增,成功扩增出了大小为 2 019 bp 的芋头过氧化物酶基因序列,该序列包含过氧化物酶4个外显子。同源性分析表明,此过氧化物酶基因为含血红素的第三类过氧化物酶。第三类过氧化物酶为典型的植物过氧化物酶,来源于高等植物的分泌型过氧化物酶,参与多种不同的生理功能[22],如细胞壁合成、组织愈伤、生长素合成与代谢,与超氧化物歧化酶相互协调配合清除过剩的自由基,使植物体内的自由基维持在一个动态的正常水平,以提高植物的抗逆性等。本研究克隆了芋头含血红素的第三类过氧化物酶基因序列,为芋头抗逆性的基因工程改良提供了一定的参考,但还需要对其组织表达和芋头植株抗疫病响应特征进行分析,同时还有必要利用转基因技术对该基因的功能作进一步研究。
[1]Misra R S,Maheshwari S K,Sriram S,et al. Integrated management of Phytophthora leaf blight disease of taro〔Colocasia esculanta(L.)Schott〕[J]. Journal of Root Crops,2007,33(2):144-146.
[2]Misra R S,Mishra A K,Sharma K,et al. Characterisation of Phytophthora colocasiae isolates associated with leaf blight of taro in India[J]. Archives of Phytopathology and Plant Protection,2011,44(6):581-591
[3]Joseph L M,Tan T K,Wong S M. Antifungal effects of hydrogen peroxide and peroxidase on spore germination and mycelial growth of Pseudocercospora species[J]. Canadian Journal of Botany,1998,76(12):2119-2124.
[4]王洪振,周晓馥,宋朝霞,等. 简并 PCR 技术及其在基因克隆中的应用[J]. 遗传,2003,25(2):201-204.
[5]Rose T M,Schultz E R,Henikoff J G,et al. Consensus-degenerate hybrid oligonucleotide primers for amplification of distantly-related sequences[J]. Nucleic Acids Research,1998,26(7):1628-1635.
[6]Staheli J P,Ryan J T,Bruce A G,et al. Consensus-degenerate hybrid oligonucleotide primers(CODEHOPs)for the detection of novel viruses in nonhuman primates[J]. Methods,2009,49(1):32-41.
[7]Rose T M,Henikoff J G,Henikoff S. CODEHOP(consensus-degenerate hybrid oligonucleotide primer)PCR primer design[J]. Nucleic Acids Research,2003,31(13):3763-3766.
[8]Boyce R,Chilana P,Rose T M. iCODEHOP:a new interactive program for designing consensusdegenerate Hybrid Oligonucleotide Primers from multiply aligned protein sequences[J]. Nucleic Acids Research,2009,37:W222-W228.
[9]Liu Y G,Whitter R F. Thermal asymmetric interlaced PCR:automatable amplification and sequencing of insert end fragment from P1 and YAC clones for chromosome walking[J]. Genomics,1995,25(3):674-681.
[10]Qin G J,Kang D M,Dong Y Y,et al. Obtaining and analysis of flanking sequences from T-DNA transformants of Arabidopsis[J]. Plant Science,2003,165(5):941-949.
[11]Antal Z,Rascle C,Fevre M,et al. Single oligonucleotide nested PCR:a rapid method for the isolation of genes and their flanking regions from expressed sequence tags[J]. Current Genetics,2004,46(4):240-246.
[12]Liu Y G,Chen Y. High-efficiency thermal asymmetric interlaced PCR for amplification of unknown f lanking sequences[J]. Biotechniques,2007,43(5):649-656.
[13]周延清,王婉珅,张喻,等. 高效热不对称交互式PCR技术克隆地黄基因[J]. 河南师范大学学报(自然科学版),2015,43(1):100-105.
[14]郭翠,张维,余桂容,等. 转G2-EPSPS基因玉米D-3侧翼序列分析与转化体特异性检测方法[J]. 作物杂志,2016(1):69-75.
[15]赵静,蔡深文,徐仲瑞,等. 海州香薷(Elsholtzia haichowensis Sun)细胞壁转化酶基因启动子(EhcwINVP)的克隆及活性分析[J]. 植物科学学报,2016,34(3):420-429.
[16]廖小芳,刁勇,邱爱华,等. 红麻细胞质雄性不育系UG93A和保持系UG93B cox1的克隆和表达分析[J]. 中国农业大学学报,2016(3):36-45.
[17]Zhou Z W,Ma H Y,Qu L J,et al. Establishment of an improved high-efficiency thermal asymmetric interlaced PCR for identification of genomic integration sites mediated by phiC31 integrase[J]. World Journal of Microbiology & Biotechnology,2012,28(3):1295 -1299.
[18]张新宇,高燕宁. PCR引物设计及软件使用技巧[J]. 生物信息学,2004,2(4):15-18,46.
[19]李运合,孙光明. 利用CODEHOP和iCODEHOP设计简并引物克隆芒果LAX基因家族片段[J]. 热带作物学报,2011,32(12):2278-2282.
[20]王龙,张凯,王旭荣,等. 利用iCODEHOP设计简并引物克隆益生菌FGM通透酶基因片段[J]. 中国畜牧兽医,2012,39(8):64-67.
[21]崔文明,刘鹭,张书文,等. 简并引物法克隆保加利亚乳杆菌β-N-乙酰氨基己糖苷酶基因[J]. 食品科学技术学报,2013,31(1):33-37.
[22]Welinder K G. Super family of plant,fungal and bacterial peroxidases[J].Current Opinion in Structural Biology,1992,2(3):388-393.
(责任编辑 崔建勋)
Cloning of peroxidase gene in taro
MO Jun-jie,LIANG Jia-xian,HU Han-qiao,HUANG Hong,CHEN Yu(College of Agronomy,Guangdong Ocean University,Zhanjiang 524088,China)
The taro peroxidase gene sequence was amplified using degenerate primers which were designed by iCODEHOP according to the taro codon usage frequency. And the peroxidase gene non-flanking sequence was amplified successfully. Then the peroxidase gene flanking sequences were amplified by hiTAIL-PCR primers which were designed on the basis of the non-flanking sequence. As a result,1 165 bp sequences were successfully cloned from taro genomic DNA,and it was affirmed that they were part of the sequences of peroxidase gene by homology analysis. Then by hiTAIL-PCR technology,the peroxidase gene flanking sequences were amplified,and 689 bp sequences were amplified. The Sequencing result could be splicedtogether with the 1 165 bp sequences. And the amplified taro peroxidase gene sequences splicedtogether with its flanking sequences were 1 854 bp. Sequence alignment with known peroxidase,we found that there were amino acid coding sequences on both sides of the amplification sequences. So,the hiTAIL-PCR technology was used as the second time,and 165 bp sequences were amplified. Splicedtogether with the 1 854 bp sequences,the amplified taro peroxidase gene sequences were 2 019 bp. With the gene on softberry prediction software analysis,it was found that the sequences contained 4 peroxidase exons. Homology analysis showed that the peroxidase gene was Class III heme-containing peroxidase.
taro;peroxidase gene;hiTAIL-PCR;cloning
S632.3;Q781
A
1004-874X(2016)09-0037-07
2016-06-21
国家公益性行业(农业)科研专项经费子课题分项目(200903017-08)
莫俊杰(1980-),男,博士,助理研究员,E-mail:104750343@qq.com
胡汉桥(1968-),男,博士,教授,E-mail:huhanqiao@sina.com

