人乳头瘤病毒16型甲基化水平与子宫颈病变程度关系的研究
王微,刘建华,王桂丽,齐特,阮强,孙峥嵘
(中国医科大学附属盛京医院1.病毒研究室;2.病毒检验科,沈阳 110004)
·论著·
人乳头瘤病毒16型甲基化水平与子宫颈病变程度关系的研究
王微1,刘建华2,王桂丽1,齐特1,阮强1,孙峥嵘1
(中国医科大学附属盛京医院1.病毒研究室;2.病毒检验科,沈阳 110004)
目的定量检测位于人乳头瘤病毒16(HPV16)L1基因3′端和LCR基因18个CpG甲基化水平,了解HPV 16甲基化与子宫颈病变的关系。方法选取本院就诊HPV16阳性正常/低度病变、高度病变、子宫颈癌标本各10例患者,采用重硫酸盐-焦磷酸测序方法分析HPVl6甲基化与子宫颈病变的关系。结果Ll基因3′端的位点7 089在正常/低度病变标本的甲基化率最高,其次是子宫颈癌,高度病变标本的甲基化率最低,3组比较差异有统计学意义(P=0.006);3组在位点7 134中甲基化率差异有统计学意义(P=0.01),与正常/低度病变标本比较,子宫颈癌及高度病变甲基化率差异均具有统计学意义(P分别为0.038、0.017)。结论HPVl6L1基因3′端甲基化位点7 089、7 134的甲基化水平与子宫颈病变程度存在相关性,HPVl6型DNA甲基化检测可用于临床上子宫颈病变筛查。
人乳头瘤病毒16;甲基化;CpG位点;子宫颈病变;焦磷酸测序
子宫颈癌是全世界高发的妇科肿瘤之一,其中发展中国家的子宫颈癌患者占全球85%以上[1]。高危型人乳头瘤病毒(human papilloma virus,HPV)感染是导致子宫颈癌的主要原因[2,3],目前已经发现的100多种亚型中有40种能入侵生殖器官,其中16、18、58、52、31、33型等高危亚型的感染是女性子宫颈癌的主要病因,尤其是16型,占所有子宫颈癌50%左右[4,5]。但仅有少部分人HPV持续性感染而导致子宫颈病变,子宫颈上皮内高度病变(highgrade squamous intraepithelial lesion,HSIL)是病毒、环境及机体共同作用的结果[6]。
由于大部分HPV感染并不会导致癌变的发生,临床上需要一种检测标记物将会发生癌变的人群区分出来进行临床治疗。传统的子宫颈病变筛查依赖于细胞学方法,该方法敏感度较低,HPV DNA定量检测具有高敏感度,但特异性比较低[7],不能确定HPV感染者是否是子宫颈病变的进一步进展人群。因此,对于子宫颈病变的早期诊断还需要其他标志物。DNA甲基化是一种常见的表观遗传学现象,基因组正常甲基化模式的改变可能会影响病毒生命周期,促使致癌过程。许多研究[8~11]显示,在子宫颈病变进程中HPV DNA发生不同程度的甲基化,尤其是L1/L2基因以及基因长调控区(long control region,LCR)变化尤为明显。本研究采用焦磷酸测序技术检测HPVl6 3′端L1及LCR区的甲基化状况,探讨HPV16甲基化水平与子宫颈病变之间的关系。
1 材料与方法
1.1材料
1.1.1标本来源:选择2014年4月到2015年5月在中国医科大学附属盛京医院就诊的HPV16型阳性患者的子宫颈拭子标本。按病理诊断结果分为3组:正常标本/子宫颈上皮内低度病变(normal/lowgrade squamous intraepithelial lesions,Normal/LSIL)组(Normal/LSIL组)、HSIL组、子宫颈鳞状细胞癌(cervical cancer,CC)组(CC组),每组各10例,共30例标本。
1.1.2主要试剂及材料:Qiagen公司的DNA提取试剂盒(QIAamp DNA Mini kit);德国QIAGEN公司转化试剂盒(EpiTect Bisulfite Kit),其他试剂采用Ta-KaRa生物工程公司试剂。
1.2方法
1.2.1DNA提取:使用DNA提取试剂盒从子宫颈拭子标本中提取DNA:向1.5 mL EP管中加入750 μL子宫颈拭子样本及750 μL异丙醇,混匀后14 000 r/min离心10 min。去掉上清后加入200 μL子宫颈拭子溶液悬浮沉淀;向加有样本的Ep管中加入20 μL蛋白酶K和200 μL缓冲液AL,漩涡振荡15 s后56℃孵育10 min;加入200 μL 100%的乙醇,漩涡振荡15 s后将溶液转移至放于2 mL收集管的柱中,6 000 g离心1 min,丢弃液体;向柱子中加入500 μL缓冲液AW2,20 000 g离心3 min,将柱子放置于另一干净的收集管中20 000 g离心1 min;将柱子置于1.5 mL Ep管中,为了提高最终DNA浓度(>50 ng/μL),用50 μL的AE缓冲液进行洗脱。
1.2.2重亚硫酸盐转化:本步实验目的是将DNA中的非甲基化C转化为U,然后通过PCR扩增转化为T。实验方法严格按照试剂盒说明书进行。
1.2.3引物设计:检测的HPV l6甲基化位点为位于L1基因3′端以及全长LCR基因的18个CpG位点:L1基因3′端包含3个位点(7 089、7 134、7 143),LCR基因(5′端)包含4个位点(7 268、7 426、7 452、7 458),增强子包含5个位点(7 532、7 550、7 673、 7 679、7 691),复制起点1个位点(7 859),E6/E7启动子包含5个位点(31、37、43、52、58),这些CpG位点在病毒整合及癌基因表达中起到决定性作用。为涵盖本研究所涉及的CpG位点,需要扩增出8条扩增子,共设计8对PCR引物,引物由上海英骏生物技术有限公司合成。见表1。
1.2.4焦磷酸测序:PCR扩增体系,5×缓冲液GC (KAPA)反应缓冲液10 μL,正向引物1 μL,反向引物1 μL,DNA 2 μL,dNTPs(10 mmol/L)1 μL,Taq酶(KAPA 5 U/μL)0.2 μL,加无菌水至50 μL;PCR扩增条件,95℃3 min;40个循环(94℃30 s,52 ℃30 s,72℃1 min);72℃7 min。所扩增的产物由上海希匹吉生物技术有限公司进行焦磷酸测序及数据分析。
1.3统计学分析
采取SPSS 13.0软件进行统计学分析,分析采用描述性统计分析方法,数据采用表示,组间比较采用方差分析。P<0.05表示差异有统计学意义。
2 结果
焦磷酸测序技术检测18个目的CpG位点的甲基化状况。测序30个标本的18个CpG位点均测序成功。
结果显示,各位点在3组子宫颈病变中表现出不同的甲基化率,位于L1区的3个位点(7 089、7 134、7 143)相对于其他位点有较高的甲基化率,启动子区位点31、37、43、52、58甲基化水平变化趋势一致,随子宫颈病变的严重程度而升高,位点7 859在3种子宫颈病变中均表现为最低甲基化水平。位点7 089、7 134 3组甲基化率比较具有统计学差异(P分别为0.006、0.01)。在位点7 134与Normal/LSIL组比较CC组的甲基化率差异有统计学意义(P=0.038),而HSIL组甲基化率差异无统计学意义(P>0.05);而此位点HSIL组与CC组的甲基化率比较差异具有统计学意义(P=0.017)。其他位点(7 143、7 268、7 426、7 452、7 458、7 532、7 550、7 673、7 679、7 691、7 859、31、37、43、52、58)3组甲基化率均没有统计学差异(P均>0.05)。见表2。
3 讨论
近年来,生物学技术上的进步使我们可以利用焦磷酸测序技术定量检测个体CpG位点的甲基化,且测序技术的可靠性及精确度很高,在甲基化研究中具有明显的优势。本研究结果显示位于L1区的位点有较高的甲基化率,其中7 089、7 134位点甲基化率在3组病变中有统计学差异(P<0.05),在HSIL中都表现出相对低甲基化率。L1基因编码病毒壳蛋白[12],该蛋白是病毒复制感染所必需,基因发生甲基化后编码蛋白质的功能受到抑制,不能编码病毒壳蛋白,因此HSIL中的低甲基化水平有利于病毒壳蛋白的表达,促进病毒的复制感染。在子宫颈癌阶段,许多病毒整合入宿主细胞中,高甲基化是宿主抵抗外源基因整合的防御机制[13]。一些关于甲基化的研究[8,12,13]发现在癌细胞中存在整合的病毒基因组,外源基因整合到宿主基因中,宿主细胞将其视为外源入侵对其进行甲基化修饰,随着整合程度的增加,甲基化程度不断增加。本研究结果显示,Normal/LSIL与CC组比较、HSIL与CC组比较7 134的甲基化率差异显著(P<0.05),但Normal/ LSIL与HSIL组比较7 134的甲基化率差异不显著(P>0.05),说明位点7 134可以对子宫颈病变做出是否为子宫颈癌的诊断,但不能将正常/低度病变与高度病变加以区分。位点7 859在3组病变中均表现为低甲基化水平,该位点位于病毒的复制起点位置,并且位于许多转录因子附近,因此低甲基化有利于维持病毒的复制[14]。启动子区的5个位点(31、37、43、52、58)甲基化水平随着病变的严重程度增加而增高,该区域是许多转录因子结合位点区,包括2个E2蛋白结合位点(E2 binding site,E2BS)。正常情况下,E2蛋白与结合位点结合从而抑制E6、E7基因的表达。随着病变进展许多病毒基因整合入宿主细胞中,E2BS发生甲基化后,E2蛋白不能正常结合,对E6、E7基因的抑制得以解除[15],E6、E7癌蛋白表达可以使宿主的抑癌蛋白p53和pRB受到抑制,从而加速癌变的进程。
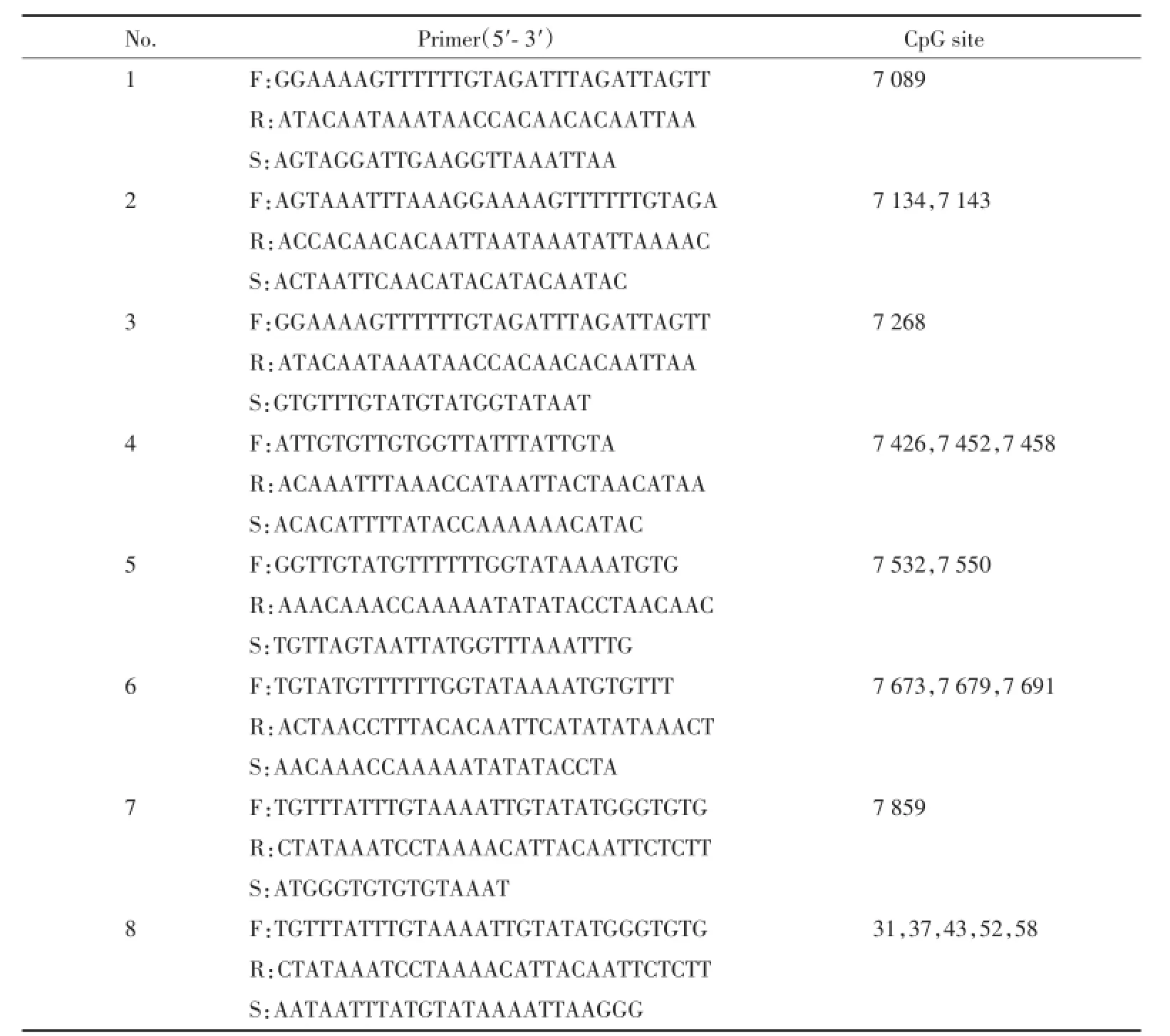
表1 HPV16L1/LCR基因引物序列及所检测的相应CpG位点Tab.1 Primer sequences and CpGs sites o f HPV16 L1/LCR region
许多研究也对HPV甲基化水平与子宫颈病变的关系做出了分析。Xi等[13]用亚硫酸盐测序的方法研究了LCR区11个CpG位点的甲基化情况,发现低度病变(CIN2-)与高度病变(CIN2+)的发生率与甲基化频率呈逆相关,并且认为在CIN2+过程中HPV低甲基化有利于病毒的复制感染,但未对子宫颈癌样本中的甲基化情况作出分析。Hublarova等[16]使用限制性内切酶-PCR法分析了正常样本、CIN1、CIN2-3、CC的甲基化情况,正常样本、CIN1甲基化率较高,CIN2-3、CC的甲基化率相对较低。Hu等[17]对30例临床样本的L1基因的3个CpG位点(7 032、7 091、7 136)进行甲基化分析发现,在位点7 032和7 091子宫颈癌中的甲基化率最高,无明显感染组中甲基化率最低,而在CIN样本中位点7 136的甲基化率最高,其次是子宫颈癌样本。出现这些研究结果不一致的原因是多方面的,可能与子宫颈病变的分类、所研究群体的种族差异及实验方法(如甲基化限制性内切酶法及亚硫酸盐测序法只能定性分析位点的甲基化情况,敏感性较低等)有关。本研究采用强有效的HPV甲基化检测方法(焦磷酸测序技术),发现位于L1基因中的位点7 089、7 134的甲基化率在3组病变中差异显著,可将子宫颈癌病程样本加以区分。
表2 3组子宫颈病变中HPVl6 3′-L1及LCR基因各检测位点的甲基化水平(%)Tab.2 Proportion methylation of each CpG site in 3′-L1/LCR o f HPV16 among three cervica l lesions(%)
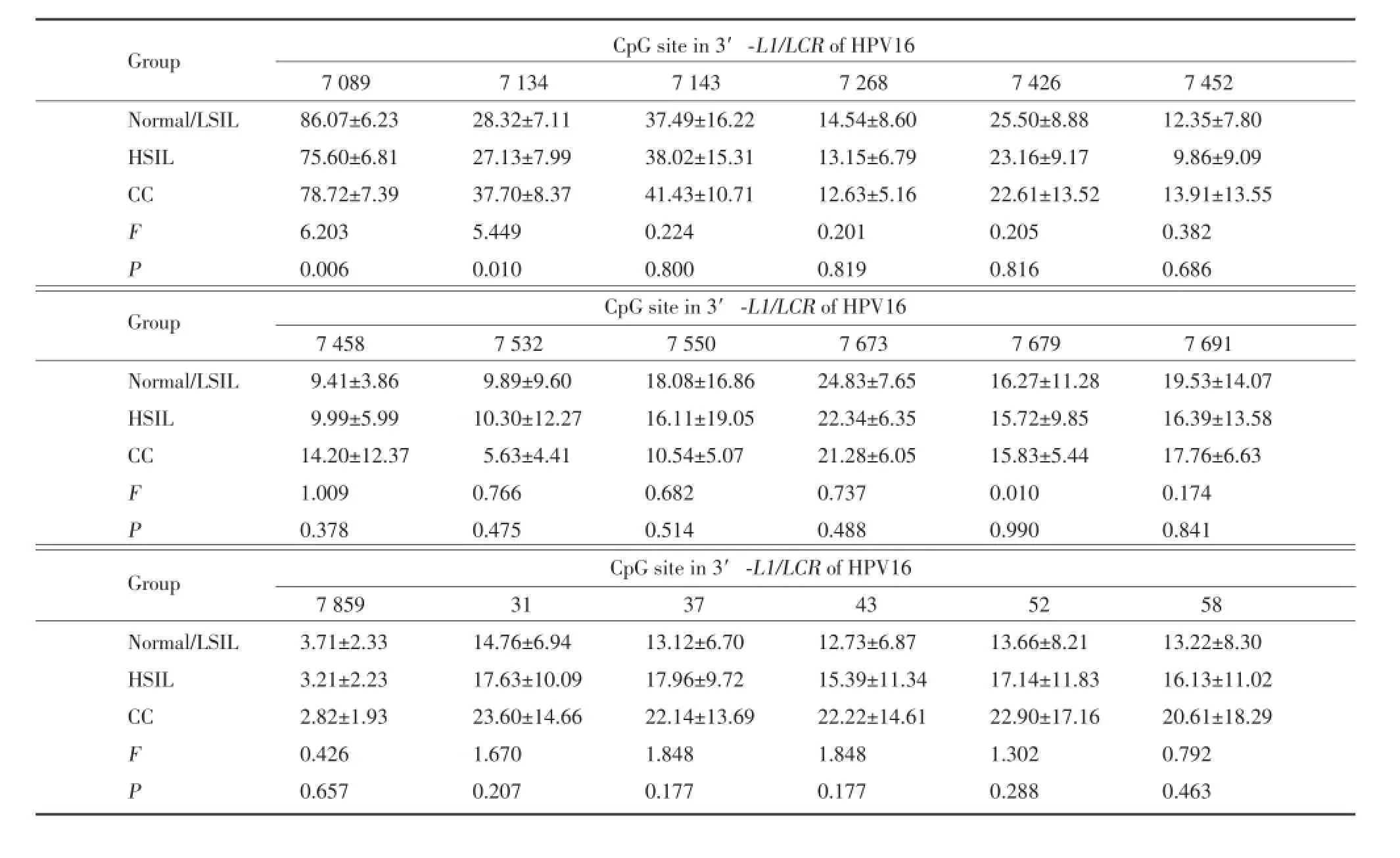
表2 3组子宫颈病变中HPVl6 3′-L1及LCR基因各检测位点的甲基化水平(%)Tab.2 Proportion methylation of each CpG site in 3′-L1/LCR o f HPV16 among three cervica l lesions(%)
Group CpG site in 3′-L1/LCR of HPV16 7 089 7 134 7 143 7 268 7 426 7 452 Normal/LSIL 86.07±6.23 28.32±7.11 37.49±16.22 14.54±8.60 25.50±8.88 12.35±7.80 HSIL 75.60±6.81 27.13±7.99 38.02±15.31 13.15±6.79 23.16±9.17 9.86±9.09 CC 78.72±7.39 37.70±8.37 41.43±10.71 12.63±5.16 22.61±13.52 13.91±13.55 F 6.203 5.449 0.224 0.201 0.205 0.382 P 0.006 0.010 0.800 0.819 0.816 0.686 Group CpG site in 3′-L1/LCR of HPV16 7 458 7 532 7 550 7 673 7 679 7 691 Normal/LSIL 9.41±3.86 9.89±9.60 18.08±16.86 24.83±7.65 16.27±11.28 19.53±14.07 HSIL 9.99±5.99 10.30±12.27 16.11±19.05 22.34±6.35 15.72±9.85 16.39±13.58 CC 14.20±12.37 5.63±4.41 10.54±5.07 21.28±6.05 15.83±5.44 17.76±6.63 F 1.009 0.766 0.682 0.737 0.010 0.174 P 0.378 0.475 0.514 0.488 0.990 0.841 Group CpG site in 3′-L1/LCR of HPV16 7 859 31 37 43 52 58 Normal/LSIL 3.71±2.33 14.76±6.94 13.12±6.70 12.73±6.87 13.66±8.21 13.22±8.30 HSIL 3.21±2.23 17.63±10.09 17.96±9.72 15.39±11.34 17.14±11.83 16.13±11.02 CC 2.82±1.93 23.60±14.66 22.14±13.69 22.22±14.61 22.90±17.16 20.61±18.29 F 0.426 1.670 1.848 1.848 1.302 0.792 P 0.657 0.207 0.177 0.177 0.288 0.463
综上所述,HPV16 L1基因的特定位点甲基化水平与子宫颈病变相关,位点7 089、7 134的甲基化可以作为子宫颈病变筛查的一个潜在性分子标记物,可作为临床上子宫颈病变诊疗的新依据。
[1]Tornesello ML,Buonaguro L,Giorgi-Rossi P,et al.Viral and cellular biomarkers in the diagnosis of cervical intraepithelial neoplasia and cancer[J].Biomed Res Int,2013,2013:519619.
[2]Fang J,Zhang H,Jin S.Epigenetics and cervical cancer:from pathogenesis to therapy[J].Tumour Biol,2014,35(6):5083-5093.
[3]Grellier N,Quero L.Cervical cancer:particularities in HIV patients [J].Bull Cancer,2014,101(11):1040-1047.
[4]Li N,Franceschi S,Howell-Jones R,et al.Human papillomavirus type distribution in 30,848 invasive cervical cancers worldwide:Variation by geographical region,histological type and year of publication[J].Int J Cancer,2011,128(4):927-935.
[5]Sun ZR,Ji YH,Zhou WQ,et al.Characteristics of HPV prevalence among women in Liaoning province,China[J].Int J Gynaecol Obstet,2010,109(2):105-109.
[6]Sun Z,Lu Z,Liu J,et al.Genetic variations of E6 and long control region of human papillomavirus type 16 from patients with cervical lesion in Liaoning,China[J].BMC Cancer,2013,13:459.
[7]Sun C,Reimers LL,Burk RD.Methylation of HPV16 genome CpG sites is associated with cervix precancer and cancer[J].Gynecol Oncol,2011,121(1):59-63.
[8]Hong D,Ye F,Lu W,et al.Methylation status of the long control re-gion of HPV 16 in clinical cervical specimens[J].Mol Med Rep,2008,1(4):555-560.
[9]Kalantari M,Osann K,Calleja-Macias IE,et al.Methylation of human papillomavirus 16,18,31,and 45 L2 and L1 genes and the cellular DAPK gene:considerations for use as biomarkers of the progression of cervical neoplasia[J].Virology,2014,448:314-321.
[10]Mirabello L,Sun C,Ghosh A,et al.Methylation of human papillomavirus type 16 genome and risk of cervical precancer in a Costa Rican population[J].J Natl Cancer Inst,2012,104(7):556-565.
[11]Murakami I,Fujii T,Dan K,et al.Methylation of human papillomavirus-52 and-58 is a candidate biomarker in cervical neoplasia [J].J Clin Virol,2013,58(1):149-154.
[12]Dutta S,Chakraborty C,Dutta AK,et al.Physical and methylation status of human papillomavirus 16 in asymptomatic cervical infections changes with malignant transformation[J].J Clin Pathol,2015,68(3):206-211.
[13]Xi LF,Jiang M,Shen Z,et al.Inverse association between methylation of human papillomavirus type 16 DNA and risk of cervical intraepithelial neoplasia grades 2 or 3[J].PLoS One,2011,6(8):e23897.
[14]Kalantari M,Calleja-Macias IE,Tewari D,et al.Conserved methylation patterns of human papillomavirus type 16 DNA in asymptomatic infection and cervical neoplasia[J].J Virol,2004,78(23):12762-12772.
[15]Munger K,Baldwin A,Edwards KM,et al.Mechanisms of human papillomavirus-induced oncogenesis[J].J Virol,2004,78(21):11451-11460.
[16]Hublarova P,Hrstka R,Rotterova P,et al.Prediction of human papillomavirus 16 e6 gene expression and cervical intraepithelial neoplasia progression by methylation status[J].Int J Gynecol Cancer,2009,19(3):321-325.
[17]胡元晶,曲芃芃.HPV16 L1基因3′端和LCR基因甲基化与其致病性的关系[J].中华妇产科杂志,2013,48(8):607-610.
(编辑武玉欣)
Correlation between Severity ofCervicalLesionsand Methylation of Human Papilloma Virus Type16DNA
WANGWei1,LIU Jian-hua2,WANGGui-li1,QITe1,RUANQiang1,SUNZheng-rong1
(1.Virus Laboratory,Shengjing Hospital,China Medical University,Shenyang 110004,China;2.Department of Clinical Laboratory,Shengjing Hospital,China MedicalUniversity,Shenyang 110004,China)
ObjectiveToquantifiablymeasure themethylation frequencyof18CpG sites in the3′region of L1 geneand long control region(LCR)gene of HPVl6 DNA,and study the relationship between HPVl6 DNA methylation and severity of cervical lesions.M ethodsA total of 10 cases Normal/low-grade squamous intraepithelial lesion(Normal/LSIL),10 cases of high-grade squamous intraepithelial lesion(HSIL),and 10 cases of cervical cancer(CC)were recruited for the study.The relationship between severity of cervical lesions and HPV16 DNA methylation was analyzed bybisultlte-pyrosequencing.Results Themethylation ratewashighestin Normal/LSILatposition7089 located in3′-L1,followed byCC.The lowest was found in HSIL.The difference in methylation percentage among the three lesions was significant(P=0.006).In 7 134,the proportion methylationwasalsodifferentamong threegroups(P=0.01),difference inmethylation percentagebetween Normal/LSILandCC,aswellasNormal/LSIL and HSILwassignificant(P=0.038,0.017).ConclusionThemethylation statusofCpG sites7 089 and 7 134 in the3′region of L1 gene isassociatedwith theseverityofcervicaldisease.Thequantificationof HPV DNAmethylation canbeused forcervicaldiseasescreening in clinicalsamples.
human papilloma virus16;methylation;CpG site;cervical lesion;pyrosequencing
R373.9
A
0258-4646(2016)04-0293-05
10.12007/j.issn.0258-4646.2016.04.002
国家自然科学基金(81171581)
王微(1989-),女,硕士研究生.
孙峥嵘,E-mail:sunzr@sj-hospital.org
2015-06-16
网络出版时间:
网络出版地址

