利用TAIL-PCR技术分析拟南芥At1g52910突变体插入位点的侧翼序列
田 蕾,郭妍君,任 丽,王 钰,孙 菲,齐海坤,马 楠,戚晓利
(佳木斯大学 生命科学学院,黑龙江 佳木斯154007)
利用TAIL-PCR技术分析拟南芥At1g52910突变体插入位点的侧翼序列
田 蕾,郭妍君,任 丽,王 钰,孙 菲,齐海坤,马 楠,戚晓利*
(佳木斯大学 生命科学学院,黑龙江 佳木斯154007)
在筛选与维管发育相关的拟南芥突变体过程中,发现拟南芥基因At1g52910突变体的花器官明显异常。TAIL-PCR分析结果表明突变体为Ds单位点插入突变,突变体后代表型出现分离现象,暗示有其它突变位点存在。
拟南芥;At1g52910;TAIL-PCR
目前利用植物基因突变的方法研究植物基因的功能是认识生命活动机理的一种有效途径。植物基因突变后功能发生改变或失去功能,有可能使某些代谢途径部分或完全发生变化,从而产生相应的可见表型变化,据此推测出突变基因的功能。基因发生突变可以通过Ds或T-DNA插入到植物基因组中诱导产生,插入的DNA序列是已知的,但与之相接的植物基因组DNA的侧翼序列并不清楚。1995年由Liu等首创并发展了交错式热不对称PCR(Thermal Asymmetric Interlaced PCR,TAIL-PCR)技术,它是一种用来分离与已知序列邻近的未知DNA序列的分子生物学技术[1]。该技术一经问世,使分子生物学研究工作者能够简易而有效地从已知序列中分离到其邻近的未知侧翼序列,解决了有关基因操作的一系列难题。
本课题组在研究与维管发育相关的基因时筛选到一花发育明显变异的拟南芥突变体(图1),属于At1g52910基因的Ds 转座突变,突变纯合体后代出现表型分离。为了探明这些表型变异是否由Ds插入所造成,通过TAIL-PCR从突变体中扩增出Ds插入位点侧翼序列,为进一步探明突变体变异位点奠定基础,也为其它转基因植物Ds插入位点的侧翼序列分析提供参考。
1 材料和方法
1.1材料
拟南芥突变体ET139购自拟南芥突变体资源中心(The Nottingham Arabidopsis Stock Centre,NASC);野生型拟南芥(ArabidopsisthalianaLandberg)种子由中国林业科学研究院林木遗传育种国家重点实验室保存。

a. 野生型拟南芥的花;b. 突变体ET139异常的花
1.2方法
1.2.1基因组DNA提取
采用CTAB法提取叶片基因组DNA,作为扩增未知侧翼序列的模板。
1.2.2引物设计
TAIL-PCR使用一套嵌套的序列特异的引物(special primer,SP)和较短的随机简并引物(Arbitrary degenerate prime,AD)组合,进行“半特异性PCR反应”。特异性引物是根据插入的DNA序列设计的,从Ds开始扩增;而随机引物是从植物基因组DNA的任意点开始的。实验分别在Ds的3′ 端和5′ 端各设计了4个嵌套的长的特异引物,其序列如表1所示,随机简并引物AD序列见表2。

表1 TAIL-PCR反应的特异性引物序列

表2 TAIL-PCR反应的特异性引物序列
1.2.3TAIL-PCR反应体系和反应条件
第1轮PCR反应体系为20.2 μL ,包含12.8 μL ddH2O,2 μL 10×Buffer,2 μL dNTPs,0.2 μL Ds primer(20μM),3 μL AD primer(20 μM),0.2 μL Taq,取19 μL加1 μL Template(50 ng植物基因组DNA)。反应完成后将产物稀释50倍,取2 μL作第2轮反应的模板。次级反应完成后产物稀释30倍,取2 μL作第3轮反应的模板。第2轮、第3轮反应的体系除模板及特异引物外,其余成分与第1轮反应相同,循环参数见表3。

表3 TAIL-PCR反应循环参数
1.2.4产物的回收与测序
TAIL-PCR扩增产物经琼脂糖凝胶电泳检测,特异条带按试剂盒(Axygen AxyPrey DNA Gel Extraction Kit)方法回收,与pGEM-Teasy vector(Promega)连接,转化DH5α感受态细胞,用T3引物和T7引物筛选成功连接的阳性菌落。对阳性菌落进行测序,将序列在NCBI用Blastn与拟南芥基因组进行比对。
2 结果和分析
2.1Ds侧翼序列的扩增
TAIL-PCR工作的原理是以基因组DNA作为模板,利用引物特异性和长短的差异,设计不对称的温度循环,通过分级反应来扩增特异引物,消除非目标产物。退火温度特异性引物要高些,任意引物要低些,退火在不同温度下进行,则二个引物或者其中一个,只有特异性引物能够很好地起作用。散布高、简并的严格循环则使靶序列的扩增优于非特异性产物。TAIL-PCR理想的电泳结果应该是第3轮产物大于300 bp且比第2轮产物略小[1-3]。产物中可能会出现多条带,但一般仅有一条最亮。
本实验采用引物组合从Ds3′ 端和5′ 分别进行TAIL-PCR扩增侧翼序列,发现在Ds3-1(Ds5-1)/Ds3-2(Ds5-2)/Ds3-4(Ds5-4)和AD组合条件下可以获得清晰的条带,且第3轮扩增产物比第2轮扩增产物偏小(图2),符合TAIL-PCR要求。随机引物选用了13种AD1~AD13,图3中显示Ds3′ 端引物与TAIL-PCR随机引物AD1~AD8 PCR电泳结果。将第3轮PCR产物经电泳检测后,回收目的条带并克隆转化测序。
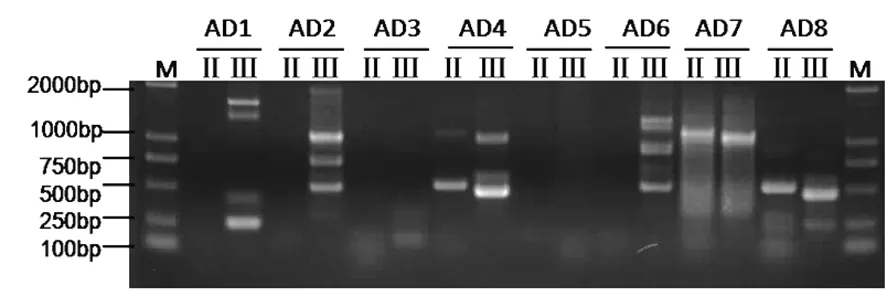
M:D2000 marker;Ⅰ、Ⅱ代表第2、3轮程序
2.2Ds插入位点侧翼序列的测序
在阳性克隆测序后获得了大小不同的侧翼序列,其结果一致,检测出的侧翼序列为At1g52910基因序列,正常表型的突变体与异常表型的突变体均插入同样的位点。将侧翼序列在NCBI用Blastn与拟南芥基因组进行比对,没有发现其它插入位点。Ds插入其第二外显子中(图3)。
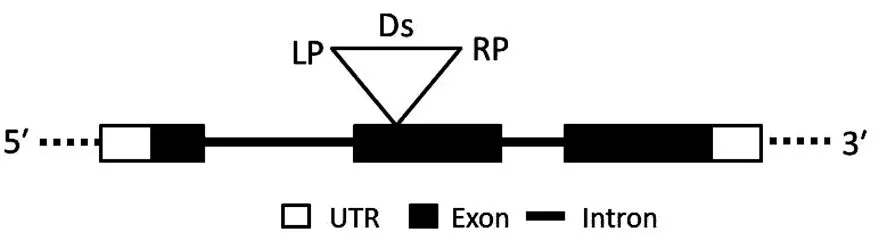
(At1g52910由3个外显子和2个内含子组成,Ds插入位点在第2外显子处)
3 讨 论
TAIL-PCR技术能快速准确分离到目标序列,对基因克隆的研究具有非常重要的意义[4]。
TAIL-PCR技术操作简单易行,对DNA纯度及浓度要求不高,只要引物设计好,以DNA为模板即可筛选到目标基因;产物的特异性高,采用简并引物和特异性嵌套引物组合及不对称的温度循环,使目标片段占优势;反应高效灵敏,本实验选用的8个Ds引物,有6个引物有效地扩增到目标片段;重复性好,快速,实验在1d内完成,可以快速地获得目标片段;若克隆的片段小可以直接用于测序,不涉及连接反应,免去了繁锁的连接克隆等手续[5]。TAIL-PCR反应需要较多的引物组合,但由于AD引物存在有限的结合位点,随机引物数目较小,对于个别的侧翼序列,即使使用不同的简并引物也难以扩增到阳性结果。本实验对拟南芥Ds转座突变选用了8个AD引物,11个随机引物,采用不同组合进行检测,为类似检测提供直接可用的方法。
TAIL-PCR对突变体ET139检测结果表明,突变体是Ds转座插入基因At1g52910第2外显子处,没有其它插入位点。突变纯合体后代出现表型分离,表明花器官的变异并非由Ds的插入而产生的,尚需通过图位克隆检测变异位点,为进一步发掘基因功能奠定基础。
[1]LIU Y G,WHITTIER R F. Thermal Asymmetric Interlaced PCR:Automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking[J]. Genomics,1995,25:674-681.
[2]LIU Y G, NORIHIRO M, TERUKO O, et al. Efficient isolation and mapping of Arabidopsis thaliana T-DNA insert junctions by thermal asymmetric interlaced PCR[J]. The plant Jouranl,1995,8(3):457-463.
[3]LIU Y G, HUANG N. Efficient amplication of insert end sequence from bacterial artificial chromosome clones by thermal asymmetric interlaced PCR[J]. Plant Molecular Biology Report, 1998, 16:175-181.
[4]罗丽娟,施季森. 一种DNA侧翼序列分离技术—TAIL-PCR[J]. 南京林业大学学报(自然科学版),2003,27(4):87-90.
[5]宋达峰,韩凝,边红武,等. 用改良TAIL-PCR技术分析转基因烟草cbf1基因插入区的侧翼序列[J]. 作物学报,2005,31(10):1377-1379.
Analysis ofAt1g52910 Mutant Flanking Sequence by TAIL-PCR
Tian Lei, Guo Yanjun, Ren Li, Wang Yu, Sun Fei, Q: Hakun, Ma Nan, Qi Xiaoli*
(College of Life Science,Jiamusi University,Jiamusi 154007,China)
In a screen of vascular development defective mutants inArabidopsis, mutantAt1g52910 was identified as a Ds transposon mutant with a mutation inAt1g52910. The insertional mutation was not co-segregated with the floral aberrance phenotype. TAIL-PCR analysis did not show additional Ds insertional sites, which suggested that there are other mutations existed.
Arabidopsisthaliana;At1g52910;TAIL-PCR;floral aberrance
10.3969/j.issn.1006-9690.2016.04.006
2015-12-20
黑龙江省大学生创新创业训练计划项目(201410222040);佳木斯大学科学技术重点项目(Sjz2012-19);黑龙江省教育厅科学研究项目(12513091);佳木斯大学校级课题(S2010-53)。
田蕾(1993—),女,生物技术专业本科生。
戚晓利(1971—),女,副教授,博士,研究方向:分子生物学教学及科研。E-mail:qixiaoli3@tom.com
S188
A
1006-9690(2016)04-0023-03

