基于SSR标记的255个枣品种亲缘关系和群体遗传结构分析
刘秀云,李 慧,2,刘志国,赵 锦,刘孟军,4
(1河北农业大学/中国枣研究中心,河北保定 071001;2宁夏农林科学院种质资源研究所,宁夏银川 750002;3河北农业大学生命科学学院,河北保定 071000;4北京林果业生态环境功能提升协同创新中心,北京 102206)
基于SSR标记的255个枣品种亲缘关系和群体遗传结构分析
刘秀云1,李 慧1,2,刘志国1,赵 锦3,刘孟军1,4
(1河北农业大学/中国枣研究中心,河北保定 071001;2宁夏农林科学院种质资源研究所,宁夏银川 750002;3河北农业大学生命科学学院,河北保定 071000;4北京林果业生态环境功能提升协同创新中心,北京 102206)
【目的】枣原产中国,种质资源丰富。对来自中国22个省区不同用途的255个枣品种进行SSR分析,揭示这些不同产地来源的枣种质资源之间的亲缘关系和群体遗传结构,为枣种质资源的科学管理和分子标记辅助育种提供参考。【方法】利用改良CTAB提取供试枣种质的基因组DNA,以前期枣基因组测序挖掘出的SSR引物为基础,进行高效率引物筛选,并利用SSR分子标记技术对255份枣种质资源的基因组DNA进行PCR扩增,然后利用8%聚丙烯酰胺凝胶电泳分离,银染后显色。根据条带有无统计数据,计算出多态性位点百分率(PIC),用NTSYS软件进行UPGMA聚类分析;利用Structure软件分析群体遗传结构,计算出最适群体数目,构建遗传结构图。【结果】从64对SSR引物中筛选出23对高效率SSR引物,在供试材料中共检测出117个多态性位点,各引物扩增的多态性位点数为2—10条,每对引物平均扩增多态位点数为5.09个,PIC值变幅为0.359—0.727,平均为0.548,这些多态性引物可应用到其他枣种质资源的研究中;建立了只需1—2个标记就可鉴别出来的部分枣品种的SSR指纹,可用于这些品种的快速分子鉴定;255个枣品种的聚类分析将所有枣品种分为15个亚类,包括4个大类和11个小类,不同品种间的相似系数范围0.71—1.00,其中北京花生枣单独聚为一类,与其他枣品种关系较远;‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’、‘陕西奶枣’和‘天津大马牙枣’的相似系数均为 1.00;结合聚类图、供试品种的用途和原产地分析,不同枣品种间的亲缘关系与品种原产地有一定相关性,但和品种用途没有显著相关性。群体结构分析中,通过绘制K与ΔK的关系图,K=15时,ΔK最大,据此将255个枣品种也同样划分为15个群体,与聚类分析结果基本一致;进一步分析表明,各群体中大部分品种血缘关系比较单一,较少品种含有其他类群的遗传成分。总体看,山西或陕西的枣品种出现在绝大部分居群中,说明这两个省的资源在不同群体间的基因交流中发挥了重要作用;南方栽培区域中来自湖南的枣品种形成了相对独立的居群,可能是其起源相对单一,且在长期栽培过程中和其他产地枣品种间基因交流较少所致。上述结果表明,供试枣品种中与来源区域相关性明显的品种由相同地域内枣品种演化而来,而另一部分与来源产地相关性不明显的品种则是由不同区域间品种经过频繁的基因交流和重组选育而来,融合了不同区域品种的特点,从而没有了明显的区域特征。【结论】不同地理环境在枣品种的群体进化中发挥了较重要的作用,影响了不同产地间枣种质资源的遗传结构组成。
枣;SSR分子标记;产地;亲缘关系;遗传结构
0 引言
【研究意义】枣(Ziziphus jujuba Mill.)原产中国,栽培历史悠久,遗传变异丰富,“同物异名”和“同名异物”现象较为突出。采用传统的形态学鉴定,费时费力,并且难以准确的鉴定性状相似的品种,而利用分子标记构建枣品种指纹图谱并进行遗传结构分析,有助于枣品种鉴定、遗传进化分析和分子辅助育种等研究。【前人研究进展】RAPD(random amplified polymorphic DNA)、AFLP(amplified fragment length polymorphism)等分子标记曾被成功用于枣的指纹图谱构建和亲缘关系分析。赵锦等[1]对枣树品种、品系及其近缘种进行了RAPD分析,乔勇等[2]利用AFLP技术对21个枣品种进行了分析,上述研究中供试材料的聚类结果与传统观点基本一致。近年来,SSR(simple sequence repeat)和SNP(single nucleotide polymorphisms)在国际植物新品种权保护联盟(UPOV)的BMT分子测试指南中被确定为构建DNA指纹数据库的标准标记方法。由于SSR标记操作简单,已成为当前各种作物建库的首选标记[3]。SSR是由1—6个碱基对组成的简单序列重复,又称微卫星DNA,已经被应用在葡萄、猕猴桃、杏、苹果等果树上进行遗传多样性分析[4-7]。2012年,麻丽颖等[8]利用12对SSR引物也构建了 36份枣品种的指纹图谱。遗传结构分析研究已在苹果[9-10]、梨[11]、杏[12-16]、毛樱桃[17]、杨梅[18]、荔枝[19]、芒果[20]、猕猴桃[21]、核桃[22-23]、石榴[24]、桃[25]中开展。在枣树研究中,李海涛[26]利用ISSR标记进行研究的结果表明,河南枣主栽品种与灰枣的总群体间分化程度较高,群体间基因交流较少,基因流可防止由遗传漂变引起的群体间的遗传分化;殷晓[27]利用ISSR标记对237份枣种质的遗传多样性进行了分析,结果表明黄河沿岸枣群体的杂合程度为重度杂合,遗传多样性丰富,变异程度较高,趋向远缘杂交。【本研究切入点】目前尚未有利用SSR技术对大规模枣种质材料进行遗传结构分析的报道,因此,在前人研究基础上,利用前期基于枣基因组测序开发的SSR引物[28],对来自全国22个省区主产地的255份枣种质进行更广范围的遗传多样性及群体结构研究。【拟解决的关键问题】分析中国枣种质资源的起源,探讨其遗传多样性水平和遗传结构,为指纹图谱构建和分子辅助育种提供理论依据。
1 材料与方法
试验于2015年在河北农业大学进行。
1.1材料
选取来自全国22个省区及主产地的255个枣树品种,供试品种信息详见表1。所有品种材料均采自山西太谷国家枣种质资源圃,于2013年6月采集供试材料的新鲜幼嫩叶片,置于冰盒中带回实验室,转入-20℃冰箱贮藏,以备提取基因组DNA。
1.2方法
基因组DNA提取方法按照肖京[29]的方法进行。
SSR引物为笔者实验室前期开发出的引物[28],SSR扩增在BIO-RAD公司生产BS97 MyCycler型的PCR仪上进行,反应体系(12.5 µL)中含有0.5 µL基因组DNA(含量10—25 ng),6.3 µL Taq MasterMix(天根),正向引物和反向引物各0.5 µL,4.7 µL ddH2O。SSR扩增程序:94℃预变性3 min,然后94℃变性30 s,57℃退火30 s,72℃延伸30 s,27个循环,最后72℃延伸7 min。扩增产物用10%聚丙烯酰胺凝胶进行分离,银染技术检测。
1.3统计分析
依据SSR扩增产物在电泳凝胶上的相对位置,对扩增条带进行记录,分别以“1”和“0”表示同一位置谱带的有无,建立原始数据阵,用NTSYS-pc 2.10e软件[30]进行后续分析。
计算多态性位点百分率P(%)=(k/n)×100,其中k为多态位点数,n为所测位点总数。SSR位点的多态性信息量(poly-morphism information content,PIC)按如下计算公式进行计算:PIC=1-ΣPi2,式中Pi表示第i个等位位点出现的频率[31]。
利用NTSYS中的Qualitative date模块计算任意两个品种间的相似系数(GS)。其计算公式为GS=2Nij/ (Ni+Nj),其中Nij为材料i和j共有的扩增片段总数,Ni为材料i中出现的扩增片段数,Nj为材料j中出现的扩增片段数。以Clustering程序中SHAN进行UPGMA(非加权组平均法)聚类分析,并通过Treeplot模块生成聚类图。
将数据转换成Structure默认的数据格式,并通过Structure软件进行群体遗传结构分析。将K值设为2 —20,采用马尔可夫链的蒙特卡洛模拟算法(markov chain monte carlo,MCMC),不作数迭代(length of burn-in period)设为10 000次,再将不作数迭代后的MCMC设为100 000次。但依此得到的数据所绘制的折线图常常不能提供真正的 K值。因此,在此基础上推算对数变化率ΔK来更为准确的反应真正的K值[32]。ΔK的计算公式为:ΔK=m(|L(K+1)-2L(K)+L(K-1)|)/s[L(K)]。L(K)为每个K对应的对数值,s为标准差,m为平均值。根据ΔK的变化规律来确定最适的群体数目,构建遗传结构图。计算群体中品种基因组变异源于该群体中的概率Q,用于分析其遗传成分[33]。
2 结果
2.1高效率 SSR引物的筛选与多态性位点百分率 P的计算
利用供试材料中30份枣品种的基因组DNA为模板,从课题组前期设计并筛选出的SSR引物[28]中进行二次引物筛选。根据扩增效果,从中选取23对多态性和重复性好的SSR引物(表2,图1),用于255份枣种质的SSR分析。
23对引物在255份供试枣种质中共扩增出117条多态性条带,多态位点百分率达 100%。各引物扩增的多态性条带数为2—10条,平均每对引物扩增5.09个多态性条带,扩增片段范围90—290 bp(图2)。BOTSTEIN等[31]提出了衡量基因变异程度高低的多态性信息量指标PIC;PIC>0.5时,该引物为高度多态性信息引物;0.25<PIC<0.5,为中度多态性信息引物;PIC<0.25,为低度多态性信息引物。本研究中筛选出的引物 PIC值变幅为 0.359—0.727(平均值0.548),均为高度或重度多态性信息引物(表2)。
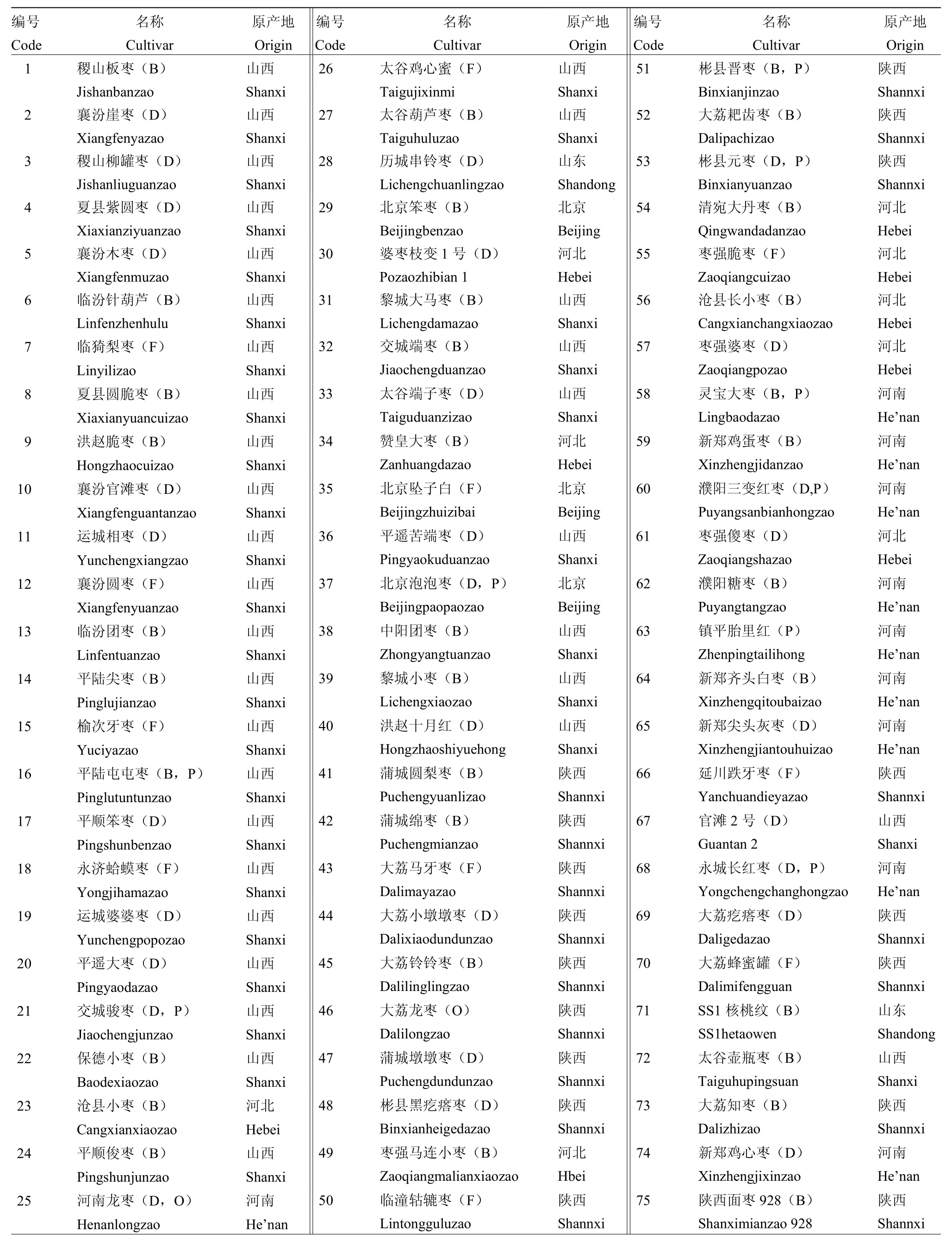
表1 供试品种材料Table 1 Cultivars used in the study
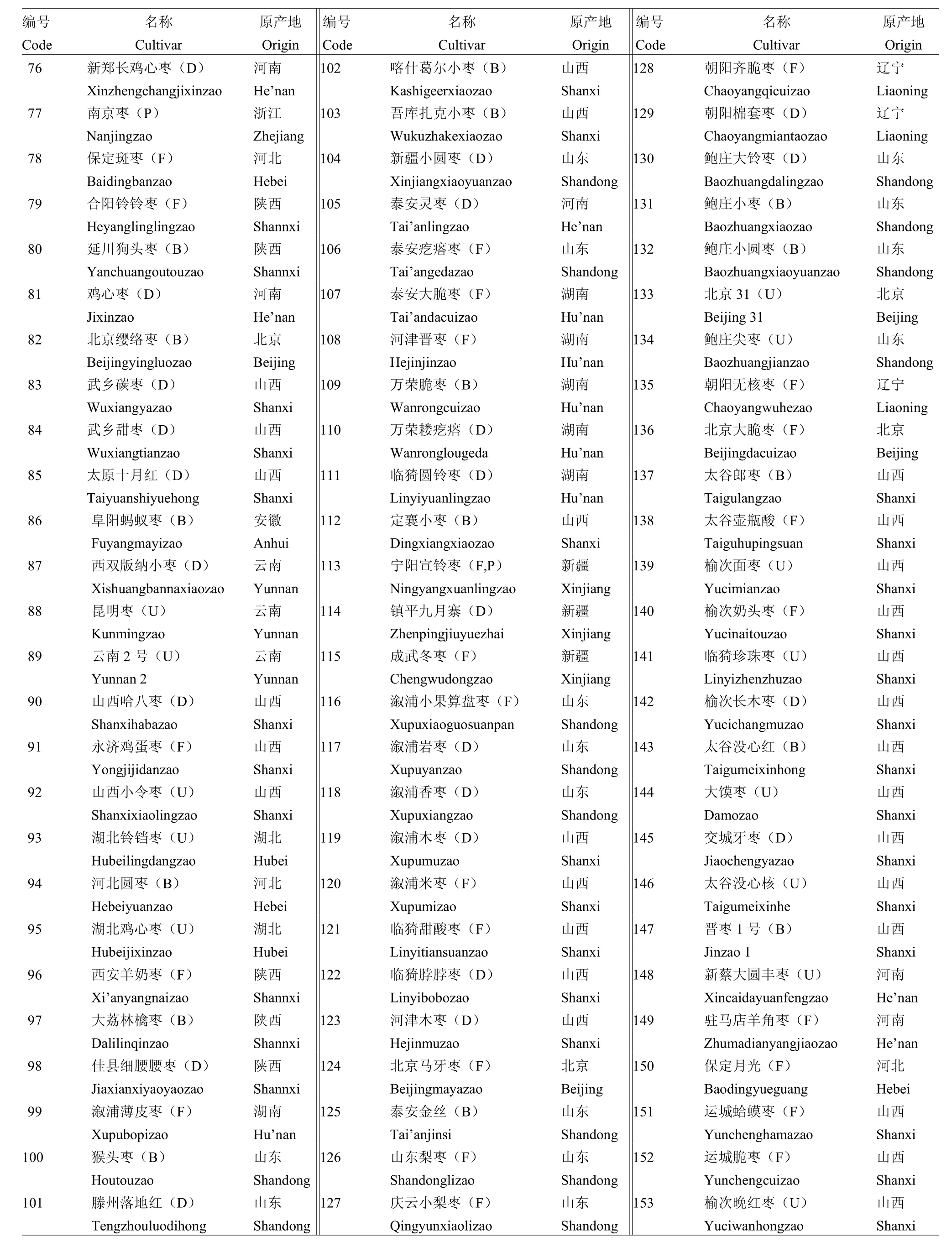
编号Code名称Cultivar原产地Origin编号Code编号Code名称Cultivar原产地Origin名称Cultivar原产地Origin 76 新郑长鸡心枣(D)Xinzhengchangjixinzao河南He’nan 102 喀什葛尔小枣(B)Kashigeerxiaozao山西Shanxi 128 朝阳齐脆枣(F)Chaoyangqicuizao辽宁Liaoning 77 南京枣(P)Nanjingzao浙江Zhejiang 103 吾库扎克小枣(B)Wukuzhakexiaozao山西Shanxi 129 朝阳棉套枣(D)Chaoyangmiantaozao辽宁Liaoning 78 保定斑枣(F)Baidingbanzao河北Hebei 104 新疆小圆枣(D)Xinjiangxiaoyuanzao山东Shandong 130 鲍庄大铃枣(D)Baozhuangdalingzao山东Shandong 79 合阳铃铃枣(F)Heyanglinglingzao陕西Shannxi 105 泰安灵枣(D)Tai’anlingzao河南He’nan 131 鲍庄小枣(B)Baozhuangxiaozao山东Shandong 80 延川狗头枣(B)Yanchuangoutouzao陕西Shannxi 106 泰安疙瘩枣(F)Tai’angedazao山东Shandong 132 鲍庄小圆枣(B)Baozhuangxiaoyuanzao山东Shandong 81 鸡心枣(D)Jixinzao河南He’nan 107 泰安大脆枣(F)Tai’andacuizao湖南Hu’nan 133 北京31(U)Beijing 31北京Beijing 82 北京缨络枣(B)Beijingyingluozao北京Beijing 108 河津晋枣(F)Hejinjinzao湖南Hu’nan 134 鲍庄尖枣(U)Baozhuangjianzao山东Shandong 83 武乡碳枣(D)Wuxiangyazao山西Shanxi 109 万荣脆枣(B)Wanrongcuizao湖南Hu’nan 135 朝阳无核枣(F)Chaoyangwuhezao辽宁Liaoning 84 武乡甜枣(D)Wuxiangtianzao山西Shanxi 110 万荣耧疙瘩(D)Wanronglougeda湖南Hu’nan 136 北京大脆枣(F)Beijingdacuizao北京Beijing 85 太原十月红(D)Taiyuanshiyuehong山西Shanxi 111 临猗圆铃枣(D)Linyiyuanlingzao湖南Hu’nan 137 太谷郎枣(B)Taigulangzao山西Shanxi 86 阜阳蚂蚁枣(B)Fuyangmayizao安徽Anhui 112 定襄小枣(B)Dingxiangxiaozao山西Shanxi 138 太谷壶瓶酸(F)Taiguhupingsuan山西Shanxi 87 西双版纳小枣(D)Xishuangbannaxiaozao云南Yunnan 113 宁阳宣铃枣(F,P)Ningyangxuanlingzao新疆Xinjiang 139 榆次面枣(U)Yucimianzao山西Shanxi 88 昆明枣(U)Kunmingzao云南Yunnan 114 镇平九月寨(D)Zhenpingjiuyuezhai新疆Xinjiang 140 榆次奶头枣(F)Yucinaitouzao山西Shanxi 89 云南2号(U)Yunnan 2云南Yunnan 115 成武冬枣(F)Chengwudongzao新疆Xinjiang 141 临猗珍珠枣(U)Linyizhenzhuzao山西Shanxi 90 山西哈八枣(D)Shanxihabazao山西Shanxi 116 溆浦小果算盘枣(F)Xupuxiaoguosuanpan山东Shandong 142 榆次长木枣(D)Yucichangmuzao山西Shanxi 91 永济鸡蛋枣(F)Yongjijidanzao山西Shanxi 117 溆浦岩枣(D)Xupuyanzao山东Shandong 143 太谷没心红(B)Taigumeixinhong山西Shanxi 92 山西小令枣(U)Shanxixiaolingzao山西Shanxi 118 溆浦香枣(D)Xupuxiangzao山东Shandong 144 大馍枣(U)Damozao山西Shanxi 93 湖北铃铛枣(U)Hubeilingdangzao湖北Hubei 119 溆浦木枣(D)Xupumuzao山西Shanxi 145 交城牙枣(D)Jiaochengyazao山西Shanxi 94 河北圆枣(B)Hebeiyuanzao河北Hebei 120 溆浦米枣(F)Xupumizao山西Shanxi 146 太谷没心核(U)Taigumeixinhe山西Shanxi 95 湖北鸡心枣(U)Hubeijixinzao湖北Hubei 121 临猗甜酸枣(F)Linyitiansuanzao山西Shanxi 147 晋枣1号(B)Jinzao 1山西Shanxi 96 西安羊奶枣(F)Xi’anyangnaizao陕西Shannxi 122 临猗脖脖枣(D)Linyibobozao山西Shanxi 148 新蔡大圆丰枣(U)Xincaidayuanfengzao河南He’nan 97 大荔林檎枣(B)Dalilinqinzao陕西Shannxi 123 河津木枣(D)Hejinmuzao山西Shanxi 149 驻马店羊角枣(F)Zhumadianyangjiaozao河南He’nan 98 佳县细腰腰枣(D)Jiaxianxiyaoyaozao陕西Shannxi 124 北京马牙枣(F)Beijingmayazao北京Beijing 150 保定月光(F)Baodingyueguang河北Hebei 99 溆浦薄皮枣(F)Xupubopizao湖南Hu’nan 125 泰安金丝(B)Tai’anjinsi山东Shandong 151 运城蛤蟆枣(F)Yunchenghamazao山西Shanxi 100 猴头枣(B)Houtouzao山东Shandong 126 山东梨枣(F)Shandonglizao山东Shandong 152 运城脆枣(F)Yunchengcuizao山西Shanxi 101 滕州落地红(D)Tengzhouluodihong山东Shandong 127 庆云小梨枣(F)Qingyunxiaolizao山东Shandong 153 榆次晚红枣(U)Yuciwanhongzao山西Shanxi

?

F:鲜食 Fresh;D:制干 Dry;B:兼用 Fresh and dry;P:加工 Processing;O:观赏 Ornamental;U:不详 Unknown
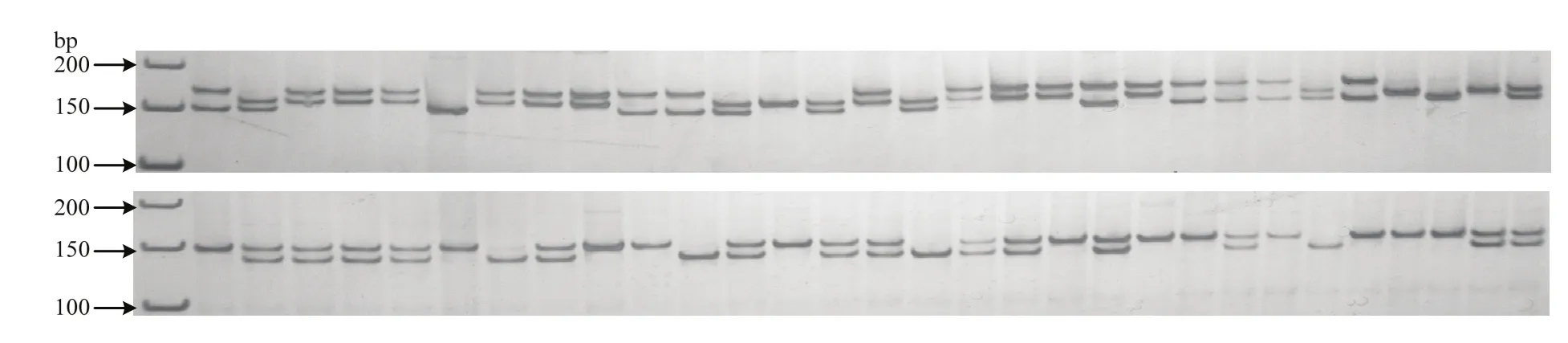
图1 筛选出的引物(JSSR97、JSSR128)对30份枣材料的扩增结果Fig.1 Amplified results of 30 jujube cultivars with selected primer pairs JSSR97 and JSSR128
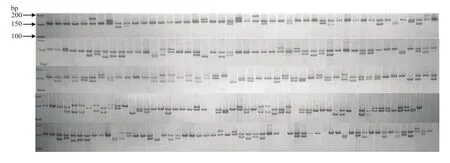
图2 引物JSSR183对255份枣材料的扩增结果Fig.2 Amplified results of 255 jujube cultivars with primer JSSR183

表2 筛选出的23对多态性SSR引物Table 2 The 23 SSR primer pairs selected for this study
2.2部分枣品种特有SSR标记的挖掘
供试枣品种的SSR分析结果以Excel表格形式保存,不同品种的SSR指纹以SSR引物名称为前缀、以该标记(扩增带)的分子量为后缀,得到每个品种在某个标记的带型编号。在255个供试品种中,有40个枣品种只需1—2个特有的SSR标记即可鉴别出来(表3)。从表1和表3可以看出,很多同一区域来源的枣品种(如同样来源于陕西省关中平原东北部蒲城县的‘蒲城直社疙瘩枣’‘蒲城直社疙瘩枣’和‘蒲城晋枣’可以被1—2个特有SSR标记区分开,这为地方品种的准确鉴定和资源管理提供了有效手段。

表3 40个枣品种的特有SSR指纹Table 3 Special SSR fingerprints of 40 jujube cultivars
2.3供试枣品种的聚类分析
利用23对引物扩增出的117条多态性条带,可将供试255个品种的251个品种分开,占98.43%,依据23对SSR引物扩增位点构建的0-1矩阵,计算出供试材料间的配对遗传相似系数(GS),其变化范围为0.71 —1.00。由聚类图(图3)可看出,在聚类阀值为0.71处,北京花生枣单独聚到一类,说明其与其他枣品种亲缘关系较远,分析原因可能是北京花生枣是独立起源的品种,也可能是其在长期栽培中与其他枣品种基因交流较少所致;其余254个品种分为14个亚类,包括4个大亚类(每类24—56个品种)和10个小亚类(1—10个品种)。在10个小亚类中,湖南的‘溆浦岩枣’和陕西的‘合阳铃铃枣’亦均单独为一个小类;同时有少部分品种没有分开,如‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’、‘陕西奶枣’和‘天津大马牙枣’之间的相似系数为1.00,表明‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’、‘陕西奶枣’和‘天津大马牙枣’的亲缘关系很近。‘奉节鸡蛋枣’和‘溆浦鸡蛋枣’分别来自重庆、湖南,这两个品种枣果的果形都是近圆形;‘陕西奶枣’和‘天津大马牙枣’分别来自陕西和天津,果形均较尖,本试验中检测出的两两品种间较近的亲缘关系也可能是不同地区间栽培引种造成的,也可能是两两品种是同物异名,或者是本研究中所用引物数量较少,尚不能将这些近缘品种完全分开。
从聚类图中还可以看出:(1)一些类群中,枣品种间的遗传关系与其原产地有明显的相关性。如I和II组的枣品种基本都是来自山西,II组的枣品种基本都来自北京,而O组的基本来自湖南;来自陕西的‘大荔铃铃枣’‘大荔蜂蜜罐’和‘大荔疙瘩枣’聚在一起,来自山东的‘泰安大脆枣’‘酥脆枣’和‘泰安疙瘩枣’也都聚在一起。(2)另一些类群中(如C、E、K组),枣品种来自于不同的产地,遗传关系与地理分布似乎没有必然的联系。这些类群相对比较分散,每一小类群中品种数量不多。如Ⅳ类群中7个枣品种来自于6个不同地区;Ⅴ类群中20个品种主要来自河北、山西和山东;Ⅵ类群中品种主要来自江苏和湖南。(3)结合聚类图和供试品种的用途和原产地分析,不同枣品种间的亲缘关系与品种原产地有一定相关性,但和品种用途没有显著相关性,例如第一大类中就包括了制干、鲜食、加工等不同用途的品种。
2.4供试枣种质的遗传结构分析
基于上述获得的SSR分析数据,利用STRUCTURE软件分析供试枣群体的遗传结构,绘制K与ΔK的关系图(图4),K=15时,ΔK最大。据此,将255个枣品种分成15个群组,进一步以K=15绘制了基于数学模型模拟的255个枣品种遗传结构的贝叶斯分配图(图5)。并计算出了15个居群中最大Q值分布(表4)。

表4 各群体Q值分布Table 4 Distribution of Q-value of fifteen groups

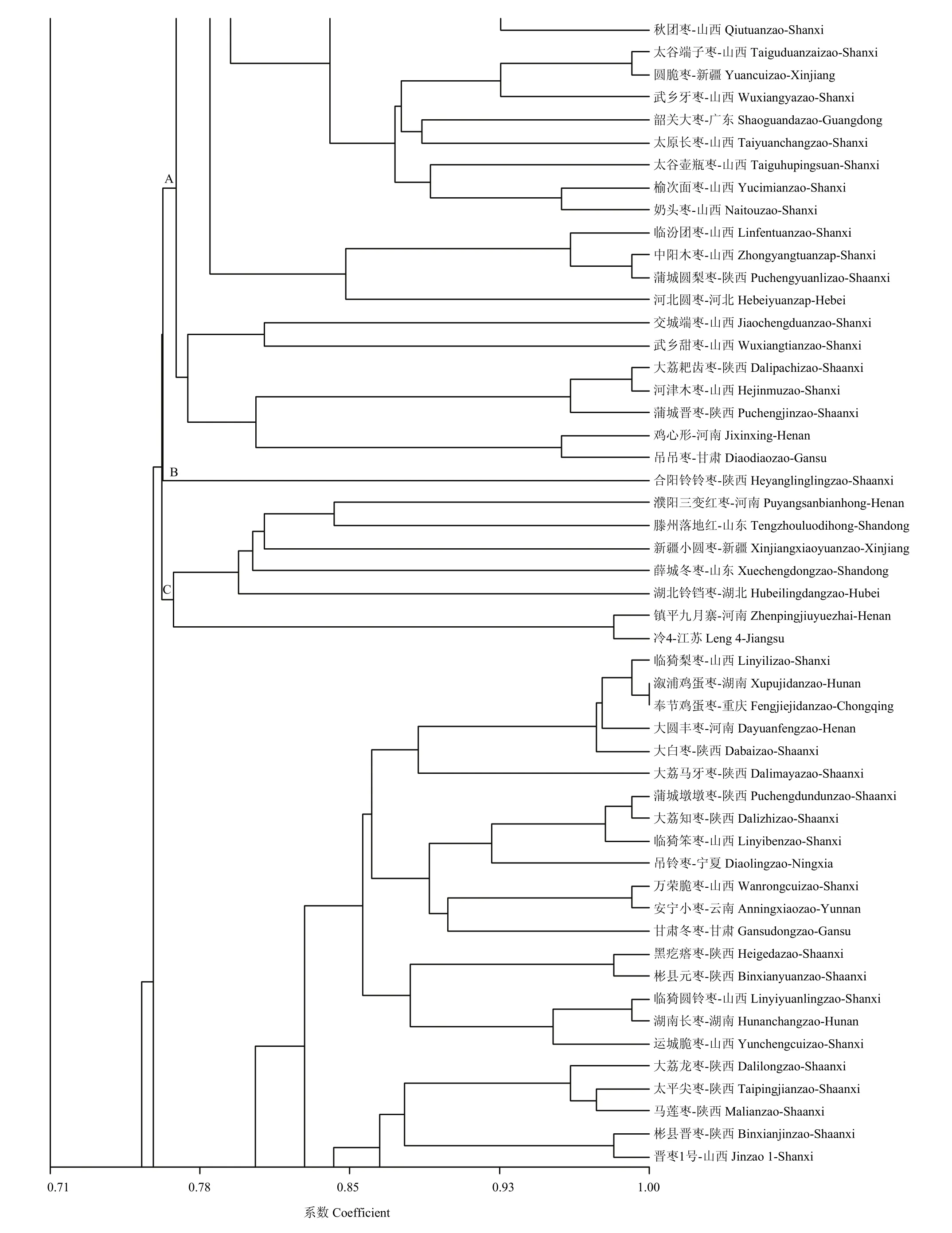

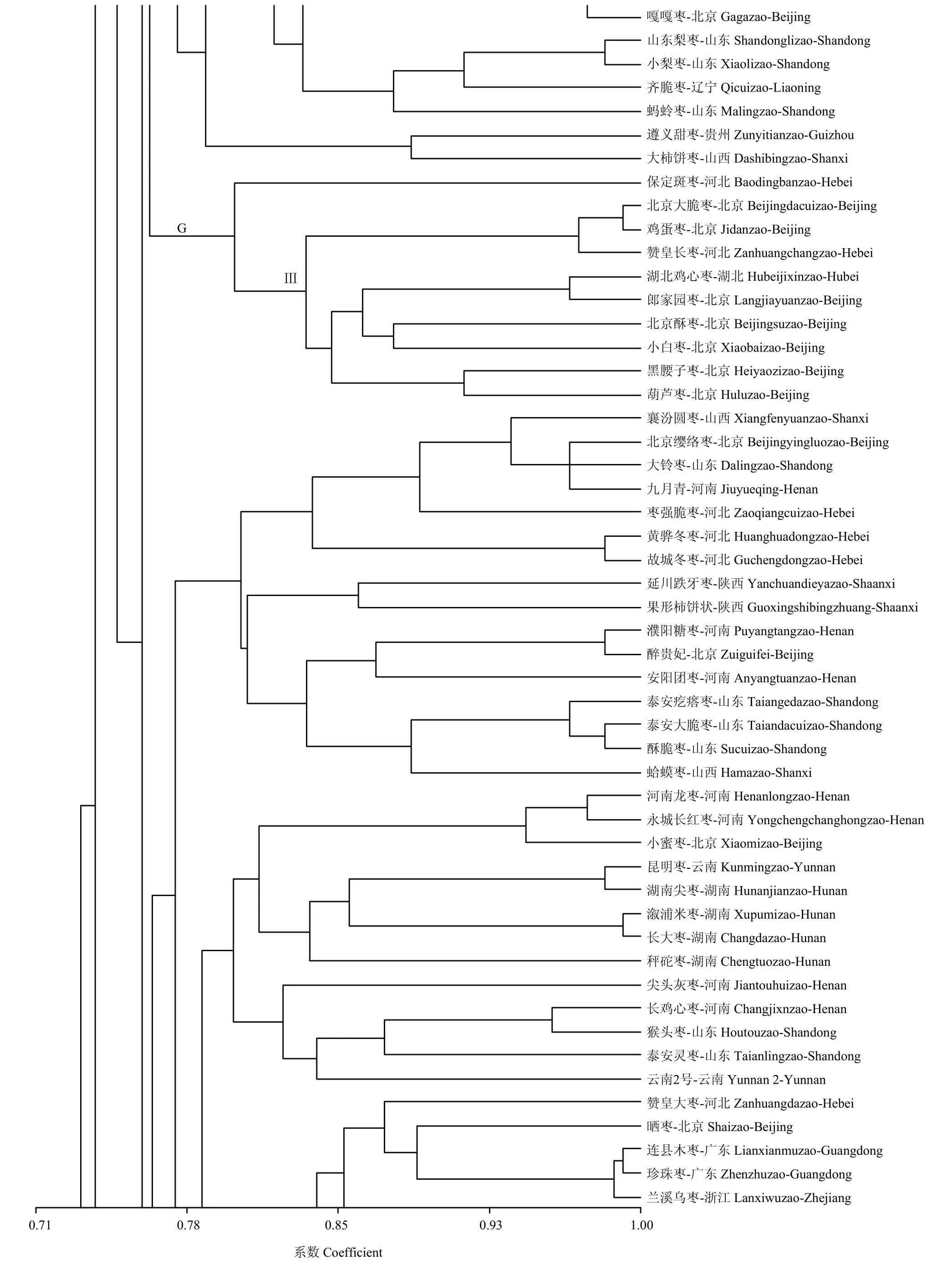

图3 基于SSR分析的255个枣品种聚类图Fig.3 Dendrogram of 255 jujube cultivars based on SSR markers

图4 枣255个品种的K值与ΔK的关系Fig.4 Graphical relationship between K and ΔK for 255 jujube cultivars

图5 255个枣品种的遗传结构分析Fig.5 Population structure of 255 jujube cultivars
图5中不同的色块代表不同的群体,纵坐标表示各居群中的品种占某居群祖先成分的比例,横坐标为样品编号。从图5中可看出,根据各个品种的混合比率,将255份供试材料分成15个居群,进一步分析表明,一些居群(如3、8、9、12、14居群)遗传背景比较一致,绝大部分来源于相同的栽培区域,混杂程度相对较低;而有的居群(如2、4、5、13居群)分配率较高,群体间混合程度相对较高,来源于不同的栽培区域,群体间的遗传物质交流相对较多。从遗传结构分析也发现,不同群体的品种和其用途没有显著相关性,但与聚类分析(图3)的结果相似。
Structure群体遗传结构分析中,当某一材料在某类群中的Q≥0.6时,则认为该材料血缘关系相对比较单一,否则认为该材料血缘关系来源复杂[33]。从根据供试材料在各个群体中的Q值分布(表4),255个供试材料中有36个品种Q值小于0.6,占所有供试材料总数的14.1%,Q值大于0.8和0.9的品种分别占68.2%和56.5%,说明各群体中大部分品种血缘关系比较单一,较少品种含有其他类群的遗传成分。整体看,来自枣主栽区域山西(居群 6、10、11)、陕西(居群14)和河北(居群1)的品种形成了以当地品种为主的居群,说明这些地区在长期的栽培演化过程中选育出了丰富的枣品种资源,其中来自山西或陕西的枣品种出现在绝大部分居群中,说明这两个省的资源在不同群体间的基因交流中发挥了重要作用;南方区域只有来自湖南的枣品种形成了相对独自的居群,可能是其起源相对单一,且在较长一段时间的栽培过程中,栽培环境比较封闭,近亲繁殖,和其他产地枣品种间基因交流较少所致(居群8)。
3 讨论
本研究选用23对SSR引物对255个枣品种进行了分析,共扩增出117条多态性谱带,多态位点比率达到100%。在枣树上,麻丽颖等[8]报道的SSR标记多态性比率也为 100%,而李莉等[34]报道的RAPD标记多态性为 99.78%,白瑞霞[35]用 AFLP标记的多态性则仅为 64.30%,谢永波[36]用 AFLP标记的多态性为78.7%,说明本研究的SSR标记的多态性效率较高。分析原因一方面是因为本研究中所用引物经过了二次筛选,另一方面也是因为供试材料多、遗传多样性丰富而使得多态位点比率较高。同时,本试验也注意到,不同 SSR引物对枣品种的鉴别能力有较大差异,并不是所有引物的多态性都较高,进一步说明筛选高效率引物的必要性。
中国枣品种繁多,不同品种间复杂的遗传关系一直是研究热点。本研究利用 SSR技术进行了 22个省区主产地 255个枣品种的遗传多样性和群体结构分析,提供了大量的多态性数据信息,为将来表型数据和基因数据的关联性分析奠定了良好的基础。聚类和群体结构两种分析结果均表明,在枣品种的群体结构中大部分品种与来源区域有一定的相关性。相关性不明显的枣品种可能是区域间品种经过频繁的基因交流或重组培育而成,从而融合了不同区域品种的特点,没有了明显的区域特征。前人利用SSR[17]、ISSR[18]、AFLP[2]进行的研究也表明,遗传多样性在一定程度上能够反映品种的地理分布特点。李海涛[26]对河南主栽枣品种的 ISSR分析表明,枣的遗传变异主要来自于居群内的变异,来自于居群间的变异贡献率较低。殷晓[27]对黄河沿岸六省48个枣品种进行的遗传多样性研究表明,地理区域内变异(93%)远大于地理区域间分化(7%)。本研究中,255个供试材料中Q值小于0.6的品种36个,仅占14.1%(表4),也进一步说明大部分品种血缘关系比较单一,以居群内或地理区域内变异为主。这些结果均表明,现有的枣种质资源可能是不同区域起源的,在物种演化过程中区域内品种间进行了频繁的基因交流和重组,进而产生了更为丰富的资源结构组成。
4 结论
利用23对SSR引物在255个供试品种中共检测出117个多态性位点,平均每对引物扩增多态位点数为5.09,PIC值变幅为0.359—0.727,平均为0.548;为其中40个枣品种找到了1—2个各自特有的SSR标记;255个枣品种的群体遗传结构与聚类分析结果基本一致,不同地理环境在枣品种的群体进化中发挥了较重要的作用,但不同枣品种的遗传关系与品种用途没有显著相关性。
References
[1] 赵锦,刘孟军. 枣树品种,品系及其近缘种的RAPD分析. 中国农业科学,2003,36(5): 590-594. ZHAO J,LIU M J. RAPD analysis on the cultivars,strains and relatedspecies of Chinese jujube (Ziziphus jujuba Mill.). Scientia Agricultura Sinica,2003,36(5): 590-594. (in Chinese)
[2] 乔勇,赵锦,杨海旭,刘孟军. 21个枣品种(系)的AFLP指纹分析. 植物遗传资源学报,2009,10(2): 205-210. QIAO Y,ZHAO J,YANG H X,LIU M J. AFLP analysis on 21 cultivars and strains of Chinese jujube. Journal of Plant Genetic Resources,2009,10(2): 205-210. (in Chinese)
[3] Union for the Protection of New Varieties of Plants. Guidelines for DNA-profiling: Molecular marker selection and database construction. Switzerland,2007: 3-4.
[4] 张萌. 基于SSR分子标记的葡萄种质资源遗传多样性分析及品种鉴定[D]. 江苏: 南京农业大学,2012. ZHANG M. Analysis of genetic diversity and cultivar identification of grapevine germplasm resources based on SSR molecular markers [D]. Jiangsu: Nanjing Agricultural University,2012. (in Chinese)
[5] 徐小彪,廖娇,黄春辉,辜青青,曲雪艳,刘善军,陈金印. 基于EST-SSR标记分析猕猴桃种质遗传关系. 果树学报,2012,29(2): 212-216. XU X B,LIAO J,HUANG C H,GU Q Q,QU X Y,LIU S J,CHEN J Y. Genetic relationships from Kiwifruit germplasms based on EST-SSR markers. Journal of Fruit Science,2012,29(2): 212-216. (in Chinese)
[6] 王玉安,欧巧明,陈建军,胡达,王发林. 甘肃地方杏品种资源的SSR遗传多样性分析. 西北农业学报,2013,22(3): 98-102. WANG Y A,OU Q M,CHEN J J,HU D,WANG F L. Analysis of genetic diversities in Gansu local apricot varieties with simple sequence repeat (SSR) markers. Acta Agriculturae Borealioccidentalis Sinica,2013,22(3):98-102. (in Chinese)
[7] 巴巧瑞. 苹果栽培品种亲缘关系的SSR和SRAP分析研究[D]. 杨陵: 西北农林科技大学,2011. BA Q R. Genetic relationship analysis of apple cultivars using SSR and SRAP markers [D]. Yangling: Northwest A & F University,2011. (in Chinese)
[8] 麻丽颖,孔德仓,刘华波,王斯琪,李颖岳,庞晓明. 36份枣品种SSR指纹图谱的构建. 园艺学报,2012,39(4): 647-654. MA L Y,KONG D C,LIU H B,WANG S Q,LI Y Y,PANG X M. Construction of SSR fingerprint on 36 Chinese jujube cultivars. Acta Horticulturae Sinica,2012,39(4): 647-654. (in Chinese)
[9] ZHANG C Y,CHEN X S,HE T M,LIU X L,FENG T,YUAN Z H. Genetic structure of Malus sieversii population from Xinjiang,China,revealed by SSR markers. Journal of Genetics and Genomics,2007,34(10): 947-955.
[10] 张春雨,陈学森,林群,苑兆和,张红,张小燕,刘崇祺,吴传金.新疆野苹果群体遗传结构和遗传多样性的 SRAP分析. 园艺学报,2009,36(1): 7-14. ZHANG C Y,CHEN X S,LIN Q,YUAN Z H,ZHANG H,ZHANG X Y,LIU C Q,WU C J. SRAP markers for population genetics structure and genetic diversity in Malus sieversii from Xinjiang,China. Acta Horticulturae Sinica,2009,36(1): 7-14. (in Chinese)
[11] 刘晶. 中国豆梨与川梨的遗传多样性和群体遗传结构研究[D]. 杭州: 浙江大学,2013. LIU J. Studies on genetic diversity and structure of Pyrus calleryana and P. pashia in China [D]. Hangzhou: Zhejiang University,2013. (in Chinese)
[12] 章秋平,刘东成,刘威生,刘硕,张爱民,刘宁,张玉萍. 华北生态群普通杏遗传多样性与群体结构分析. 中国农业科学,2013,46(1): 89-98. ZHANG Q P,LIU D C,LIU W S,LIU S,ZHANG A M,LIU N,ZHANG Y P. Genetic diversity and population structure of the north China populations of apricot (Prunus armeniaca L.). Scientia Agricultura Sinica,2013,46(1): 89-98. (in Chinese)
[13] 张俊环,王玉柱,孙浩元,杨丽. 不同用途杏品种群的AFLP分析.果树学报,2011,28(4): 610-616. ZHANG J H,WANG Y Z,SUN H Y,YANG L. Genetic analysis of apricot (Armeniaca) by fluorescent-AFLP markers. Journal of Fruit Science,2011,28(4): 610-616. (in Chinese)
[14] YUAN Z H,CHEN X S,HE T M,FENG J R,FENG T,ZHANG C Y. Population genetic structure in apricot (Prunus armeniaca L.)cultivars revealed by fluorescent-AFLP markers in southern Xinjiang,China. Journal of Genetics and Genomics,2007,34(11): 1037-1047.
[15] 何天明,陈学森,高疆生,张大海,徐麟,吴燕. 新疆栽培杏群体遗传结构的SSR分析. 园艺学报,2006,33(4): 809-812. HE T M,CHEN X S,GAO J S,ZHANG D H,XU L,WU Y. Using SSR markers to study population genetic structure of cultivated apricots native to Xinjiang. Acta Horticulturae Sinica,2006,33(4): 809-812. (in Chinese)
[16] 苑兆和,陈学森,张春雨,何天明,冯建荣,冯涛. 普通杏群体遗传结构的荧光AFLP分析. 园艺学报,2008,35(3): 319-328. YUAN Z H,CHEN X S,ZHANG C Y,HE T M,FENG J R,FENG T. Population genetic structure in apricot (Armeniaca L.) cultivars revealed by fluorescent-AFLP markers. Acta Horticulturae Sinica,2008,35(3): 319-328. (in Chinese)
高含硫天然气净化厂中控室暖通系统技术优化与应用评价…………………………………………………………(1):110
[17] 宛甜,蔡宇良,冯瑛,张雪,何恒流. 野生毛樱桃 SSR遗传多样性和遗传结构分析. 西北植物学报,2013,33(8): 1544-1550. WAN T,CAI Y L,FENG Y,ZHANG X,HE H L,WAN T. Genetic diversity and genetic structure of wild Prunus tomentosa Thub. based on simple sequence repeats markers. Acta Botanica Boreali-Occidentalia Sinica,2013,33(8): 1544-1550. (in Chinese)
[18] 赖恭梯,刘炜婳,张梓浩,冯新,林玉玲,刘生财,祁芳斌,赖钟雄.戴云山野生杨梅自然群体遗传结构的 ISSR分析. 热带作物学,2013,34(10): 1863-1870. LAI G T,LIU W H,ZHANG Z H,FENG X,LIN Y L,LIU S C,QI F B,LAI Z X. Genetic structure of natural populations revealed by ISSR in Myrica rubra from daiyunshan mountains. Chinese Journal of Tropical Crops,2013,34(10): 1863-1870. (in Chinese)
[19] 昝逢刚,吴转娣,曾淇,张惠云,李明芳,郑学勤. 荔枝种质遗传多样性的SRAP分析. 分子植物育种,2009,7(3): 562-568. ZAN F G,WU Z D,ZENG Q,ZHANG H Y,LI M F,ZHENG X Q. Genetic diversity analysis of litchi germplasm by SRAP markers. Molecular Plant Breeding,2009,7(3): 562-568. (in Chinese)
[20] 余贤美,艾呈祥. 杧果野生居群遗传多样性ISSR分析. 果树学报,2007,24(3): 329-333. YU X M,AI C X. Genetic diversity of wild Mangifera indica populations detected by ISSR. Journal of Fruit Science,2007,24(3): 329-333. (in Chinese)
[21] 刘亚令,李作洲,姜正旺,刘义飞,黄宏文. 中华猕猴桃和美味猕猴桃自然居群遗传结构及其种间杂交渐渗. 植物生态学报,2008,32(3): 704-718. LIU Y L,LI Z Z,JIANG Z W,LIU Y F,HUANG H W. Genetic structure and hybridization introgession in natural populations of two closely related Actinidia species,A. Chinensis and A. Deliciosa. Journal of Plant Ecology (Chinese Version),2008,32(3): 704-718. (in Chinese)
[22] 刘晓丽,陈学森,张美勇,陈晓流,何天明,张立杰,张春雨. 普通核桃(Juglans regia) 3个群体遗传结构的SSR分析. 果树学报,2008,25(4): 526-530. LIU X L,CHEN X S,ZHANG M Y,CHEN X L,HE T M,ZHANG L J,ZHANG C Y. Population genetic structure analysis of Juglans regia using SSR markers. Journal of Fruit Science. 2008,25(4): 526-530. (in Chinese)
[24] YUAN Z H,YIN Y L,QU J L,ZHU L Q,LI Y. Population genetic diversity in Chinese pomegranate (Punica granatum L.) cultivars revealed by fluorescent-AFLP markers. Journal of Genetics and Genomics,2007,34(12): 1061-1071.
[25] 吕志江,李疆,吾买尔夏提·塔汉,曾斌,罗淑萍. 新疆野扁桃种质资源遗传多样性的ISSR分析. 果树学报,2010,27(6): 918-923. LÜ Z J,LI J,OMIR S T,ZENG B,LUO S P. ISSR analysis for genetic diversity of Amygdalus ledebouriana germplasm from xinjiang,China. Journal of Fruit Science,2010,27(6): 918-923. (in Chinese)
[26] 李海涛. 河南枣主栽品种及灰枣群体遗传变异分析[D]. 郑州: 河南农业大学,2008. LI H T. Analysis of genetic variation in Henan main cultivated Ziziphus jujuba varieties and Z. ‘Huizao’ population [D]. Zhengzhou: Agricultural University of Henan,2008. (in Chinese)
[27] 殷晓. 基于 SSR标记的中国枣遗传多样性研究[D]. 杨凌: 西北农林科技大学,2013. YIN X. Genetic diversity and population structure of Chinese jujube analysed by SSR markers [D]. Yangling: Northwst A & F University,2013. (in Chinese)
[28] XIAO J,ZHAO J,LIU M J,LIU P,DAI L,ZHAO Z H. Genome-wide characterization of simple sequence repeat (SSR) loci in Chinese jujube and jujube SSR primer transferability. PLoS One,2015,10(5): e0127812.
[29] 肖京. 枣基因组 SSR位点特征分析及引物开发[D]. 保定: 河北农业大学,2014. XIAO J. Characterization of SSR loci in jujube genome and development of SSR primers [D]. Baoding: Agricultural University of Hebei,2014. (in Chinese)
[30] ROHLF F J. NTSYS-PC: Numerical taxonomy and multivariate analysis system version 1.80. Setauket,New York: Distribution by Exeter SoftWare,1994.
[31] BOTSTEIN D,WHITE R L,SKOLNICK M,DAVIS R W. Construction of a genetic linkage map in man using restriction fragment length polymorphism. American Journal of Human Genetics,1980,32(3): 314-331.
[32] EVANNO G,REGNAUT S,GOUDET J. Detecting the number of clusters of individuals using the software structure: A simulation study. Molecular Ecology,2005,14(8): 2611-2620.
[33] 吴承来,张倩倩,董炳雪,李圣福,张春庆. 我国部分玉米自交系遗传关系和遗传结构解析. 作物学报,2010,36(11): 1820-1831.WU C L,ZHANG Q Q,DONG B X,LI S F,ZHANG C Q. Analysis of genetic structure and genetic relationships of partial maize inbred lines in China. Acta Agronomica Sinica,2010,36(11): 1820-1831. (in Chinese)
[34] 李莉,彭建营,白瑞霞. 中国枣属植物亲缘关系的RAPD分析. 园艺学报,2009,36(4): 475-480. LI L,PENG J Y,BAI R X. Studies on the phylogenetic relationship of Chinese Ziziphus by RAPD technique. Scientia Agricultura Sinica,2009,36(4): 475-480. (in Chinese)
[35] 白瑞霞. 枣种质资源遗传多样性的分子评价及其核心种质的构建[D]. 保定: 河北农业大学,2008. BAI R X. Studies on genetic diversity and core collection construction of Ziziphus jujuba germsplasm resources using AFLP and SRAP markers. Baoding: Agricultural University of Hebei,2008. (in Chinese)
[36] 谢永波. 枣属种质资源形态学评价及品种 AFLP鉴定[D]. 泰安:山东农业大学,2014. XIE Y B. Morphological evaluation on germplasm resources jujube and cultivar identification by AFLP [D]. Tai’an: Shangdong Agricultural University,2014. (in Chinese)
(责任编辑 赵伶俐)
Genetic Diversity and Structure of 255 Cultivars of Ziziphus jujuba Mill.
LIU Xiu-yun1,LI Hui1,2,LIU Zhi-guo1,ZHAO Jin3,LIU Meng-jun1,4
(1Reserch Center of Chinese Jujube,Agricultural University of Hebei,Baoding 071001,Hebei;2Germplasm Resources Institute of Ningxia Academy of Agriculture and Forestry Sciences,Yinchuan 750002;3College of Life Science,Agricultural University of Hebei,Baoding 071001,Hebei;4Beijing Collaborative Innovation Center for Eco-Enviromental Improvement with Forestry and Fruit Trees,Beijing 102206)
【Objective】 There are abundant jujube germplasm resources in China. A total of 255 cultivars of Ziziphus jujuba Mill. from 22 provenances were used as materials to reveal their genetic diversity and phylogenetic relationship by SSR analysis,and the results of analysis would help us to manage jujube germplasm resources and offer references for molecular maker-assisted breeding. 【Method】 Good genomic DNA was extracted from young leaves of jujube germplasm resources following the improved CTAB method,and then were amplified by simple sequence repeat molecular markers to analyze genetic diversity and genetic structure with the selected high-efficiency primer pairs which were excavated based on the genome sequencing. Separation of the amplified fragments was performed on 8% denaturing polyacrylamide gels and the gels were stained with AgNO3for visualizing the SSR fragments. The data were counted by presence or absence of the band and the percentage of polymorphic loci (PIC) was calculated. UPGMA cluster analysis was carried by software NTSYS,the optimal number of groups and population genetic structure was analyzed by software Structure.【Result】Totally,117 polymorphic alleles were revealed with 23 primer pairs which was selected from 64 primer pairs,each primer amplified polymorphic loci ranged from 2 to 10,with an average of 5.09 for each primer pairs. Polymorphism information content (PIC) values for the primer pairs ranged from 0.359 to 0.727,with an average of 0.548,these polymorphisms primers could be further applied to other study. The fingerprint for some jujube cultivars was established with 1-2 markers,providing a reference for the management of jujube germplasm. Meanwhile,based on the UPGMA cluster analysis,255 cultivars were divided into fifteen subgroups,which included four big groups and eleven small groups. Similarity coefficients among the cultivars were between 0.71 to 1.00,‘Beijinghuashengzao’ was clustered into one separate group,which has a distant relationship with other cultivars. The similarity coefficients of ‘Fengjiejidanzao’ and ‘Xupujidanzao’,‘Shannxinaizao’ and ‘Tianjindamayazao’ were both 1.00. In some subgroups the genetic relationship between cultivars and their provenances has a significant positive correlation,but the cultivars and their uses has no significant correlation. Based on K and ΔK values,255 jujube cultivars were also divided into fifteen populations by the population genetic structure analysis. The kinship among cultivars in the same population was relatively simple,and a few cultivars contained genetic component of other groups. The cultivars from Shanxi or Shannxi were distributed in most populations,indicating jujube cultivars of the two provinces played important roles in the gene exchange among populations. The jujube cultivars from Hunan of the South region formed a relatively alone population,indicating that the cultivars might be from the same source,or in the long-term cultivation few times of gene exchange were happened in Hunan cultivars with other populations. Different geographical environment played a key role in the evolution of jujube germplasm populations,some cultivars were selected from the same geographical environment and the others were selected by genetic recombination among those cultivars from the various geographical environment. Meanwhile,the consistency of the two different methods was further verifed the accuracy of the results,which provide useful clues and reference for the genetic diversity and structure of jujube germplasm.【Conclusion】Geographical environment play significant roles in the population evolution of jujube cultivars,affecting the genetic structure composition between different habitats.
Chinses jujube; SSR markers; provenances; genetic diversity; population structure
2016-01-18;接受日期:2016-04-13
“十二五”国家科技支撑计划(2013BAD14B03)、北京市2011协同创新中心项目(PXM2016_014207_000038)、河北农业大学青年学术带头人项目
联系方式:刘秀云,E-mail:jingxianyu705@126.com。李慧,E-mail:1063979927@qq.com。刘秀云和李慧为同等贡献作者。通信作者刘孟军,E-mail:lmj1234567@aliyun.com。通信作者赵锦,E-mail:zhaojinbd@126.com

